Histoplasma capsulatum es el agente causal de la histoplasmosis, micosis asociada principalmente a pacientes inmunocomprometidos. La rápida identificación del hongo a partir del cultivo permite el tratamiento temprano.
ObjetivoEvaluar un sistema de PCR para dos dianas específicas de H. capsulatum en lisados acuosos de cultivos.
MétodosSe utilizaron dos técnicas de PCR previamente descritas que, en reacciones independientes, amplifican fragmentos específicos de 111 y 279 pb del gen AgM de H. capsulatum. Se analizaron 248 cepas de H. capsulatum y 68 de otras especies fúngicas.Para la amplificación se partió de un lisado acuoso (que contenía el ADN), obtenido por tres ciclos de hervido/enfriamiento rápido a 0°C. En casos particulares se obtuvo ADN purificado y/o se secuenció el producto de la amplificación.
ResultadosLas técnicas de PCR amplificaron las dos bandas a partir del lisado acuoso de 239 cepas de H. capsulatum; las 9 restantes sólo mostraron bandas de amplificación a partir de ADN purificado. No se observó amplificación específica a partir de lisado acuoso ni de ADN purificado de 66 cepas de especies distintas de H. capsulatum. Dos cepas de Emmonsia crescens presentaron ambas bandas de amplificación cuyas secuencias resultaron tener una homología superior al 97% con secuencias de H. capsulatum. El tiempo total de la prueba no superó las 7h con un 96% de sensibilidad, 97% de especificidad y un valor predictivo positivo de 99%.
ConclusionesEl método es rápido, económico y puede ser utilizado como una alternativa para identificar presuntivamente H. capsulatum en lisados de cultivo sin purificar.
Histoplasma capsulatum is the agent of histoplasmosis, a deep mycosis mainly afflicting immunocompromised patients. Rapid identification of the fungus isolated from clinical specimens allows timely administration of specific treatment.
AimTo assess the ability of a dual PCR system targeting specific H. capsulatum DNA sites to identify fungal species in simple aqueous lysates from cultured fungi.
MethodsWe analysed the performance of two independent PCR reactions designed to amplify fragments of 111 and 279bp included in H. capsulatum-specific gene AgM. We used 248 H. capsulatum strains and 68 isolates of other fungal species. Reaction templates consisted of aqueous lysates of cultured fungi (either in mycelial or yeast phase) obtained after three cycles of boiling and immediate cooling at 0°C. Selected strains were submitted to conventional DNA extraction and/or sequencing.
ResultsBoth PCR systems performed identically. Amplification from aqueous lysates was achieved from 239 H. capsulatum strains; the remaining 9 strains only showed specific bands when purified DNA was used as template. Of all other fungal species tested, only 2 Emmonsia crescens strains amplified H. capsulatum-specific bands and sequences of their amplified PCR products matched > 97% with H. capsulatum sequences. Total test time did not exceed 7h with 96% sensitivity, 97% specificity and 99% positive predictive value.
ConclusionsThe assay is fast, accurate and economical, and can be an alternative method for presumptive identification of H. capsulatum in simple culture lysates.
Histoplasma capsulatum var. capsulatum es un hongo termodimorfo, agente etiológico de la histoplasmosis. Las personas adquieren la infección por inhalación de conidios y pequeños fragmentos de hifas que el hongo desarrolla en la naturaleza. En las últimas décadas hubo un incremento de la enfermedad asociada a pacientes inmunocomprometidos14.
El diagnóstico se realiza a través del examen microscópico de los especímenes clínicos previa coloración de Giemsa, que permite observar las levaduras intracelulares características de H. capsulatum. Esto requiere de un observador experimentado, depende de una buena toma de muestra, y no deja de ser un método presuntivo, puesto que otras especies fúngicas como Candida glabrata9,18 y Sporothrix schenkii19 pueden confundirse con H. capsulatum en los materiales clínicos. El diagnóstico definitivo requiere el desarrollo e identificación del agente causal en cultivo. A 25-28°C se observan conidios característicos de la fase micelial y a 35-37°C se desarrollan levaduras. Además de la morfología microscópica de los cultivos, la termoconversión de la fase micelial a levaduriforme o viceversa y la detección de exoantígenos H y M específicos, son pruebas que completan la identificación definitiva de la especie. La aparición de conidios en la fase micelial requiere de 2 a 4 semanas de desarrollo (media de 20 días)14,19. La termoconversión es laboriosa y no siempre exitosa, puesto que en algunos casos no es completa. Por su parte, la detección de exoantígenos requiere entre 4 semanas y 3 meses16. El hallazgo cada vez más frecuente de aislamientos aberrantes, que no presentan macro y/o micromorfología característica21,27,29, la ausencia de la termoconversión17,24 y la producción lenta de exoantígenos aumenta la dificultad en el diagnóstico de la histoplasmosis. Además, hongos miceliales de los géneros Sepedonium, Chrysosporium, Corynascus y Renispora pueden producir macroconidios tuberculados rememorando la micromorfología de H. capsulatum en su fase micelial1,7,8,14. De lo expuesto, se infiere que la identificación microbiológica clásica de H. capsulatum puede ser dificultosa y requiere a veces de tiempos prolongados.
Un avance importante en la identificación fue el AccuProbe Histoplasma (Gen-Probe, Inc., San Diego, CAm EE.UU.)10. La dificultad de este método radica en que pueden observarse reacciones falsas positivas con especies de Chrysosporium2, Aspergillus10 e incluso Nocardia asteroides25; además, la sonda quimioluminiscente posee una baja vida media (seis meses), y el instrumental necesario para la lectura de la reacción posee un elevado costo11.
Recientemente, se comenzaron a desarrollar técnicas utilizando diferentes variantes de la técnica de PCR. Existen métodos basados en los perfiles de amplificación de secuencias repetitivas del ADN cromosómico23, de las secuencias ITS19, y del antígeno M (proteína con actividad catalasa)8.
El objetivo de este trabajo fue evaluar la utilidad de un sistema de dos reacciones de PCR descritas por Guedes et al13 en lisados acuosos de cultivos para identificar cultivos de H. capsulatum.
Materiales y métodosCepasSe estudiaron 245 cepas de H. capsulatum incluidas en la colección de cultivos del Departamento de Micología del INEI-ANLIS Dr. Carlos G. Malbrán, Argentina. Los hongos fueron recibidos en el centro para su identificación entre los años 1988 y 2009. Se incluyeron tres cepas de referencia: Downs (ATCC 38904), G186B (ATCC 26030) y G217B (ATCC 26032). Algunos aislamientos poseían características atípicas desde el primocultivo (falta de conidiación, morfología aberrante o pigmento poco característico).
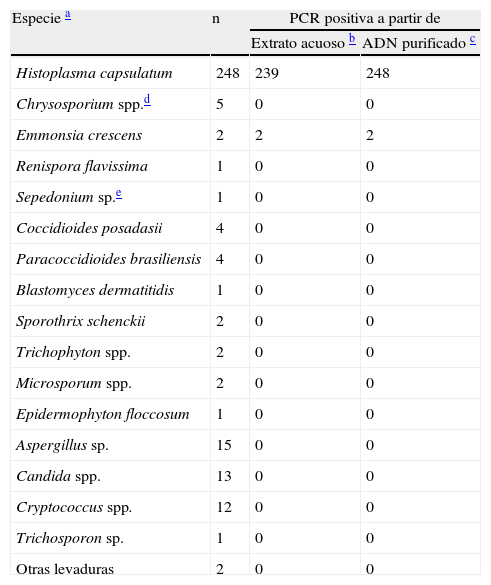
Además se analizaron 68 cepas de diversas especies comúnmente involucradas en patologías fúngicas y especies saprófitas que por su morfología pueden confundirse con H. capsulatum (tabla 1). El ADN purificado de las especies diferentes de H. capsulatum fue gentilmente cedido por los laboratorios de Hongos Miceliales y de Levaduras del Departamento de Micología.
Especies fúngicas analizadas utilizando ADN purificado y extracto acuoso.
| Especie a | n | PCR positiva a partir de | |
| Extrato acuoso b | ADN purificado c | ||
| Histoplasma capsulatum | 248 | 239 | 248 |
| Chrysosporium spp.d | 5 | 0 | 0 |
| Emmonsia crescens | 2 | 2 | 2 |
| Renispora flavissima | 1 | 0 | 0 |
| Sepedonium sp.e | 1 | 0 | 0 |
| Coccidioides posadasii | 4 | 0 | 0 |
| Paracoccidioides brasiliensis | 4 | 0 | 0 |
| Blastomyces dermatitidis | 1 | 0 | 0 |
| Sporothrix schenckii | 2 | 0 | 0 |
| Trichophyton spp. | 2 | 0 | 0 |
| Microsporum spp. | 2 | 0 | 0 |
| Epidermophyton floccosum | 1 | 0 | 0 |
| Aspergillus sp. | 15 | 0 | 0 |
| Candida spp. | 13 | 0 | 0 |
| Cryptococcus spp. | 12 | 0 | 0 |
| Trichosporon sp. | 1 | 0 | 0 |
| Otras levaduras | 2 | 0 | 0 |
La identificación se realizó por métodos fenotípicos y en algunos casos se complementó con métodos moleculares.
Sensibilidad 96%, especificidad 97%, valor predictivo positivo (VPP): 99%, valor predictivo negativo (VPN): 88%.
Todas las especies fueron identificadas fenotípicamente, y cuando fue necesario se recurrió a la identificación genotípica5,12,15,28.
Extracción y purificación de ADNPara estandarizar la PCR a las condiciones de nuestro laboratorio se utilizó ADN purificado de las cepas Downs y 073061, esta última aislada de un paciente argentino con histoplasmosis. El ADN se obtuvo a partir de un cultivo con agitación por 72h en 100mL de caldo GYE (glucosa 2%, extracto de levadura 1%) a 36°C. A partir de allí se siguió el protocolo de Möller et al20 modificado. Brevemente, las células fúngicas se separaron por centrifugación, se lavaron con buffer TES (0,1M TRIS-ClH pH=8,0; 10mM EDTA pH=8,0; 10mM de DTT) y se resuspendieron en solución de lisis (TES-SDS 2%). Se trituraron mecánicamente por agitación en vórtex con esferas de vidrio de 500μm de diámetro. Se agregó proteinasa K (concentración final 100μg/mL) y se incubó durante 1h a 55°C. Posteriormente, se realizó una extracción fenólica, el ADN se purificó por tratamiento con RNAsa (100μg/mL) y proteinasa K (0,2mg/mL) y se repitió la extracción fenólica. El ADN se resuspendió en agua y se conservó a –20°C hasta su uso.
Los ácidos nucleicos de Emmonsia crescens, Sepedonium sp., Chrysosporium spp. y Renispora flavissima se extrajeron especialmente para este estudio. El inóculo inicial de 105-106 conidios se preparó a partir de un cultivo de 7 días en PDA (extracto acuoso de papa con glucosa al 30%, agar 1,5%). Cinco mililitros de este inóculo se trasvasaron a caldo PYG (peptona 2%, glucosa 2%, extracto de levadura 1%) y se incubó en agitación a 120rpm durante 48h a 28°C. El micelio obtenido se separó por filtración, se lavó con agua destilada estéril y se liofilizó. Una porción del micelio liofilizado se colocó en un tubo tipo Eppendorf y se trituró con un pistillo. Se le agregó solución de lisis (0,05M TRIS-ClH pH=8,0; 0,05M EDTA pH=8,0; SDS 3%; beta mercaptoetanol 1%) y se incubo durante 1h a 65°C. Posteriormente, el ADN se extrajo y se purificó como se indica en el párrafo anterior.
Lisados de cultivos sin purificarComo fuente de ADN de la reacción de PCR se utilizaron los lisados de los cultivos sin purificar, obtenidos por tratamiento con calor/frío de micelio y/o levadura. Brevemente, se colocaron aproximadamente 100mg de levadura y/o micelio en un 1mL de agua Milli Q. La suspensión se calentó en baño a 100°C durante 5min y se enfrió rápidamente en hielo (0°C). Este proceso se repitió tres veces. Se dejó reposar 10min y se separó el sobrenadante.
Reacción de PCRSe utilizaron los primers diseñados por Guedes et al8; estos reconocen un fragmento específico del gen que codifica para el antígeno M de H. capsulatum. El par Msp1F (ACA AGA GAC GAC GGT AGC TTC ACG) y Msp1R (GCG TTG GGG ATC AAG CGA TGA GCC) origina una banda de 111 pb, y el Msp2F (CGG GCC GCG TTT AAC AGC GCC) y Msp2R (ACC AGC GGC CAT AAG GAC GTC) una banda de 279 pb. Los primeros se emplearon en dos reacciones independientes.
Para evaluar la utilidad de los lisados acuosos como fuente de ADN para la prueba de PCR se utilizaron las cepas Downs y 073061 como controles positivos y la cepa de R. flavissima (073100) como control negativo.
Las 248 cepas de H. capsulatum y las 68 de otras especies fúngicas se evaluaron usando como fuente de ADN de la reacción el lisado acuoso de los cultivos. En cada reacción de PCR se incluyó un control de inhibición; para ello cada prueba se realizó por duplicado y a uno de los tubos se le agregó 1μl de ADN purificado de H. capsulatum (5 ng/μl). Cuando el resultado de alguna PCR fue negativo en el control de inhibición; se repitió la PCR utilizando ADN purificado del aislamiento correspondiente. Además, se evaluaron los ácidos nucleicos purificados de las especies diferentes de Histoplasma.
Interpretación y análisis de resultadosLa presencia de una o ambas bandas fue indicativo de H. capsulatum. En los casos en que se obtuvieron productos de amplificación de las cepas diferentes de H. capsulatum, éstos se purificaron con PureLink PCR Purification kit (Invitrogen Life-Technologies, CA, EE.UU.) y se secuenciaron las bandas para ambas hebras. Utilizando el programa BLAST se buscó la homología de las secuencias con las depositadas en GenBank y con las secuencias de los productos de amplificación obtenidos de la cepa Downs y dos cepas autóctonas de H. capsulatum (062776 y 073061).
Los resultados fueron volcados a una tabla de dos por dos y se analizó la sensibilidad, la especificidad, el valor predictivo positivo (VPP) y el valor predictivo negativo (VPN).
Condiciones de bioseguridadLa manipulación de los hongos se realizó según recomendaciones del CDC4. La identificación presuntiva de los primocultivos de hongos dimórficos (observación de las características microscópicas y obtención de lisados de cultivos para PCR), así como la extracción y purificación de ácidos nucleicos se realizó en un laboratorio de nivel de bioseguridad 2 (BSL2) dentro de una cabina de seguridad biológica clase ii. La propagación y manipulación de cultivos para las pruebas de dimorfismo y producción de exoantígenos de H. capsulatum y otros hongos dimórficos se realizó en BSL3.
ResultadosLas reacciones de PCR presentaron las bandas de 111 y 279 pb a partir de los ácidos nucleicos purificados y de los lisados acuosos sin purificar de las cepas Downs y 073061. Las reacciones realizadas a partir del ADN purificado y del lisado acuoso de R. flavissima (073100) resultaron negativas.
Ambas técnicas de PCR realizadas sobre los lisados acuosos fueron positivas para 239 de las 248 cepas de H. capsulatum (sensibilidad 96%) (tabla 1). Los lisados acuosos que no produjeron amplificados por PCR correspondieron a 9 cepas; tres eran de años recientes (2005 en adelante) y las seis restants tenían una larga historia en la colección de cultivos e incluían dos cepas de referencia (G186B y G217B). La sensibilidad aumento al 100% cuando se repitió la metodología con el ADN purificado de estas 9 cepas.
Entre las 68 especies diferentes de H. capsulatum evaluadas, 66 no produjeron amplificados ni a partir de los lisados acuosos ni a partir del ADN purificado (especificidad 97%). Dos cepas de E. crescens (083307 y 062869) presentaron las bandas de 111 y 279 pb, y una cepa de Sepedonium sp. una banda de ≈ 200 pb con el primer MSP1F-MSP1R.
Las secuencias de las bandas de 111 y 279 pb de las cepas de E. crescens tuvieron una homología mayor al 97% cuando fueron comparadas en BLAST, con la secuencia de H. capsulatum depositada en el GenBank (AF026268.2). Una homología similar se observó con los fragmentos secuenciados de la cepa Downs, 062776 y 073061 (fig. 1). La secuencia de la banda de Sepedonium sp. resultó de 207 pb, y en el análisis con el programa BLAST no mostró similitud con ninguna secuencia depositada en el GenBank ni con las bandas de las cepas de H. capsulatum secuenciadas en este estudio. Esta reacción fue considerada negativa.
Secuencias de las bandas de 111 (a) y de 279 pb (b) de H. capsulatum (Hc): cepa ATCC 38904 (Downs), dos cepas clínicas autóctonas (062776 y 073061) y de las cepas de E. crescens (Ec) (062869 y 083307). Estas secuencias están comparadas con la secuencia AF026268.2, depositada en Gen Bank.
El tiempo consumido desde la obtención del lisado acuoso hasta la visualización de los productos amplificados varió entre 5 y 7h.
Las técnicas de PCR evaluadas sobre los lisados acuosos tuvo un VPP de 99% y un VPN de 88%. Cuando se utilizó ADN purificado el VPP se mantuvo en un valor similar: 99,2%, mientras que el VPN aumentó a 100%.
En 85 de las 248 cepas de H. capsulatum se realizaron las técnicas de PCR a partir del lisado de cultivo sin purificar antes de identificarlas morfológica y fisiológicamente. En estas cepas se documentaron los tiempos de termoconversión y de producción de exoantígenos. La termoconversión insumió en promedio 72 días (mediana de 40 días) y la producción de exoantígenos 60 días (mediana de 45 días). En este grupo de aislamientos, 3 tuvieron micelio estéril, uno produjo el exoantígeno a los 52 días y la termoconversión insumió 35 subcultivos (más de un año). En otras dos cepas la termoconversión fue incompleta hasta el momento en que finalizó este estudio.
DiscusiónEl diagnóstico de la histoplasmosis requiere el aislamiento y la identificación de H. capsulatum a partir de los cultivos de especímenes clínicos3. Las técnicas clásicas de identificación conllevan tiempos de hasta 4 semanas debido al lento desarrollo del hongo en los medios de cultivo3 y demandan trabajar en BSL34.
El método aquí evaluado es económico, de fácil implementación y permite la identificación rápida de H. capsulatum a partir de lisados de cultivos. En coincidencia con los autores que describieron inicialmente las técnicas de PCR8, los dos sistemas produjeron resultados equivalentes, lo que confirma que la presencia de una de las bandas es suficiente para identificar presuntivamente H. capsulatum. En nuestro estudio, ninguno de los dos sistemas de PCR ensayados permitió diferenciar las cepas de E. crescens de las de H. capsulatum; inclusive, en el análisis de las secuencias de los fragmentos amplificados (fig. 1). En estudios complementarios, enfrentando el antígeno de las dos cepas de E. crescens con suero anti-Histoplama (IMMY H60110 Immuno Mycologics, Norman, OK, EE.UU.) se formaron bandas de identidad parcial en las pruebas de inmunodifusión (datos no mostrados). No sabemos si otras especies genéticamente relacionadas a H. capsulatum, no incluidas en este trabajo, son capaces de producir las bandas de amplificación con los primers ensayados. Probablemente sea necesario investigar un número mayor de cepas de especies relacionadas con el género Emmonsia. Las cepas de E. crescens aquí analizadas fueron aisladas: una de una biopsia perianal de un paciente HIV positivo (062869), y la otra de un lavado broncoalveolar (083307). Ambas fueron identificadas sin inconvenientes por su morfología y la presencia de adiasporas en los cultivos a 36°C. Es importante considerar en este punto que, si bien la adiaspiromicosis y la histoplasmosis presentan cuadros clínicos similares, la enfermedad provocada por E. crescens es extremadamente rara en humanos, y tanto en el tejido como en cultivos a 36°C produce adiasporas5. Otros autores observaron reacciones cruzadas entre especies utilizando métodos moleculares. Dot et al6 utilizaron ADN de H. capsulatum como control positivo de una reacción de PCR para diagnosticar una adiaspiromicosis. La reacción de PCR positiva en las cepas de E. crescens probablemente se deba a que el agente de la adiaspiromicosis y el de la histoplasmosis son ascomicetes. Ambos géneros tienen su estado teleomorfo en el género Ajellomyces (Onygenaceae, Onygenales) y comparten un ancestro común evolutivamente cercano5,22,26.
Valores del 100% de sensibilidad y especificidad fueron referidos por otros autores que trabajaron sobre lisados de cultivos de H. capsulatum sin purificar utilizando PCR en tiempo real basándose en dianas ITS19, sin embargo, estos autores no detallan si incluyeron cepas de E. crescens en su estudio.
A partir de 2005, los aislamientos que llegaron al laboratorio como presuntos hongos dimórficos fueron analizados por la metodología aquí descrita. Cuatro aislamientos no arrojaron amplificados por PCR y posteriormente se identificaron como Coccidioides posadasii, Paracoccidioides brasiliensis, Aspergillus sp. y R. flavissima; estas cepas están incluidas como control de especificidad en la tabla 1.
Otros tres aislamientos de pacientes con sospecha de histoplasmosis y cultivo con micelio estéril dieron como primer resultado indicativo de H. capsulatum amplificados por PCR a partir del lisado; estos resultados fueron confirmados en un tiempo superior a los 30 días por termoconversión y producción de exoantígenos.
Otros métodos rápidos de identificación como el AccuProbe Histoplasma requiere instrumental específico, a lo que se suma el elevado coste del reactivo11. Por su parte, el análisis de las secuencias ITS, consideradas dianas específicas y eficaces para identificar hongos patógenos como Histoplama12,13, requiere equipos de secuenciación. Muchos centros asistenciales de mediana y alta complejidad cuentan con BSL2 con cabinas de seguridad biológica clase ii y están aptos para manejar primocultivos de Histoplasma. En estos laboratorios, la metodología aquí descrita puede convertirse en un método alternativo, ya que en general cuentan con laboratorios de biología molecular, pero no cuentan con servicio de secuenciación para utilizar las dianas ITS.
Entre las ventajas del método aquí propuesto destacamos que solo necesita un termociclador convencional, reduce los tiempos de identificación (5-7 horas), y en consecuencia reduce los costes de la determinación. Aun teniendo que obtener ADN purificado, el método es ventajoso puesto que se extendería a no más de 6 días entre el desarrollo del hongo y la extracción de ADN. Es importante recalcar que aunque el VPP se mantuvo, la sensibilidad y el VPN aumentaron al 100% cuando se utilizó ADN purificado.
Independiente de la edad del cultivo, la fase de desarrollo utilizada, la contaminación bacteriana asociada y el medio de cultivo (algunos extractos acuosos se obtuvieron a partir de H. capsulatum crecido en el medio de Lowestein Jensen) las reacciones de PCR a partir de los lisados acuosos de cultivos sin purificar permitieron identificar H. capsulatum con muy buena sensibilidad y especificidad.
El método aquí ensayado se propone como una alternativa al AccuProbe Histoplasma y a la secuenciación de ITS, para aquellos laboratorios con presupuestos limitados, que poseen termociclador pero no tienen acceso a equipos de secuenciación. Aunque las reacciones cruzadas con Emmonsia podrían ser una limitación, la identificación de esta especie a partir de materiales clínicos no es dificultosa. Es importante tener en cuenta que, según los criterios vigentes, la identificación definitiva de H. capsulatum requiere de la termoconversión dimórfica y/o la detección de exoantígenos específicos en el sobrenadante del cultivo2,16.
AgradecimientosLos autores agradecen a la Dra. Viviana Ritacco las sugerencias realizadas durante la elaboración de este manuscrito.