El género Armillaria, y más concretamente la especie Armillaria mellea, constituye un problema muy importante en el sector vitivinícola de Galicia, lo que ha ocasionado importantes reducciones del rendimiento de los viñedos en los últimos 15 años. Este hongo ataca al sistema radicular, causando un descenso del vigor y, finalmente, la muerte de la planta. Hasta la fecha, no se conoce ningún método químico o biológico que resulte efectivo contra el patógeno una vez que ha entrado en la planta.
ObjetivosEl objetivo principal del presente trabajo fue conocer la incidencia y distribución del género Armillaria mediante técnicas moleculares en las distintas zonas de cultivo de vid en Galicia, abarcando las cinco denominaciones de origen (DO) vitivinícolas gallegas (Rías Baixas, Ribeiro, Ribeira Sacra, Valdeorras y Monterrei).
MétodosSe analizaron 624 muestras (483 de suelo y 141 de plantas de vid sintomáticas) mediante nested-PCR/RFLP, PCR-RFLP y análisis filogenéticos.
ResultadosArmillaria mellea está ampliamente distribuida en viñedos de las cinco DO, con mayor incidencia en la DO del Ribeiro.
ConclusionesEs necesario establecer medidas para reducir el avance de la podredumbre radicular causada por Armillaria mellea en las cinco DO de Galicia.
The genus Armillaria, specifically Armillaria mellea, is an important phytopathological problem in the wine sector in Galicia (NW Spain), having caused yield reductions in vineyards for the last 15 years. The fungus attacks the root system, resulting in a decrease in vigour, and eventually in the death of the plant. Up to now, there is no chemical or biological method really effective against the pathogen once it has infected the plant.
AimsThe main objective of this work was to study the incidence and distribution of the genus Armillaria across the five Galician protected designation of origin (DO) wines (namely Rías Baixas, Ribeiro, Ribeira Sacra, Valdeorras and Monterrei) through the application of molecular techniques.
MethodsA total of 624 samples (483 soils and 141 symptomatic vines) were analyzed by nested-PCR/RFLP, PCR-RFLP and phylogeny.
ResultsArmillaria mellea is widely distributed in vineyards of the five DO wines, with the highest incidence in the Ribeiro DO.
ConclusionsPreventive control measures against Armillaria mellea must be established in the five DO wines of Galicia, in order to reduce the advance of white root rot.
La podredumbre blanca de la raíz, causada por el género Armillaria, es una de las enfermedades más importantes que afectan al viñedo, especialmente en áreas de clima templado. La especie más polífaga y agresiva es Armillaria mellea, si bien otras especies, como Armillaria gallica y Armillaria cepistipes, se han aislado también de vides enfermas1,2. Los síntomas específicos de esta podredumbre se localizan en el sistema radicular y cuello de la planta, donde se observa un micelio blanco-amarillento que cubre, a modo de fieltro, la zona situada entre la madera y la corteza. A veces, mezcladas con el micelio, aparecen unas hifas diferenciadas con forma de cordones, que reciben el nombre de rizomorfos y que se extienden en el suelo, favoreciendo la diseminación de la enfermedad6,19,20.
Una viña puede sobrevivir durante varios años después de haber sido infectada, pero, una vez que la mayoría de las raíces han sido atacadas por el hongo, la planta generalmente muere. Las características definitorias del hongo, como su carácter saprofito, la presencia de rizomorfos, su localización en el suelo, a veces a profundidades considerables, y que el micelio se encuentre protegido debajo de la corteza, hacen muy difícil su control. De hecho, en la práctica no hay un método que resulte realmente efectivo para eliminar el patógeno una vez que ha entrado en la planta.
El sector vitivinícola constituye una de las actividades agrarias e industriales más importantes en España, tanto a nivel económico como cultural. España es uno de los países con mayor extensión de viñedo de la Unión Europea y del mundo, con casi un millón de hectáreas cultivadas, de las que el 98% se destinan a vinificación, el 1,5% a uva de mesa, el 0,3% a la elaboración de pasas, y el 0,2% restante a viveros15. En Galicia hay 24.942ha de viñedo dedicado a uva de vinificación, la mayoría localizadas en Pontevedra (12.123ha, casi el 50% del total) y Ourense (8.419ha)25. La producción de uva en Galicia en 2011 fue de 218.538t, de las que el 57,6% se produjeron en Pontevedra (125.895t) y un 28% en Ourense, provincias donde se ubican la mayor parte de los viñedos para la producción de vinos con denominación de origen (DO) en Galicia15.
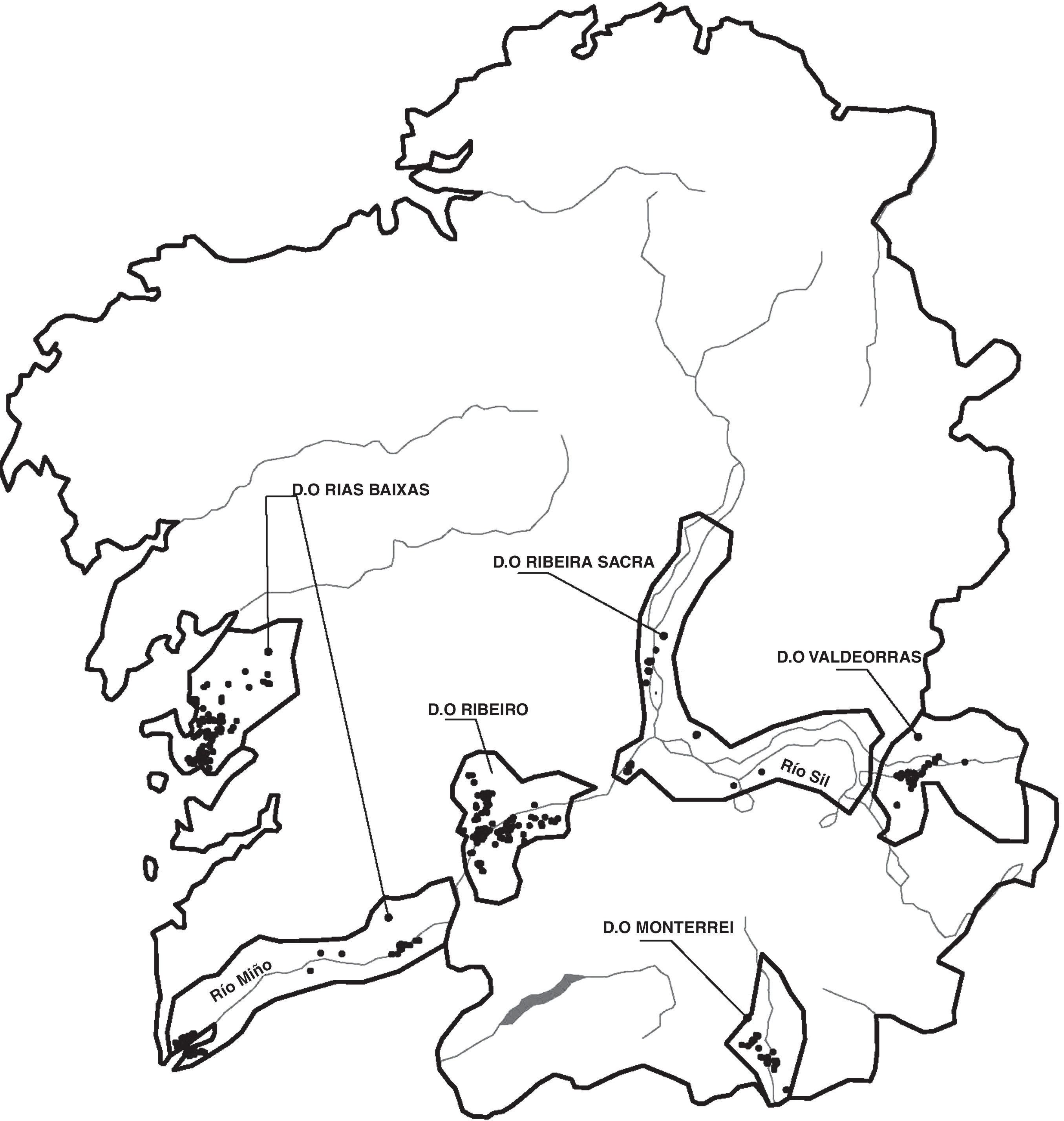
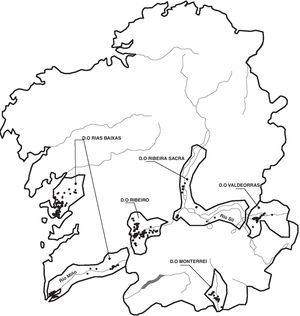
En la actualidad hay cinco denominaciones de origen de vinos gallegos: Rías Baixas en Pontevedra, que incluye las subzonas Salnés, Condado, Rosal, Sotomaior y Ribeira do Ulla (esta última, con algunas hectáreas en el sur de la provincia de A Coruña), Ribeiro, Valdeorras y Monterrei en Ourense, y Ribeira Sacra en Lugo. En los últimos años, el rendimiento de algunos viñedos en todas las DO de Galicia ha sufrido fuertes disminuciones, que podrían estar relacionadas con la presencia de la enfermedad causada por Armillaria. A este respecto, solo existe alguna información relativa a la DO Rías Baixas, obtenida hace más de 15 años1.
El objetivo de este trabajo fue estudiar la incidencia y distribución de la podredumbre blanca radicular causada por especies del género Armillaria en viñedos de las cinco DO de Galicia, a través de su detección en suelo y planta.
Material y métodosDurante el año 2011 se estudió la presencia de Armillaria en suelo y la incidencia de podredumbre blanca en planta en un 5% de la superficie total de viñedo adscrita a cada una de las DO gallegas.
Muestreo y aislamiento de especiesDentro de cada DO, se seleccionaron viñedos de aproximadamente 1ha, inspeccionándose 182 fincas en Rías Baixas, 137 en Ribeiro, 68 en Valdeorras, 62 en Ribeira Sacra y 34 en Monterrei, analizando un total de 483 viñedos (fig. 1). En todos los viñedos se recogieron submuestras de suelo hasta 30cm de profundidad para obtener una muestra compuesta de un 1kg. En cada viñedo se hizo una inspección visual de las plantas para detectar aquellas con síntomas de enfermedad, como reducción del crecimiento, marchitamiento, clorosis y disminución del tamaño de las hojas. Estos síntomas son inespecíficos, por lo que, para determinar si había síntomas específicos de podredumbre blanca radicular, se cavó una pequeña zanja alrededor de cada planta sintomática para poder detectar micelio o rizomorfos en cuello y raíces superficiales, separando con cuidado la corteza. Cuando se observó la presencia de estructuras fúngicas se tomaron muestras de la raíz.
En siete viñedos de la DO Ribeiro se observaron cuerpos de fructificación de morfología similar a los del género Armillaria, tomando también muestras de los mismos.
Las muestras de suelo, raíz y basidiocarpos se dispusieron en bolsas individuales, debidamente etiquetadas y codificadas, y se mantuvieron a 4°C hasta su análisis en laboratorio.
Detección e identificación de Armillaria en sueloDe cada muestra de suelo de los viñedos, se cogieron 2 submuestras de 1g. De cada submuestra se extrajo el ADN fúngico mediante el kit comercial UltraClean® Soil DNA Isolation Kit (MO BIO Laboratories, Inc.) siguiendo el protocolo recomendado por el fabricante. Con el método de Lochman et al.13, con modificaciones realizadas por Escofet et al.5, se amplificó la región IGS del ADN extraído mediante nested-PCR; el fragmento obtenido en la segunda PCR se digirió con las enzimas de restricción Mbo I y Hinf I (Takara Bio, Inc., Japón).
Detección e identificación de Armillaria en raícesLa detección e identificación de especies de Armillaria en las 141 muestras de raíces recogidas se llevó a cabo mediante 2 métodos: directamente de la muestra utilizando la técnica PCR-RFLP, y mediante la obtención a partir del material vegetal infectado de aislamientos fúngicos en cultivo y posterior análisis filogenético de secuencias de ADN.
Reacción en cadena de la polimerasa-polimorfismos de longitud de fragmentos de restricciónSe siguió la metodología propuesta por Harrington y Wingfield9 y Pérez et al.16, pero con las modificaciones propuestas por Mansilla et al.14 para permitir la identificación del patógeno fúngico directamente a partir del material vegetal, sin necesidad de obtener previamente un cultivo puro del hongo correspondiente. En cada muestra de raíz se recogieron 20-40mg de micelio, a partir de los cuales se extrajo el ADN fúngico mediante el kit EZNA® Fungal DNA Mini Kit (OMEGA Bio-Tek), siguiendo el protocolo corto propuesto en el manual del fabricante. La región IGS del ADN molde obtenido se amplificó mediante PCR utilizando los primers LR12R y O-1, recomendados para Armillaria4. Todas las muestras se amplificaron por duplicado. Se incluyó un control negativo en todas las reacciones para confirmar la ausencia de contaminación. Los productos de la amplificación se digirieron con la enzima de restricción Alu I (Takara Bio, Inc.). Los fragmentos digeridos se analizaron mediante electroforesis en gel de agarosa. Las imágenes del gel se analizaron mediante el programa de densitometría 1D-Manager (TDI, Madrid). La identificación fúngica se llevó a cabo comparando el patrón de restricción de cada muestra para las especies europeas y norteamericanas de Armillaria9,16.
Análisis filogenético del ácido desoxirribonucleico de aislamientos en cultivoSe cortaron pequeños fragmentos de raíz con micelio de cada muestra y se sometieron a desinfección superficial, que consistió en un primer lavado de 2min con lejía, seguido por lavado en agua destilada durante 15min. A continuación se introdujeron en alcohol al 50% durante 10min y se pasaron por agua destilada. Las muestras se dejaron secar bien sobre papel de filtro hasta su siembra en placa Petri sobre los medios de cultivo agar-extracto de malta y benomilo-dicloran-estreptomicina8,12,24. La siembra se llevó a cabo disponiendo por placa 4-5 fragmentos desinfectados de raíz de cada muestra. Para cada muestra, hubo 2 placas por medio de cultivo.
Las placas se mantuvieron en estufa de cultivo en oscuridad a una temperatura de 24±1°C. Se obtuvieron aislamientos que formaron colonias típicas de especies de Armillaria y otros cuyas colonias no eran conocidas. Se tomaron entre 20-40mg de micelio de cada aislamiento fúngico obtenido para extraer su ADN y amplificarlo mediante PCR, utilizando los primers O-1 y LR12R, que amplifican la región IGS del ADN, en el caso de las colonias con morfología compatible con Armillaria, siguiendo el método de Harrington y Wingfield9, y los primers universales ITS1F/ITS423 para las demás.
Todos los productos de PCR se purificaron con el kit High Pure PCR Product Purification (Roche Applied Science) y se secuenciaron. Las secuencias de ADN obtenidas se analizaron con el programa Sequencing Analysis 5.1 (AP Biotech) y se compararon con secuencias relacionadas disponibles en la base de datos del National Center for Biotechnology Information utilizando la aplicación Basic Local Alignment Search Tool3. Las secuencias de aislamientos representativos de cada especie fúngica encontrada en este estudio se depositaron en el GenBank (los números de acceso de estos aislamientos se indican en los árboles filogenéticos correspondientes). Se hicieron 2 alineamientos de secuencias: uno de la región IGS para especies del género Armillaria, y otro de la región ITS para los demás aislamientos. En ambos casos las secuencias se ensamblaron y se alinearon mediante el programa ClustalX versión 1.83. Los alineamientos se ajustaron manualmente utilizando el programa BioEdit versión 7.0.9. Se realizaron análisis filogenéticos mediante el método del vecino más próximo, utilizando el programa Molecular Evolutionary Genetics Analysis v. 5.122. Para el análisis de distancia (vecino más próximo) se usó el modelo de sustitución nucleotídica de 2 parámetros de Kimura. La consistencia de cada filogenia se determinó mediante el análisis de 1.000 réplicas.
Identificación de Armillaria mediante análisis molecular del ácido desoxirribonucleico aislado a partir de basidiocarposSe pesaron 20-40mg de láminas del sombrero de cada muestra de basidiocarpos para la extracción de ADN mediante el kit comercial EZNA® Fungal DNA Mini Kit. Mediante PCR, se amplificó la región ITS del ADN obtenido con los primers ITS1F/ITS4, secuenciando el amplicón y procediendo con el análisis de secuencias como se describió anteriormente para las secuencias obtenidas a partir de aislamientos en cultivo.
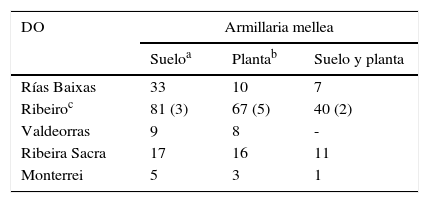
ResultadosDetección e identificación de especies del género Armillaria en suelo mediante reacción en cadena de la polimerasa-polimorfismos de longitud de fragmentos de restricciónEl análisis de los patrones de restricción obtenidos con las enzimas Mbo I y Hinf I, tras amplificación del fragmento de aproximadamente 700pb de la región IGS del ADN fúngico extraído de muestras de suelo, permitió identificar 3 especies: A. mellea, A. gallica y A. cepistipes. A. mellea se encontró en suelos de las cinco denominaciones de origen, A. gallica se identificó en Rías Baixas y Ribeira Sacra, y A. cepistipes en un solo suelo de Rías Baixas (tabla 1).
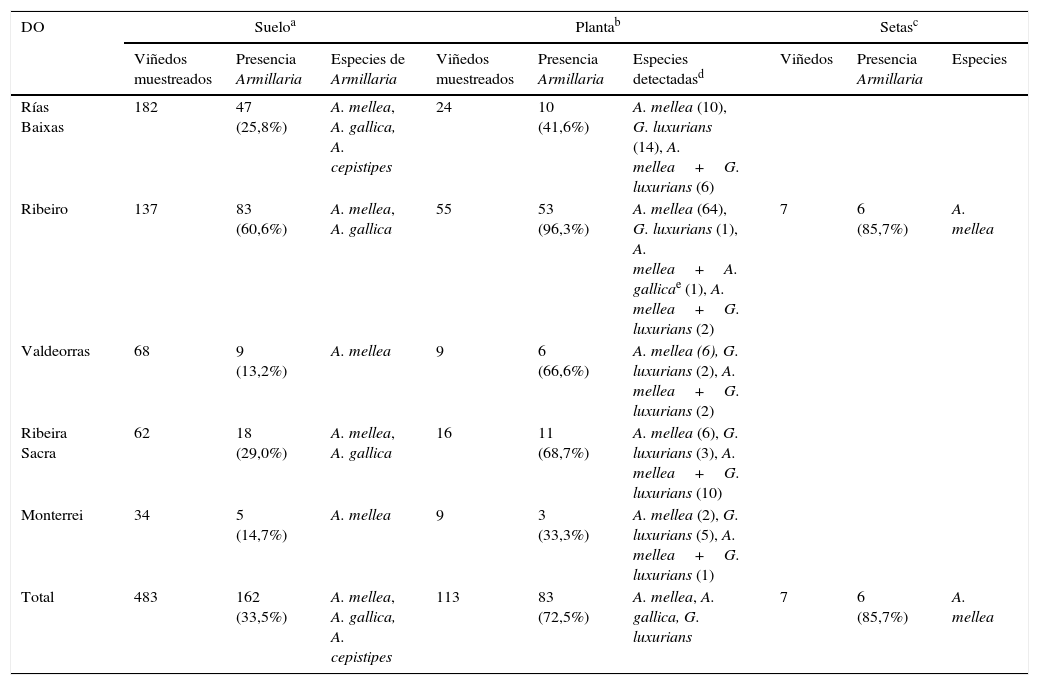
Número de viñedos muestreados, número de muestras con presencia de Armillaria, especies del género detectadas, y presencia de Gymnopus luxurians en las cinco denominaciones de origen de vid en Galicia en suelo, planta y setas
| DO | Sueloa | Plantab | Setasc | ||||||
|---|---|---|---|---|---|---|---|---|---|
| Viñedos muestreados | Presencia Armillaria | Especies de Armillaria | Viñedos muestreados | Presencia Armillaria | Especies detectadasd | Viñedos | Presencia Armillaria | Especies | |
| Rías Baixas | 182 | 47 (25,8%) | A. mellea, A. gallica, A. cepistipes | 24 | 10 (41,6%) | A. mellea (10), G. luxurians (14), A. mellea+G. luxurians (6) | |||
| Ribeiro | 137 | 83 (60,6%) | A. mellea, A. gallica | 55 | 53 (96,3%) | A. mellea (64), G. luxurians (1), A. mellea+A. gallicae (1), A. mellea+G. luxurians (2) | 7 | 6 (85,7%) | A. mellea |
| Valdeorras | 68 | 9 (13,2%) | A. mellea | 9 | 6 (66,6%) | A. mellea (6), G. luxurians (2), A. mellea+G. luxurians (2) | |||
| Ribeira Sacra | 62 | 18 (29,0%) | A. mellea, A. gallica | 16 | 11 (68,7%) | A. mellea (6), G. luxurians (3), A. mellea+G. luxurians (10) | |||
| Monterrei | 34 | 5 (14,7%) | A. mellea | 9 | 3 (33,3%) | A. mellea (2), G. luxurians (5), A. mellea+G. luxurians (1) | |||
| Total | 483 | 162 (33,5%) | A. mellea, A. gallica, A. cepistipes | 113 | 83 (72,5%) | A. mellea, A. gallica, G. luxurians | 7 | 6 (85,7%) | A. mellea |
La presencia de Armillaria se registró en 162 muestras de suelo, un 33% de los suelos de viñedo estudiados. En la gran mayoría (un 95,7%), la única especie detectada fue A. mellea. La mayor incidencia de estos hongos correspondió a los suelos de la DO Ribeiro (un 60% del total) y, la menor, a los de las DO Monterrei y Valdeorras, con un 15 y un13% de los suelos con presencia de A. mellea, respectivamente.
Detección e identificación de especies del género Armillaria a partir de raíces mediante reacción en cadena de la polimerasa-polimorfismos de longitud de fragmentos de restricción y de aislamientos en cultivo mediante análisis filogenéticoEl 62% de las vides sintomáticas de podredumbre blanca radicular presentaron en sus raíces, bajo la corteza, un micelio de color blanco, dispuesto en capas en forma de abanico y asociado a rizomorfos, características típicas de Armillaria. Sin embargo, en las raíces de un 12% de las plantas sintomáticas se observó un micelio blanco amarillento, diferente al de Armillaria, compacto, pulverulento, que se extendía desde la raíz hacia arriba, por encima del cuello de la planta, provocando descortezamiento. En otro 12% de las vides con síntomas se encontraron ambos tipos de micelio, y en el 14% restante no fue visible ningún micelio.
Cuando se cultivó micelio blanco procedente de raíces sintomáticas, con la excepción de un aislamiento que mostró una colonia costrosa con un tono rojizo más intenso y rizomorfos poco abundantes, cilíndricos, de ramificación monopódica y color rojizo, que se corresponden con las que forma A. gallica, todos los demás formaron colonias características de A. mellea: algodonosas-costrosas, con manchas rojizas y rizomorfos abundantes, planos, de color blanco, que presentaban ramificación dicotómica.
El cultivo del micelio amarillento dio lugar a colonias de color blanco al principio y amarillento con posterioridad, con numerosas fíbulas características de los basidiomicetos.
El análisis de los patrones de restricción obtenidos tras amplificación del ADN fúngico procedente de las raíces en las que el micelio mostraba la morfología típica de A. mellea confirmó la identificación de este patógeno, encontrándose los perfiles de restricción mel 1 (320, 180, 155pb) y mel 2 (320, 155pb)16 para A. mellea: un 70% de los perfiles de A. mellea se correspondió con el descrito para mel 1, y el 30% restante para mel 2. Ambos perfiles se detectaron en las cinco DO.
El aislamiento de Armillaria que había formado colonias costrosas características de A. gallica dio lugar al patrón gal 1 (400, 240, 190pb) de A. gallica16. El aislamiento de A. gallica se encontró en una sola muestra de raíz, afectada también por A. mellea, en la DO Ribeiro (tabla 1).
El análisis comparativo de secuencias mediante la aplicación Basic Local Alignment Search Tool para la región IGS de aislamientos de este estudio produjo entre un 99-100% de homología con secuencias del GenBank correspondientes a A. mellea y A. gallica. De la misma manera, la comparación de las secuencias ITS obtenidas con aislamientos de este trabajo dio lugar a un 99-100% de homología con secuencias del GenBank pertenecientes a Gymnopus luxurians.
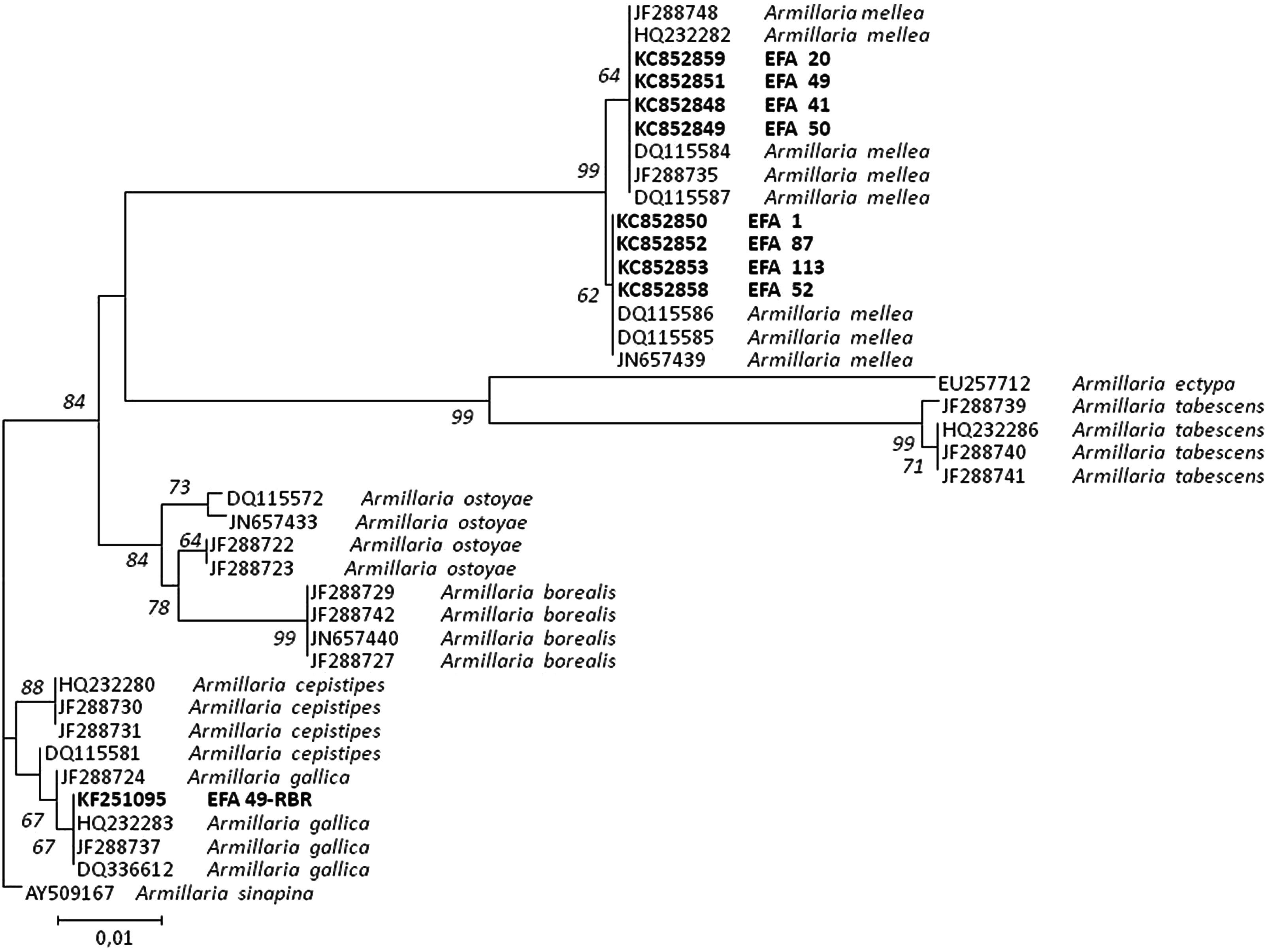
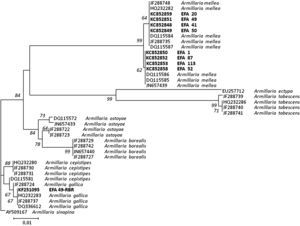
El análisis filogenético de la región IGS incluyó 8 aislamientos de A. mellea, 4 correspondientes al patrón mel 1 y 4 al mel 2, y uno de A. gallica perteneciente al patrón gal 1, obtenidos en este estudio, 29 secuencias incluidas en el GenBank y una secuencia de Armillaria sinapina (n.o GenBank AY509167) como grupo externo (outgroup).
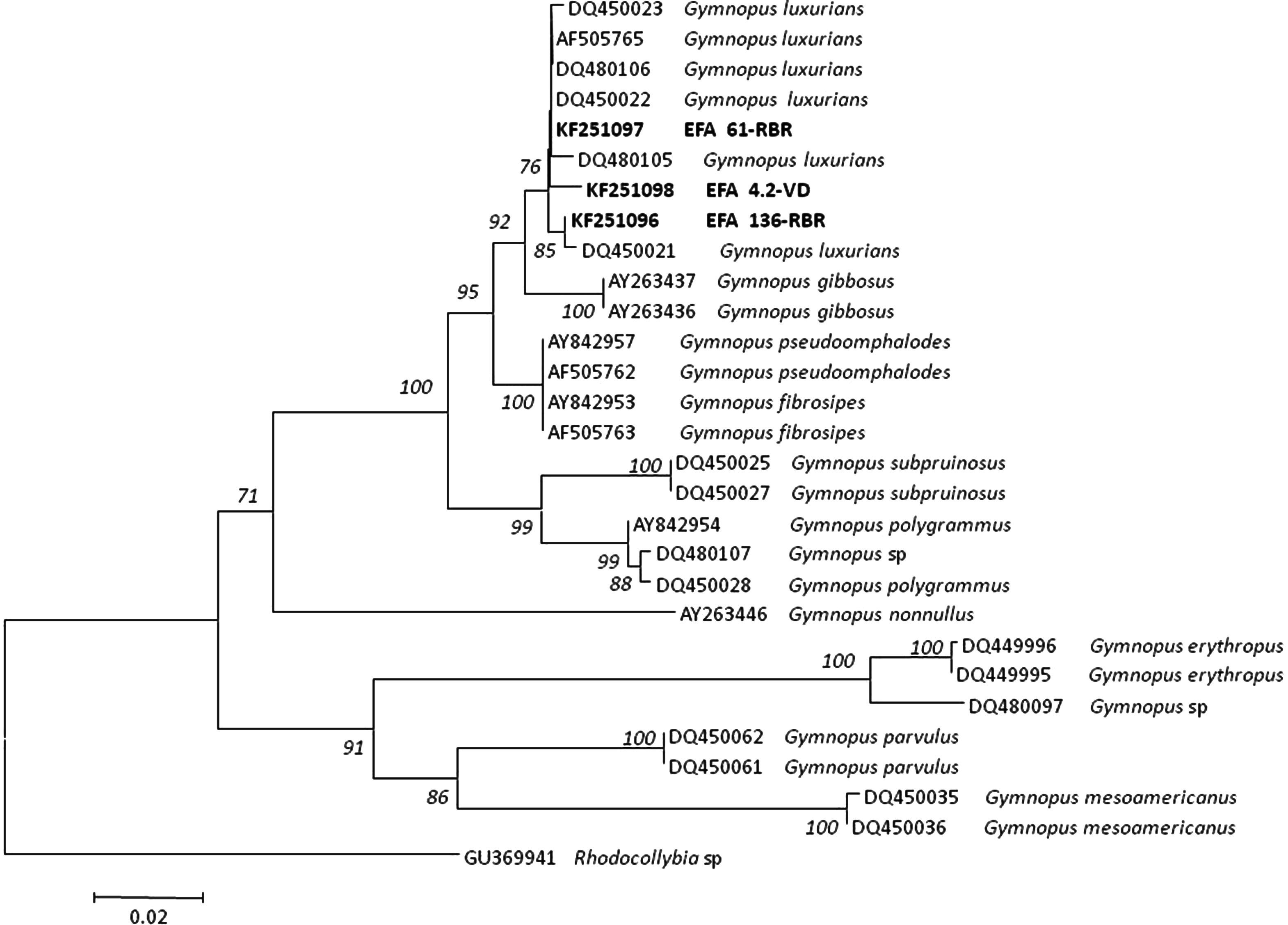
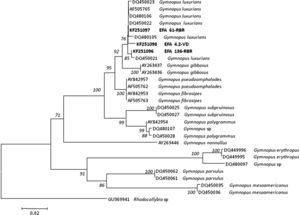
El análisis filogenético de la región ITS incluyó 3 aislamientos de Gymnopus luxurians obtenidos en este estudio, 25 secuencias incluidas en el GenBank y una secuencia de Rhodocollybia sp. (n.o GenBank GU369941) como grupo externo.
El árbol vecino más próximo óptimo para las especies de Armillaria y Gymnopus se presenta en las figuras 2 y 3, respectivamente. En cada árbol solo se indican los valores del número de repeticiones del análisis (bootstrap) superiores al 50%, considerando valores significativos los superiores al 70%10.
Árbol del vecino más cercano mostrando la relación de 39 secuencias de Armillaria de la región IGS del ADNr usando el modelo de sustitución nucleotídica de dos parámetros de Kimura. Los valores del número de repeticiones del análisis (bootstrap) superiores al 50% se indican en los nodos del árbol. La secuencia IGS de Armillaria sinapina (GenBank Accession: AY509167) se usó como grupo externo (outgroup). Las secuencias generadas en este estudio, junto con el número de acceso del GenBank, se indican en negrita. La longitud de las ramas es proporcional al número estimado de sustituciones nucleotídicas. La escala se corresponde con 0,01 sustituciones nucleotídicas por sitio.
Árbol del vecino más cercano mostrando la relación de 29 secuencias de Gymnopus de la región ITS que incluye ITS1, 5.8S ADNr e ITS2 del ADNr usando el modelo de sustitución nucleotídica de 2 parámetros de Kimura. Los valores del número de repeticiones del análisis (bootstrap) superiores al 50% se indican en los nodos del árbol. La secuencia ITS de Rhodocollybia sp. (GenBank Accession: GU369941) se seleccionó como grupo externo (outgroup). Las secuencias generadas en este estudio, junto con el número de acceso del GenBank, se indican en negrita. La longitud de las ramas es proporcional al número estimado de sustituciones nucleotídicas. La escala se corresponde con 0,02 sustituciones nucleotídicas por sitio.
En la tabla 2 se presenta la correspondencia en las cinco DO en la detección de Armillaria en suelo y en planta. No siempre que se detectó Armillaria en suelo se observaron plantas con síntomas de enfermedad, sin embargo, el nivel de detección en suelo del patógeno fue superior en las cinco DO a aquel en planta.
Identificación de Armillaria mediante análisis molecular de basidiocarposEl análisis molecular a partir de ADN extraído directamente de setas procedentes de viñedos de la DO Ribeiro indicó que la mayoría (6 de 7) pertenecían a A. mellea (tabla 1).
DiscusiónEn este trabajo se ha constatado la presencia de especies de Armillaria en suelos de viñedo de las cinco DO de vinos de Galicia. En la mayoría de los casos se encontró A. mellea, una de las especies más agresivas y polífagas del género, que puede causar podredumbre blanca radicular en la vid. La presencia de este patógeno fue particularmente alta en la DO Ribeiro, ya que A. mellea se detectó en casi el 50% de los suelos de viñedo de la DO, y más baja en las DO Ribeira Sacra y Rías Baixas (27 y 22% de suelos con A. mellea, respectivamente) y, sobre todo, en las DO Monterrei (15%) y Valdeorras (13%).
Se encontraron plantas con podredumbre radicular causada por A. mellea en viñedos de las cinco DO, si bien no hubo siempre correspondencia entre la detección del patógeno en suelo y la presencia de plantas con síntomas de la enfermedad, es decir, hubo viñedos en los que el patógeno se detectó en suelo, pero no en planta. Esto indicaría la utilidad del diagnóstico en suelo previo a la detección en planta, lo que ayudaría a la toma de decisiones para poder minimizar el efecto del patógeno. La DO con mayor proporción de plantas enfermas fue la DO Ribeiro, ya que 53 de los 137 viñedos inspeccionados tenían al menos una planta afectada. A esta DO le siguió, en incidencia de A. mellea en planta, la DO Ribeira Sacra, donde se detectaron 16 viñedos (de un total de 62) con alguna planta enferma. En las demás DO (Rías Baixas, Valdeorras y Monterrei), el porcentaje de viñedos con plantas afectadas por la podredumbre causada por A. mellea fue de alrededor del 10%.
Guillaumin7, Prospero et al.18, Aguín et al.1,2, Escofet et al.5 y Pertot et al.17 también detectaron principalmente A. mellea en viñedos, lo que evidencia que esta especie es la responsable de las mayores pérdidas económicas en las áreas de cultivo europeo de vid.
Es importante destacar la presencia de 2 perfiles diferentes de A. mellea en planta, caracterizados como mel 1 y mel 2, siendo el patrón mel 1 el más abundante y presente en las cinco DO de vino en Galicia. Ambos perfiles ya habían sido detectados en viñedos de una de las denominaciones de origen, Rías Baixas, observando también una mayor abundancia del patrón mel 12. No se sabe si esta variación intraespecífica podría estar relacionada con diferencias de patogenicidad, por lo que sería necesario llevar a cabo investigaciones comparando cepas de ambos perfiles.
El análisis de los patrones de restricción de suelo también indicó que en 6 muestras (correspondientes a viñedos de las DO Rías Baixas, Ribeiro y Ribeira Sacra) estaba presente A. gallica, y en 3 (de Rías Baixas), A. cepistipes. Estas 2 especies muestran un comportamiento y características morfológicas muy parecidas. La presencia de A. gallica y A. cepistipes en viña ya había sido descrita por Aguín et al.1,2 y Pertot et al.17 en planta. A. gallica se considera un saprofito o parásito de debilidad, y es frecuente encontrarla en especies arbóreas de hoja ancha21. A. cepistipes también se considera un parásito de debilidad, y se encuentra en especies de hoja ancha y coníferas11,12.
Es importante resaltar la detección de un número alto de viñas con una podredumbre blanca radicular similar a la causada por Armillaria, pero que el análisis molecular demostró que era ocasionada por Gymnopus luxurians. Ambos géneros (Armillaria y Gymnopus) tienen un origen monofilético común. Hasta el momento no existe información acerca de este hongo como patógeno ni en vid ni en otros cultivos. En la bibliografía se describe como un saprofito ampliamente distribuido en el suelo26. El efecto de este hongo en los viñedos gallegos necesita ser investigado.
Los resultados obtenidos aconsejan establecer de forma inmediata un plan de control preventivo para evitar el avance de la enfermedad en las DO gallegas. Las medidas consistirían, en primer lugar, en reducir aquellos factores que favorecen el desarrollo del patógeno (exceso de humedad y materia orgánica en el suelo, pH ácido), en eliminar todo aquel material infectado para minimizar la presencia de inóculo en suelo y en aplicar organismos antagonistas y simbióticos que puedan contribuir a la protección del viñedo frente al ataque de Armillaria.
FinanciaciónEste trabajo ha sido financiado mediante un convenio de colaboración entre la Diputación de Pontevedra y el Centro Tecnológico Agroalimentario de Lugo (CETAL).
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.
Los autores queremos agradecer a Paula Barreiro y a Jacobo Dalama (EFA) su trabajo en el procesamiento de muestras, y a los propietarios y Consellos Reguladores de cada una de las Denominaciones de Origen por su interés y ayuda en el muestreo.