Candida auris fue aislada por primera vez en Japón en el año 2009, como agente causal de una infección de oído7. Desde 2016 diversos organismos nacionales e internacionales de salud han alertado sobre la aparición y expansión de este hongo6.
C. auris está filogenéticamente relacionada con Candida haemulonii y es identificada erróneamente por técnicas bioquímicas y de proteómica5. La mayoría de las plataformas de diagnóstico comerciales no ofrecen una identificación fiable, por lo que C. auris no debería descartarse cuando un aislamiento es identificado por API® 20C AUX como Rhodotorula glutinis o Candida sake; por MicroScan® como Candida famata, Candida lusitaniae, Candida guilliermondii o Candida parapsilosis; por BD Phoenix™ como C. haemulonii o Candida catenulata; por Vitek 2® como C. haemulonii, o cuando no se obtiene una identificación por espectrómetro de masas, MALDI-TOF Vitek-MS® (bioMérieux)1,4.
Artículos recientes sobre el brote de C. auris que tuvo lugar en España mencionan la posibilidad de confundir C. auris con C. haemulonii y C. lusitaniae por MALDI-TOF Vitek-MS®, y con Saccharomyces cerevisiae por métodos basados en la asimilación de azúcares como el AUXACOLOR®6. Ante la posibilidad de una incorrecta identificación, y dada la importancia de una respuesta a corto plazo sobre la presencia de C. auris, se explica a continuación el uso de una técnica convencional (restriction fragment length polymorphism [RFLP]) para su correcta identificación. Se recopilaron ocho especies (seis de ellas pertenecientes al género Candida) de las nueve que pueden ser confundidas con C. auris: C. haemulonii, S. cerevisiae, R. glutinis, C. famata, C. sake, C. lusitaniae, C. guilliermondii y C. parapsilosis. C. catenulata no se aisló de ninguna muestra hospitalaria por lo que nos basamos en datos bibliográficos2. Todas ellas fueron identificadas mediante MALDI-TOF Vitek-MS®.
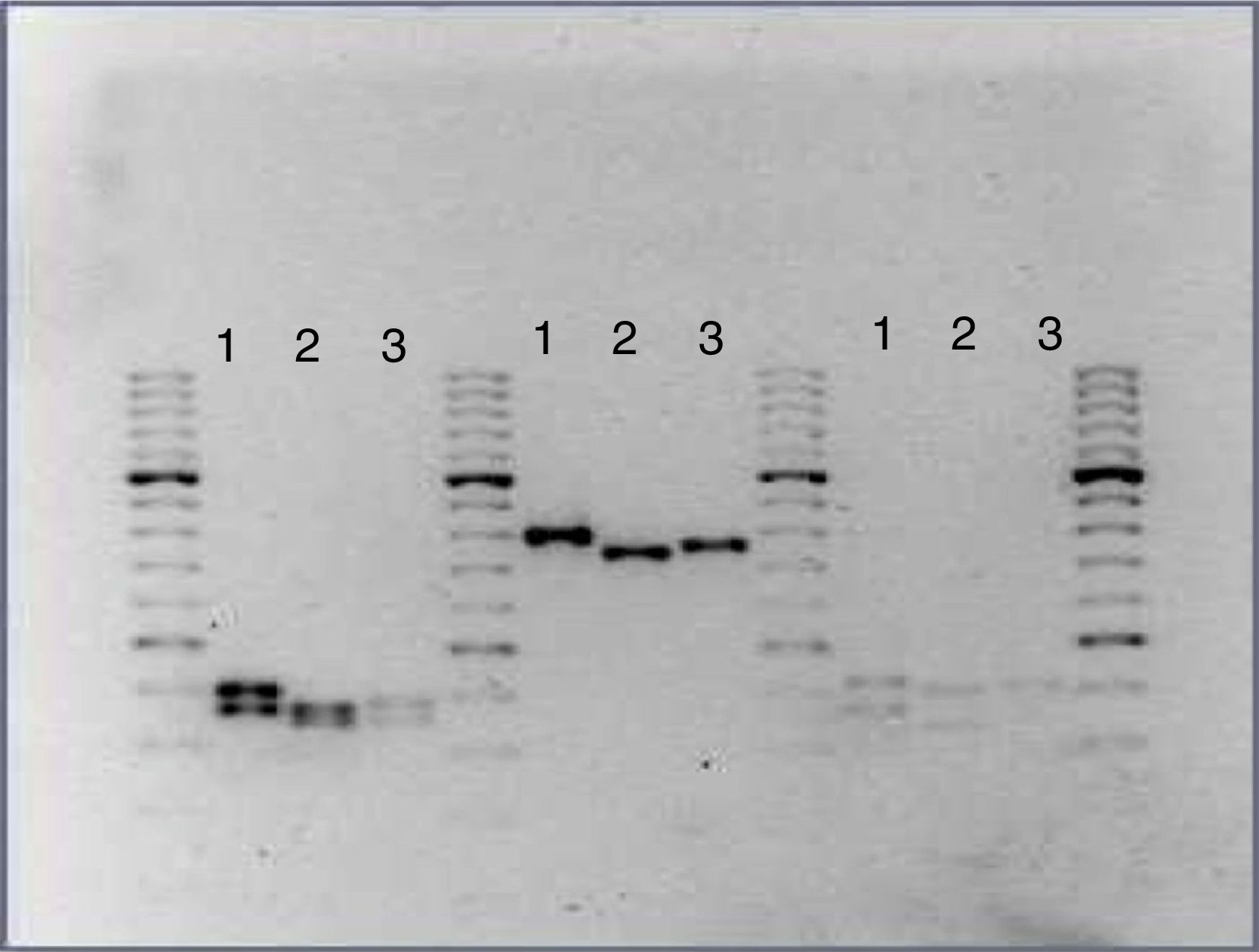
A partir de algunas colonias de las diferentes especies cultivadas en el medio cromogénico CAN2 (bioMérieux) para C. albicans se obtuvieron eluidos de ADN mediante el sistema de extracción automatizado MagCore®. Una vez hecha la extracción del ADN se realizó la amplificación de las regiones ITS1 e ITS2, y del 5,8S rRNA, según lo descrito por Esteve-Zarzoso et al.3. El producto obtenido fue digerido a una temperatura de 37°C mediante las enzimas de restricción HaeIII y Hinfl durante 2h, y durante 9h con la enzima HhaI. Una vez terminada la digestión, el producto de PCR obtenido se visualizó mediante una electroforesis en gel de agarosa al 2% con el empleo de SYBR® Safe como fluoróforo. Para la realización del control interno de la técnica se incluyeron controles positivos y negativos. Como controles positivos se emplearon dos cepas de C. auris previamente secuenciadas en el Hospital Universitario y Politécnico de La Fe, Valencia.
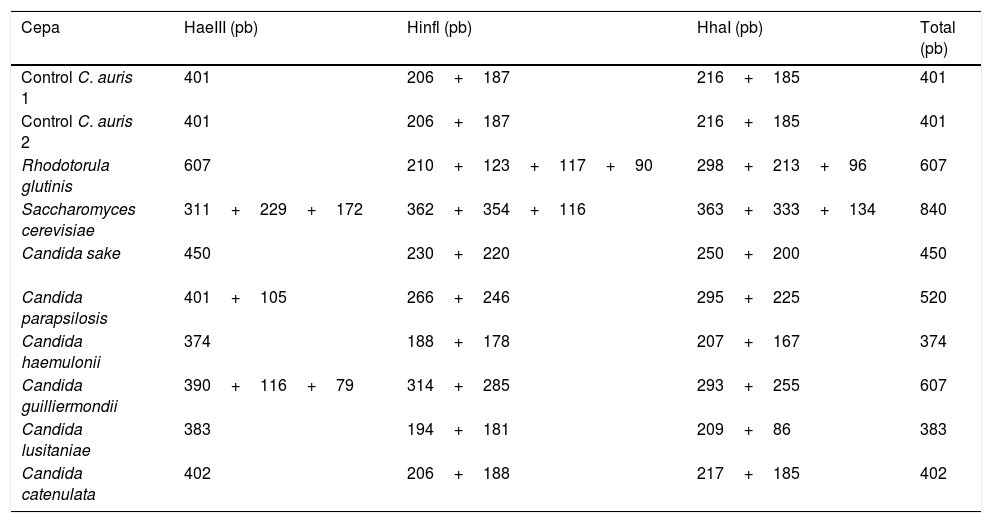
Mediante la amplificación fue posible diferenciar C. auris (401 pares de bases [pb]) de siete de las especies estudiadas; fueron indiferenciables C. lusitaniae y C. catenulata debido al similar peso molecular de los fragmentos obtenidos tras la digestión (383 y 402pb, respectivamente). Los aislamientos de C. auris presentaron un tamaño de 401pb con la enzima de restricción HaeIII, dos fragmentos de 206 y 187pb con la enzima de restricción Hinfl, y dos fragmentos de aproximadamente 216 y 185pb con la enzima de restricción HhaI (fig. 1). Todos los aislamientos estudiados presentaron patrones de restricción característicos y fácilmente diferenciables por esta técnica, excepto C. catenulata (tabla 1). La identificación correcta de C. auris puede llevarse a cabo por métodos moleculares como la secuenciación de las regiones ITS o D1-D2 y también mediante MALDI-TOF si incluye las librerías RUO.
Patrones de restricción (fragmentos en pb) de los aislamientos estudiados
| Cepa | HaeIII (pb) | Hinfl (pb) | HhaI (pb) | Total (pb) |
|---|---|---|---|---|
| Control C. auris 1 | 401 | 206+187 | 216+185 | 401 |
| Control C. auris 2 | 401 | 206+187 | 216+185 | 401 |
| Rhodotorula glutinis | 607 | 210+123+117+90 | 298+213+96 | 607 |
| Saccharomyces cerevisiae | 311+229+172 | 362+354+116 | 363+333+134 | 840 |
| Candida sake | 450 | 230+220 | 250+200 | 450 |
| Candida parapsilosis | 401+105 | 266+246 | 295+225 | 520 |
| Candida haemulonii | 374 | 188+178 | 207+167 | 374 |
| Candida guilliermondii | 390+116+79 | 314+285 | 293+255 | 607 |
| Candida lusitaniae | 383 | 194+181 | 209+86 | 383 |
| Candida catenulata | 402 | 206+188 | 217+185 | 402 |
pb: pares de bases.
Con los resultados obtenidos y considerando el caso falso positivo del aislamiento de C. catenulata (aunque el resultado fuese teórico y no real) la sensibilidad y especificidad de la técnica fueron de un 100% (intervalo de confianza [IC] del 95%: 15,8-100%) y 85,7% (IC 95%: 42,1-99,6%), respectivamente. No obstante, hay que considerar que el número de aislamientos estudiados fue muy limitado. El RFLP de las regiones ITS1/ITS2 permite la caracterización de C. auris, diferenciándola del resto de especies, excepto de C. catenulata. Esta técnica puede considerarse una alternativa para aquellos laboratorios que no dispongan de los sistemas que permitan su correcta identificación.