Bio-Oss (Geistlich) está formado por hueso bovino esterilizado y desproteinizado compuesto por carbonato de apatita deficiente en calcio y es idéntico al hueso humano desde el punto de vista fisicoquímico1-8. Bio-Oss tiene una fuerza de compresión de 35 MPa y su naturaleza porosa (75 al 80 % del volumen total) permite incrementar en gran medida la superficie del material. Este aumento de la superficie proporciona un substrato para incrementar la angiogénesis y constituye un armazón para la formación de hueso1-8. Se ha descrito que Bio-Oss favorece la osteogénesis y tiene una tasa de reabsorción muy baja. Se ha utilizado con frecuencia en casos de elevación del suelo del seno maxilar9-16. En algunos casos puede ser ventajoso utilizar un material que presenta muy poca degradación, como Bio-Oss. Cuando se utiliza, el hueso crece en dirección ascendente desde el hueso preexistente en el suelo sinusal hacia la zona injertada, manteniendo el espacio, ayudando a prevenir la indeseada reabsorción ósea y sin muestras de reacción inflamatoria. El éxito de Bio-Oss en el aumento de seno maxilar también se confirmó en un estudio a largo plazo17.
Puesto que se no se conoce por completo el mecanismo por el cual Bio-Oss estimula la actividad de los osteoblastos para favorecer la formación ósea, los autores trataron de obtener más información mediante el análisis de los microARN (miARN).
La transcripción es la síntesis de ARN mensajero (ARNm) a partir del ADN, mientras que la traducción es la síntesis de proteína a partir del ARNm. Los miARN constituyen una clase de ARN pequeños, funcionales y no codificantes de 19 a 23 nucleótidos que regulan la traducción de los ARNm en proteínas18,19. Así pues, los miARN regulan la cantidad de ARNm específicos en las vías metabólicas de silenciación del ARN postranscripcional al inducir la degradación de los ARNm diana20-26.
Para investigar el efecto de Bio-Oss en los miARN se utilizó un microchip conteniendo cientos de secuencias de miARN (Invitrogen). Esta técnica se denomina micromatriz miARN o perfil de expresión génica, porque permite que el técnico analice de forma simultánea los cambios en cientos de miARN. Al analizar 329 secuencias de miARN humano repartidas por la matriz (portaobjetos), los autores compararon las expresiones de los miARN en las células osteoblastoides humanas (MG63) tratadas con Bio-Oss frente a las células (MG63) no tratadas.
Materiales y métodos
Cultivos celulares
La células osteoblastoides (MG63) se cultivaron en pocillos Falcon para cultivos celulares estériles (Becton Dickinson) que contenían un medio mínimo esencial de Eagle suplementado con un 10 % de suero fetal bovino (Sigma Chemical) y antibióticos (100 mg/ml de penicilina y 100 mg/ml de estreptomicina, Sigma Chemical). Los cultivos se mantuvieron a 37 ºC en una atmósfera humidificada de dióxido de carbono al 5 %. Se obtuvieron las células MG63 y se sembraron con una densidad de 1 x 105 cél/ml en pocillos de 9 cm2 (3 ml) utilizando tripsina al 0,1 % y ácido etilendiaminotetraacético al 0,02 % en tampón de Eagle sin Ca2+ ni Mg2+ para liberar las células. Se añadió Bio-Oss en un grupo de pocillos a una concentración de 100 mg/10 ml. Transcurridas 24 h, cuando los cultivos alcanzaron la subconfluencia, se procesaron las células para extraer el ARN.
Micromatrices de miARN
El miARN se extrajo de las células utilizando el PureLink miRNA Isolation Kit (Invitrogen). Se tomaron 400 ng de miARN de cada muestra (tratada y control) y se utilizaron para hibridar el NCode Multi-Species miRNA Microarray, un portaobjetos conteniendo 329 secuencias humanas de miARN por duplicado. Se utilizó un NCode miRNA Labeling System (Invitrogen) para marcar e hibridar el miARN de la micromatriz, según las instrucciones del fabricante. En pocas palabras, se añadió una cola de poli(A) a cada miARN utilizando una poli(A) polimerasa y un tampón reactivo optimizado. Posteriormente, la secuencia de captura se ligó al miARN utilizando un oligo(dT) intermediario. Tras la purificación, los miARN marcados se hibridaron con las micromatrices y se incubaron durante toda la noche.
Tras un período de incubación de 18 a 20 h, la matriz se lavó y se hibridó con el reactivo de captura Alexa Fluor 3 (para las células control) y Alexa Fluor 5 (para las células tratadas). La matriz se lavó de nuevo y luego se exploró utilizando un escáner de micromatrices estándar (Axon Instruments).
Tras el escaneado, se identificaron todos los puntos gracias a un archivo GenePixR Array List descargado de www.invitrogen.com/ncode que enumera la identidad y la localización de todas las sondas impresas en la matriz. Las imágenes se cuantificaron utilizando el programa GenePix 6.0 (Axon Instruments). La intensidad de la señal de cada punto se calculó restando el fondo local de la intensidad total. Los datos se normalizaron utilizando los paquetes DNMAD y Preprocessing27,28, que generaron un valor promedio de las dos réplicas de cada miARN. Para seleccionar los miARN expresados de forma diferencial se analizaron los datos obtenidos utilizando el paquete Significance Analysis of Microarray29.
Para las predicciones y validaciones diana, los miARN se procesaron utilizando miRBase Target30, un recurso de la red desarrollado por Enright Lab en el Wellcome Trust Sanger Institute. Esta fuente utiliza un algoritmo denominado miRanda para identificar los posibles lugares de unión de un determinado miARN en la secuencia genómica.
La lista de genes diana se procesó mediante FatiGO31, una interfaz de la red que realiza la extracción de datos sencillos utilizando Gene Ontology32. La extracción de datos consiste en asignar el término Gene Ontology más característico a cada grupo de genes regulados.
Resultados
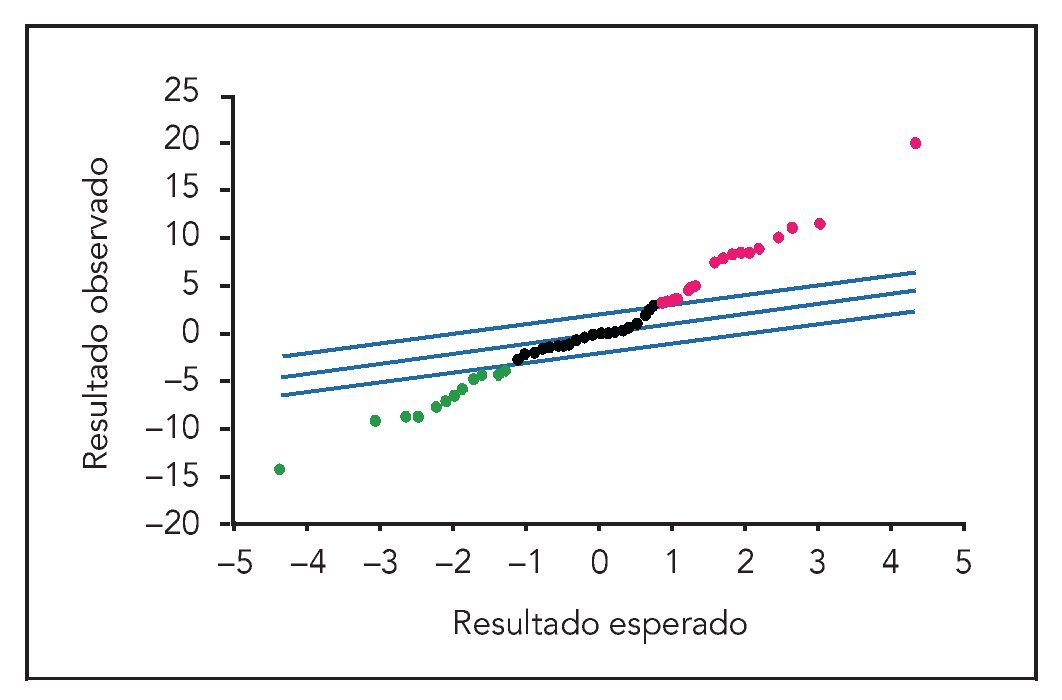
La hibridación del miARN (obtenido de las células MG63 cultivadas con Bio-Oss a una concentración de 100 mg/10 ml) con las secuencias seleccionadas en el portaobjetos permitieron que los autores realizaran un análisis sistemático de los miARN y proporcionaron información primordial sobre la regulación del proceso de traducción inducido por Bio-Oss. Existieron nueve miARN regulados al alza (mir-423, mir-492, mir-191, mir-23a, mir-377, mir-494, mir-214, mir-139b, mir-320) y cuatro miARN regulados a la baja (mir-27a, mir-24, mir-188 y let-7c), para una tasa de descubrimientos falsos igual a 0 y una puntuación > 7. La figura 1 presenta el gráfico del análisis estadístico de la micromatriz y muestra la expresión diferencial de los miARN. Puesto que el miARN tiene potencia para regular miles de genes, los autores seleccionaron solamente los genes relacionados con la osteogénesis y la remodelación ósea regulados de la misma forma por dos miARN casi independientes (tabla 1). Se excluyeron los genes con regulaciones opuestas.
Figura 1 Análisis estadístico del gráfico de la micromatriz de células MG63 tratadas durante 24 h con Bio-Oss a una concentración de 100 mg/10 ml. Los miARN regulados a la baja se localizan en la zona inferior izquierda; los miARN regulados al alza se sitúan en la zona superior derecha. Las líneas paralelas trazadas desde la parte inferior izquierda a la parte superior derecha son los valores de corte.
Discusión
Los informes histológicos muestran que la mayoría de las partículas de Bio-Oss estaban rodeadas por hueso maduro, compacto, de reciente formación, con osteonas bien organizadas2,5,7,8. En algunos campos se observaron osteoblastos en proceso de aposición ósea directamente en la superficie de la partícula. No existieron vacíos en la interfaz partículahueso y éste se mantuvo en contacto estrecho con las partículas en todo momento9-13. No se observó ningún infiltrado de células inflamatorias alrededor de las partículas o en la interfaz con el hueso. Sin embargo, apenas se conoce la manera en que este biomaterial altera la actividad de los osteoblastos a nivel genético para favorecer la formación ósea. Por tanto, los autores trataron de obtener más información analizando los miARN de los osteoblastos expuestos a Bio-Oss frente a los osteoblastos no expuestos.
Los miARN son un tipo de moléculas de ARN no codificante, de pequeño tamaño (19 a 23 nucleótidos) recién descubiertas. Desempeñan un papel importante en el proceso de la regulación postranscripcional. Los miARN no se traducen en proteínas, en lugar de ello regulan la expresión de otros genes al segmentar o reprimir la traducción de sus ARNm diana.
La hibridación del miARN obtenido de las células MG63 cultivadas con 100 mg/10 ml de Bio-Oss con las secuencias seleccionadas en el portaobjetos permitió a los autores realizar un análisis sistemático de los miARN y proporcionó información primordial con respecto a la regulación de la traducción inducida por Bio-Oss (tabla 1).
La amplia mayoría de los genes detectados se regula a la baja, y entre ellos existieron varios genes homeobox (genes que regulan la morfogénesis de todo un segmento del organismo). Los nogging (NOG) inactivan a los miembros de la superfamilia del factor de crecimiento transformante b de proteínas de señalización, como la proteína morfogenética ósea 4. Al difundirse por las matrices extracelulares de forma más eficiente que los miembros de la superfamilia del factor de crecimiento transformante b, los NOG tienen un papel importante en la creación de gradientes morfogenéticos33. El homólogo humano engrailed 1 (EN1) interviene en el control de la formación de patrones durante el desarrollo de las extremidades34. El gen homeobox distal-less 5 (DLX5) se regula al alza; es un miembro de la familia homeobox que regula el factor de transcripción e interviene en el desarrollo óseo y la consolidación de las fracturas. La mutación de este gen está asociada con la malformación ectrodactilia de manos y pies35. Otro grupo de genes regulados a la baja son las hormonas, como el factor de crecimiento insulinoide tipo 2 (IGF2), que es miembro de la familia insulínica de los factores de crecimiento polipeptídicos implicados en el desarrollo y el crecimiento36.
Los genes estudiados en este artículo son solamente unos cuantos de los descritos en la tabla 1 que están regulados de forma diferencial por el miARN. Los autores analizaron brevemente aquellos cuyo funcionamiento se conoce mejor y que estuvieron relacionados directamente con la formación ósea, el desarrollo esquelético, la remodelación del cartílago y la producción ósea. Además, el hecho de que varios genes relacionados con la formación ósea tengan un control negativo de la traducción puede estar relacionado con una fase temprana del análisis (es decir, las células MG63 cultivadas durante 24 h hasta la subconfluencia). Esta fase se caracteriza por una cinética celular elevada y una baja diferenciación y producción de matriz extracelular.
Vale la pena destacar que MG63 es una línea celular y no un cultivo celular primario de osteoblastos. No obstante, las ventajas de utilizar una línea celular están relacionadas con el hecho de que la reproducibilidad de los datos es más elevada, ya que no existe variabilidad en el paciente estudiado. Los cultivos celulares primarios constituyen una fuente de células normales, pero también contienen diferentes tipos de células contaminantes y células en diferentes estadios de diferenciación. Es más, los autores eligieron realizar el experimento después de 24 h para obtener información sobre los estadios iniciales de la estimulación, un momento crítico en cualquier procedimiento de injerto. Son necesarios más investigadores, con otras líneas celulares osteoblastoides, cultivos primarios y diferentes momentos puntuales para obtener una comprensión global de los sucesos moleculares relacionados con Bio-Oss. No obstante, se detectó un efecto global en la formación de hueso y el modelo de estudio puede ser útil para investigar el uso combinado de citocinas como la proteína morfogenética ósea 4.
Agradecimientos
Este trabajo ha sido financiado por becas de la Universidad de Ferrara, Italia (FC), PRIN 2005 prot. 2005067555-002 (FC). Fondazione CARIFE (F ) y la Fondaziones CARISBO (FP).
Correspondencia: Dr Francesco Carinci,
Department of DMCCC, Section of Maxillofacial Surgery, University of Ferrara, Corso Giovecca, 203, 44100 Ferrara, Italy;
fax: +39 0532 291582;
e-mail: crc@unife.it.