El presente trabajo tiene por objetivo mostrar algunas aplicaciones recientes de aprendizaje automático en el área de la salud. El aprendizaje automático o machine learning es una rama de la inteligencia artificial que ha logrado grandes avances en la extracción de patrones y análisis predictivo obteniendo el estado del arte en varias tareas. Por lo mismo, esta tecnología es utilizada en varios sistemas dentro de hospitales y clínicas. Este trabajo introduce a la temática de aprendizaje automático y algunos de sus usos en salud. Posteriormente, se muestran algunas aplicaciones divididas según los tipos de datos que utilizan.
This work aims to show recent applications of machine learning in health. Machine learning is a branch of artificial intelligence that has produced excellent results in pattern recognition and predictive analysis achieving state-of-the-art performance in several tasks. For that reason, this technology is used in multiple hospital systems. This article introduces machine learning, important algorithms, and ethical considerations as well as commenting on various applications in health sorted by the type of data used in those studies.
El aprendizaje automático está presente en la vida cotidiana con sus diversas aplicaciones; desde la recomendación de una serie de televisión hasta la predicción del tiempo de viaje de transporte. El aprendizaje automático corresponde a una rama de la inteligencia artificial que permite la extracción de patrones significativos a partir de un conjunto de datos. La inteligencia artificial es un término general para referirse a los sistemas informáticos que imitan la inteligencia humana1.
En salud, el aprendizaje automático y la inteligencia artificial han contribuido al desarrollo de diversas aplicaciones como el diagnóstico basado en imágenes, la predicción de resultados clínicos, el monitoreo de pacientes, entre otros. Sin embargo, este camino no ha estado exento de desafíos, comenzando por la disponibilidad de datos de calidad para entrenar los modelos y la infraestructura tecnológica para alojar estos sistemas, hasta las consideraciones éticas del uso de datos y modelos2.
Este trabajo tiene por objetivo mostrar las aplicaciones del aprendizaje automático en salud. Para ello se introducirán definiciones y conceptos necesarios para entender cómo funciona el aprendizaje automático y una pequeña discusión sobre la ética asociada al área. A continuación, se detallarán aplicaciones en salud, comenzando por la utilización de datos estructurados, como los registros médicos electrónicos y datos administrativos, para continuar con datos no estructurados, como las imágenes médicas y el texto clínico.
2DefinicionesEl aprendizaje automático se define como el campo que estudia los algoritmos computacionales que mejoran a partir de la experiencia3. En términos simples, se refiere a la generación de programas computacionales que mejoran en alguna métrica de evaluación con el uso de datos.
Algunos conceptos que se usan comúnmente en el área de aprendizaje automático pueden ser desconocidos para el personal médico. A continuación, introducimos las definiciones de los conceptos más relevantes1,4:
Datos etiquetados: se refiere a los ejemplos con sus variables correspondientes que tienen etiquetas o valores reales para su variable objetivo. Por ejemplo, imágenes de células con la etiqueta de cancerígenas o benignas.
Conjunto de entrenamiento: subconjunto de datos etiquetados que se utiliza para entrenar un modelo.
Conjunto de prueba: subconjunto de los datos etiquetados que se utiliza para calcular métricas de evaluación del rendimiento del modelo. Este subconjunto no es utilizado para entrenar el modelo.
Modelo: se refiere al conjunto de parámetros entrenados a partir de los datos del conjunto de entrenamiento. El modelo puede ser de diversos tipos (ecuaciones matemáticas, árboles o grafos, reglas lógicas, entre otros) y debe ser definido previamente.
Algoritmo: serie de pasos o rutina que permiten entrenar un modelo, es decir, seleccionar los valores de los parámetros a partir de los datos del conjunto de entrenamiento.
Enfoques de aprendizaje automático
Dos importantes enfoques de aprendizaje que ayudan a comprender los tipos de aplicaciones existentes son el aprendizaje supervisado y no supervisado.
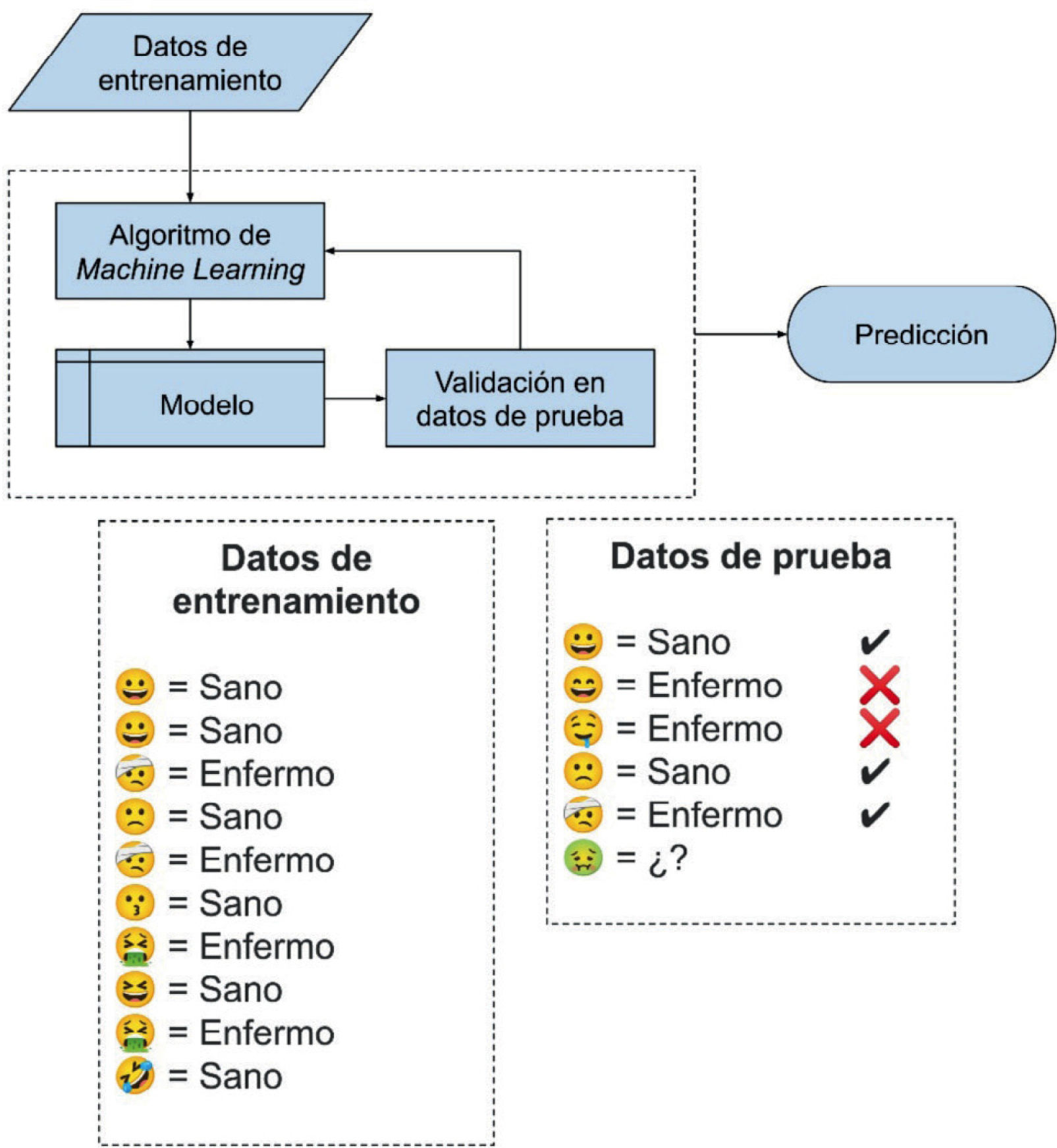
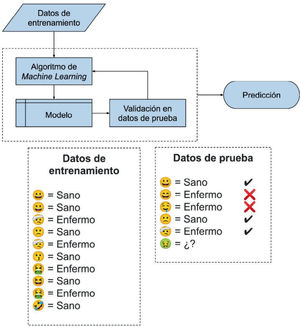
Aprendizaje supervisado: generación de un modelo que utiliza un conjunto de entrenamiento para predecir una variable objetivo. Un ejemplo de aprendizaje supervisado es utilizar imágenes de fondo de ojo para predecir retinopatía diabética en pacientes. Para entrenar un modelo que pueda “aprender” a predecir esta tarea, se requiere un conjunto de imágenes de fondo de ojo con etiquetas de condición normal y con retinopatía diabética (Fig. 1).
Esquema de aprendizaje supervisado. Los datos de entrenamiento son utilizados por el algoritmo de machine learning para generar un modelo, que es validado en los datos de prueba. Es posible iterar el proceso hasta obtener resultados satisfactorios. Finalmente, el modelo entrenado es utilizado para realizar predicciones.
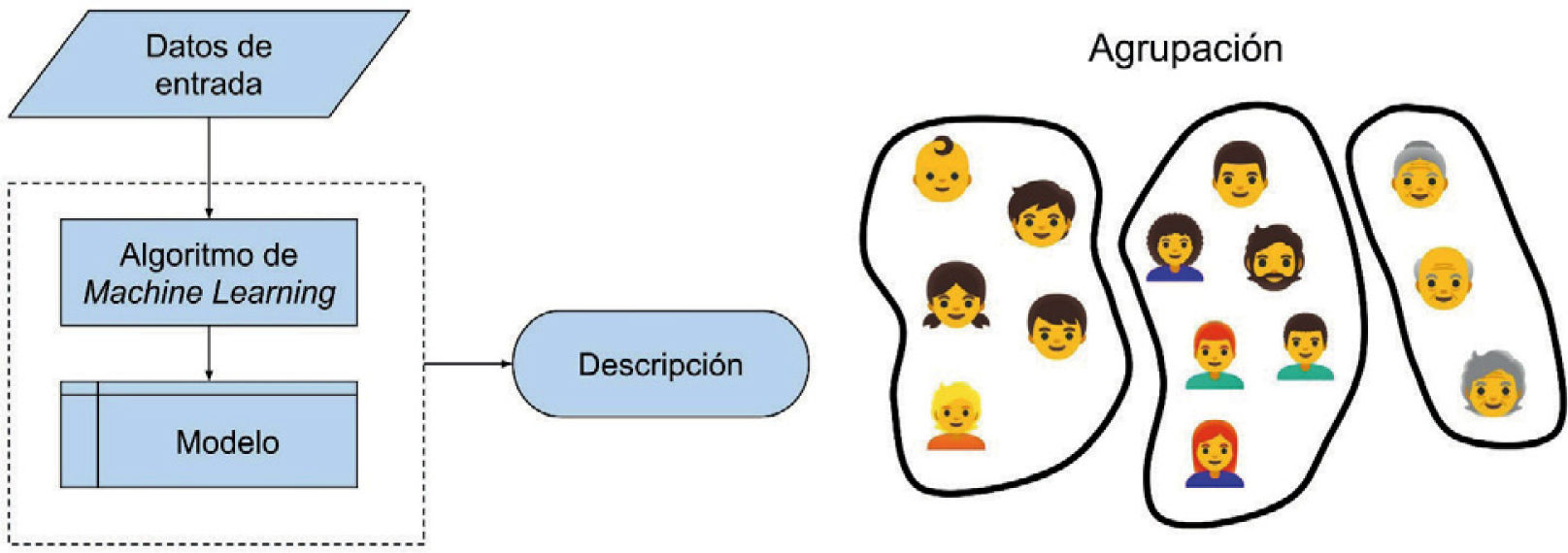
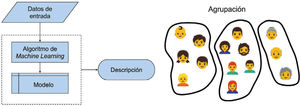
Aprendizaje no supervisado: entrenamiento de un modelo que utiliza datos sin etiquetas para realizar análisis descriptivo de los mismos (agrupación, asociaciones o detección de anomalías). Un ejemplo de aprendizaje no supervisado sería generar grupos o clúster de pacientes de acuerdo a un conjunto de variables, que permita focalizar ciertos tratamientos (Fig. 2).
3Técnicas de aprendizaje automáticoAlgunos algoritmos que se han utilizado extensivamente para realizar modelos de aprendizaje automático han sido Random Forest, Support Vector Machines y Redes Neuronales. Adicionalmente, el Deep Learning o aprendizaje profundo se utiliza en aplicaciones más actuales.
Random Forest: se refiere a un algoritmo que genera un modelo de varios árboles de decisión y cada uno de ellos tiene un voto en la predicción final. En la generación de árboles se utiliza un subconjunto de variables escogidas aleatoriamente para obtener variabilidad entre ellos.
Support Vector Machines: es un algoritmo que genera un hiperplano separador entre los datos con sus respectivas etiquetas. El objetivo es encontrar un hiperplano con la máxima separación y que cometa la menor cantidad de errores.
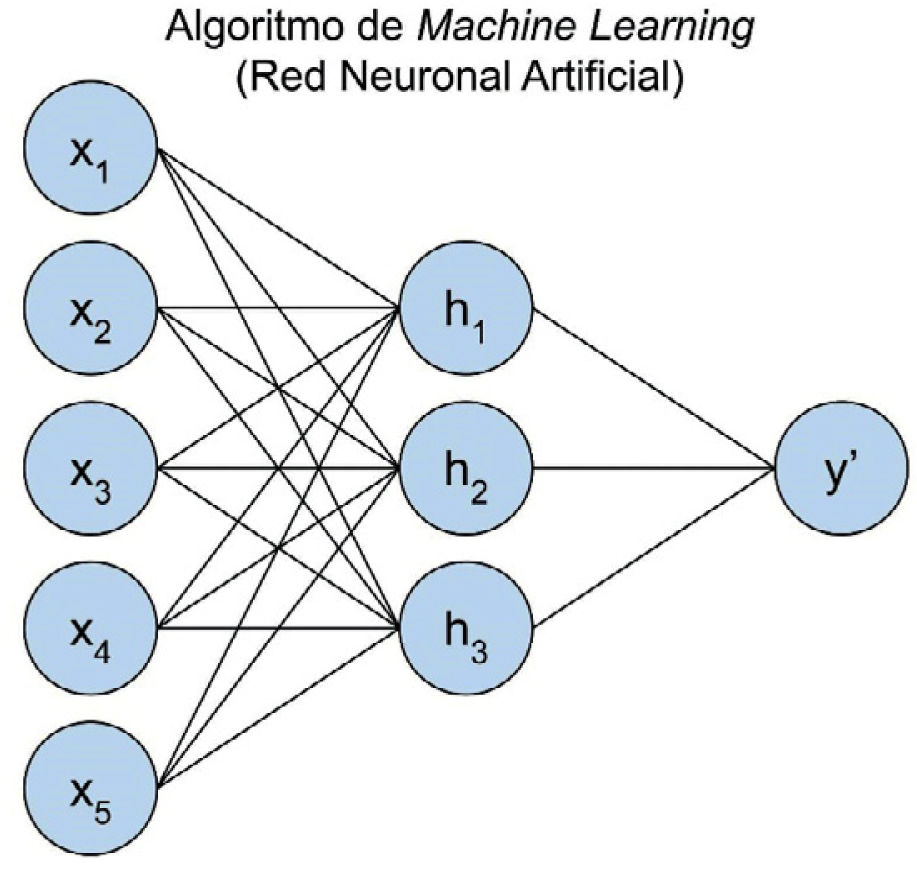
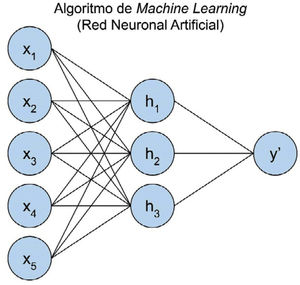
Redes Neuronales: técnica que genera un modelo basado en nodos o neuronas interconectadas, donde en cada una de ellas se aplica una función de activación a la multiplicación entre los parámetros y los valores de entrada (Fig. 3).
Deep Learning: Durante la última década el uso de redes neuronales se fue haciendo intensivo y nuevas arquitecturas comenzaron a surgir. La mayoría de ellas tienen una gran cantidad de neuronas y con varias capas de profundidad, por lo cual esta nueva sub-área fue denominada deep learning. Dentro de esta subárea de estudio se encuentran la mayoría de los avances actuales en torno a modelos predictivos en imágenes, texto, audio y video. Algunos ejemplos de arquitecturas relevantes son las Redes Neuronales Convolucionales (CNN, por sus siglas en inglés), utilizadas ampliamente en procesamiento de imágenes, o Transformers, utilizados en procesamiento de lenguaje natural.
3.1Base de datos en saludPara entrenar modelos de aprendizaje automático es necesario contar con datos. En el área de la salud son varias las iniciativas que buscan recopilar datos para que posteriormente puedan ser utilizados en investigación o aplicaciones, en particular, para aprendizaje automático. Dentro de las iniciativas existentes es posible destacar al banco de datos de registros clínicos y administrativos de la Universidad de Washington5 y el UK Biobank6. Adicionalmente, existen esfuerzos en la unificación de la terminología para que sea posible integrar datos de distintas fuentes. Uno de estos esfuerzos es SNOMED CT7, que busca ser la terminología clínica más comprehensiva del mercado.
4APLICACIONES DE APRENDIZAJE AUTOMÁTICO EN SALUD4.1Registros médicos electrónicos y datos administrativosEn la era de los grandes datos, el uso de registros médicos electrónicos y datos administrativos de salud se ha convertido en una fuente valiosa de información para entrenar modelos que puedan realizar predicciones. Los registros médicos electrónicos de buena calidad pueden ayudar a predecir la mortalidad intrahospitalaria, la readmisión de pacientes, el diagnóstico de enfermedades, y otras problemáticas. Por su parte, los datos administrativos pueden ayudar a generar modelos para mejorar y optimizar la gestión hospitalaria, como la predicción de inasistencia de pacientes, organización de horas médicas y de pabellón, etc. En esta sección se presentan algunas investigaciones que trabajan tanto con registros médicos electrónicos como con datos administrativos.
4.1.1Mortalidad intrahospitalariaLa mortalidad intrahospitalaria es definida como la muerte de un paciente una vez que se ha hecho un ingreso al hospital donde es atendido. Diversos estudios han intentado realizar una estimación certera de la mortalidad intrahospitalaria utilizando técnicas de aprendizaje automático. En general, los estudios intentan realizar una predicción con una anticipación entre 24 y 48 horas desde el ingreso del paciente hasta el deceso de este.
El trabajo de Ye et al.9 por ejemplo, tuvo como objetivo monitorear el riesgo de mortalidad con un sistema de alerta temprana en tiempo real en dos hospitales de Estados Unidos. Para entrenar distintos modelos de aprendizaje automático utilizaron datos de 42.484 pacientes entre 2015 y 2016, y la evaluación de los modelos se realizó con datos de 11.762 pacientes durante el 2017. A partir de la evaluación se identificó el algoritmo Random Forest como el de mejor desempeño. Las variables utilizadas para la predicción no solo incluyeron datos tradicionales, tales como signos vitales y datos de laboratorio, sino que también incluyeron historial médico como diagnósticos médicos e indicadores de utilización clínicos. El sistema logró predecir 99 pacientes del grupo de evaluación como de alto riesgo, de los cuales 40 fallecieron dentro de las 24 horas siguientes y 68 dentro de los 7 días siguientes. Estos resultados son comparativamente mejores que sistemas similares. Los autores mencionan que el sistema será integrado a los hospitales donde se realizó el estudio.
Di Castelnuovo et al.10, analizaron los factores de riesgo cardiovasculares y la mortalidad intrahospitalaria en cerca de 3.900 pacientes con COVID-19 en Italia. Mediante técnicas de aprendizaje automático se identificaron factores de riesgo asociados a la mortalidad. Para ello se entrenó un modelo de Random Forest con variables como edad, género, comorbilidades crónicas, proteína c-reactiva (CRP, por sus siglas en inglés), tasa de filtración glomerular estimada (eGFR por sus siglas en inglés), entre otras. Con ello fue posible identificar que las variables más relevantes para predecir la mortalidad intrahospitalaria son la eGFR y CRP, seguidas por la edad. Este estudio fue uno de los primeros realizados con grandes cantidades de datos al inicio de la pandemia del COVID-19, lo que permitió alertar a la comunidad científica de factores de riesgo en mortalidad intrahospitalaria.
4.1.2Diagnóstico de enfermedadesEl uso de registros médicos electrónicos con aprendizaje automático para determinar un puntaje de riesgo o diagnosticar enfermedades es cada vez más común en los sistemas de salud. Existen diversos estudios que implementan modelos con datos clínicos para dichas tareas. Por ejemplo, en el caso de la Diabetes Mellitus tipo 2, varios estudios se han focalizado en screening y diagnóstico para apoyar la toma de decisiones médicas11. Uno de estos estudios12 obtiene resultados con un excelente rendimiento. La investigación utiliza diversos grupos de variables extraídas de los registros médicos electrónicos, y posteriormente elimina variables correlacionadas y unifica algunas de ellas para generar 5 variables que son utilizadas para entrenar varios algoritmos de aprendizaje automático, siendo Random Forest el que obtiene el mejor resultado detectando 98% de los casos con la enfermedad. Las variables son extraídas de reportes de comunicación, reportes de alta, reportes de diagnóstico, exámenes y prescripciones médicas.
4.1.3Inasistencia de pacientesLos datos administrativos de salud también son útiles para resolver problemas que se presentan en la gestión de la salud. Uno de esos problemas es la inasistencia de pacientes a sus horas médicas. En una investigación13 se probaron diversos algoritmos de aprendizaje automático para lograr predecir el fenómeno. El mejor modelo logra detectar 82% de las citas con inasistencia utilizando el clasificador Naive Bayes. Las variables más relevantes para la predicción fueron la cantidad de días entre la programación de la hora y la realización de la cita, horas perdidas por el paciente con anterioridad, número de días desde la última hora, entre otras.
En Chile, la magnitud del problema de inasistencia de pacientes alcanza el 16,5% de las citas. Un estudio realizado bajo el proyecto FONDEF, “Soluciones tecnológicas, basadas en técnicas matemáticas avanzadas de aprendizaje de máquinas, para aumentar la eficiencia en la gestión hospitalaria”, logró detectar un 65% de las citas no atendidas en cuatro hospitales del Servicio de Salud Metropolitano Sur entre el 2018 y 202014.
4.2Imágenes médicasLa inteligencia artificial en el área de análisis de imágenes médicas está mostrando un papel importante y disruptivo. Los continuos avances tecnológicos han permitido introducir nuevas modalidades en consultas médicas, como la tomografía computarizada (TC), resonancia magnética (RM), tomografía por emisión de positrones (TEP), TEP/TC y TEP/RM. El análisis de grandes cantidades de datos de imagen de alta complejidad creados por estas modalidades se ha convertido en un gran desafío, tanto por la demanda en aumento de estos exámenes, y por el elevado tiempo dedicado en su análisis debido a la escasez de radiólogos15.
La disponibilidad de estas imágenes médicas ofrece diversas oportunidades, como por ejemplo, la caracterización de variaciones (normales y anormales) entre sujetos16, detección de anomalías en pacientes, descubrimiento temprano de progresión de enfermedades (biomarcadores)17, selección de tratamiento y predicción de estos (radiómica)18, y correlación de hallazgos en genotipo y fenotipo19. El análisis de estas imágenes, asistido por un computador, permite extraer y cuantificar la información relevante para apoyar la interpretación clínica20.
El análisis de imágenes médicas implica el cálculo de mediciones, mediante la cuantificación de sus diversas propiedades. La estimación de estos cálculos de forma manual por parte de personal médico puede estar sujeta a variabilidad de los observadores, lo que podría afectar significativamente la interpretación de resultados, reproducibilidad en estudios frente a otras variables o la sensibilidad que se requiere. Ante la necesidad de métodos más eficaces, fiables y validados, que también puedan emplear una cantidad masiva de datos, surgen los algoritmos de aprendizaje automático como un apoyo al análisis de imágenes médicas.
4.2.1Patología digitalLa patología digital incorpora la obtención, gestión, intercambio e interpretación de información patológica. Las imágenes corresponden a las capturas de tejido fijado sobre portaobjetos de vidrio a través del escaneo de alta resolución. Una aplicación de machine learning en esta área es la identificación del carcinoma de células escamosas de orofaringe21. Este trabajo se realizó con el objetivo de identificar células escamosas de orofaringe relacionado con el virus del papiloma humano, en donde los pacientes desarrollan enfermedades recurrentes como metástasis en el 10% de los casos, y los restantes pueden presenciar una morbilidad importante por el tratamiento, por lo que es fundamental poder identificar la presencia de tumores agresivos o indolentes. Los datos con los que se trabajaron corresponden a escaneos de portaobjetos teñidos con hematoxilina y eosina de una cohorte de micromatrices de casos. El clasificador denominado “QuHblC” predijo correctamente los resultados de 140 pacientes obteniendo un 87,5% de precisión. Se concluyó que, con pequeñas aplicaciones de hematoxilina y eosina, el clasificador QuHblC puede predecir fuertemente el riesgo de recurrencia. Con una validación prospectiva, este clasificador puede ser útil para estratificar a los pacientes en diferentes grupos de tratamiento.
4.2.2RadiologíaRadiómica es un método de extracción masiva de características de imágenes médicas nacido del trabajo de Lambin et al.22. Esta extracción se realiza a través de algoritmos de caracterización de datos de exámenes radiológicos como TC, RM, TEP/TC o TEP/RM. Este método tiene como objetivo descubrir patrones y/o características que a simple vista no son notorias para predecir pronósticos o respuestas terapéuticas para varios tipos de cáncer.
Un ejemplo de aplicación es un trabajo realizado en 2019 en cáncer de pulmón23. Los métodos manuales y semiautomáticos de segmentación de tumores para la extracción de características también están sujetos a la variabilidad de los observadores, lo que conduce a una sobreestimación o subestimación de las propiedades del tumor. Este trabajo tuvo como objetivo el uso de las características radiómicas centradas en cada píxel de los datos radiológicos obtenidos a partir de exámenes TC en 8 pacientes. Se empleó un set de datos con 25 características de textura para el entrenamiento de modelos de árboles de decisión. Los principales resultados indican que el modelo predice con precisión la ubicación del tumor en el conjunto de prueba (área bajo la curva ROC (AUC)= 83,9%).
4.3Texto clínicoEn la atención clínica del paciente, el texto juega un rol de suma importancia. Uno de los medios que el profesional de salud tiene para persistir el conocimiento generado en el proceso de cuidado del paciente es el texto en el registro clínico, siendo este medio el único por el cual el profesional personalmente puede volcar el conocimiento adquirido en el proceso de anamnesis y examen clínico24. El texto clínico es la fuente más rica de información existente en el registro clínico, por ende, es fundamental el desarrollo de herramientas para extraer información desde estas fuentes de datos. El texto es difícil de analizar, porque al contrario de las fuentes de datos estructuradas, como, por ejemplo, los resultados de laboratorio, no existe un modelo subyacente que gobierne la estructura de estos datos25.
El Procesamiento de Lenguaje Natural (PLN) es un área de intersección entre las ciencias de la computación y la lingüística que busca analizar de manera automática el lenguaje humano. Esta área se utiliza principalmente para extracción automática de información, traducción y transcripción automáticas de voz25. Para resolver estas tareas y tomando en cuenta la naturaleza no estructurada de los datos de texto, el estado del arte en PLN está basado en deep learning.
Los principales avances del PLN clínico se han originado en el lenguaje inglés y para poder transferir estos conocimientos a otros lenguajes se necesita una recopilación masiva de datos de texto del dominio en el idioma deseado26. El acceso a datos de texto clínico es un desafío en sí mismo, dada la naturaleza privada de los datos clínicos25. Por lo mismo, se necesitan esfuerzos conjuntos entre el área académica y la industria de la salud para poder avanzar en el conocimiento del PLN clínico al idear formas de extraer información clínica sin atentar contra la privacidad de los pacientes.
En general, las tareas más importantes que resuelve el PLN y que tienen aplicaciones importantes en medicina son la extracción de información, la clasificación de documentos y el análisis de voz24.
En la tarea de extracción de información buscamos sistematizar el conocimiento contenido en datos de texto para extraer piezas importantes que puedan aportarnos información sobre un fenómeno27. Dentro de esta tarea podemos destacar el reconocimiento de entidades médicas, en donde buscamos piezas de texto que pertenecen a una categoría predeterminada, llámese enfermedades, partes del cuerpo o cualquier otra categoría. Específicamente, alrededor de esta tarea se han organizado competencias que buscan seleccionar el modelo que mejor se comporte en un desafío específico y es desde estas tareas donde nacen muchas aplicaciones interesantes en medicina. La competencia ProfNER, por ejemplo, buscaba encontrar ocupaciones médicas dentro de texto libre no estructurado, específicamente en redes sociales28. En MEDDOCAN, por su parte, se buscaba detectar información sensible dentro de texto clínico no estructurado para realizar la anonimización automática de los documentos29. Finalmente, en eHealth-KD se buscaba detectar toda la información importante desde el punto de vista semántico, dentro del texto clínico y las relaciones entre cada mención30.
En la tarea de clasificación, buscamos asociar automáticamente una categoría a un documento de texto. Esta es una de las tareas más simples y más utilizada en el análisis de texto clínico31. Utilizando técnicas de clasificación de texto podemos analizar distintos tipos de texto clínico, tales como reportes de radiología para detectar patologías en reportes de tomografías computarizadas de cabeza32, certificados de defunción para detectar causas de muerte33, tuits para detectar infecciones por influenza34, entre otros.
El análisis de voz busca información relevante dentro de datos de audio de discurso humano. Con estas técnicas de procesamiento de lenguaje natural podemos detectar biomarcadores de voz para la enfermedad de Parkinson35, analizar disartria36, detectar COVID-19 a través de dispositivos móviles37, entre otras.
5APLICACIONES EN CHILEEn Chile, se han realizado avances significativos en pos de empujar los límites del estado del arte en PLN clínico en español. Para apoyar la optimización de la lista de espera, se desarrolló un detector de clasificaciones erróneas de pacientes con problemas de salud pertenecientes a las garantías explícitas de salud que estaban en la lista de espera incorrecta38. Para simplificar el análisis de los datos de texto libre no estructurado de la lista de espera chilena, se aplicó un método de resumen a través de nubes de palabras del contenido de las interconsultas incluidas en la lista de espera39. Se estimó la incidencia de psoriasis al extraer datos de diagnósticos desde los atributos de texto libre de la lista de espera chilena40. Desde nuestro país se han hecho aportes significativos al desafiante problema que es el PLN clínico en español.
Entre otros trabajos realizados en Chile se destaca una herramienta de detección automática de retinopatía diabética (RD) denominada DART desarrollada en 202141,42, en donde se analizan fotografías del fondo del ojo mediante IA para la implementación en el programa nacional chileno de detección de RD. Este estudio se realizó con una muestra de 1.123 exámenes oculares para diabéticos bajo un protocolo diseñado por una comisión compuesta por el Ministerio de Salud y especialistas de retinas, utilizando la metodología de doble ciego para oftalmólogos y la herramienta diseñada con IA.
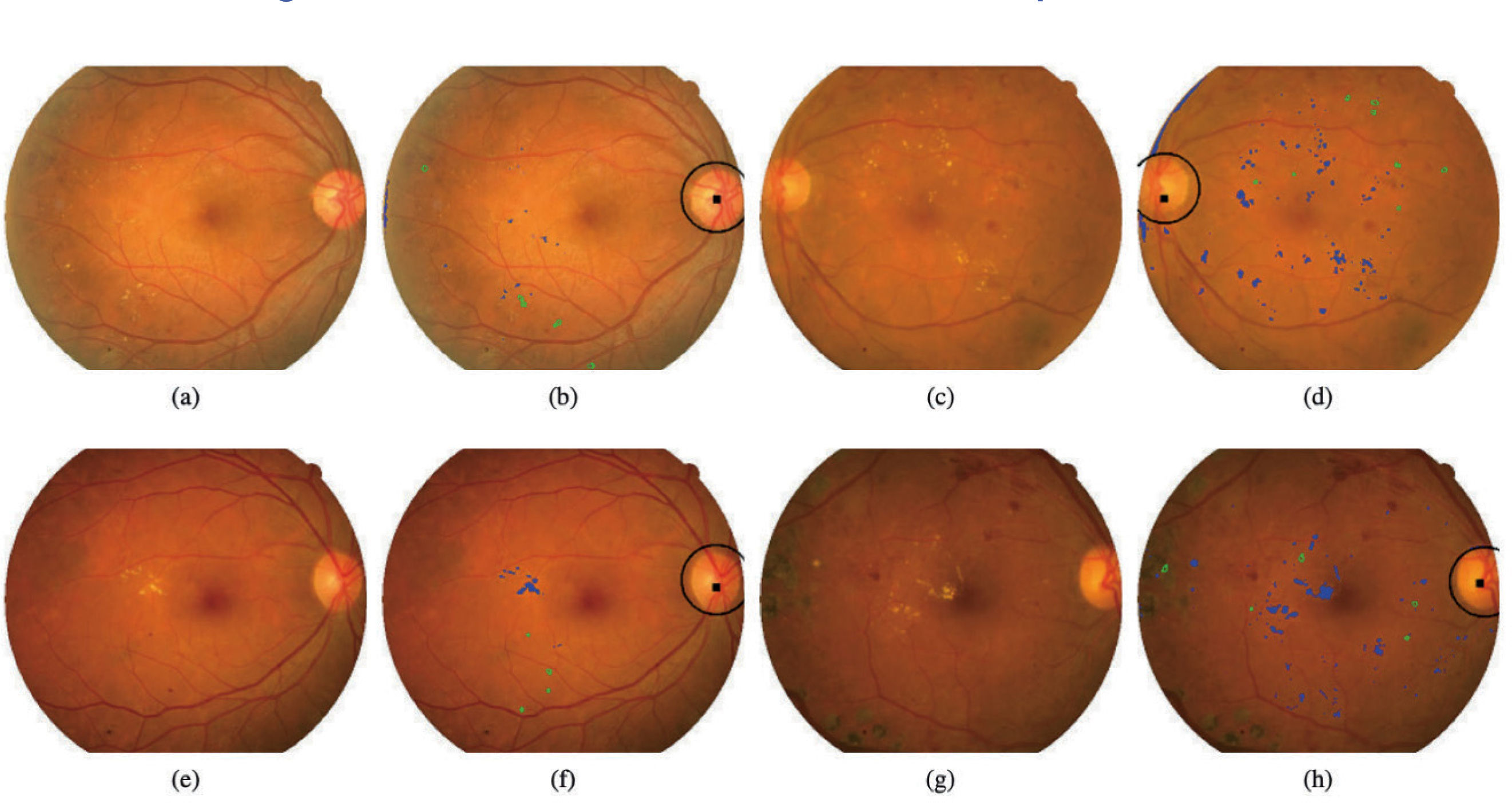
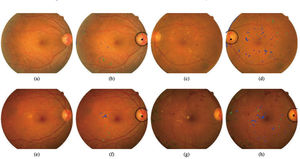
El trabajo consistió en la detección precisa de anomalías en las imágenes del fondo del ojo a través de 2 CNN. La primera CNN se utilizó para la detección de signos de edema macular diabético (EMD) y la otra CNN se utilizó para detectar signos de RD, en donde finalmente se ponderan ambas salidas para obtener la probabilidad de RD por ojo. Se obtuvo como resultado una sensibilidad de 94,6%, especificidad del 74,3% y valor predictivo negativo del 98,1%, concluyendo que DART es una herramienta válida para implementarse en el sistema de salud chileno. En la actualidad DART es una herramienta del sistema de salud chileno del modelo de atención en salud denominado Hospital Digital, donde busca prevenir la principal causa de ceguera en Chile en todos los pacientes con diabetes mellitus en control en los centros de atención primaria del país (Fig. 4).
Resultados de detección de retinopatía diabética. Las imágenes (a), (e), (c) y (g) corresponden a imágenes originales y las imágenes (b), (f), (d) y (h) muestran: localización de disco óptico y posición real (objeto circular); detección de lesiones (azul); detección de lesiones rojas (verde). Fuente: Arenas-Cavalli JT. et al.24.
El presente trabajo introdujo el área del aprendizaje automático, mostrando diversas aplicaciones en salud. Como se puede notar, existen investigaciones que utilizan aprendizaje automático y logran rendimientos considerables en tareas que antes estaban relegadas a ser realizadas por humanos. Este hecho abre una ventana de posibilidades para mejorar la eficiencia de los servicios y prestaciones de salud, brindando un apoyo a la decisión clínica.
No obstante, es importante considerar los aspectos éticos relacionados al uso de estas herramientas. Como indica el documento preparado por la Organización Mundial de la Salud43, se pueden identificar seis aspectos claves para un enfoque ético del uso de IA en salud:
- 1.
Proteger la autonomía humana (humanos deben mantener el control de los sistemas y la toma de decisiones)
- 2.
Promover el bienestar humano, la seguridad y el interés público (IA no debería producir daño y debería incluir medidas de mejoramiento continuo)
- 3.
Asegurar la transparencia e inteligibilidad (debería ser entendible para desarrolladores, clínicos, pacientes y supervisores. El diseño y despliegue debe ser suficientemente documentado)
- 4.
Responsabilidad (humanos deberían ser capaces de asegurar la calidad a partir de un conocimiento claro de las tareas que la IA está realizando)
- 5.
Inclusividad y equidad (garantizar un uso que no discrimine por edad, género, etnia, orientación sexual, u otra característica protegida por los derechos humanos)
- 6.
Sustentabilidad (consistente con los esfuerzos mundiales de reducir el impacto humano en el medio ambiente, así como anticiparse a posibles pérdidas de trabajo debido a la automatización)
Kohli y Geis8 establecen que los aspectos éticos de IA en salud pueden estar asociados a tres áreas principales: los datos, los algoritmos y las prácticas. En el caso de los datos utilizados, se debe considerar el consentimiento informado, la privacidad, la propiedad, la objetividad y la brecha de acceso a la información entre los que tienen o no los recursos para la gestión y análisis de estos. Por el lado de los algoritmos, se debe considerar la seguridad (mantener la confianza de los sistemas), transparencia (interpretabilidad de los modelos) y alineación de valores con el fin de optimizar los resultados en pos de los pacientes. Por último, considerar la ética en las prácticas, informando el código de conducta de las personas y organizaciones que participan en todo el ciclo de vida del desarrollo de estas herramientas, la innovación realizada, la investigación, el diseño, la construcción, la implementación, su paso a producción y la descontinuación de esta.
Esperamos que este artículo permita un acercamiento de profesionales de la salud al aprendizaje automático. Esta área es intrínsecamente interdisciplinaria44, y para potenciar su uso responsable se requiere un entendimiento común tanto de quienes desarrollan las aplicaciones como de quienes las usan y legislan sobre ellas.
FinanciamientoEste trabajo ha sido financiado por la ANID a través de los Fondos Basales para Centros de Excelencia FB210005 (Centro de Modelamiento Matemático), Fondecyt de Iniciación 11201250 (J. Dunstan) y Beca de Doctorado Nacional 21211659 (C. Aracena) y 21220200 (F. Villena). Además, la investigación conducida por J. Dunstan es apoyada por los Institutos Milenio ICN2021_004 (iHealth) e ICN17_002 (IMFD).
Declaración de conflicto de interésLos autores declaran no tener conflictos de intereses.