La ubicuitina (Ub) es una proteína pequeña la cual es utilizada por los organismos eucariontes para marcar proteínas, en la mayoría de los casos para que éstas sean posteriormente degradadas. La ubicuitinación ocurre en tres pasos sucesivos los cuales requieren de la acción de una enzima activadora, una enzima conjugadora y una ligasa de Ub. Una vez ubicuitinada la proteína seguirá un destino diferente de acuerdo a la topología de la ubicuitinación. Muchas de las proteínas marcadas por ubicuitinación serán degradadas por un complejo proteínico de 2.5 Mda conocido como el proteasoma 26S. Las plantas emplean ampliamente este mecanismo de degradación regulada de proteínas para modular procesos de crecimiento y desarrollo o bien, para responder ante situaciones adversas como puede ser una baja disponibilidad de agua o el ataque por patógenos. Durante la evolución las plantas han desarrollado diversas estrategias para defenderse ante la agresión por patógenos, sin embargo, estos organismos han logrado implementar herramientas que les permiten contrarrestar los mecanismos de defensa de las plantas, entre otras formas, los patógenos han logrado manipular el sistema Ub/proteasoma para poder infectarlas eficientemente.
Ubiquitin (Ub) is a small protein used to label proteins by eukaryotic organisms; in most cases labeled proteins will be degraded afterwards. Ubiquitination occurs in three sequential steps through reactions requiring the action of the ubiquitin-activating enzyme, the ubiquitin-conjugating enzyme and the ubiquitin ligase. After this, the target protein will follow a different fate according to ubiquitination topology. Most ubiquitinated proteins will be degraded in the 2.5 M protein complex termed the 26S proteasome. Plants employ this mechanism of regulated protein degradation to modulate developmental and growing processes as well as to respond to detrimental situations, like water deficit or pathogen attack. During the evolution plants have developed different strategies to cope against pathogen infection, however these organisms have acquired tools that allow them to counteract plant defense mechanisms. Among different ways to undermine plant resistance pathways, pathogens have now the ability to manipulate the Ub/proteasome system to efficiently infect them.
Debido a su naturaleza sésil, las plantas están constantemente sujetas a situaciones medioambientales adversas, debido a ello, han desarrollado mecanismos que les permiten subsistir cuando se presentan condiciones de crecimiento desfavorables. Gran parte de estos mecanismos de defensa involucran la activación de genes cuyas funciones contribuyen a contender con medios hostiles. La activación de genes de defensa ante diferentes tipos de estrés inicia con la percepción de un estímulo particular que se transmite a través de una cascada de señalización de diferentes componentes que incluyen la movilización de iones, la generación de especies de oxígeno reactivas, la fosforilación de proteínas y la activación de factores de transcripción, cuya consecuencia final es la síntesis de proteínas que le permitirán a la planta contender con un medio adverso. Las respuestas a diferentes tipos de estrés en las plantas transitan por vías comunes de señalización. Por ejemplo, el análisis del perfil de transcripción de genes inducidos por estrés biótico muestra un gran número de genes comunes inducidos por varios tipos de estrés abiótico[1], sugiriendo una compleja red regulatoria de interacciones en la respuesta de las plantas a distintas clases de estrés ambiental. El control de los procesos celulares está mediado en gran parte a través de modular la concentración de proteínas regulatorias. Esto puede ocurrir, o bien, controlando su síntesis, pero también regulando su degradación. A diferencia del estudio de la síntesis y ensamblaje de proteínas que ha ocurrido por un largo tiempo, el correspondiente a los mecanismos de degradación de estas moléculas es más reciente y sólo hasta los últimos años del siglo pasado y los primeros de éste se ha empezado a reconocer la importancia de estos procesos catabólicos para la vida y la muerte de las células. Uno de los sistemas más importantes en el control de la degradación de proteínas es el sistema ubicuitina/proteasoma (SUP), este sistema regula múltiples procesos de desarrollo y respuestas al medio ambiente en los eucariontes[2,3]. La ubicuitinación es una forma de modificación postraduccional que regula, no sólo la estabilidad, sino que también la actividad y la localización de proteínas.
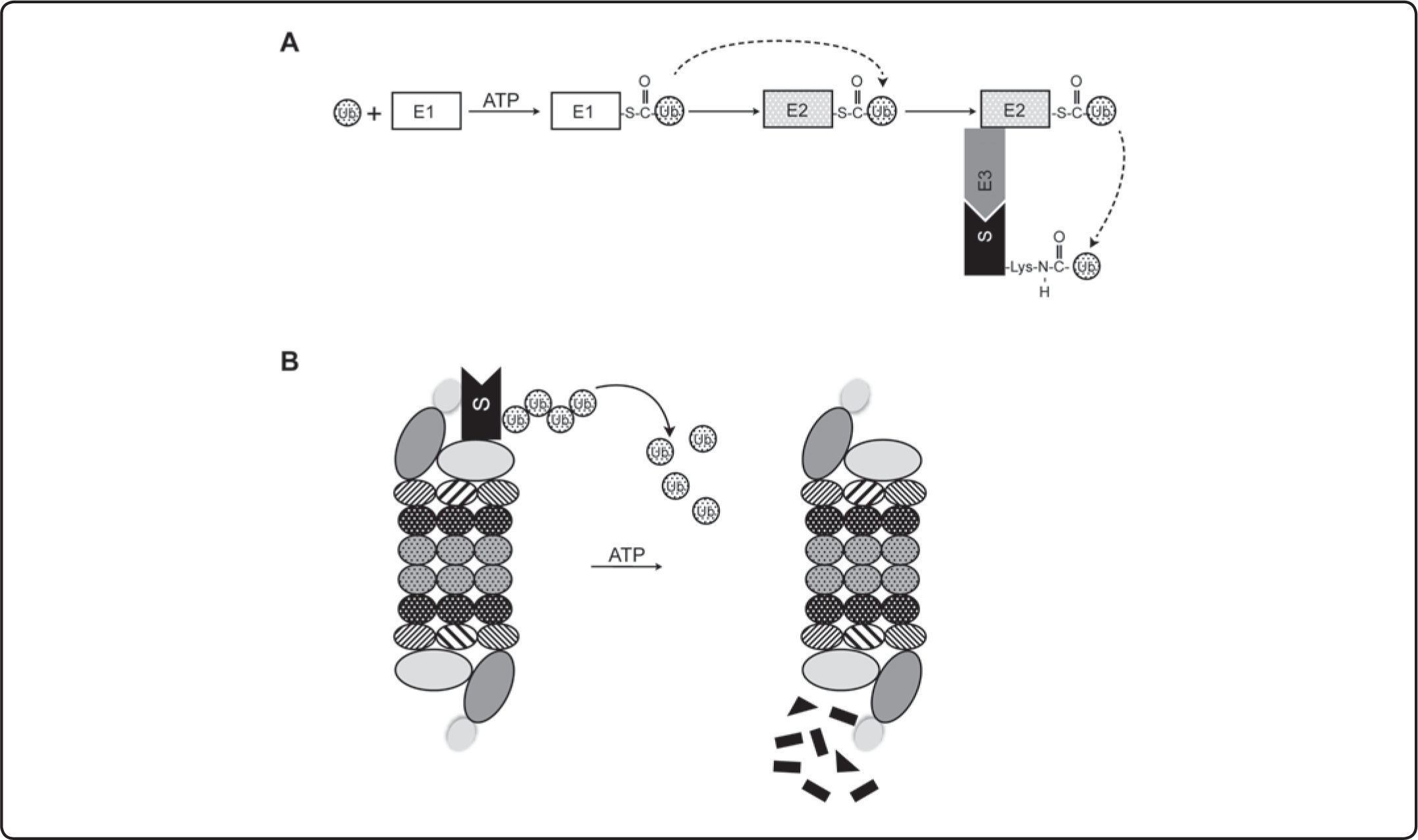
La degradación de proteínas por el SUP ocurre a través de dos pasos sucesivos: primero la proteína a degradar es conjugada al polipéptido ubicuitina (Ub) (Figura 1A), posteriormente la proteína poliubicuitinada es reconocida por una proteasa multicatalítica denominada proteasoma 26S, quien la degrada (Figura 1B)[4,5].
El sistema ubicuitina/proteasoma. A) Cascada de ubicuitinación (ver descripción en el texto). B) Degradación de la proteína poliubicuitinada por el proteasoma 26S. Subunidades específicas del proteasoma tienen la capacidad de reconocer proteínas poliubicuitinadas y también de remover la cadena de Ub. Finalmente, la proteína es degradada por las tres actividades proteolíticas. La energía proporcionada por el ATP se utiliza para desdoblar y transportar a la proteína que será degradada.
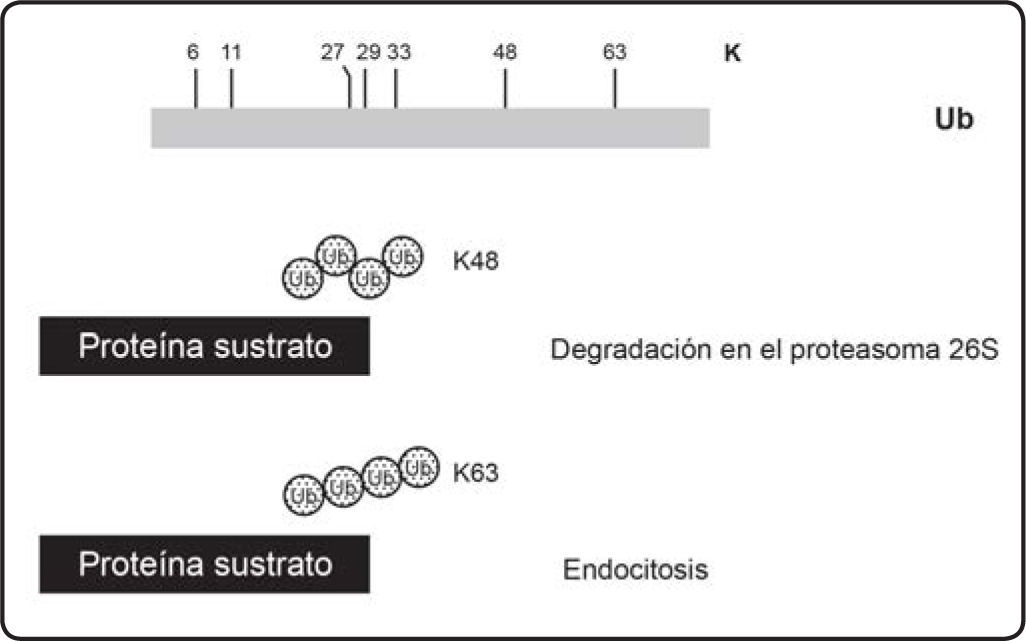
La Ub es una proteína pequeña de sólo 76 aminoácidos (8 kDa) y como su nombre lo indica esta molécula se encuentra presente y muy conservada en todos los eucariontes en los que hasta ahora se le ha buscado[6]. Además de su papel en la degradación regulada de proteínas, la Ub también es una señal en otros procesos como el transporte vesicular de proteínas, la internalización de receptores localizados en la membrana plasmática, la modificación de histonas y la reparación del ADN, etc.[7] La Ub puede ser conjugada a la proteína blanco, ya sea como monómero o bien como cadenas de Ub de longitud variable. Cada residuo de Ub tiene la capacidad de formar un enlace isopeptídico con otra Ub en el cual la glicina del extremo carboxilo se une covalentemente con una lisina de otra molécula de Ub. Como la molécula de Ub tiene 7 lisinas (posiciones 6, 11, 27, 29, 33 48 y 63), las cadenas de Ub pueden adoptar diferentes topologías dependiendo de la unión de la glicina a cualquiera de las 7 lisinas (Figura 2)[7].
La cadena de poliubicuitina puede tener diferentes topologías. La molécula de Ub tiene 7 lisinas (K), las cuales tienen la capacidad de formar un enlace isopeptídico con la glicina C-terminal de otra Ub. La topología de la cadena de poliubicuitina determinará el destino de la proteína blanco. Cadenas de lisina 48 serán preferentemente degradadas en el proteasoma y aquéllas con modificaciones de lisina 63 serán, en una gran proporción de los casos, dirigidas a procesos endocíticos.
En los animales se han detectado la formación de cadenas de todas las lisinas[7], al igual que en la planta modelo Arabidopsis thaliana, con excepción de la Lys27[8]. Es posible la generación de cadenas homogéneas en las cuales se forma un solo tipo de enlace y también es posible que se construyan cadenas ramificadas formadas por enlaces de varios tipos. El destino final de la proteína blanco dependerá de la conformación de la cadena de Ub[7]. Por ejemplo, cadenas conectadas a través de lisina 48 marcan proteínas que serán degradadas en el proteasoma 26S y las marcadas por lisina 63 son dirigidas a procesos de endocitosis (Figura 2)[9] o se emplearán en la señalización intracelular y la reparación del ADN[10]. Para el caso de las proteínas que serán degradadas en el proteasoma al menos 4 moléculas de Ub son necesarias para su reconocimiento por éste[11]. En las células de mamíferos se ha encontrado que existen factores solubles que impiden que las proteínas marcadas con cadenas de Ub lisina 63 se asocien al proteasoma 26S. Por otra parte, otros factores promueven la asociación al proteasoma de proteínas marcadas con Ub lisina 48. De esta forma la célula es capaz de regular el modo en que habrán de degradar sus proteínas[12].
Ubicuitinación de proteínasLa Ub es unida al grupo å-NH2 de una lisina interna de las proteína blanco en una cascada de conjugación que ocurre en tres pasos sucesivos (Figura 1A). La Ub es primero activada en una reacción dependiente de ATP por la enzima activadora de Ub o E1. Una cisteína catalítica en E1 es usada para formar un intermediado Ub-S-E1 con un enlace tioéster de alta energía entre la cisteína y el grupo carboxilo de la glicina del C-terminal de la Ub. Mediante una reacción de trans-esterificación el complejo Ub-S-E1 transfiere la Ub a la cisteína del sitio activo de la enzima conjugadora de Ub, también llamada E2, formando un complejo E2-S-Ub. Finalmente, E2-S-Ub transfiere la Ub a la proteína sustrato (S) en una reacción facilitada por la ligasa de Ub o E3[5]. Esta cascada de conjugación de Ub puede entonces repetirse múltiples veces hasta formar una cadena de poli-Ub.
Las plantas codifican solamente para una o dos proteínas E1 que se expresan en la mayoría de los tejidos[13]. El número de proteínas E2 es mayor, por ejemplo en A. thaliana existen 36 isoformas que forman 12 grupos[14]. Esto indica un mayor grado de especificidad con respecto al patrón de expresión específico de tejido o en respuesta a un estado de desarrollo y también en cuanto al reconocimiento de diferentes E3. Por otra parte, la diversidad de E3 en las plantas es enorme, contándose alrededor de 1,500 de éstas en el genoma de A. thaliana[15]. Esta gran diversidad de E3 sugiere que A. thaliana y, en general las plantas, utilizan la ubicuitinación para regular un gran número de procesos durante su vida. Baste con decir para apoyar esto último que más del 5% del genoma de A. thaliana codifica para proteínas del SUP[8]. De acuerdo a lo anterior, se ha demostrado la participación del SUP durante el desarrollo y crecimiento, en la respuesta a hormonas y en un gran número de respuestas al medio ambiente.
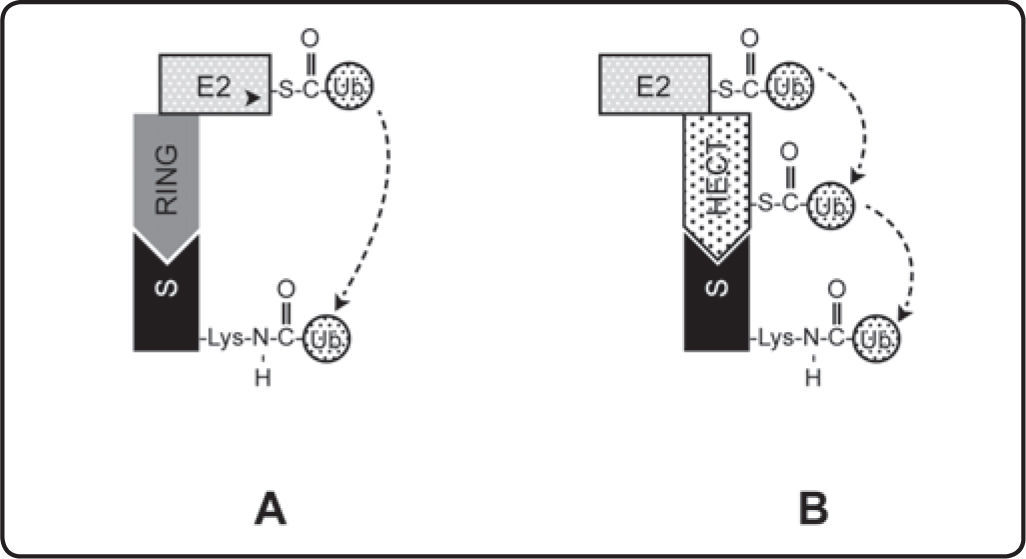
Existen tres clases principales de E3s: RING (Really Interesting New Gen), HECT (Homology to E6-Associated Carboxyl-Terminus) y la de tipo caja U. Además de sus características estructurales estas E3 se diferencian por el mecanismo de transferencia de Ub al sustrato (Figura 3). Las E3 de tipo RING y caja U no parecen tener actividad catalítica y funcionan como puentes para la transferencia de la Ub directamente del complejo E2-S-Ub a la proteína sustrato (Figura 3A). El motivo denominado RING consta de 40-60 aminoácidos y es un tipo particular de dedo de zinc que tiene residuos de cisteína e histidina que coordinan 2 átomos de zinc[16]. El dominio RING es esencial para la unión a E2. Por su parte el dominio caja U está relacionado estructuralmente al dominio RING, pero en lugar de los sitios de unión a zinc existen residuos cargados y polares muy conservados que mantienen la estructura mediante la formación de puentes de hidrógeno. Al igual que el dominio RING, el dominio caja U tiene la capacidad de unir a E2. A diferencia de las E3 de tipo RING o U-box, las de tipo HECT son aceptores de la Ub del complejo E2-S-Ub, formándose primero un complejo E3-S-Ub, que transfiere la Ub a la proteína blanco (Figura 3B)[17]. Las E3 de tipo RING a su vez se dividen en monoméricas y, como lo indica su nombre, están formadas por una única proteína con la capacidad de unir a E2 y a la proteína sustrato.
Diferentes formas de transferencia de la Ub al sustrato. A) Ligasas de Ub del tipo RING (o del tipo caja U) sirven sólo como puentes para la transferencia de la Ub de E2 a la proteína sustrato. B) Las ligasas de Ub del tipo HECT primero reciben a la Ub de E2 y posteriormente la transfieren a la proteína sustrato.
El otro grupo de E3 de tipo RING lo conforman las llamadas multiméricas o también ligasas Culina-RING (CRLs por su nombre en inglés: Cullin-RING ligases). Éstas reciben este último nombre porque además de una proteína con dominio RING, poseen alguna de las diferentes culinas descritas. La culina es una especie de andamio que permite el ensamblaje del complejo E3[15]. El genoma de A. thaliana codifica para 5 culinas funcionales pertenecientes a tres tipos: culina 1 y culina 2, ambas del tipo 1, culina 3a, culina 3b y culina 4. Existen distintos complejos CRLs formados, por al menos una proteína con el dominio RING, una culina y por una proteína adaptadora capaz de dar especificidad al complejo por un sustrato particular. Por ejemplo el complejo CRL denominado SCF, está constituido por 4 proteínas: SKP1 (ASK1 en Arabidopsis), culina 1, una proteína con caja F y Rbx1 (la proteína con el motivo RING). La proteína con la caja F es la adaptadora y, por tanto, la responsable de reclutar a la proteína que será ubicuitinada[4,5]. La función de la proteína SKP1 es de servir de puente entre culina 1 y la proteína con caja F. En las plantas se han descrito varias proteínas con caja F que participan en el control de procesos importantes, entre ellos las respuestas de defensa ante el estrés, la respuesta a hormonas, así como a múltiples procesos de desarrollo[3]. Un análisis de posibles proteínas con caja F en el genoma de A. thaliana revela la presencia de 724 de ellas[18].
En las plantas además del complejo SCF, que como se dijo contiene a la culina1, existen otros CRLs basados en culina 3 y culina 4. El complejo BTB está formado por culina 3, una proteína adaptadora del tipo BTB/POZ y Rbx1. En el genoma de A. thaliana se han identificado 80 diferentes proteínas adaptadoras BTB/POZ. Por su parte el complejo DWD está compuesto por culina 4, una proteína adaptadora de tipo DWD, una proteína que conecta a culina 4 con la proteína DWD, además de Rbx1. Un análisis del genoma de A. thaliana nos revela que existen 85 genes que codifican para las proteínas adaptadoras DWD[15]. Como se puede ver, el número de proteínas adaptadoras en A. thaliana es enorme lo cual sugiere que un número similar de proteínas debe ser regulada por un proceso de ubicuitinación.
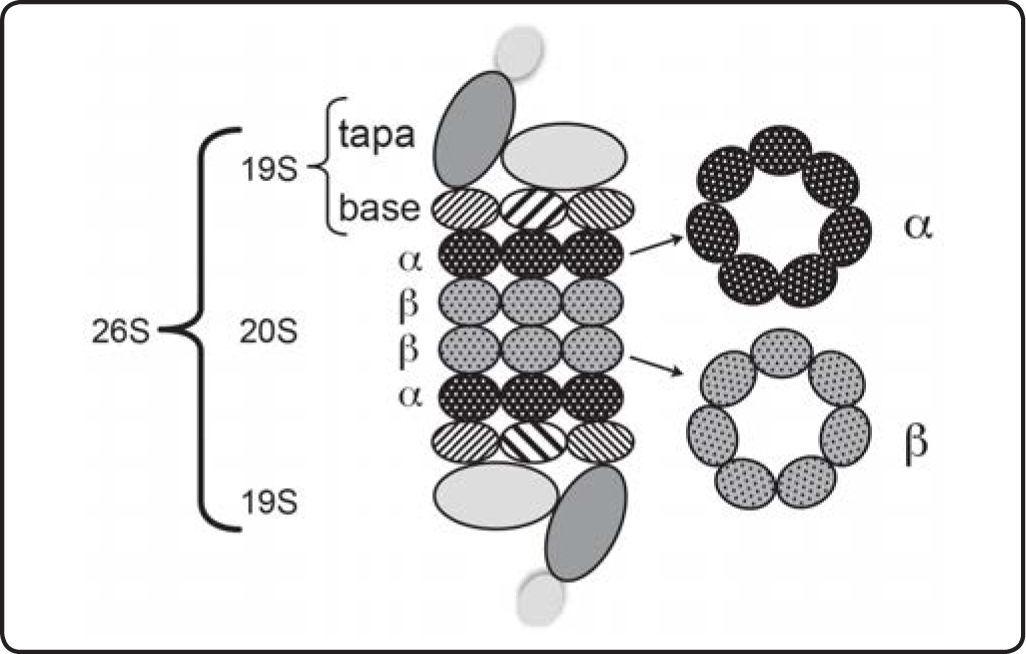
El proteasoma 26SEl proteasoma 26S es un enorme complejo de 2.5 Mda que se localiza en el núcleo y el citoplasma de todas las células eucariontes y su estructura se encuentra altamente conservada entre estos organismos. El proteasoma 26S está compuesto por dos subcomplejos: una partícula central 20S (CP) en donde se lleva a cabo la proteólisis, y una partícula reguladora 19S (RP) que prepara al sustrato para ser internado y degradado en la CP (Figura 4). La CP es una estructura con forma de barril compuesta por cuatro anillos heptaméricos apilados que forman una cámara proteolítica. Los anillos externos están formados por las subunidades α y los centrales por subunidades β[19,20]. La unión de los cuatro anillos forma una cavidad central en la cual tres de las subunidades β presentan actividad proteolítica: β1, β2 y β5[20]. Las subunidades α están encargadas de controlar la entrada de la proteína blanco en la cavidad proteolítica y de regular las interacciones entre la CP y la RP. Para la degradación de los sustratos ubicuitinados se necesita que la RP esté unida a la CP. La RP a su vez, está formada por la base y la tapa. La base está conformada por 9 subunidades, 6 de ellas con actividad de ATPasa, con una función parecida a la de las chaperonas de desdoblar a las proteínas que serán degradadas. Por su parte, la tapa está constituida por 12 subunidades que interaccionan con las subunidades α de la CP y funcionan abriendo la cavidad central e introduciendo los sustratos en ésta[19,21]. La RP realiza además las funciones de reconocer la cadena de poliubicuitina y unir selectivamente a las proteínas a degradar y de remover las cadenas de Ub con su actividad de desubicuitinasa[19]. Además de las subunidades ya mencionadas, otras proteínas se asocian al proteasoma 26S en condiciones específicas para regular su estructura y su función. La consecuencia de esto es que la población de proteasomas en la célula es diversa y dinámica[21,22]. En las plantas la existencia de más de un gen capaz de codificar para la mayoría de las subunidades que lo conforman es un factor adicional que agrega complejidad al proteasoma 26S en estos organismos[21].
Estructura del proteasoma 26. El proteasoma 26S está formado por 2 subunidades 19S y una 20S. La subunidad 19S a su vez está formada por una base y una tapa. La subunidad 20S está formada por 4 anillos, los externos contienen 7 subunidades α (á1- α7) y los internos 7 subunidades β (β1-β7). Las proteínas que serán degradadas son primero desubicuitinadas y desdobladas por la subunidad 19S para posteriormente llegar a la cavidad central formada por los cuatro anillos de la subunidad 20S en donde son degradadas.
Como se ha mencionado, cuando las plantas se enfrentan a situaciones medioambientales adversas deben de responder activando mecanismos que les permitan adaptarse y/o defenderse ante estas condiciones hostiles. También se dijo que entre los mecanismos empleados por las plantas para contender con diversos tipos de estrés se incluye el control de la abundancia de ciertas proteínas. Esto último tendrá como consecuencia el cambio en los patrones de expresión de ciertos genes, lo que al final ayudará a incrementar las oportunidades de sobrevivencia de la planta. En esta parte se revisarán algunos ejemplos de vías de defensa de las plantas que involucran al SUP para su regulación.
Mecanismos de defensa de las plantasLas plantas son constantemente atacadas por distintos organismos como hongos, bacterias, insectos, nematodos, virus, etc. Para defenderse de éstos, las plantas son capaces de desplegar una serie de mecanismos de defensa, algunos específicos y otros generales. En la actualidad existe suficiente evidencia para involucrar al SUP como parte de los mecanismos de defensa. Cabe aquí agregar que los patógenos han adquirido también la habilidad para utilizar y/o manipular el SUP de la planta en su propio beneficio.
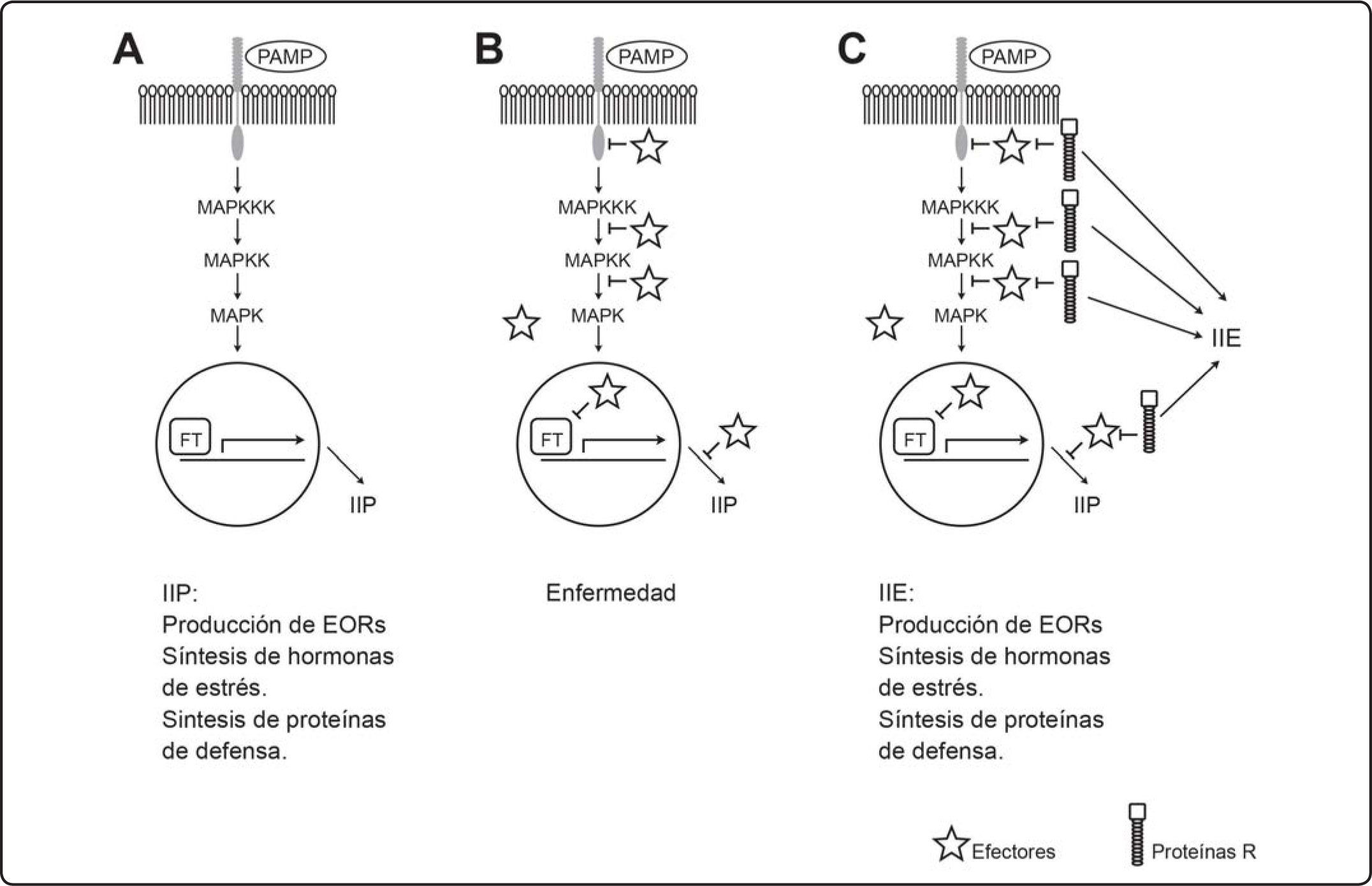
Entre los mecanismos empleados por las plantas se encuentran las barreras químicas o físicas formadas previamente al ataque del patógeno como pueden ser compuestos con actividad tóxica en contra del organismo agresor o la presencia de espinas, etc. También existen mecanismos cuya activación depende de la presencia del patógeno. Las plantas poseen dos maneras de reconocerlo, inicialmente la planta es capaz de detectar ciertas moléculas esenciales del mismo conocidas como patrones moleculares asociados al patógeno (PAMPs por su nombre en inglés: pathogen-associated molecular patterns), entre éstos se encuentran la flagelina bacteriana o la quitina presente en la pared de los hongos[23]. Los PAMPs son reconocidos por receptores en la membrana plasmática de la célula vegetal, muchos de estos receptores, además de un dominio extracelular para el reconocimiento del PAMP, poseen un dominio intracelular con actividad de cinasa de proteínas (Figura 5). El evento de reconocimiento activa una primera respuesta de defensa conocida como “inmunidad inducida por PAMPs” (IIP) o también como resistencia basal, ésta consiste en la activación de una cascada de eventos de fosforilación de proteína, influjo de Ca2+, la producción de especies de oxígeno reactivas (EORs), la síntesis de hormonas asociadas a las respuestas a estrés como ácido salicílico (SA), ácido jasmónico (JA) o etileno (Et) y, finalmente, la transcripción de genes que codifican para proteínas de defensa. Por su parte, los patógenos han desarrollado mecanismos para evadir la respuesta de defensa de las plantas, para ello cuentan con proteínas denominadas efectores, introducidas de diversas formas a la célula vegetal, una vez dentro los efectores pueden interferir con la IIP y, de esta manera, provocar los síntomas de enfermedad en la planta (Figura 5).
Percepción de patógenos y despliegue de mecanismo de defensa en las plantas. A) Mediante receptores en la membrana plasmática que reconocen componentes esenciales del patógeno (PAMPs) la célula vegetal detecta al organismo agresor, esto desencadena una cascada de señalización que culmina con la respuesta de defensa basal o IIP. B) Los patógenos interfieren con la IIP mediante la introducción de efectores en la célula vegetal y de esta forma adquieren la capacidad de volverse virulentos. C) Si la planta posee proteínas de resistencia (R) específicas de un efector particular, reconocen la interacción del efector con su blanco celular y de esta manera inducen la IIE, esta última es una respuesta más rápida y robusta que la IIP.
Como respuesta a la acción de los efectores las plantas han desarrollado otro mecanismo de defensa denominado “inmunidad inducida por efectores” (IIE). Esta vía de defensa depende de la presencia de proteínas de resistencia (R) capaces de reconocer la presencia de un efector específico (Figura 5). Esto implica que la planta debe tener a la proteína R específica capaz de reconocer a un efector particular[23,24], entonces se dice que la interacción es incompatible, que el patógeno es avirulento y la planta resistente. Si por el contrario, la planta no posee dicha proteína R, la interacción será compatible, el patógeno virulento y la planta susceptible. Los eventos desencadenados en la IIE son semejantes a aquéllos producidos en la IIP, sin embargo, la IIE es más prolongada y más robusta, además casi siempre incluye lo que se conoce como la respuesta de hipersensibilidad (RH) que es un tipo de muerte celular programada en donde las células más cercanas al sitio de infección mueren con la finalidad de evitar la propagación del patógeno. El proceso de muerte celular no es exclusivo de la IIE, ya que se ha reportado que también algunos PAMPs son capaces de inducir muerte celular, sin embargo, es más común encontrarlo en la IIP[24].
Participación del SUP en la resistencia de las plantas a patógenosEl primer tipo de evidencia que apoya la participación del SUP en la respuesta de defensa de las plantas es la referente al incremento en los niveles de mensajeros y/o proteínas de genes que codifican para proteínas del SUP. Por ejemplo, la infección por el virus del mosaico del tabaco o el tratamiento con hormonas involucradas en la respuesta a patógenos como SA, JA o Et induce un incremento en la acumulación de E1 en tabaco[25]. También los transcritos de distintas E3 muestran incrementos bajo este tipo de tratamientos[26,27]. La lista de genes que codifican para componentes del SUP y de sus correspondientes proteínas que responden al estrés biótico es muy grande e incluye a prácticamente todos los participantes en este sistema desde genes de Ub, del sistema de ubicuitinación, hasta de las subunidades del proteasoma. Por ejemplo, criptogein, un polipéptido elicitor secretado por el oomiceto Phytophthora cryptogea el cual induce respuestas de defensa en tabaco es capaz de inducir la acumulación de las proteínas del proteasoma α3, α6 y β1, así como de sus correspondientes mensajeros[28]. Otro tipo de evidencia proviene del análisis de mutantes, por ejemplo se encontró que la deleción del gen UBA1, que codifica para una de las dos E1 presentes en A. thaliana, provoca una mayor susceptibilidad a patógenos[29]. Todo esto nos sugiere que ante el ataque de patógenos la célula vegetal requiere de los componentes del SUP para contender exitosamente contra organismos agresores.
Como se ha mencionado, las E3 son los componentes más diversos del SUP y los que van a seleccionar a la proteína para ser ubicuitinada, esto implica entonces que la participación de E3 en la especificidad del proceso debe ser preponderante sobre la que pudieran tener los otros componentes en la cascada de ubicuitinación. No es de extrañar entonces la abundancia de estudios sobre el papel de las E3 en las respuestas de defensa de las plantas, aquí revisaremos algunos de los ejemplos de E3 que participan en los distintos pasos del proceso.
En algunos casos sólo se ha identificado la posible participación de E3s porque los niveles de sus transcritos se incrementan bajo estrés biótico. Este es el caso del gen AtFBS1 de A. thaliana y su ortólogo PvFBS1 de frijol. Los mensajeros de ambos genes se acumulan por la infección por patógenos de las respectivas plantas, sin embargo también lo hacen por la aplicación de distintos tipos de estrés como el salino o el osmótico, así como por la herida. Incluso el tratamiento con hormonas implicadas en las respuestas a estrés induce la acumulación de estos mensajeros y sugiere que las proteínas con caja F AtFBS1 o PvFBS1 participan en una respuesta general al estrés biótico y abiótico en las plantas[26].
Existen casos también en donde, además del incremento en la cantidad de transcrito, se han podido analizar fenotipos relacionados al estrés en plantas con alteraciones en la expresión de genes que codifican para E3s. Así tenemos el caso del gen OsDFR1 de arroz que codifica para una proteína con caja F y es inducido tanto por la infección del hongo Magnaporthe grisea[30] como por el tratamiento de la planta con la fitohormona ácido abscísico la cual tiene un papel importante en la respuesta de las plantas ante el estrés relacionado con la baja disponibilidad de agua. Cuando este gen es sobreexpresado en tabaco, las plantas transgénicas generadas tienen una mayor resistencia al virus del mosaico del jitomate (ToMV) y a la bacteria Pseudomonas syringae pv tabaci, e indica que OsDFR1 participa en una vía general de defensa en contra de patógenos y posiblemente en una respuesta general al estrés. Un hipotético blanco de esta proteína con caja F podría ser un represor de la respuesta de defensa, ya que en las plantas transgénicas la aplicación de SA o la infección con ToMV produce una mayor elevación en la expresión de algunos genes de defensa cuando se compara con plantas no transgénicas bajo los mismos tratamientos[30]. Otro caso interesante es el de la proteína ACIF1 que también tiene una caja F que ha sido implicada como un regulador positivo de la RH y de la resistencia mediada por varias proteínas R en tabaco y jitomate. El transcrito de ACIF1 se acumula en respuesta al efector Avr9 de Cladosporium fulvum y también en respuesta a la herida. El silenciamiento del gen de tabaco inhibe la RH inducida por varios efectores, así como la resistencia a TMV mediada por la proteína de resistencia N[31].
Otro ejemplo con características diferentes a las anteriores por su localización subcelular son las proteínas CaRING1 de chile (Capsicum annum)[32] y la proteína RING1 de A. thaliana[33], ambas se localizan en la membrana plasmática y parecen ser requeridas para las respuestas de defensa a patógenos, incluida la RH. El mensajero de CaRING1 se acumula en respuesta a la infección por la bacteria Xanthomonas campestris pv vesicatoria (Xcv). Se ha demostrado que su actividad de ligasa de Ub se requiere para la inducción de la RH. Plantas de chile silenciadas para CaRING1 son más susceptibles a la infección por Xcv y tienen una RH disminuida, así como una menor expresión de genes de defensa. Como se esperaría, la sobreexpresión de CaRING1 en Arabidopsis induce un incremento en la resistencia a patógenos. Por su parte el transcrito de RING1 se encuentra en bajos niveles en todos los tejidos de A. thaliana y se incrementa por la infección de una cepa avirulenta de la bacteria P. syringae o el tratamiento con la toxina fúngica fumonisina B1 FB1. El silenciamiento de RING1 resulta en hipersensibilidad a FB1 y la disminución en la expresión de genes de defensa[33].
En algunos casos, además de fenotipos asociados con la alteración de los niveles del mensajero de una E3, se han podido identificar los sustratos de éstas, así tenemos el ejemplo de la ligasa de Ub del tipo RING de A. thaliana llamada BOI1. Esta E3 interactúa con el factor transcripcional BOS1 en el núcleo y es capaz de promover su ubicuitinación in vitro. BOS1 es necesario para la resistencia a algunos patógenos. Plantas mutantes en el gen BOS1 son más susceptibles a infecciones por patógenos. La acumulación de la proteína BOS1 se observa sólo en plantas tratadas con el inhibidor del proteasoma MG132, lo cual sugiere su degradación por éste[34]. No obstante plantas mutantes en el gen BOI1, en las que se esperaría una acumulación del factor transcripcional BOS1, son más susceptibles a infecciones por hongos, por tanto aún no es muy claro el significado biológico de la posible ubicuitinación de BOS1 mediada por BOI1, no obstante el hecho de encontrar una susceptibilidad incrementada a patógenos en las mutantes en BOI1 y el que la expresión de este gen se incremente por la infección del hongo Botrytis cinerea, apoya el papel de esta ligasa de Ub en la defensa de la planta[34].
Existen otras ligasas de Ub cuyo papel no es el de activar la respuesta de defensa, sino por el contrario, tienen una función de represión de ésta, en particular en situaciones en las que la planta no se encuentra enfrentada a un patógeno. Esto último tiene un gran valor adaptativo, ya que las respuestas de defensa son costosas en términos energéticos y, por lo mismo, tienen una gran repercusión en el crecimiento y el desarrollo de la planta. Por lo tanto, con el propósito de mantener las respuestas de defensa apagadas cuando no existe la presencia de un patógeno, además de otros mecanismos, las plantas emplean el SUP. Por ejemplo, se ha encontrado que la proteína con caja F CPR1 de A. thaliana es un regulador negativo de las proteínas R SNC1 y RPS2[35,36]. Una mutante en el gen CPR1 muestra respuestas de defensa constitutivas y los niveles de SNC1 son elevados. Si se muta ahora también el gen SNC1 se suprime el fenotipo de la mutante en CPR1. Por otra parte, la aplicación del inhibidor del proteasoma MG132 en las plantas silvestres estabiliza a SNC1, apoyando que esta proteína es degradada por el proteasoma. El mensajero de CPR1 se incrementa cuando las plantas de Arabidopsis son infectadas con P. syringae, lo cual sugiere que esta E3 se requiere también para evitar que la planta reaccione excesivamente ante el ataque de un patógeno[36]. Otro ejemplo de regulación negativa de componentes del sistema de defensa es el del factor transcripcional MYB30, éste es un regulador positivo de la defensa y la RH en A. thaliana. MYB30 es ubicuitinado por MIEL1 una ligasa de Ub de tipo RING, y degradado por el proteasoma[37]. Por lo tanto, la función de MIEL1 es atenuar la muerte celular y la respuesta de defensa mediante la degradación de MYB30. Cuando la planta es infectada, la acumulación del transcrito de MIEL1 disminuye sólo en la zona infectada, esto a su vez permite la acumulación de MYB30 y en consecuencia la inducción de la RH. Como la expresión de MIEL1 no cambia en la zona adyacente a la infección es posible que la degradación de MYB30 mediada por MIEL1 pudiera contribuir a evitar que la RH se esparciera más allá del sitio de infección[31]. Esto permite un ajuste fino de la transcripción de genes involucrados en la respuesta de defensa y el control de la muerte celular.
En los casos anteriores, las E3 tenían un papel en mantener apagada la respuesta de defensa en condiciones en las cuales no era requerida, sin embargo, existen también mecanismos en los que también participa el SUP para atenuar la respuesta de defensa una vez que ésta se ha dado, de tal forma que no se prolongue excesivamente y, en consecuencia, tenga efectos perjudiciales para la planta. Como se mencionó anteriormente, la flagelina es un PAMP y su presencia es percibida por el receptor FLS2. La interacción de flagelina con su receptor induce la asociación de éste con un correceptor denominado BAK1. Cuando las plantas de A. thaliana son tratadas con flagelina, las proteínas con caja U PUB12 y PUB13, probablemente a través de su interacción previa con BAK, se asocian al FLS2[38]. Para que esta asociación ocurra, PUB12/13 tienen que ser fosforiladas por BAK1. Una vez unidas a este complejo PUB12/13 serán capaces de ubicuitinar el dominio citosólico de FLS2 y esto promueve su translocación a vesículas en donde será degradado[39]. La internalización de receptores a través de endocitosis, y su posterior degradación, es un mecanismo muy utilizado por distintos organismos para atenuar estímulos provenientes del exterior. Como se mencionó previamente, la ubicuitinación además de ser utilizada como una señal para el reconocimiento por el proteasoma 26S, es también una señal para el proceso de endocitosis. Otro ejemplo relacionado al anteriormente descrito es el de tres ligasas de Ub, también del tipo caja U denominadas PUB22, PUB23 y PUB24, las cuales son reguladores negativos de la resistencia basal. Al infectar las plantas de A. thaliana con P. syringae pv tomato (Pst) o el oomiceto Hyaloperonospora arabidopsis hay un incremento de los mensajeros de los tres genes. Las plantas mutantes en estos tres genes muestran un incremento en la cascada de fosforilación y la síntesis de EORs inducidas por la flagelina, así como de la expresión de genes de defensa en respuesta a la infección por Pst[40]. Estas plantas mutantes tienen también una resistencia aumentada a la infección por patógenos. La respuesta a otros PAMPs está también aumentada en esta triple mutante, lo cual sugiere un mecanismo compartido de represión de la señalización en la IIP en respuesta a PAMPs a través de PUB22-24. Más recientemente se ha demostrado que PUB22 promueve la ubicuitinación para su degradación en el proteasoma 26S de la proteína Exo70B2, la cual es una subunidad de uno de los complejos involucrados en el proceso de exocitosis[41]. La exocitosis, entre otras cosas, contribuye a remodelar membranas en respuesta a la alteración de las condiciones medioambientales. Por tanto, Exo70B2 podría contribuir al reciclaje de proteínas que participan en la señalización mediada por PAMPs y que se localizan en la membrana plasmática, entre las cuales se incluiría al receptor de la flagelina FLS2. Se demostró también que Exo70B2 se requiere para la respuesta inducida por distintos PAMPs, así como para la respuesta de defensa ante distintos patógenos. PUB22 al igual que muchas otras ligasas de Ub se regula de manera autocatalítica, esto es, las E3 monoméricas o en el caso de las multiméricas, las proteínas adaptadoras, son capaces de autoubicuitinarse y de esta manera promover su degradación por el proteasoma. Cuando un PAMP es percibido PUB22 se estabiliza, y de esta manera provoca que los niveles de Exo70B2 disminuyan, esto entonces sería otro mecanismo para la atenuación de la respuesta de defensa de la planta.
Los patógenos interfieren con el SUP de la plantaHasta aquí se han descrito algunos ejemplos de cómo las plantas utilizan el SUP durante su relación con organismos patógenos, éstos últimos por su parte han aprendido durante la evolución a utilizar a la maquinaria celular vegetal para infectar más eficientemente. Existen descritas varias estrategias empleadas por los patógenos para alterar el SUP, desde la producción de compuestos que inhiben al proteasoma, hasta la síntesis de ligasas de Ub que luego son transferidas a la célula vegetal para favorecer la infección. En seguida se describirán algunos ejemplos de esto.
Un ejemplo en el cual el patógeno inhibe directamente al proteasoma lo encontramos en la bacteria P. syringae pv syringae. Este organismo secreta un péptido llamado siringolina A, se ha demostrado que éste es un factor de virulencia, ya que cepas bacterianas que no lo sintetizan son menos virulentas[42]. Se demostró que la siringolina A es capaz de inhibir irreversiblemente la actividad proteolítica del proteasoma. Esto apoya lo mencionado previamente respecto a la importancia del proteasoma en las vías de defensa de las plantas.
También se han encontrado patógenos que aprovechan los mecanismos de defensa de la planta para infectar. Cuando se tratan con inhibidores del proteasoma protoplastos de tabaco infectados con el virus del mosaico del tabaco (TMV) se observa que formas poliubicuitinadas de la proteína de movimiento viral se acumulan en las células. La proteína de movimiento viral facilita el paso del virus de una célula a otra a través del plasmodesmo[43]. La primera conclusión que se podría tener de este hallazgo es que estas proteínas virales serían degradadas mediante el SUP como una forma de protección de la planta, sin embargo, podría ser también un mecanismo empleado por el virus para mantener niveles apropiados de la proteína de movimiento, ya que un exceso de ésta es perjudicial para la planta y, por lo tanto, para la propagación eficiente del virus. A este respecto, se ha demostrado que la inhibición de la ubicuitinación incrementa la resistencia de la planta al TMV, lo cual sugeriría entonces que la degradación de la proteína de movimiento es benéfica para el virus[43].
Existen también otros mecanismos en los cuales el patógeno utiliza efectores que, una vez dentro de la célula vegetal, son capaces de manipular el SUP en beneficio del organismo agresor. Por ejemplo, el oomiceto P. infestans produce una proteína llamada infestina 1 (INF1), con propiedades semejantes a los PAMPs, ya que induce la respuesta de defensa en las plantas, incluyendo la muerte celular. Para neutralizar el reconocimiento de la INF1 por la planta, el oomiceto también sintetiza al efector de AVR3a, el cual es detectado por la proteína de resistencia R3a. En la búsqueda del mecanismo de acción del efector AVR3a se encontró que éste es capaz de interactuar con la E3 de papa, CMPG1, que es una proteína con caja U necesaria para la RH ocurrida en respuesta a algunos efectores y para la muerte celular mediada por la INF1[44]. Al igual que otras E3, CMPG1 es normalmente degradada por el proteasoma y esto ocurre durante el proceso de muerte celular inducido por la INF1. Al unirse AVR3A a CMPG1 lo estabiliza y de esta manera suprime la muerte celular inducida por la INF1, la cual es necesaria para la defensa de la planta, permitiendo de este modo la proliferación del patógeno.
A diferencia del último ejemplo descrito en donde un efector tiene como blanco a una ligasa de Ub existen efectores que son componentes del SUP. Un primer caso es el del efector GrUBCEP12 producido por el nematodo Globodera rostochiensis[45]. GrUBCEP12 consiste de un péptido señal, un dominio de Ub y una extensión de 12 aminoácidos (GrCEP12) en el extremo carboxilo del efector. El péptido señal es removido del resto de la proteína antes de que el nematodo introduzca el resto de la molécula en la célula vegetal. Cuando el gen que codifica a este efector es silenciado, se reduce la capacidad infecciosa de G. rostochiensis, por el contrario cuando se sobreexpresa en plantas de papa la porción que es secretada, Gr(DSP)UbCEP12, estas plantas se vuelven más susceptibles a la infección. Gr(DSP)UbCEP12 dentro de la planta es cortada, liberando Ub y GrCEP12. GrCEP12 tiene la capacidad de suprimir la IIP y también la muerte celular asociada a la IIE, esto sugiere que el blanco GrCEP12 podría ser un componente común en las vías de señalización de la IIP y la IIE. Además de esto, las plantas que sobreexpresan Gr(DSP) UbCEP12 tienen una reducción importante en los niveles del mensajero del gen RPN2, este gen codifica para una de las subunidades de la partícula 19S del proteasoma. Es probable que la Ub secretada por el nematodo podría afectar los niveles celulares locales de Ub y esto, a su vez, tendría el efecto de alterar los niveles de los componentes del proteasoma. De esta forma, suprimiría la función del proteasoma del hospedero con el fin de incrementar su capacidad de infección[45].
Existen también efectores que son ligasas de Ub con blancos específicos en la célula vegetal. Un ejemplo sin lugar a dudas interesante es el del efector AvrPtoB de P. syringae pv tomato. AvrPtoB interactúa con la cinasa de proteínas Pto y esto activa la IIE en jitomate. AvrPtoB, es una proteína con caja U y se ha demostrado, que al igual que sus contrapartes producidas por la planta, tiene actividad de ligasa de Ub. Mutaciones que suprimen la actividad de E3 también elimina la capacidad de este efector de inducir la RH en presencia de su proteína R correspondiente y la virulencia en ausencia de ésta[46]. Se han encontrado dos blancos para AvrPtoB, el primero es la proteína R llamada Fen, que sufre un proceso de ubicuitinación dependiente de AvrPtoB[47]. El segundo es el receptor de la flagelina FLS2, que también es ubicuitinado por el efector[48], por lo tanto, AvrPtoB tiene la capacidad de suprimir tanto la IIP como la IIE.
Otro ejemplo de ligasa de Ub codificada en el genoma bacteriano lo encontramos en Agrobacterium tumefaciens. Este organismo es una bacteria del suelo que causa la enfermedad de la agalla de la corona que consiste en la formación de una especie de tumor debida a la transmisión e integración de un segmento de ADN bacteriano conocido como T-ADN al genoma vegetal. El T-ADN codifica para proteínas que promueven el crecimiento incontrolado de las células vegetales al interferir con el balance hormonal de éstas. En la infección por A. tumefaciens además del T-ADN, varias proteínas son transferidas a la célula vegetal, entre ellas la proteína VirF la cual contiene una caja F. Una vez dentro de la célula forma un complejo SCF que promueve la ubicuitinación y posterior degradación de la proteína de la planta VIP1, así como de la proteína bacteriana VirE2, asociada a VIP1. Estas dos proteínas son necesarias para el transporte nuclear del T-ADN, sin embargo, para la integración de éste al genoma vegetal es necesaria su remoción[49].
ConclusionesEl SUP es una maquinaria esencial en el desarrollo y las respuestas al medio ambiente en los organismos eucariontes, ya que permite la degradación de una manera regulada de proteínas que han sido mal plegadas o bien de proteínas que ya no son requeridas para el funcionamiento celular. Las plantas utilizan el SUP para regular múltiples respuestas entre las que se incluyen aquéllas que les permiten defenderse ante el ataque de patógenos. Es así que el SUP participa en las cascadas de señalización que culminan con la síntesis de proteínas de defensa o bien es empleado para regular que dichas proteínas de defensa se sinteticen en el momento adecuado y en el lugar preciso en donde está ocurriendo el ataque por un patógeno. Los patógenos por su parte han también desarrollado estrategias para evadir la respuesta de defensa de las plantas, entre estas estrategias podemos encontrar la interferencia con la habilidad del SUP para prevenir la infección o bien la síntesis de componentes del SUP que tienen blancos específicos en la célula vegetal.