La identificación rápida de microorganismos es crítica, en especial en pacientes sépticos hospitalizados. La espectrometría de masas conocida como matrix-assisted laser desorption/ionization time-of- flight mass spectrometry (MALDI-TOF MS) permite la identificación directa desde botellas de hemocultivos positivos en forma rápida y sencilla.
Este estudio evaluó el desempeño del procedimiento basado en el sistema MALDI Biotyper que utiliza el kit comercial MALDI Sepsityper de Bruker Daltonics (en adelante, MS) frente a uno artesanal (en adelante, HF). Se procesaron 840 botellas de hemocultivos positivos con HF y 542 de estas fueron evaluadas también con MS. Se logró la identificación de los microorganismos en 670 (79,76%) y 391 (72,14%) botellas, respectivamente (p = 0,0013). Se demostró la efectividad de ambos procedimientos para la identificación de microorganismos desde frascos de hemocultivos positivos. Sin embargo, el procedimiento HF fue superior al MS, en especial frente a bacterias gram positivas.
Rapid identification of microorganisms is critical in hospitalized infected patients. Blood culture is currently the gold standard for detecting and identifying microorganisms causing bacteremia or sepsis. The introduction of mass spectrometry by matrix-assisted laser desorption/ionization time-of-flight (MALDI-TOF MS) in microbiology laboratories, especially in microorganisms growing in blood culture bottles, provides rapid identification.
This study evaluates the performance of the Maldi Sepsityper Biotyper procedure (hereinafter, MS) compared to that of an in-home method (hereinafter, HF). Eight hundred and forty (840) positive blood culture bottles were processed using the HF procedure, 542 of which were also processed using MS. The organisms were identified in 670 (79.76%) and 391 (72.14%) bottles respectively (p = 0,0013). This study demonstrates the effectiveness of both procedures for identifying microorganisms directly from positive blood culture bottles. However, the HF procedure proved to be more effective than MS, especially in the presence of Gram positive organisms.
La identificación rápida de los microorganismos causantes de bacteriemia es importante para optimizar el manejo clínico y la terapia antimicrobiana1. Esta se realiza mediante algoritmos que incluyen pruebas manuales, uso de sistemas automatizados y la eventual confirmación por secuenciación de genes, cuando es necesario.
El desarrollo de la técnica de espectrometría de masas con el equipo MALDI-TOF MS (Bruker Daltonics) ha significado un cambio revolucionario en la identificación de microorganismos en los laboratorios de microbiología clínica. Se trata de un procedimiento rápido, fácil, de alto rendimiento y bajo costo si se dispone de la aparatología5. Esta técnica es eficiente en materia de identificación, ya que proporciona resultados precisos en más del 95% de los organismos aislados y permite adelantar el tiempo de identificación microbiana entre 24-48h, comparado con lo que demanda la identificación bioquímica clásica4.
El MALDI-TOF MS utiliza para la identificación el cálculo del tiempo de migración («tiempo de vuelo») de cada fragmento de una molécula a través de un trayecto predeterminado tras la desorción/ionización por un láser de la molécula en una matriz determinada. Cada bacteria analizada entregará siempre el mismo espectro de masa. La identificación se realiza mediante la comparación del resultado de una bacteria con todos los espectros de masas que contiene la base de datos15. La capacidad del equipo de asignar un puntaje o score ≥2 en la identificación de una bacteria y/o levadura a nivel de especie depende de la existencia de un espectro de proteínas propio del microorganismo en estudio en la base de datos del fabricante. Según algunos trabajos científicos, se puede considerar válida una identificación en género y especie cuando el score es ≥1,74,6,18.
La identificación de microorganismos en forma directa desde las botellas de hemocultivo positivas mediante esta técnica requiere de un proceso de preparación de la muestra, ya que las proteínas presentes en los glóbulos rojos y blancos y en el suero pueden originar picos espectrales que interfieren en la identificación8.
Este estudio tuvo como objetivo evaluar el desempeño de un procedimiento artesanal (HF) versus el del sistema MALDI Biotyper usando el kit comercial MALDI Sepsityper (Bruker Daltonic) para la identificación de microorganismos directamente desde las botellas de hemocultivos positivas. El MALDI Sepsityper es un kit que contiene los reactivos necesarios para la lisis de los glóbulos rojos y un protocolo específico para estandarizar la separación de microorganismos de frascos de hemocultivos antes de realizar el análisis espectrométrico.
Materiales y métodosSe estudiaron los hemocultivos positivos registrados entre el 1/11/2013 y el 30/4/2014 en la Sección Microbiología Clínica del Hospital General de Agudos «Dr. Juan A. Fernández». Se consideró un episodio por paciente y se incluyeron los hemocultivos positivos confirmados por la presencia de bacterias en la tinción de Gram. Para este trabajo se descartaron los hemocultivos que originaron cultivos polimicrobianos. Las botellas analizadas correspondieron al sistema automatizado de detección microbiana BACTEC FX (BD): Plus Aerobic/F, Plus Anaerobic/F y Peds Plus//F para cultivo aeróbico, anaerobio y de especímenes pediátricos.
El procesamiento de las botellas se realizó según el protocolo establecido por el fabricante. Se tomó una alícuota de 1ml de sangre para realizar ambos procedimientos, el MS y el HF.
La interpretación de los resultados fue de acuerdo al score proporcionado por el equipo. Se consideraron válidas en forma preliminar las identificaciones con score ≥1,7 para género y especie, según los criterios aceptados por la literatura internacional4,6,18.
Los resultados fueron confirmados con el análisis de una colonia aislada en agar sangre con 24h de incubación por el MALDI-TOF MS con un score ≥2 por pruebas bioquímicas convencionales manuales o automatizadas con el equipo Phoenix BD (EE. UU.) o de las 2 maneras. Todos las identificaciones de estreptococos del grupo viridans y Streptococcus pneumoniae fueron confirmadas con pruebas bioquímicas convencionales.
Para el procesamiento de los datos obtenidos se empleó la versión 3.1 del MALDI Biotyper Software (Bruker Daltonik, Bremen, Alemania).
El proceso de preparación incluyó mantener las muestras y los reactivos a temperatura ambiente antes de ser utilizados. Todas las botellas se analizaron en forma idéntica en un período de tiempo que no superó las 8h desde su positivización.
El procesamiento para el análisis por MS se efectuó de acuerdo a la guía que incluye el kit proporcionada por el fabricante (Guide Maldi Sepsityper, 2010). Se utilizó 1ml del hemocultivo positivo en un tubo Eppendorf con 200μl de buffer de lisis. La centrifugación para obtener el pellet fue de 1min a 13000x g. Se agregaron 300μl de agua (Fluka, Suiza), se agitó en vórtex 1min, se agregaron 900μl de etanol absoluto (Sigma Aldrich, EE. UU.), se agitó nuevamente en vórtex 1min y se centrifugó 5min a 13000x g. Al sedimento se le añadieron 50μl de ácido fórmico (Fluka, Alemania) y 50μl de acetonitrilo (Sigma Aldrich, EE. UU.), se agitó en vórtex y se centrifugó 3min a 13000x g. Para el procedimiento HF, se utilizó 1ml de hemocultivo positivo en un tubo seco con 2ml de agua destilada estéril. Se agitó en vórtex 2min y se centrifugó durante 15min a 3000x g. El sedimento se extrajo con hisopo y se pasó a un tubo Eppendorf. A partir de este momento se continuó con el protocolo de extracción del MS. En ambos procedimientos se colocó 1μl de pellet por duplicado en la placa reutilizable del sistema. Una vez seca la muestra, se añadió sobre cada una 1μl de solución de matriz ácido α-ciano-4-hidroxicinámico (HCCA, Bruker Daltonik, Alemania). La placa se dejó secar a temperatura ambiente y se la colocó dentro del equipo. La calibración se realizó con un calibrador BTS (Bruker Daltonik, Alemania), según el manual de procedimientos, y se ingresaron los datos de acuerdo a lo requerido por el software SPSS 17.0 IBM, Illinois y STATISTIX 7.0.
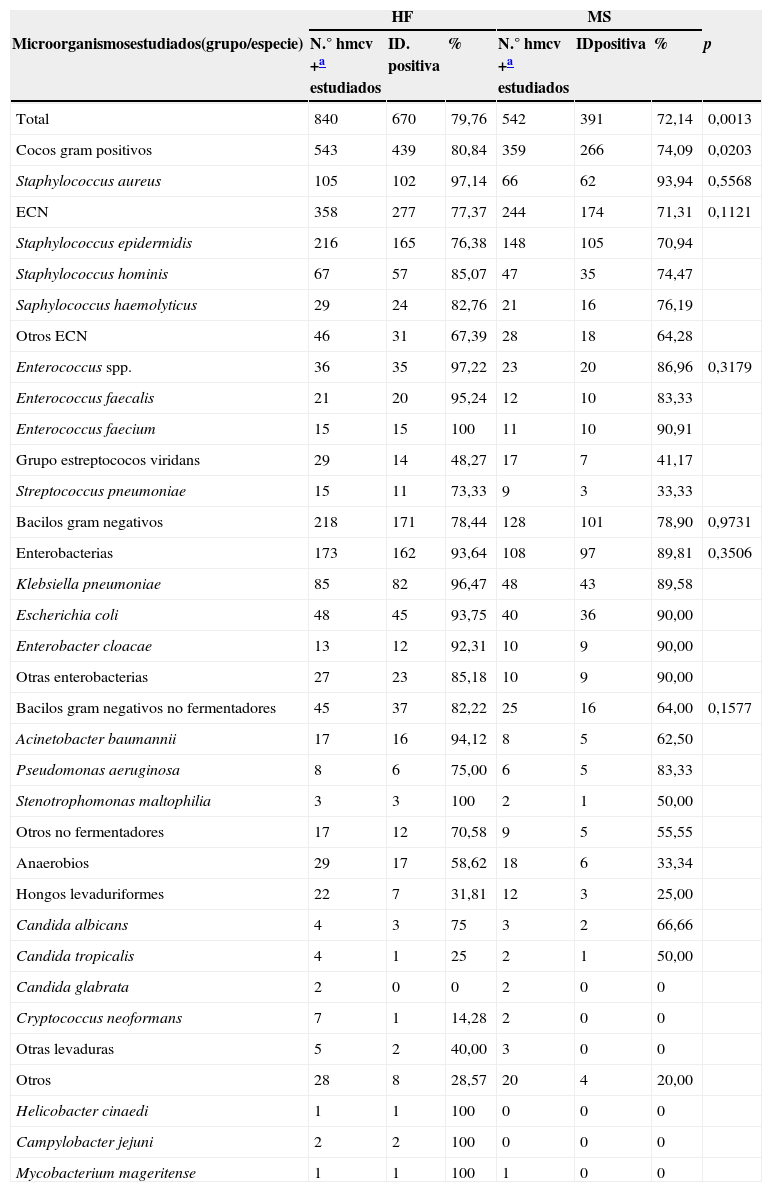
ResultadosSe abordó la identificación directa de los microorganismos en un total de 840 hemocultivos positivos con un HF; en 542 frascos además se ensayó en simultáneo el procedimiento MALDI Sepsityper/Biotyper (MS). Se llegó a establecer el género y la especie en un total de 670 hemocultivos (79,76%) con el procedimiento HF y de 391 hemocultivos (72,14%) con el procedimiento MS.
La tabla 1 resume los resultados obtenidos. Estos se presentan por grupos de microorganismos; se indican las especies más frecuentes de cada grupo.
Desempeño comparativo en la identificación de agentes causales de bacteriemia a partir de frascos de hemocultivos positivos con un procedimiento artesanal (HF) y con un kit comercial para análisis por espectrometría de masas MALDI TOF (MS)
| HF | MS | ||||||
|---|---|---|---|---|---|---|---|
| Microorganismosestudiados(grupo/especie) | N.° hmcv +a estudiados | ID. positiva | % | N.° hmcv +a estudiados | IDpositiva | % | p |
| Total | 840 | 670 | 79,76 | 542 | 391 | 72,14 | 0,0013 |
| Cocos gram positivos | 543 | 439 | 80,84 | 359 | 266 | 74,09 | 0,0203 |
| Staphylococcus aureus | 105 | 102 | 97,14 | 66 | 62 | 93,94 | 0,5568 |
| ECN | 358 | 277 | 77,37 | 244 | 174 | 71,31 | 0,1121 |
| Staphylococcus epidermidis | 216 | 165 | 76,38 | 148 | 105 | 70,94 | |
| Staphylococcus hominis | 67 | 57 | 85,07 | 47 | 35 | 74,47 | |
| Saphylococcus haemolyticus | 29 | 24 | 82,76 | 21 | 16 | 76,19 | |
| Otros ECN | 46 | 31 | 67,39 | 28 | 18 | 64,28 | |
| Enterococcus spp. | 36 | 35 | 97,22 | 23 | 20 | 86,96 | 0,3179 |
| Enterococcus faecalis | 21 | 20 | 95,24 | 12 | 10 | 83,33 | |
| Enterococcus faecium | 15 | 15 | 100 | 11 | 10 | 90,91 | |
| Grupo estreptococos viridans | 29 | 14 | 48,27 | 17 | 7 | 41,17 | |
| Streptococcus pneumoniae | 15 | 11 | 73,33 | 9 | 3 | 33,33 | |
| Bacilos gram negativos | 218 | 171 | 78,44 | 128 | 101 | 78,90 | 0,9731 |
| Enterobacterias | 173 | 162 | 93,64 | 108 | 97 | 89,81 | 0,3506 |
| Klebsiella pneumoniae | 85 | 82 | 96,47 | 48 | 43 | 89,58 | |
| Escherichia coli | 48 | 45 | 93,75 | 40 | 36 | 90,00 | |
| Enterobacter cloacae | 13 | 12 | 92,31 | 10 | 9 | 90,00 | |
| Otras enterobacterias | 27 | 23 | 85,18 | 10 | 9 | 90,00 | |
| Bacilos gram negativos no fermentadores | 45 | 37 | 82,22 | 25 | 16 | 64,00 | 0,1577 |
| Acinetobacter baumannii | 17 | 16 | 94,12 | 8 | 5 | 62,50 | |
| Pseudomonas aeruginosa | 8 | 6 | 75,00 | 6 | 5 | 83,33 | |
| Stenotrophomonas maltophilia | 3 | 3 | 100 | 2 | 1 | 50,00 | |
| Otros no fermentadores | 17 | 12 | 70,58 | 9 | 5 | 55,55 | |
| Anaerobios | 29 | 17 | 58,62 | 18 | 6 | 33,34 | |
| Hongos levaduriformes | 22 | 7 | 31,81 | 12 | 3 | 25,00 | |
| Candida albicans | 4 | 3 | 75 | 3 | 2 | 66,66 | |
| Candida tropicalis | 4 | 1 | 25 | 2 | 1 | 50,00 | |
| Candida glabrata | 2 | 0 | 0 | 2 | 0 | 0 | |
| Cryptococcus neoformans | 7 | 1 | 14,28 | 2 | 0 | 0 | |
| Otras levaduras | 5 | 2 | 40,00 | 3 | 0 | 0 | |
| Otros | 28 | 8 | 28,57 | 20 | 4 | 20,00 | |
| Helicobacter cinaedi | 1 | 1 | 100 | 0 | 0 | 0 | |
| Campylobacter jejuni | 2 | 2 | 100 | 0 | 0 | 0 | |
| Mycobacterium mageritense | 1 | 1 | 100 | 1 | 0 | 0 | |
HF: procedimiento artesanal; MS: procedimiento basado en el uso del kit comercial MALDI Sepsityper/Biotyper de Bruker Daltonic.
Las diferencias en la capacidad de identificación con HF vs. MS fueron significativas (p = 0,0013). Asimismo, la diferencia entre ambos procedimientos fue significativa (p = 0,020) para la caracterización de bacterias gram positivas.
Los porcentajes de identificación de Staphylococcus aureus fueron 97,14% y 93,94% con los procedimientos HF y MS, respectivamente (p = 0,5568). Los correspondientes a las especies de enterococos fueron de 97,22% y 86,96% (p = 0,379). Los porcentajes de identificación fueron menores de 80% en los estafilococos coagulasa negativos (ECN) con ambos procedimientos y no hubo diferencias significativas (p = 0,1121), y menores del 50% en el grupo estreptococos viridans. Con respecto a S. pneumoniae, el número de muestras en las que se identificó a este microorganismo fue bajo (n=15 con HF y n=9 con MS), de manera que no es posible evaluar a estos métodos comparativamente, aunque cabe indicar que se detectó un número mayor con el procedimiento HF que con el MS. Si consideramos todos los bacilos gram negativos en conjunto (no fermentadores y enterobacterias), la tasa de identificación fue 78,44% vs. 78,90% mediante HF y MS, respectivamente; esta diferencia no fue significativa (p = 0,9731). Tampoco fueron significativas las diferencias entre HF y MS (93,64% vs. 89,81% (p = 0,3506) en la identificación de Enterobacteriaceae.
En cuanto a hongos levaduriformes y anaerobios, el número de microorganismos estudiados con ambos procedimientos fue demasiado bajo como para extraer conclusiones.
Con respecto a otros microorganismos, solo destacamos aquellos que consideramos de muy baja frecuencia y de difícil diagnóstico.
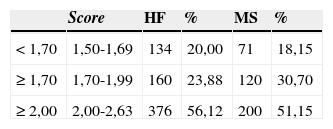
En la tabla 2 se presentan las categorías de scores y cómo se distribuyeron los resultados de la identificación en esas categorías para los 2 procedimientos evaluados.
Distribución del score obtenido en la identificación de agentes causales de bacteriemia a partir de frascos de hemocultivos positivos con un procedimiento artesanal (HF) y con un kit comercial para análisis por espectroscopía de masa MALDI TOF (MS)
| Score | HF | % | MS | % | |
|---|---|---|---|---|---|
| <1,70 | 1,50-1,69 | 134 | 20,00 | 71 | 18,15 |
| ≥1,70 | 1,70-1,99 | 160 | 23,88 | 120 | 30,70 |
| ≥2,00 | 2,00-2,63 | 376 | 56,12 | 200 | 51,15 |
HF: procedimiento artesanal; MS: procedimiento basado en el uso del kit comercial MALDI Sepsityper/Biotyper de Bruker Daltonic.
Las bacteriemias están asociadas a una alta mortalidad, un largo tiempo de estadía hospitalaria y, por consiguiente, a un aumento de los costos. La identificación del microorganismo seguida de la prueba de sensibilidad es crítica para el manejo de los pacientes con bacteriemia. En este estudio, el procedimiento basado en la técnica de MALDI-TOF MS identificó a más del 80% de los microorganismos investigados en los hemocultivos por ambos procedimientos, con un score ≥1,70. Nuestros resultados coincidieron con lo que informa la literatura en general. Existen diferencias con respecto a la interpretación de los scores obtenidos en hemocultivos. Mientras que algunos autores se ajustan a los puntajes convencionales aceptados de forma general (en el caso de Bruker Daltonics, ≥2,00 para identificación a nivel de especie y ≥1,70 para identificación a nivel de género), otros consideran puntos de corte más bajos (scores de 1,5 o incluso 1,3) si es que se obtienen repetidamente valores superiores a estos para la misma identificación en el mismo análisis o en análisis distintos2.
En un estudio sobre más de 500 hemocultivos, La Scola et al.13 llegaron a la identificación correcta en el 66% de los hemocultivos evaluados (91% con bacterias gram negativas y 49% con gram positivas). Por su parte, Prod’hom et al.16 lograron una identificación correcta en 79% de las muestras (90% con bacterias gram negativas y 73% con gram positivas), aunque con un número de hemocultivos inferior (n=122).
Stevenson et al.17 analizaron un total de 212 hemocultivos positivos obteniendo una identificación correcta en el 76,4% de ellos. Kok et al. estudiaron 507 hemocultivos positivos con el kit Sepsityper y lograron la identificación correcta de 379 de ellos (74,8%); se identificaron 132 (67,7%) de 195 microorganismos gram positivos y 149 (91,4%) de 163 gram negativos9.
Con respecto al género Staphylococcus, encontramos diferencias entre los ECN y S. aureus. En el caso de los ECN, con el kit MALDI Sepsityper Kit se logró la identificación en el 79,33% de los hemocultivos vs. 77,30% con el HF (p = 0,1121); el porcentaje de identificación de acuerdo a la bibliografía internacional es de 71,9% a 84%7,10,14,16; se advierte que nuestros resultados fueron comparables con estos.
Resaltamos los valores obtenidos en cuanto a la identificación de S. aureus: 93,75% y 97,17% con MS y HF, respectivamente.
Al igual que lo informado por otros autores3,9,13,16, las mayores coincidencias se obtuvieron en relación con los bacilos gram negativos pertenecientes a la familia Enterobacteriaceae, la cual fue del 88,18% con el MS y del 93,69% con el HF (p = 0,3506).
Stevenson et al.17 concluyen que realizar el MALDI-TOF-MS directo de hemocultivos positivos reduce en forma notoria el tiempo de identificación de especies, lo cual se asoció con un 11,3% de incremento en la proporción de pacientes que recibieron terapia antibiótica adecuada dentro de las 24h de la positivización del hemocultivo.
La reducción del tiempo necesario para llevar a cabo el diagnóstico etiológico de la bacteriemia se ve reflejada en un aumento de la supervivencia de los pacientes, en los cuales la adecuada administración de antibióticos es esencial para una evolución favorable10,12, ya que cada hora de retraso en el inicio del tratamiento se asoció con una disminución de la tasa de supervivencia de casi 8%11.
Consideramos que la superioridad del procedimiento HF podría deberse al mayor tiempo de centrifugación para concentrar el pellet; al empleo de un tubo en lugar de un Eppendorf, el que proporciona una mayor superficie para la separación, y a la extracción del pellet con hisopo. También es posible que el agregado de 2ml de agua destilada sea más efectivo que los 200μl de buffer de lisis del kit, por el mayor volumen para lisar y luego separar las macromoléculas contenidas en la sangre que podrían interferir con los espectros de los microorganismos.
Por otro lado, como el procedimiento HF emplea agua destilada en lugar de buffer de lisis este tiene un costo muy inferior al kit comercial que se emplea para la MS, que es de 5,15 dólares americanos por determinación12. Es importante mencionar que el costo del equipo MALDI-TOF es elevado y no resulta accesible para instituciones de baja o mediana complejidad.
En 30min se puede identificar el género de un microorganismo y la especie presunta o definitiva con el método MALDI-TOF y esto es de fundamental importancia para el manejo del paciente con bacteriemia. Estas tecnologías continuarán desarrollándose, especialmente para identificar mejor a los miembros del grupo de estreptococos viridans, a otros estreptococos y a las levaduras, a fin de lograr una mayor sensibilidad.
Responsabilidades éticasProtección de personas y animalesLos autores declaran que para esta investigación no se han realizado experimentos en seres humanos ni en animales.
Confidencialidad de los datosLos autores declaran que en este artículo no aparecen datos de pacientes.
Derecho a la privacidad y consentimiento informadoLos autores declaran que en este artículo no aparecen datos de pacientes.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.
Agradecemos a BD Argentina por su asesoramiento y entrega de kits para la realización de este trabajo.