A partir del descubrimiento de la presencia de ácidos nucleicos fetales libres circulantes en el plasma materno se ha generado un gran interés sobre su origen, naturaleza y posibles usos médicos. En este artículo de revisión realizamos un amplio y conciso resumen sobre los resultados de los estudios de ADN y ARN fetal libre en plasma materno, sus perspectivas futuras, principalmente orientadas hacia el diagnóstico prenatal no invasivo, área donde se espera un impacto importante en el futuro inmediato. También se reportan resultados prometedores en la evaluación de la función placentaria y como marcadores predictores y de severidad en complicaciones del embarazo.
The discovery of circulating free fetal nucleic acids in maternal plasma has sparked wide interest in their origin, characteristics and possible medical uses. This review provides a comprehensive and concise summary of the results of studies of free fetal DNA and RNA in maternal plasma and discusses future possibilities for their use, mainly aimed at non-invasive prenatal diagnosis, an area where this discovery is expected to have a major impact in the very near future. Promising results have been reported in the assessment of placental function and in the use of these nucleic acids as predictive markers of the severity of pregnancy complications.
Hoy en día el diagnóstico prenatal requiere de muestras de células fetales obtenidas por vías invasivas, como amniocentesis y toma de muestras de vellosidad coriónica. Estos procedimientos invasivos representan un riesgo para la madre y el feto1. Para evitar este riesgo potencial, se han desarrollados métodos de genética para el diagnóstico prenatal. Estudio previos han encontrado ácidos nucleicos libres en el plasma de sujetos normales2, en pacientes con cáncer3 y mujeres embarazadas4,5. Este último proveniente de células trofoblásticas6 debido al tráfico celular entre el feto y la madre7. Estos ácidos nucleicos pueden permitir una alternativa segura y precisa de métodos de diagnóstico no invasivo. En los últimos años, se ha realizado un gran progreso en esta área. En particular, los estudios se han encaminado en detectar ADN y ARN del feto, en el plasma materno, además la utilización de la técnica de PCR en tiempo real (QRT-PCR) ha permitido detectar niveles muy bajos de estas moléculas8. En este artículo de revisión, hacemos un breve resumen de los estudios que se han llevado a cabo en esta área y las ventajas que representa.
ADN fetal libre en plasma maternoA raíz del descubrimiento del paso de células fetales a la sangre materna9, se comenzó a estudiar si además de células también se podía aislar material genético libre en plasma materno. En 1948 se publicó la primera evidencia de ácidos nucleicos en plasma y orina10. Este trabajo no fue valorado por muchos años hasta que otros investigadores encontraron estas moléculas plasmáticas en pacientes con lupus eritematoso sistémico (LES)11,12, artritis reumatoidea13 y cáncer3,14–18. Todavía se desconoce el mecanismo por el cual el ADN es liberado al suero y plasma. Ha sido postulado que en LES, las células multinucleadas sensibilizadas liberan su ADN endógeno aumentando el ADN circulante en plasma. Adicionalmente, los neutrófilos apoptóticos circulantes podrían contribuir al aumento en los niveles de ADN de doble cadena libre19. Similarmente en malignidad, el ADN libre en plasma probablemente proviene de la apoptosis de células cancerígenas y normales. Otra teoría postula que la destrucción celular debido a la radioterapia podría liberar el ADN al plasma20.
En 1997, Lo et al lograron aislar ADN libre circulante en el plasma de mujeres embarazadas4. Estos estudios abrieron una nueva posibilidad de realizar un diagnóstico prenatal no invasivo donde ni el feto ni la madre puedan correr ningún peligro21. El mecanismo por el cual el ADN de origen feto-placentario es liberado al plasma es desconocido, aunque se piensa que la muerte celular programada (apoptosis) puede estar involucrada22,23.
En los últimos años, se han desarrollado nuevos ensayos para aislar y analizar ADN fetal en plasma materno obteniéndose un avance rápido en estas áreas4,24–26. Existen varias ventajas al utilizar ADN fetal libre en plasma. La primera ventaja es que sorprendentemente este se encuentra en muy altas concentraciones durante el embarazo4,24. La alta concentración de ADN fetal en plasma materno hace del plasma materno una fuente de fácil acceso para el diagnóstico prenatal no invasivo. En segundo lugar, la desaparición del ADN fetal libre en circulación materna es extremadamente rápida. La vida media del ADN fetal libre se ha determinado en 16,3min (4-30min)27. Por lo tanto, el ADN fetal en plasma materno podría ser utilizado como un marcador en tiempo real para monitorización fisiológica y patológica tanto del feto como de la madre. Tercero, métodos como la PCR y sus derivados son altamente sensibles para la detección de ADN plasmático libre4,25. Cuarto, se ha demostrado que este ADN puede ser detectado desde la 5.ª semana de embarazo, lo que lo haría útil en diagnóstico de patologías fetales mucho antes de que puedan ser detectadas por ultrasonido o posterior al nacimiento28. Recientemente se ha utilizado la técnica de reacción en cadena de la polimerasa cuantitativa en tiempo real (QRT-PCR) para medir la cantidad de ADN fetal libre en plasma. Es así como con esta técnica se ha podido determinar el sexo fetal en mujeres con fetos masculinos, evaluando el gen SRY4,24,29, hallazgo de mucha utilidad en el diagnóstico de las enfermedades ligadas al sexo. Una de las utilidades más importantes es en la determinación del genotipo Rh D fetal en mujeres Rh D negativas30, esta prueba ya ha sido introducida como un servicio de rutina por el Servicio Nacional de Sangre Británico desde 200131. También se ha utilizado en la detección de polimorfismos en los cromosomas 13, 18 y 21 para el diagnóstico de trisomías32, diagnóstico prenatal de enfermedades autosómicas recesivas (fibrosis quística33 e hiperplasia adrenal congénita34) y de autosómicas dominantes (distrofia miotónica, acondroplasia y enfermedad de Huntington)35–37. Cabe resaltar que se ha logrado detectar ADN fetal en el plasma de mujeres que han tenido hijos varones muchos años después inclusive décadas después del parto38.
En los últimos años se han desarrollado varias estrategias para aislar el ADN fetal en plasma materno para ser utilizado como marcador en diagnóstico prenatal no invasivo. Se ha descubierto que el tamaño del ADN fetal es menor que el materno39. Este descubrimiento ha permitido un mejor aislamiento del ADN a partir del plasma materno. Por otro lado, también se ha establecido que existen diferencias en la metilación del ADN fetal con respecto al ADN de origen materno. Como es el caso del gen SERPINB5, el cual está hipometilado en la placenta e hipermetilado en células de sangre materna40. Por lo tanto, el SERPINB5 hipometilado puede utilizarse como un marcador fetal específico en plasma materno. Debido a que el gen se encuentra en el cromosoma 18, se ha demostrado que el análisis de la razón alélica de un polimorfismo de un solo nucleótido (SNP) en la forma hipometilada del SERPINB5 puede ser utilizada como un marcador en el plasma materno para el diagnóstico de trisomía 1841. También se ha demostrado que diferencias en los patrones de metilación pueden ser utilizadas para diagnóstico prenatal no invasivo de trisomía 21 utilizando plasma materno42. Otra aproximación hacia la detección de aneuploidías es el desarrollo de métodos cuantitativos altamente discriminatorios, los cuales permiten la detección de secuencias de ADN específicas, incluso si el ADN fetal representa una proporción baja del ADN total libre circulante. Lo et al demostraron que la PCR digital es una muy buena herramienta para este fin43. Es importante destacar que el ADN fetal ha podido ser amplificado y secuenciado con muy buenos resultados44–46.
Adicionalmente, se ha observado que en algunas patologías del embarazo existen anomalías en la concentración de ADN fetal circulante en plasma. La primera enfermedad que fue asociada con estas anomalías fue la preeclampsia, donde se encontró que la concentración de ADN en las gestantes con preeclampsia estaba incrementada cinco veces en comparación con los controles47, al parecer por diferencias en el aclaramiento del ADN fetal circulante plasmático48. Otras enfermedades donde se ha visto involucrada la variación en concentración de ADN son: algunas aneuploidías49, parto pretérmino50, hiperémesis gravídica51 y placenta invasiva52.
ARN fetal libre en plasma maternoA partir de la detección de ADN derivado de tumores en plasma se inició la búsqueda de ARN en el plasma y suero de pacientes con cáncer53,54. Debido a que muchas veces no es posible determinar el origen del ADN encontrado en el plasma de mujeres embarazadas y se puede confundir con el ADN materno, se planteó el interrogante si se podría aislar ARN de origen feto-placentario en el plasma materno y así determinar la expresión de genes específicos de la placenta. Poon et al fueron los primeros en publicar la presencia de ARN fetal en el plasma de mujeres embarazadas, mediante la detección de ARNm del gen ZFY que se encuentra en el cromosoma Y. Las tasas de detección de ARN fetal en plasma materno al inicio y final de los embarazos fueron 22 y 63%, respectivamente. La menor tasa de detección de ARN al inicio del embarazo con respecto a etapas finales sugiere que la concentración de ARN plasmático fetal es menor en etapas tempranas del embarazo. Esta observación es similar a los resultados que indican que la concentración de ADN fetal en plasma materno se incrementa con el progreso del embarazo55. Al estudiar este ARN se encontró que era mucho más estable de lo que se pensaba. Tsui et al investigaron esta aparente paradoja en detalle y reportaron que la moléculas de ARN de plasma endógeno eran altamente estables a 4°C por 24 horas en comparación con ARN tisular extraído y purificado2. Además al pasar el plasma por filtros de 0,45μm se encontraban diferencias estadísticamente significativas al medir la expresión del ARNm de la subunidad beta de la hormona gonadotrofina coriónica humana (βhCG), lactógeno placentario humano (hPL) y griceraldehido-6-fosfato deshidrogenasa (GAPDH), en comparación con plasma no filtrado. Esto sugiere que las moléculas de ARN podrían estas asociadas a algún tipo de estructura celular o membranal que lo protege de su degradación8. Adicionalmente, demostró que los transcritos de ARNm derivados de placenta son detectables en plasma materno durante el embarazo y desaparecen rápidamente después del parto. Estos resultados demuestran que la placenta es un órgano importante para liberar ARN fetal en plasma materno. Luego Ng et al demostraron que esas moléculas de ARN de plasma de pacientes con cáncer estaban asociadas con cuerpos apoptóticos que podrían protegerlo de la degradación de las RNasas56,57, esto podría dar una explicación a la estabilidad de este ARN. A pesar de esta aparente estabilidad, estudios demostraron que la molécula no es un transcrito intacto la cual mostró una mayor degradación en el extremo 3′ comparado con el extremo 5′58.
A raíz de estos descubrimientos se empezaron a desarrollar diferentes estrategias metodológicas para la evaluación y la medición de transcritos derivados de placenta. Con el fin de buscar un marcador de ARN fetal independiente del género, Ng et al recientemente demostraron que el ARNm expresado en placenta es fácilmente detectable en plasma materno, utilizando ARNm codificante para hPL y ßhCG por la técnica de QRT-PCR8. En 1999, Vogelstein y Kinzler propusieron una técnica innovadora llamada «PCR digital» para cuantificar con una alta precisión una sola molécula de ARN59. Esta nueva técnica se ha utilizado para estudios en pacientes con cáncer de ovario60 y trisomía 21 fetal43.
En el 2004 Tsui et al compararon los perfiles de expresión génica entre los tejidos de la placenta y de sangre periférica correspondiente de las mujeres embarazadas en su primer y tercer trimestre, por análisis de microarray61. Esta es una técnica de análisis de ARN plasmático que puede representar una nueva herramienta para el análisis no invasivo de perfiles fetales de expresión génica. Los resultados obtenidos de los estudios de ARN fetal en plasma materno a través de la técnica de microarrays son claves para escoger los transcritos a ser estudiados en diversas patologías del embarazo.
El estudio del ARN fetal circulante en plasma materno se ha encaminado al desarrollo de estrategias para el diagnóstico prenatal de patologías como preeclampsia y algunas aneuploidías62. Se espera que se desarrollen nuevos marcadores que permitan acercarnos cada vez más a establecer un diagnóstico prenatal no invasivo utilizando marcadores de ARNm.
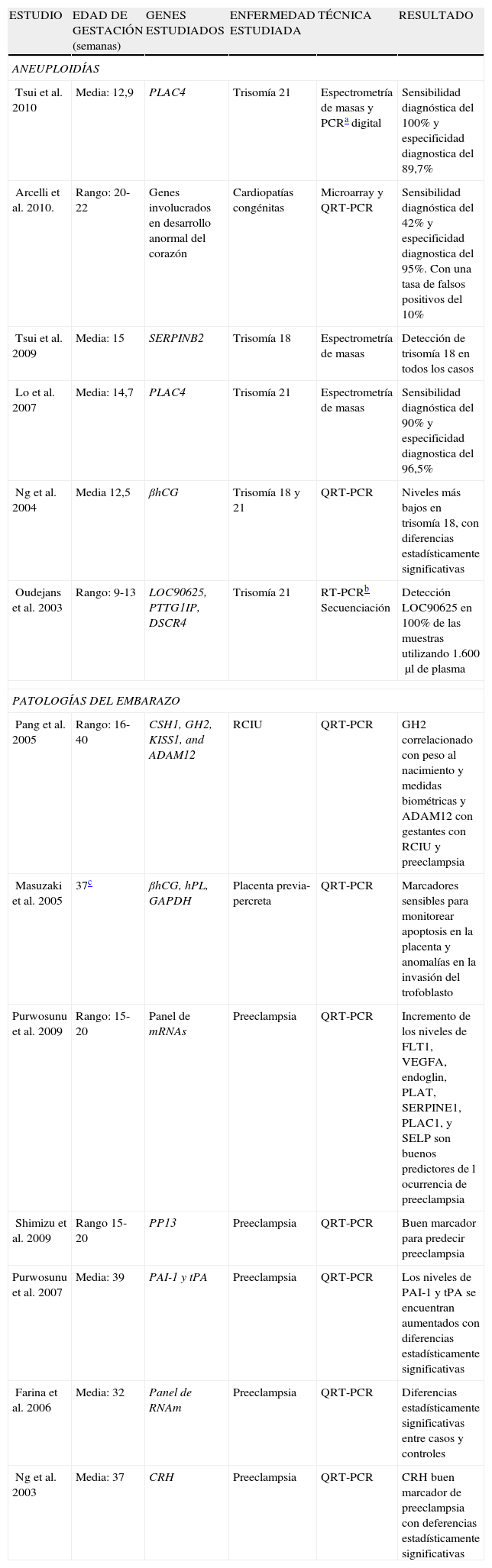
ARN de origen feto-placentario en plasma materno en el diagnóstico prenatal de patologías fetalesPara desarrollar marcadores para diagnóstico prenatal de trisomía 21 se estudió el gen LOC90625 el cual se encuentra en el cromosoma 21 y se detecta en un 100% cuando se trabaja con 1600μl de plasma en el primer trimestre, por lo tanto puede ser utilizado como marcador para diagnóstico prenatal de síndrome de Down63. Adicionalmente, se ha estudiado si las concentraciones de ARNm en plasma podrían ser un marcador para trisomías. Por este motivo Ng et al realizaron un estudio en donde se evaluaron los niveles de ARNm de la ßhCG en mujeres con fetos con trisomía 18 y 21 durante el primer trimestre del embarazo. Los resultados mostraron que había diferencias estadísticamente significativas entre las concentraciones de ARNm en plasma de mujeres con trisomía 18 y controles (p<0,05), con concentraciones 9,4 veces menores en mujeres con fetos con trisomía 1862. Por lo tanto, las concentraciones de ARNm varían en algunas patologías y podrían servir como marcadores para un diagnóstico prenatal no invasivo o inclusive como factores predictores de la ocurrencia y severidad de enfermedades del embarazo (tabla 1).
Estudios que reportan el uso de mRNA circulante derivado de placenta para estudio de aneuploidías y complicaciones del embarazo
| ESTUDIO | EDAD DE GESTACIÓN (semanas) | GENES ESTUDIADOS | ENFERMEDAD ESTUDIADA | TÉCNICA | RESULTADO |
| ANEUPLOIDÍAS | |||||
| Tsui et al. 2010 | Media: 12,9 | PLAC4 | Trisomía 21 | Espectrometría de masas y PCRa digital | Sensibilidad diagnóstica del 100% y especificidad diagnostica del 89,7% |
| Arcelli et al. 2010. | Rango: 20-22 | Genes involucrados en desarrollo anormal del corazón | Cardiopatías congénitas | Microarray y QRT-PCR | Sensibilidad diagnóstica del 42% y especificidad diagnostica del 95%. Con una tasa de falsos positivos del 10% |
| Tsui et al. 2009 | Media: 15 | SERPINB2 | Trisomía 18 | Espectrometría de masas | Detección de trisomía 18 en todos los casos |
| Lo et al. 2007 | Media: 14,7 | PLAC4 | Trisomía 21 | Espectrometría de masas | Sensibilidad diagnóstica del 90% y especificidad diagnostica del 96,5% |
| Ng et al. 2004 | Media 12,5 | βhCG | Trisomía 18 y 21 | QRT-PCR | Niveles más bajos en trisomía 18, con diferencias estadísticamente significativas |
| Oudejans et al. 2003 | Rango: 9-13 | LOC90625, PTTG1IP, DSCR4 | Trisomía 21 | RT-PCRb Secuenciación | Detección LOC90625 en 100% de las muestras utilizando 1.600μl de plasma |
| PATOLOGÍAS DEL EMBARAZO | |||||
| Pang et al. 2005 | Rango: 16-40 | CSH1, GH2, KISS1, and ADAM12 | RCIU | QRT-PCR | GH2 correlacionado con peso al nacimiento y medidas biométricas y ADAM12 con gestantes con RCIU y preeclampsia |
| Masuzaki et al. 2005 | 37c | βhCG, hPL, GAPDH | Placenta previa-percreta | QRT-PCR | Marcadores sensibles para monitorear apoptosis en la placenta y anomalías en la invasión del trofoblasto |
| Purwosunu et al. 2009 | Rango: 15-20 | Panel de mRNAs | Preeclampsia | QRT-PCR | Incremento de los niveles de FLT1, VEGFA, endoglin, PLAT, SERPINE1, PLAC1, y SELP son buenos predictores de l ocurrencia de preeclampsia |
| Shimizu et al. 2009 | Rango 15-20 | PP13 | Preeclampsia | QRT-PCR | Buen marcador para predecir preeclampsia |
| Purwosunu et al. 2007 | Media: 39 | PAI-1 y tPA | Preeclampsia | QRT-PCR | Los niveles de PAI-1 y tPA se encuentran aumentados con diferencias estadísticamente significativas |
| Farina et al. 2006 | Media: 32 | Panel de RNAm | Preeclampsia | QRT-PCR | Diferencias estadísticamente significativas entre casos y controles |
| Ng et al. 2003 | Media: 37 | CRH | Preeclampsia | QRT-PCR | CRH buen marcador de preeclampsia con deferencias estadísticamente significativas |
Una estrategia que se desarrolló recientemente es la determinación de la razón alélica para diagnóstico de algunas aneuploidías. Esta estrategia es llamada aproximación de la «razón alélica de un SNP de ARN» y se realiza con espectrometría de masas; los resultados han mostrado una alta sensibilidad y especificidad estudiando el gen PLAC4 en pacientes con fetos con trisomía 2164,65 y el gen SERPINB2 en trisomía 1866. La limitación que tiene esta técnica es que únicamente se pueden analizar fetos heterocigotos para el SNP analizado. Para eliminar esta limitación se deberían analizar múltiples SNP.
Adicionalmente, Alcelli et al estudiaron la expresión de genes involucrados en el desarrollo anormal del corazón encontrando diferencias estadísticamente significativas entre los niveles encontrados en plasma de mujeres con fetos diagnosticados con cardiopatías congénitas comparados con controles normales67.
ARN de origen feto-placentario en plasma materno en complicaciones del embarazoTambién se han reportado diferencias en los niveles de ARN fetal en plasma materno en complicaciones del embarazo como placenta previa, restricción de crecimiento intrauterino (RCIU) y preeclampsia.
Debido a estudios realizados en preeclampsia con ADN fetal plasmático, se pensó que el ARNm también podría estar involucrado en esta patología. Por este motivo, Ng et al estudiaron la expresión del gen de la hormona liberadora de corticotropina (CRH) en mujeres con preeclampsia en el tercer trimestre. Se encontró que la expresión de CRH se encontraba disminuida en mujeres con preeclampsia comparado con los controles68. En el 2006, Farina et al69 estudiaron la expresión de un panel de ARNm y encontraron diferencias estadísticamente significativas en comparación con los controles en los transcritos de hPL, inhibina A, KISS-1, proteína plasmática asociada a embarazo (PAPP-A), inhibidor del activador de plasminógeno-1 (PAI-1), selectina-P y receptor del factor del crecimiento vascular endotelial (VEGFR) y a raíz de estos resultados se analizó si era posible predecir la preeclampsia en mujeres asintomáticas midiendo la expresión de la proteína placentaria 13 (PP13), dando como resultado diferencias estadísticamente significativas entre mujeres asintomáticas que desarrollaron preeclampsia y controles sanos70. Adicionalmente, se han estudiado los genes involucrados en la coagulación activador de plasminógeno tisular (tPA) y PAI-1, y se encontró que su expresión está significativamente incrementada en pacientes con preeclampsia (p<0,0001)71. Purwosunu et al (2009) evaluaron los niveles de ARNm de PAI-1, tPA, factor de crecimiento endotelial vascular, receptor 1 del factor de crecimiento endotelial vascular, endoglina, gen específico de placenta-1 y selectina P en mujeres con y sin preeclampsia. Se encontró que la expresión de todos los genes evaluados estaba aumentada en el plasma de pacientes con preeclampsia y la expresión génica de todos los genes evaluados se correlaciono positivamente con la severidad de la enfermedad72.
Adicionalmente, en RCIU se ha reportado que la concentración de ARNm de la hormona del crecimiento (GH2) se correlaciona de manera estadísticamente significativa con el peso al nacer y las medidas biométricas fetales. Además, la concentración de ARNm de ADAM12 se encontró aumentada en gestantes con RCIU y preeclampsia con diferencias estadísticamente significativas comparado con las pacientes normales73.
Masuzaki et al encontraron que los niveles de ARNm de los genes ßhCG y hPL disminuyen y aumentan, respectivamente, en pacientes con placenta previa74.
DiscusiónEl estudio de los ácidos nucleicos fetales libres circulantes en el plasma materno abre nuevas posibilidades para estrategias en la detección de condiciones fetales que predispongan a enfermedad, diagnóstico prenatal no invasivo, determinación de riesgo para sufrir complicaciones del embarazo y severidad de la patología. También muy importante es el impacto que ha tenido en la generación de conocimiento sobre la fisiología de la placenta y la regulación de la unidad madre-feto-placenta, es probable que este conocimiento derive en nuevas estrategias en la prevención, diagnóstico, pronóstico y tratamiento de patologías fetales y del embarazo. Finalmente, cabe resaltar que el estudio de ARN fetal libre en plasma materno tiene la ventaja de poder determinar perfiles de expresión génica mientras que el análisis de ADN fetal libre en plasma materno tiene la ventaja de poder evaluar epigenéticamente al feto.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.