Introducción
La shigelosis es una infección invasiva del colon, ocasionada por Shigella spp., que origina un espectro de manifestaciones clínicas que van desde procesos leves de diarrea autolimitada a cuadros de disentería bacilar. Existen 4 especies de Shigella: S. sonnei, S. flexneri, S. boydii y S. dysenteriae. S. sonnei suele producir casos menos severos que las otras especies y en nuestro medio representa más del 80% de los aislamientos del género1. En España esta infección ha experimentado un marcado descenso, pasando de cifras de aislamiento en torno al 1-5% en la década de 19801,2 a resultar actualmente un hallazgo casi excepcional (en el momento presente un porcentaje elevado de las cepas de Shigella sp. aisladas en nuestro medio, lo son como causa de diarrea en viajeros internacionales). Ocasionalmente se detectan brotes de origen hídrico3 o alimentario4. También son posibles los brotes de transmisión interpersonal, especialmente en guarderías y escuelas infantiles5,6. El objetivo de este trabajo es describir un brote de gastroenteritis ocasionado por S. sonnei en un colegio de Madrid en el que se realizó la tipificación molecular de los aislamientos por electroforesis en gel de campos pulsantes (PFGE), análisis de las secuencias de diversos genes implicados en el metabolismo general de la bacteria (MLST) y estudio del perfil de plásmidos.
Métodos
Ante la notificación el 2 de noviembre de 2004 de 2 casos de infección por S. sonnei en 2 hermanos que acudían a un mismo colegio se inició una investigación epidemiológica. Tras contactar con el centro escolar se comprobó que desde la segunda quincena de septiembre se habían producido al menos una decena de casos similares. Con objeto de confirmar el posible brote se confeccionó una encuesta, dirigida a los padres, que recogió información sobre antecedentes recientes de sintomatología gastrointestinal entre los 469 alumnos del colegio así como el posible contacto con otras personas que hubieran sufrido una clínica similar, tanto en el entorno familiar como extrafamiliar.
Así mismo se efectuó una visita al centro escolar para conocer la distribución de las aulas y los aseos correspondientes a cada nivel educativo, los menús servidos en el comedor escolar los días previos y la posible existencia de alteraciones en los conductos de distribución de agua potable. Se consideró caso probable a todo alumno/a que desde el día 15 de septiembre de 2004 padeció diarrea de comienzo brusco y duración igual o superior a 3 días acompañada, al menos, de dos de los siguientes síntomas o signos: emisión de sangre y/o moco en heces, fiebre o dolor abdominal. El criterio de confirmación de caso incluyó el aislamiento de S. sonnei en coprocultivo. Se realizaron coprocultivos en 5 pacientes distintos siguiendo una metodología estándar. La susceptibilidad de los aislamientos frente a ampicilina, amoxicilina/ácido clavulánico, cefotaxima, cefalotina, ácido nalidíxico, ciprofloxacino, cloranfenicol, cotrimoxazol, sulfonamidas, estreptomicina, gentamicina, kanamicina y tetraciclina se determinó mediante difusión en disco. La tipificación molecular de las cepas mediante PFGE se llevó a cabo utilizando la enzima de restricción XbaI (Roche, España) y según el protocolo formulado por PulseNet Europe (www.pulsenet-europe.org). Los patrones obtenidos fueron comparados usando el coeficiente de Dice, con una tolerancia del 1,5%. Para la construcción del dendrograma se utilizó el algoritmo UPGMA (unweighed pair group matching analysis) contenido en el programa Fingerprinting II Informatix (BioRad, España). En la ejecución del MLST se siguió el protocolo propuesto por www.shigatox.net. Cada uno de los genes estudiados (aspC, clpX, fadD, icdA, lysP, mdh y uidA) fue amplificado por reacción en cadena de la polimerasa (PCR) utilizando los iniciadores recomendados en http://shigatox.net/stec/mlst-new y a continuación secuenciado en ambas direcciones. La extracción de plásmidos se efectuó mediante el método de Kado y Liu7. Para valorar la capacidad discriminativa de estas técnicas se incluyeron en el estudio 8 cepas de S. sonnei de la colección del Laboratorio Nacional de Referencia de Salmonella y Shigella (LNRSS).
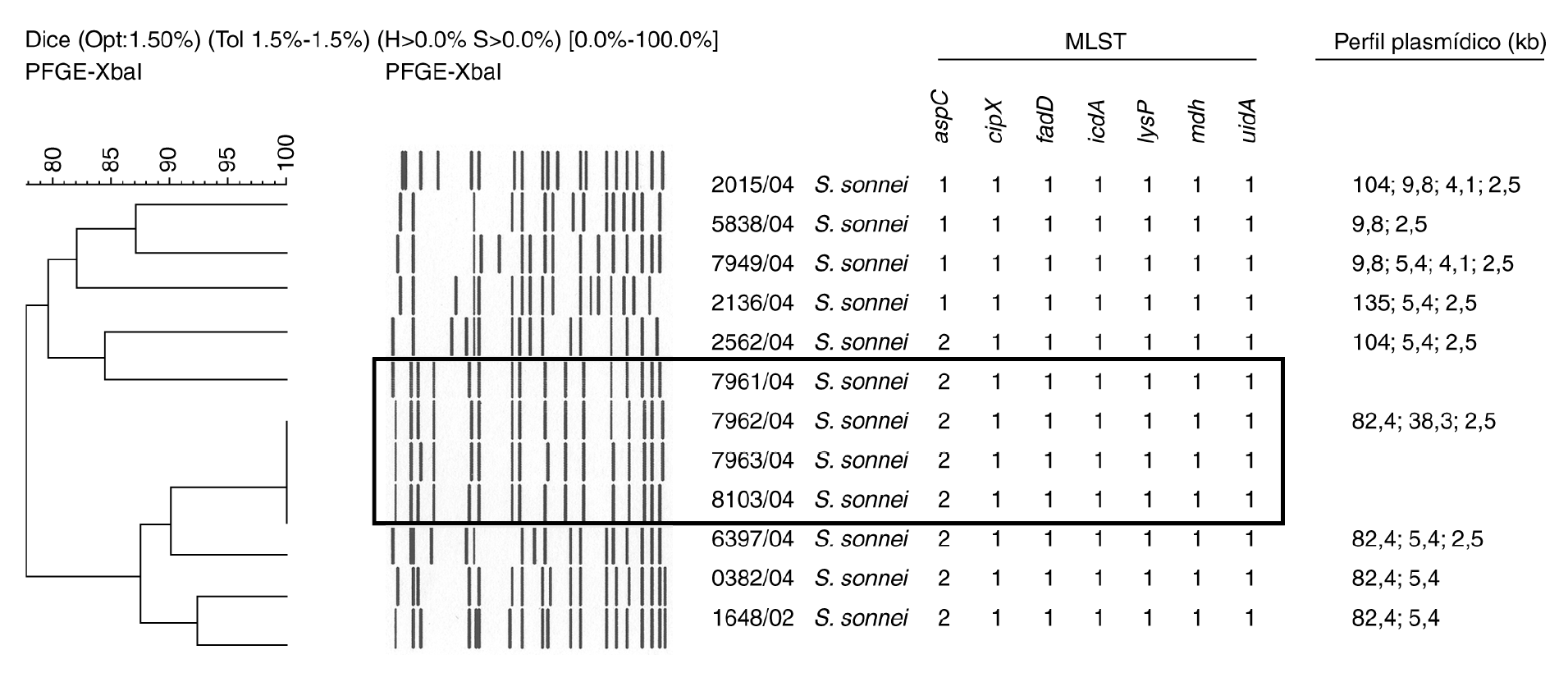
Resultados
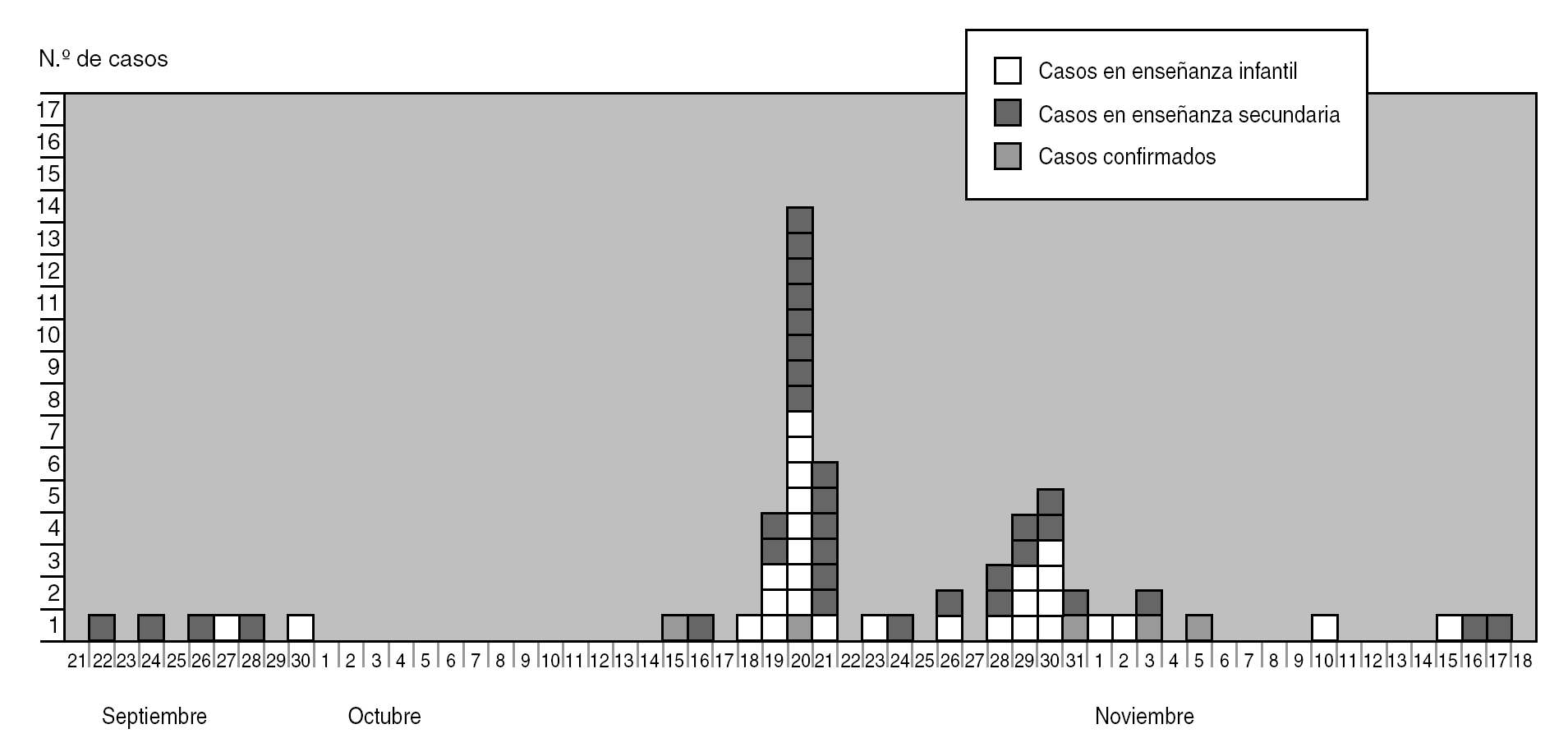
Se obtuvo respuesta en 407 (87%) de las 469 encuestas distribuidas y se detectaron en total 67 casos (tasa de ataque del 16,5% sobre el total de encuestas cumplimentadas y del 14,3% sobre el total de alumnos del colegio). Se observó una asociación significativa (p < 0,05) entre mayor tasa de ataque y menor edad (31 casos en 156 alumnos de enseñanza infantil [19,9%] frente a 36 casos en 313 alumnos de secundaria [11,5%]). El brote se prolongó por 56 días. Entre el 22 de septiembre y el 18 de octubre aparecieron los primeros cuadros clínicos y entre los días 19 de octubre y 3 de noviembre se acumularon un 80% del total de casos (fig. 1). Un total de 11 casos se dieron entre hermanos (también alumnos del mismo colegio) de otros escolares sintomáticos. No se detectó ningún caso ajeno al colegio ni en el entorno familiar ni en el extrafamiliar. Los síntomas más frecuentes (aparte de la diarrea) fueron fiebre superior a 38 °C (97%), dolor abdominal (89,6%), náuseas/vómitos (83,6%) y emisión de sangre/moco en heces (44,8%). La duración de la clínica fue de 4 a 17 días con una media de 6,5. El 56,7% de los casos ocurrieron en niñas. La mayoría de los pacientes recibieron tratamiento sintomático y fueron diagnosticados clínicamente. El primer caso confirmado apareció el 15 de octubre. En 5 pacientes se aisló S. sonnei. Cuatro de estas cepas pudieron recuperarse para ser caracterizadas. Las cuatro eran resistentes frente a ampicilina, estreptomicina, sulfonamidas y cotrimoxazol, y sensibles frente al resto de los antibióticos probados. La tipificación molecular por los tres métodos empleados mostró un patrón común (las 4 cepas exhibían una homología del 100% por PFGE, poseían la misma combinación de plásmidos [82 y 38 kb] y revelaban el mismo perfil alélico por MLST). La tipificación por PFGE y la determinación del perfil de plásmidos diferenciaron las 4 cepas del brote de otras 8 cepas control de S. sonnei pertenecientes a la colección del LNRSS y provenientes de casos esporádicos. MLST sólo reveló polimorfismo en el gen aspC, identificándose 2 alelos aspC1 y aspC2, que diferían en un único nucleótido que daba lugar a un cambio en un aminoácido. A pesar de este polimorfismo, no se logró diferenciar a las 4 cepas del brote de otras 4 cepas de la colección del LNRSS (fig. 2). Dada la demora en la detección del brote no fue posible obtener muestras de alimentos de los menús servidos en el comedor escolar durante los días de mayor incidencia (18 al 20 de octubre). Se comprobó que los alumnos de preescolar (3-5 años) no siempre iban acompañados al baño por profesorado del colegio. Además utilizaban aseos distintos de los de los niños más mayores. Estos aseos disponían de una sola toalla de uso común para el secado de manos. No se detectaron anomalías en la red de distribución de agua potable del edificio.
Figura 1. Distribución de los casos de shigelosis según el día de inicio de los síntomas.
Figura 2. Análisis de los patrones de PFGE, MLST y perfil plasmídico de las cepas de Shigella sonnei del estudio.
Discusión
Los brotes causados por S. sonnei pueden surgir esporádicamente entre niños de edad escolar y preescolar5,6. Los menores de 4 años son los más susceptibles en estas situaciones5. En el brote aquí descrito la sintomatología resultó más grave que en otros brotes de shigelosis3, con disentería en casi la mitad de los pacientes, pero comparable a la detectada en escolares de edad similar6. La mayoría de los casos se agruparon en pocos días. Este hecho podría apuntar a una exposición a una fuente común. En algunos brotes asociados con el consumo de alimentos el origen de la infección puede estar mediado por una persona colonizada4. Aunque no se llegó a confirmar la forma de introducción de la cepa en el colectivo, el brote podría haberse iniciado a partir de un individuo infectado. La aparición de varios casos previos al pico de máxima incidencia (uno de ellos confirmado 3 días antes del día que reunió mayor número de casos) respalda esta posibilidad. La desviación hacia la derecha de la curva epidémica indica que se produjo una propagación de persona a persona. La distribución de los casos según los niveles educativos apunta a diferentes grados de exposición relacionados con hábitos higiénicos deficitarios en edades tempranas6. La aparición de casos entre miembros de la misma familia (hermanos también escolarizados en el colegio) revela una asociación cercana entre casos. Entre las limitaciones metodológicas del estudio de este brote hay que señalar que la encuesta epidemiológica no se recabó información específica sobre antecedentes recientes de viajes al extranjero de los escolares o sus contactos. Por estos motivos no se puede descartar un origen importado del brote. En este brote, las 4 cepas aisladas en pacientes distintos mostraron un patrón análogo de sensibilidad antibiótica y los resultados de la tipificación molecular sugirieron una identidad común8. En consonancia con otros estudios de similares características9 la determinación del perfil de plásmidos y PFGE demostraron tener un alto nivel de discriminación. Sin embargo, el perfil plasmídico no debe considerarse una herramienta especialmente apropiada para la diferenciación entre cepas. Si bien es probable que cepas de un brote compartan el mismo perfil, también es posible que cepas no relacionadas entre sí, pero originarias de la misma zona, incorporen los mismos plásmidos (como consecuencia de fenómenos de conjugación) o alberguen otros plásmidos diferentes pero de tamaño indiferenciable. En la actualidad PFGE se considera la técnica de elección en el estudio de enteropatógenos10. El tipado mediante MLST, aunque ha sido descrito con potencial variabilidad entre las cepas de Escherichia coli diarreagénicos y Shigella sp. (www.shigatox.net), resultó de escasa utilidad como marcador epidemiológico en S. sonnei, ya que tan sólo en un gen (aspC) se identificó más de una variante alélica que, además, no permitió diferenciar inequívocamente a las cepas responsables del brote.
Correspondencia:
Dr. J.C. Sanz.
Laboratorio Regional de Salud Pública.
General Oraa, 15. 28006 Madrid. España.
Correo electrónico: juan.sanz@salud.madrid.org
Manuscrito recibido el 17-8-2006; aceptado el 12-12-2006.