Introducción
Los enterococos resistentes a vancomicina (ERV) han producido en los últimos años múltiples brotes en unidades de cuidados intensivos (UCI). Según datos de los Centros de Control de Enfermedades de EE.UU. (CDC), el porcentaje de ERV durante un período de 10 años se ha incrementado de 0,5 a 25% del total de Enterococcus spp. aislados de UCI1.
Los mecanismos de transmisión estudiados para este patógeno serían las manos del personal de salud, la contaminación ambiental y los reservorios animales2. Según la bibliografía especializada, los ERV son recuperados de las manos del personal de salud entre el 10 y el 43%3 y pueden sobrevivir en ellas durante más de 60 min tras la adquisición del microorganismo4. Hay estudios que señalan que los trabajadores de UCI que se encuentran colonizados con ERV aumentan el riesgo de transmisión a los pacientes2,5.
La contaminación con ERV de camas, instrumentales hospitalarios y objetos cercanos a los pacientes ha sido documentada6 en distintas áreas, y la UCI es una de las principales.
Los estudios de vigilancia de este patógeno permiten la detección temprana de brotes epidémicos y el control de la infección realizando las medidas de aislamiento correspondientes de pacientes infectados y/o colonizados con dicho patógeno. Por otro lado, está demostrado que los hospitales que no realizan vigilancia activa tienen el doble de riesgo de bacteriemias por ERV2,3.
En nuestro país el primer aislamiento de E. faecium resistente a vancomicina fue documentado en el año 19967. En 1997 se documentó un brote que involucró a 3 pacientes8 y en el mismo año se aislaron 2 cepas de E. faecalis con resistencia a vancomicina; en una de ellas se confirmó la presencia del gen vanB9. Desde ese período hasta la actualidad se han publicado en Argentina otros aislamientos de ERV10,11 pero, según nuestro conocimiento, no se ha realizado la búsqueda de dicho patógeno en el ambiente hospitalario y en manos de personal de salud.
En nuestra institución se realizó un estudio de vigilancia activa entre junio y agosto de 2001. No se detectaron pacientes colonizados con ERV en UCI de ambos centros hospitalarios pertenecientes al Hospital Universitario CEMIC. En dicho estudio se tomaron 134 muestras rectales de todos los pacientes que ingresaron en UCI y a los 3 y 7 días del ingreso. El 20,5% de los pacientes incluidos habían recibido antibióticos durante los 2 meses previos a la toma de la muestra12.
Ante el surgimiento de este patógeno en el año 2003, los objetivos propuestos para este trabajo fueron:
1.Caracterizar el primer brote de ERV en nuestra institución.
2.Analizar las características clínico-epidemiológicas de los pacientes con ERV.
3.Detectar reservorios ambientales y realizar la búsqueda en manos del personal.
4.Caracterizar el gen responsable de la resistencia a glucopéptidos.
5.Establecer la relación clonal entre los aislamientos.
Materiales y métodos
El Hospital Universitario CEMIC posee dos centros de hospitalización (Centro Médico Hospital I [CM 1] y Centro Médico Hospital II [CM 2]) a una distancia de 20 km entre sí. Cada centro dispone de una unidad de cuidados intensivos (UCI 1 y UCI 2) con 7 camas disponibles en UCI 1 (a, b, c, d, e, f y g) y 4 camas disponibles en UCI 2 (A, B, C y D).
En el mes de agosto del año 2003 se detectó el primer caso de ERV en nuestra institución, en el que el microorganismo se recuperó de una muestra de líquido pleural de un paciente con insuficiencia renal crónica y diabetes tipo II internado en UCI 1. Tras confirmar dicho aislamiento, se implementaron cultivos de vigilancia activa a todos los pacientes que ingresaban a la UCI (113 pacientes) durante el período comprendido entre agosto y diciembre del año 2003. Las muestras fueron conservadas y transportadas en medio de transporte de Stuart.
Entre los períodos de noviembre y diciembre se realizaron tres muestreos de cultivos de muestras ambientales y de manos de personal de salud. Dichas muestras fueron obtenidas con gasas estériles e incubadas en 10 ml de caldo cerebro-corazón. Durante el primer muestreo ambiental se tomaron 19 muestras en total en UCI 1: barandas de camas (14), monitor (1), respirador (1), bomba (1), bipap (1) y picaporte (1). En UCI 2 se tomaron 12 muestras ambientales: barandas de cama (8), monitor (2) y respirador (2).
En el segundo muestreo ambiental se tomaron 16 muestras en total en UCI 1: de barandas de camas (14), respiradores (1) y bomba (1). En UCI 2 se tomaron 7 muestras ambientales: barandas de camas (6) y respirador (1). Durante el tercer muestreo se tomaron sólo muestras de UCI 1: barandas de cama (14) y respirador (1).
Simultáneamente se tomaron 23 muestras de manos del personal de salud, 14 en UCI 1 (5 enfermeros, 2 médicos de planta, 5 médicos residentes y 2 contactos cercanos) y 9 en UCI 2 (3 enfermeros, 4 médicos residentes y 2 contactos cercanos).
Las muestras provenientes de pacientes, ambientales y del personal de salud fueron incubadas durante 18 h a 35 °C y subcultivadas en agar bilis esculina azida sódica (ABEA) suplementado con vancomicina (6 μg/ml). El cultivo se consideró positivo cuando crecieron colonias negras, correspondientes a cocos grampositivos en cadena, catalasa negativos y productores de pirrolidonil arilamidasa (PYR) y leucina amonipeptidasa (LAP). Se completó la identificación sobre la especie utilizando los esquemas de identificación bioquímica según Murray et al13.
Los estudios de sensibilidad antibiótica se realizaron por el método de difusión por discos según National Committee for Clinical Laboratory Standard y Clinical and Laboratory Standard Institute (NCCLS/ CLSI)14 para ampicilina (10 μg), ciprofloxacina (5 μg), estreptomicina de alta carga (300 μg), gentamicina de alta carga (120 μg), vancomicina (30 μg), teicoplanina (30 μg) y linezolid (30 μg). La resistencia a vancomicina y teicoplanina se confirmó utilizando placas de agar que contenían 6 μg/ml de vancomicina14 y se determinó la concentración inhibidora mínima (CIM) según recomendaciones del NCCLS/CLSI15.
La presencia del gen vanA fue confirmada por la amplificación de un fragmento de 766 pb del gen vanA mediante la técnica de reacción en cadena de la polimerasa (PCR) utilizando cebadores específicos, VANAF (59 -GCTATTCAGCTGTACTC3-9) y VANAR (59 -CAGCGGCCATCATACGG-39)16.
La relación genética entre los aislamientos de ERV provenientes de muestras de pacientes y del ambiente fueron evaluadas por la técnica de electroforesis de campo pulsado (PFGE: Pulsed-field gel electrophoresis). Tras la lisis de ADN cromosómico embebido en bloques de agarosa se procedió a la restricción utilizando la enzima SmaI y posterior electroforesis, según condiciones estandarizadas por el fabricante17. Los patrones de macrorrestricción se interpretaron siguiendo los criterios de Tenover et al18.
Con el objeto de analizar qué factores de riesgo anteriormente documentado en otros estudios19,20 pudieron asociarse a la colonización y/o infección con ERV, se determinaron las características clínico-epidemiológicas de los pacientes tales como edad, sexo, motivo de internación, enfermedad de base, procedencia de otra institución, tratamientos antibióticos previos, cantidad de aislamientos de ERV y tiempo de permanencia en el hospital.
En nuestro estudio, se definió como infección por ERV el aislamiento del germen de un sitio estéril y/o la presencia de fiebre o foco clínico atribuible al mismo. En las restantes situaciones se lo asumió como colonizante.
En el análisis de los datos se determinó la media de edad y el tiempo de hospitalización de los pacientes analizados21.
Resultados
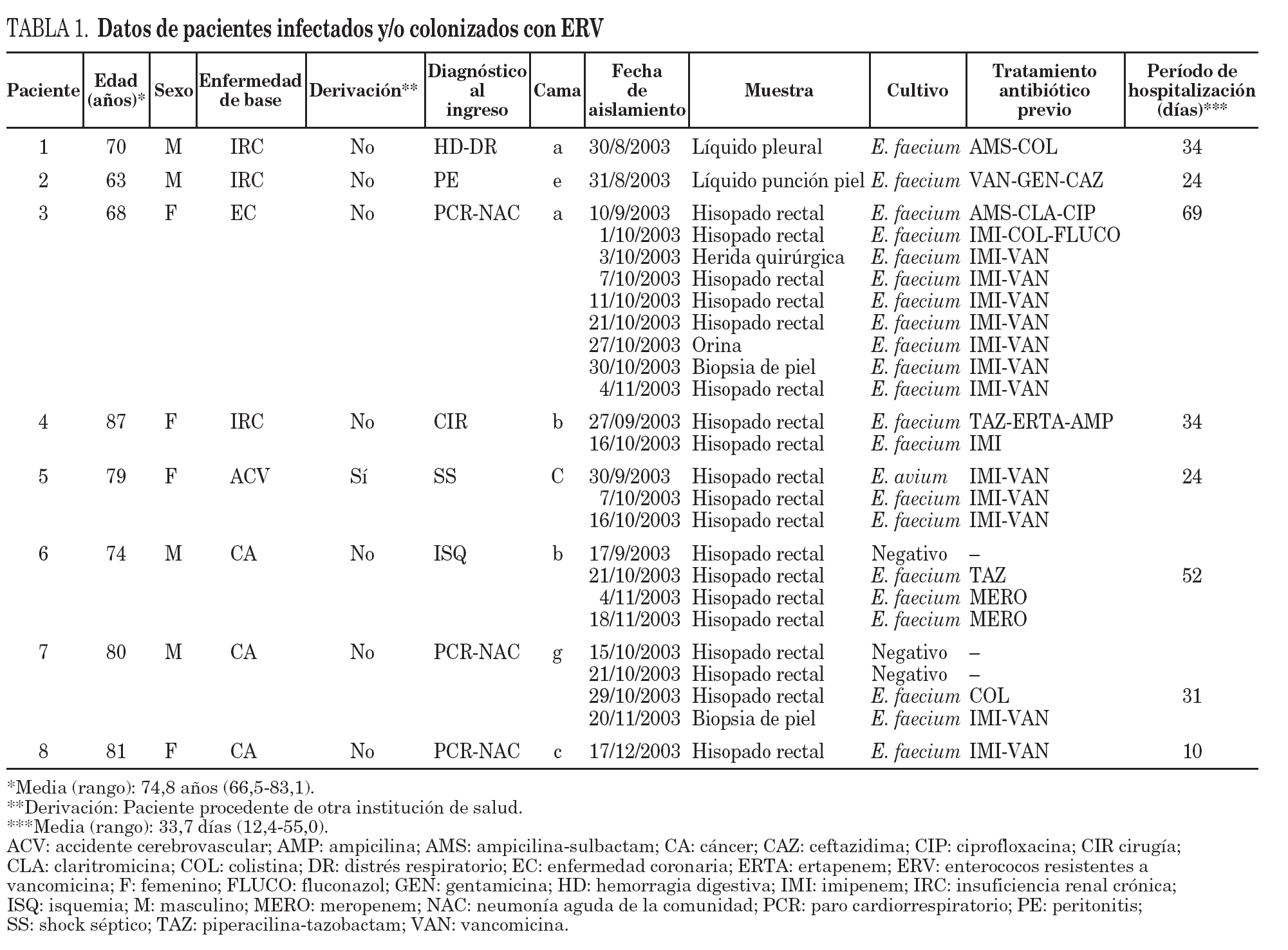
Durante el período de estudio (de agosto a diciembre) se recuperaron un total de 23 aislamientos de ERV (1 de orina, 2 de líquidos de punción, 1 de herida quirúrgica, 2 de biopsias de piel y 17 de muestras rectales tomadas con hisopo) pertenecientes a 8 pacientes de un total de 113 pacientes estudiados (7,1%).
Los aislamientos obtenidos de líquido pleural (1), líquido de punción de piel (1) y biopsia de piel (2) fueron considerados clínicamente como infecciones y los aislamientos obtenidos de herida quirúrgica (1), orina (1), hisopado rectal (17) fueron considerados colonizaciones.
De acuerdo con los datos obtenidos, la mayor parte de los pacientes infectados y/o colonizados con ERV pertenecían a UCI 1, que fue donde se registró el caso índice, y todos habían ingresado directamente a UCI 1. El análisis de las características clínico-epidemiológicas de los pacientes reveló que la enfermedad de base en los pacientes estudiados fue insuficiencia renal crónica (3), cáncer (3), enfermedad coronaria (1) y accidente cerebrovascular (1) (tabla 1).
Con respecto a los antibióticos utilizados previamente en aquellos pacientes con ERV, se observa que todos habían recibido esquemas de antibióticos de amplio espectro incluyendo carbapenemes, cefalosporinas de amplio espectro, aminoglucósidos, y 4 de ellos habían recibido vancomicina. El período de ingreso de los pacientes con ERV osciló entre 10 y 34 días con una media de 33,7 días (tabla 1).
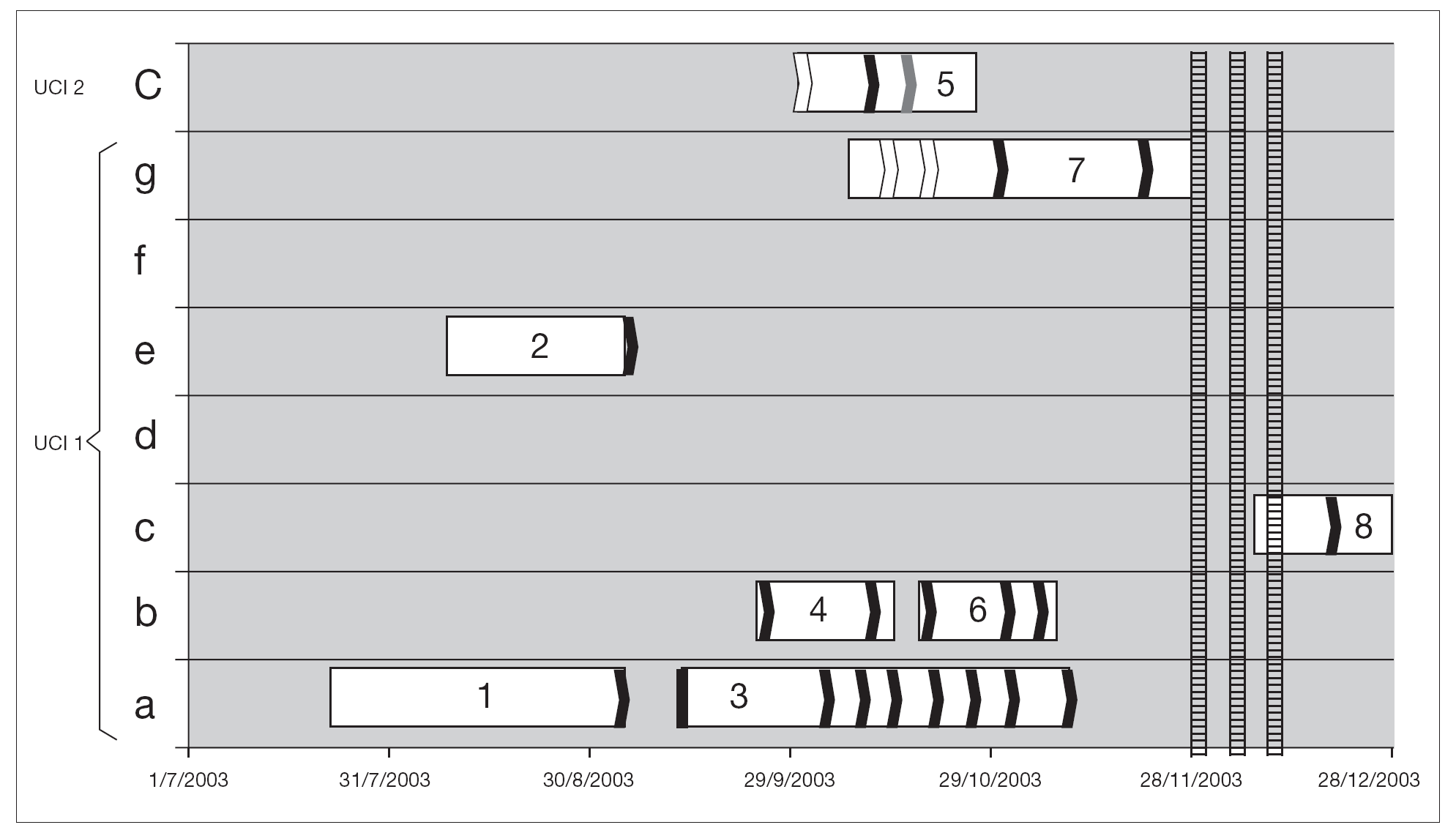
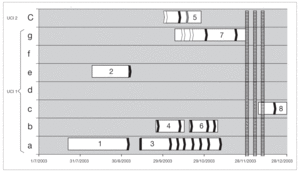
De los 8 pacientes involucrados en el brote, 7 de ellos estuvieron internados en UCI 1 y 1 en UCI 2 (fig. 1).
Figura 1. Distribución de pacientes infectados y portadores de ERV. A cada paciente se le ha asignado un número dependiendo del orden cronológico de aislamiento. El eje de abcisas muestra el tiempo de ingreso a UCI y en la ordenada la cama ocupada. Las fechas blancas señalan cultivos negativos, las negras son aislamientos de E. faecium resistente a vancomicina y la flecha gris, un aislamiento de E. avium resistente a vancomicina. Las 3 líneas rayadas muestran los 3 períodos de búsqueda ambiental.
En el estudio ambiental y de personal de salud se obtuvieron 7 aislamientos de ERV (baranda derecha [2], baranda izquierda [2], respirador [1], bomba [1], bipap [1]), todos recuperados durante el primer muestreo ambiental en UCI 1. Durante el segundo y tercer muestreo, todas las muestras ambientales fueron negativas y no se obtuvo crecimiento de ERV en las muestras de manos del personal. Las medidas de control establecidas en la institución tras los primeros aislamientos de ERV fueron: aislamientos de los portadores e infectados, limpieza y desinfección de las áreas implicadas, personal exclusivo para cada paciente y uso de instrumental desechable.
Todos los aislamientos fueron sensibles a linezolid y resistentes a ampicilina, ciprofloxacina, estreptomicina de alta carga, gentamicina de alta carga, vancomicina y teicoplanina. La CIM a vancomicina fue superior a 256 μg/ml y la CIM a teicoplanina fue de 32-64 μg/ml.
Mediante la reacción de PCR se reveló la presencia del gen vanA en todos los aislamientos de ERV.
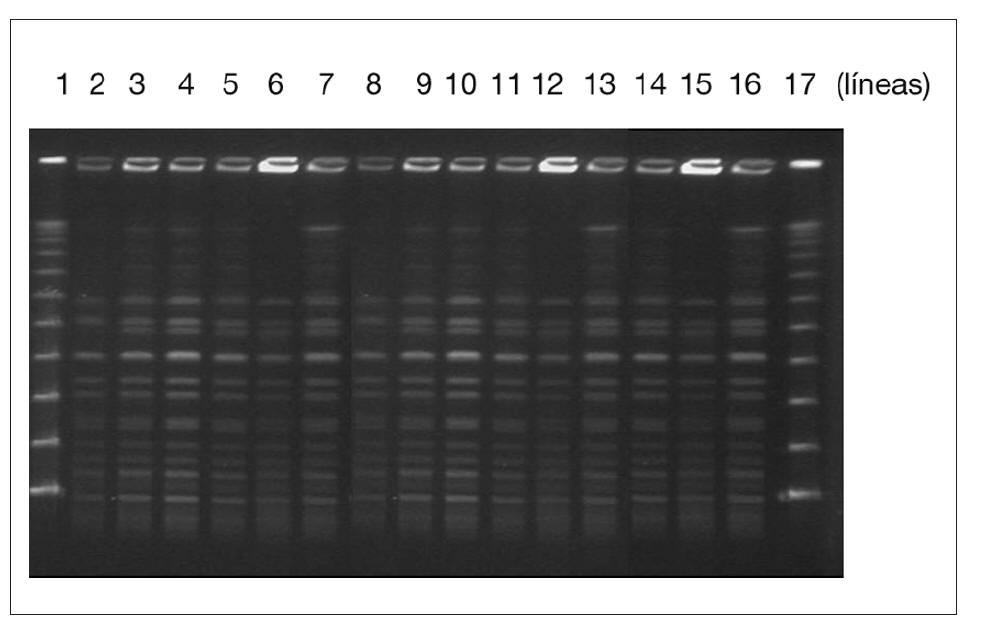
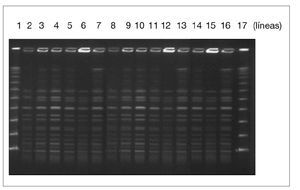
Los resultados de la técnica de PFGE evidenciaron que los aislamientos clínicos y ambientales de E. faecium presentaban el mismo patrón de bandas, documentando la presencia de un mismo clon circulante (fig. 2).
Figura 2. Patrón de macrorrestricción de los aislados de E. faecium resistentes a vancomicina de pacientes (líneas 2-9) y muestras del ambiente (líneas 10-16). Marcador de peso molecular (líneas 1 y 17).
Discusión
Clásicamente se consideraba que las infecciones producidas por Enterococcus spp. eran de origen endógeno. Sin embargo, la transmisión exógena puede ser causada a través de las manos del personal de salud, favoreciendo la circulación dentro del ambiente hospitalario y la colonización de los pacientes. En este tipo de infecciones exógenas se han aislado la mayor parte de las cepas resistentes a múltiples antimicrobianos, incluyendo vancomicina (2).
En EE.UU. los Enterococcus spp. son considerados microorganismos emergentes producto de la presión de selección del medio hospitalario y en Europa se atribuye su origen al uso de antibióticos como promotores en el engorde de animales22,23.
En Argentina se han realizado estudios de colonización/infección por ERV, pero hasta nuestro conocimiento no existen estudios que incluyan muestras obtenidas del ambiente o de manos de personal de salud.
En este trabajo se documenta el primer brote de ERV detectado en nuestra institución, a partir del cual se realizó la búsqueda de este patógeno en el ambiente hospitalario y en manos del personal.
Entre los factores de riesgo para adquirir una infección por ERV, en la literatura se hace referencia a edad avanzada, enfermedad de base (inmunosupresión, diabetes, cáncer, etc), cirugías previas, internación prolongada, hospitalización en una unidad quirúrgica oncohematológica, o de cuidados intensivos, y el uso de múltiples esquemas antibióticos de amplio espectro principalmente cefalosporinas, antibióticos antianaeróbicos y vancomicina22,23. En los pacientes analizados en nuestra institución sólo 1 provenía de otro centro de atención y 6 de ellos tenían como enfermedad de base insuficiencia renal crónica o cáncer (tabla 1). Estos antecedentes sugerirían realizar un estudio de casos y controles para detectar los factores de riesgo para colonización e infección por ERV en nuestra institución y luego evaluar la necesidad de realizar estudios de vigilancia de ERV en las salas de hemodiálisis y/o salas de tratamientos ambulatorios de pacientes oncológicos según corresponda.
Los estudios moleculares ponen en evidencia la presencia de uno o múltiples clones circulantes; esta información es útil para definir la epidemiología de un microorganismo en el hospital. En el presente estudio se aisló un único clon, lo que permitiría demostrar la transmisión horizontal nosocomial de este patógeno y el papel del ambiente hospitalario como reservorio, obstaculizando la completa erradicación de ERV durante un brote.
Aunque es muy difícil establecer cuáles son los elementos de dispersión, en el brote que describimos hay una clara coincidencia temporo-espacial, entre todos los pacientes. Siete pacientes estaban internados en UCI 1 (al igual que el caso índice), y 1 paciente de UCI 2, y se documenta transmisión cruzada entre los dos centros (fig. 1). Si bien no se realizó el estudio ambiental al inicio del brote, la presencia en el ambiente de ERV durante el mismo sugiere que las medidas de control de infecciones no fueron lo suficientemente efectivas para erradicar el mismo. La ausencia de aislamiento de este patógeno en las manos del personal en la muestra obtenida en este estudio no descarta un posible papel de los mismos en la perpetuación del brote y transmisión cruzada entre los dos centros hospitalarios.
En conclusión, nuestros resultados sugieren la necesidad de una mejor adherencia a las medidas de control de infecciones y destaca la decontaminación ambiental para evitar la transmisión de ERV, analizar los esquemas empíricos empleados, minimizar el tiempo de hospitalización y generar recursos que permitan continuar con los estudios de vigilancia para poder evaluar, en el futuro, la eficacia de las acciones educativas implementadas.
Agradecimientos
Los autores agradecen la colaboración de Andreia Penteado, Thaís Gimaraes y Fernanda Marques por la colaboración técnica en la realización del trabajo.
Correspondencia: Dra. M.S. Zárate.
Güemes, 3066, 1.º 4. 1425 Buenos Aires. Argentina.
Correo electrónico: *sole_z@hotmail.com.
Manuscrito recibido el 4-10-2006; aceptado el 13-2-2007.