Legionella pneumophila es un bacilo gramnegativo perteneciente a la familia Legionellaceae, que incluye únicamente el género Legionella, el cual engloba 48 especies y más de 70 serogrupos. L. pneumophila origina más del 90% de las infecciones, siendo el serogrupo 1 el más frecuentemente aislado en pacientes (más del 80% de los casos confirmados)1,2.
El cultivo en medio sólido buffered charcoal yeast extract (BCYE) agar es la técnica diagnóstica de referencia y, aunque su sensibilidad es baja, posibilita tanto los estudios de epidemiología molecular como los estudios de susceptibilidad1. El aislamiento en cultivo de este microorganismo presenta una serie de dificultades, como son la pérdida progresiva de viabilidad de la bacteria, el enmascaramiento de las colonias cuando se procesan muestras contaminadas con microbiota orofaríngea y el largo tiempo requerido para la obtención de los resultados susceptibilidad3. La inmunofluorescencia directa (IFD) permite detectar con rapidez la presencia de cualquier serogrupo de L. pneumophila en muestras clínicas y, aunque tiene una buena especificidad, su sensibilidad es muy baja (25–75%)3. Las pruebas serológicas presentan la limitación que supone detectar la seroconversión (se requieren al menos 9 semanas) después de haberse iniciado el cuadro clínico1. La detección del antígeno en orina permite establecer el diagnóstico etiológico de forma casi inmediata en el 80–85% de los casos, aunque solo detecta el serogrupo 14. La PCR en tiempo real permite realizar un diagnóstico directo muy sensible en menos de 2h, y cuantificar el número de bacterias en la muestra3. Presentamos el caso de un paciente diagnosticado y monitorizado mediante PCR en tiempo real.
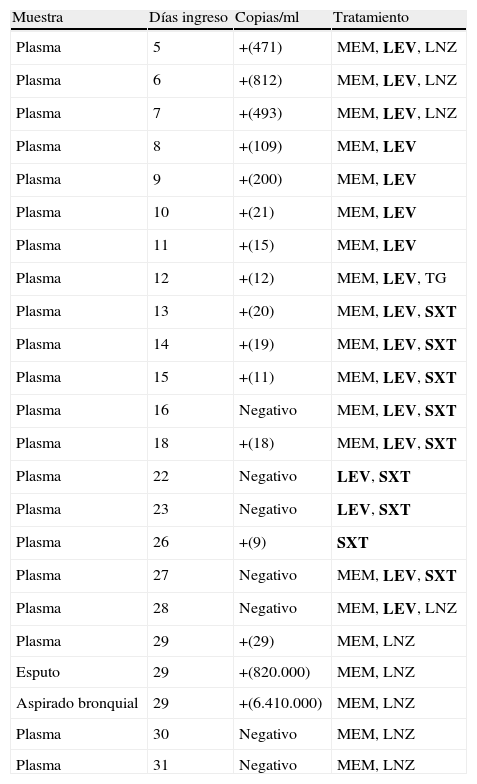
El paciente estudiado era una mujer de 69 años, inmunodeprimida, que ingresó en nuestro centro para resección de glioblastoma multiforme frontal. La placa de tórax, parámetros y analítica previos fueron normales. La paciente presentó fiebre intraoperatoria, por lo que se tomaron cultivos (aspirado bronquial tras intubación, esputo y urocultivo). Se realizó una placa de tórax en el postoperatorio inmediato, objetivándose un aumento de densidad bien delimitado en lóbulo medio. Se realizó antigenuria urgente de L. pneumophila y Streptococcus pneumoniae que fue positiva para L. pneumophila. El cultivo de aspirado bronquial fue positivo para L. pneumophila a los 21 días de incubación. Las pruebas serológicas fueron negativas. Se realizó PCR en tiempo real que detectaba el gen 16S codificante de ARNr, según el protocolo descrito por Reichl en 20025 en el equipo LightCycler 2.0 (Roche, Mannheim, Alemania) de las muestras respiratorias, que fueron positivas. La paciente recibió tratamiento antimicrobiano endovenoso (azitromicina, piperacilina-tazobactam y levofloxacino) durante 5 días, y 9 días más de levofloxacino 500mg/12h vía oral para completar los 14 de tratamiento. Ante la mejoría clínica, recibió el alta. En febrero de 2009, la paciente ingresó de nuevo por fiebre, astenia y deterioro del estado general, con neutropenia grado IV febril, por lo que se inició tratamiento antibiótico empírico con meropenem, levofloxacino y linezolid (tabla 1). Al ingreso, la paciente no podía expectorar, por lo que no fue posible obtener muestras respiratorias para cultivo. Se realizó la determinación de antígeno de L. pneumophila en orina, que fue positiva. Se recogió una muestra de suero para análisis serológico de L. pneumophila, que mostró unos títulos de IgG de 256 e IgM <10. Se recogieron muestras de plasma cada 24–72h hasta el día 33 de ingreso, día en que falleció. Se utilizaron dichas muestras de plasma para el estudio de la presencia de L. pneumophila mediante PCR en tiempo real (tabla 1).
Seguimiento del segundo ingreso del paciente mediante PCR gen codificante ARNr 16S en muestras de plasma y respiratorias (en negrita tratamientos efectivos frente a L. pneumophila)
| Muestra | Días ingreso | Copias/ml | Tratamiento |
| Plasma | 5 | +(471) | MEM, LEV, LNZ |
| Plasma | 6 | +(812) | MEM, LEV, LNZ |
| Plasma | 7 | +(493) | MEM, LEV, LNZ |
| Plasma | 8 | +(109) | MEM, LEV |
| Plasma | 9 | +(200) | MEM, LEV |
| Plasma | 10 | +(21) | MEM, LEV |
| Plasma | 11 | +(15) | MEM, LEV |
| Plasma | 12 | +(12) | MEM, LEV, TG |
| Plasma | 13 | +(20) | MEM, LEV, SXT |
| Plasma | 14 | +(19) | MEM, LEV, SXT |
| Plasma | 15 | +(11) | MEM, LEV, SXT |
| Plasma | 16 | Negativo | MEM, LEV, SXT |
| Plasma | 18 | +(18) | MEM, LEV, SXT |
| Plasma | 22 | Negativo | LEV, SXT |
| Plasma | 23 | Negativo | LEV, SXT |
| Plasma | 26 | +(9) | SXT |
| Plasma | 27 | Negativo | MEM, LEV, SXT |
| Plasma | 28 | Negativo | MEM, LEV, LNZ |
| Plasma | 29 | +(29) | MEM, LNZ |
| Esputo | 29 | +(820.000) | MEM, LNZ |
| Aspirado bronquial | 29 | +(6.410.000) | MEM, LNZ |
| Plasma | 30 | Negativo | MEM, LNZ |
| Plasma | 31 | Negativo | MEM, LNZ |
LEV: levofloxacino; LNZ: linezolid; MEM: meropenem; SXT: cotrimoxazol; TG: tigeciclina.
El día 29 de ingreso, la paciente fue trasladada a UCI y se obtuvieron muestras de esputo y de aspirado bronquial, a partir de las cuales el cultivo y la PCR en tiempo real fueron positivos para L. pneumophila serogrupo 1. En los dos episodios de legionelosis se realizó aislamiento de L. pneumophila en cultivo de la muestra respiratoria, aunque la obtención de esta en el segundo evento se demoró aproximadamente un mes desde la fecha del ingreso, debido a que la paciente no podía expectorar. La PCR ARNr 16S de muestras de plasma permitió detectar el ADN hasta el día 15 de ingreso, de manera similar a lo descrito anteriormente por otros autores6. Los días posteriores se observó que la detección de ADN en plasma era fluctuante y cercana al límite de detección de la técnica, ya que la paciente continuaba infectada, como se pudo confirmar tras la obtención de las muestras respiratorias días antes de fallecer. Se enviaron las cepas al Instituto de Salud Carlos III para tipificación por métodos moleculares. Los 2 aislamientos se trataban de la misma cepa de L. pneumophila serogrupo 1 serotipo Pontiac. Esto sugería que se trataba de una reactivación, aunque no se puede descartar una reinfección por la misma cepa.
El diagnóstico de laboratorio de la infección por Legionella spp., debería basarse en la detección del antígeno urinario combinado con una PCR en muestra respiratoria o plasma. Aunque el cultivo de muestras respiratorias es necesario para estudios epidemiológicos, esta podría ser la mejor estrategia diagnóstica inicial, que proporcionaría la máxima sensibilidad y especificidad para detectar L. pneumophila. Además, permitiría tener resultados en pocas horas que orientaría a un manejo clínico adecuado. Debido a que una vez iniciado el tratamiento antimicrobiano el cultivo de L. pneumophila puede ser negativo y a que la detección de antígeno en orina puede mantenerse positiva durante un año, la PCR a tiempo real se presenta como una posible herramienta para la monitorización de pacientes con legionelosis.