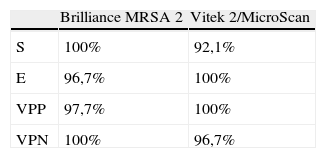
Durante los últimos 10 años, Staphylococcus aureus resistente a meticilina (SARM) se ha convertido en el patógeno nosocomial de mayor relevancia1. La septicemia por S. aureus se asocia a altas tasas de mortalidad, hospitalización prolongada e incremento de costes2. En estos casos, la instauración temprana de un tratamiento empírico apropiado es esencial para la curación de los pacientes3. Actualmente están comercializados ensayos que detectan SARM directamente del hemocultivo positivo mediante amplificación de ácidos nucleicos; sin embargo, las restricciones económicas que estamos viviendo actualmente impiden, en algunos casos, la implementación de esta tecnología en la rutina de los laboratorios. Por esta razón, es interesante conocer la eficacia de métodos fenotípicos alternativos bastante más baratos. La finalidad de este estudio fue evaluar la eficacia del medio Brilliance MRSA 2 (Oxoid) para la detección de SARM directamente desde el frasco de hemocultivo positivo. Se seleccionaron 113 hemocultivos positivos de distintos pacientes con observación microscópica compatible con estafilococos de manera que el número de SARM, de S. aureus sensible a meticilina (SASM) y de estafilococos coagulasa negativos resistentes a meticilina (SCNRM) fuera similar (n≥30 para cada uno de ellos). Estos se sembraron en agar Columbia (Becton-Dickinson, Sparks, MD, Estados Unidos) y medio cromogénico Brilliance MRSA 2 (Oxoid, Reino Unido). La identificación y el antibiograma de los 123 aislamientos recuperados se hicieron con Vitek 2 (tarjetas ID-GP, AST-P588) (BioMérieux, Francia) y/o MicroScan (panel PC31) (Siemens, Alemania) y los resultados se interpretaron de acuerdo con los criterios del CLSI4. Los casos en que hubo discrepancias entre el antibiograma y el crecimiento en la placa cromogénica se resolvieron mediante la detección de la proteína PBP2′ por aglutinación (Slidex MRSA Detection, BioMérieux, Francia) y PCR del gen mecA5. La distribución de estafilococos por especies fue: 72 S. aureus, 51 estafilococo coagulasa negativo (36 S. epidermidis, 2 S. hominis, 8 S. haemolyticus, 1 S. warneri, 2 S. saprophyticus, 2 S. lugdunensis). Todos los estafilococos identificados como SARM crecieron de color azul intenso en las placas Brilliance MRSA 2, independientemente de que estuvieran en cultivo puro o no. Solo hubo 2 aislamientos de S. epidermidis en los que las colonias presentaron color azul más oscuro parecido al propio de SARM. Es importante recordar que se recomienda efectuar la lectura de las placas a las 24h para favorecer el desarrollo del color característico, evitando así falsos negativos de color más claro. Tres de los aislamientos de S. aureus fenotípicamente identificados como sensibles a meticilina crecieron en el medio cromogénico y fueron reconocidos como SARM por métodos moleculares. Pensamos que se trata de poblaciones heterorresistentes en las que la proporción de la población resistente es muy pequeña, por lo que, utilizando inóculos bajos, puede no ser detectada; sin embargo, con un inóculo de mayor tamaño (hemocultivo directo) en un medio selectivo (Brilliance MRSA 2) la población resistente es evidenciada. De hecho, crecieron pocas colonias en Brilliance MRSA 2. Según estos resultados, la sensibilidad (100%) y el valor predictivo negativo (100%) del medio Brilliance MRSA 2 para la detección de la resistencia a meticilina fueron superiores a la de los 2 sistemas para antibiogramas automatizados (tabla 1).
Sensibilidad (S), especificidad (E), valor predictivo positivo (VPP) y valor predictivo negativo (VPN) de los métodos fenotípicos empleados respecto a los métodos de referencia (aglutinación PBP2′ y PCR del gen mecA)
| Brilliance MRSA 2 | Vitek 2/MicroScan | |
| S | 100% | 92,1% |
| E | 96,7% | 100% |
| VPP | 97,7% | 100% |
| VPN | 100% | 96,7% |
En conclusión, consideramos que la siembra directa en medio Brilliance MRSA 2 de los hemocultivos positivos con cocos tipo estafilococo en la observación microscópica adelanta 24h, con gran fiabilidad, la posibilidad de emitir un informe preliminar, de bacteriemia por SAMR a un precio asequible para todos los laboratorios de microbiología clínica. No obstante, para la emisión del informe definitivo consideramos necesario realizar la identificación y el antibiograma por medios convencionales a partir de las colonias obtenidas de la siembra del hemocultivo.