El presente documento intenta reflejar y actualizar las principales tareas y cometidos que un laboratorio de microbiología debería tener para realizar el diagnóstico y seguimiento de los pacientes infectados por el VIH. Se distribuye en 3 apartados: en el primero se trata el diagnóstico serológico, que en los últimos años ha sufrido una importante renovación, y en el que hemos procurado adecuarnos a las demandas diagnósticas y epidemiológicas actuales, para que desde los laboratorios podamos contribuir a no perder oportunidades de diagnóstico. En una segunda parte se describe la determinación de la carga viral plasmática, y se hace una exhaustiva revisión de los avances tecnológicos y de las recomendaciones actuales, además de abordar un tema de enorme interés clínico, la significación de la viremia persistente de bajo grado. Finalmente, en el tercer apartado se desarrolla el tema de las resistencias a los fármacos antirretrovirales tanto en pacientes naive como en fracaso, analizando la transcriptasa reversa, la proteasa y la integrasa, e incorporando como novedades las técnicas de determinación del tropismo viral, y el papel de las variantes minoritarias.
This document attempts to update the main tasks and roles of the Clinical Microbiology laboratory in HIV diagnosis and monitoring. The document is divided into three parts. The first deals with HIV diagnosis and how serological testing has changed in the last few years, aiming to improve diagnosis and to minimize missed opportunities for diagnosis. Technological improvements for HIV Viral Load are shown in the second part of the document, which also includes a detailed description of the clinical significance of low-level and very low-level viremia. Finally, the third part of the document deals with resistance to antiretroviral drugs, incorporating clinical indications for integrase and tropism testing, as well as the latest knowledge on minority variants.
En esta década se han cumplido los 30 años de la infección VIH-sida. Durante este tiempo los laboratorios de microbiología han tenido que hacer un gran esfuerzo para adaptarse a la demanda clínica que requieren estos pacientes. Durante los años 80 y 90 se tuvieron que desarrollar e implantar nuevas técnicas para el diagnóstico microbiológico de las infecciones oportunistas, en la segunda mitad de los 90 se generalizó el uso de las determinaciones de la carga vírica plasmática, lo que supuso el amanecer de la microbiología molecular, y en la primera década de este siglo se implementaron las técnicas de secuenciación para la detección y análisis de las mutaciones que confieren resistencias a los fármacos antirretrovirales. El presente artículo resume la tercera edición del procedimiento del Diagnóstico microbiológico de la infección por el VIH y en él se han intentado reflejar y actualizar las principales tareas y cometidos que un laboratorio de microbiología debería tener para realizar la atención integra al paciente infectado por el VIH. Se han respetado algunos contenidos y el objetivo de la segunda edición, orientar de forma conceptual y práctica la metodología empleada en el diagnóstico y seguimiento de la infección por el VIH. Se mantiene su estructura en los 3 apartados anteriores: diagnóstico serológico, determinación de la carga viral plasmática (CVP) y análisis de las resistencias a los fármacos antirretrovirales. Se ha incorporado un cuadro resumen con las «ideas clave» en cada uno de los capítulos, que esperamos sean de utilidad para todos los lectores del procedimiento.
Diagnóstico serológicoEl diagnóstico serológico en la presente actualización ha sufrido una importante renovación, intentando adecuarnos a las demandas diagnósticas y epidemiológicas actuales, y a contribuir a disminuir las oportunidades perdidas en cuanto al diagnóstico. El diagnóstico de la infección por el VIH se realiza mediante la detección de anticuerpos específicos en el suero o plasma del paciente. La elección de la técnica apropiada y del algoritmo diagnóstico dependerán del objetivo a alcanzar.
Pruebas de cribadoEl diagnóstico de infección por el VIH se hace generalmente en función de la serología, es decir, la detección de anticuerpos VIH-1/2 o la detección simultánea de anticuerpos VIH-1/2 y del antígeno p24 del VIH-1. Los ensayos serológicos para esta determinación pueden ser de cribado (screening) o de confirmación. Los ensayos de cribado identifican las muestras reactivas y deben tener una sensibilidad superior, y los ensayos de confirmación permiten conocer si las muestras reactivas con un ensayo de cribado contienen anticuerpos específicos para el VIH-1/2 y deben tener una especificidad superior.
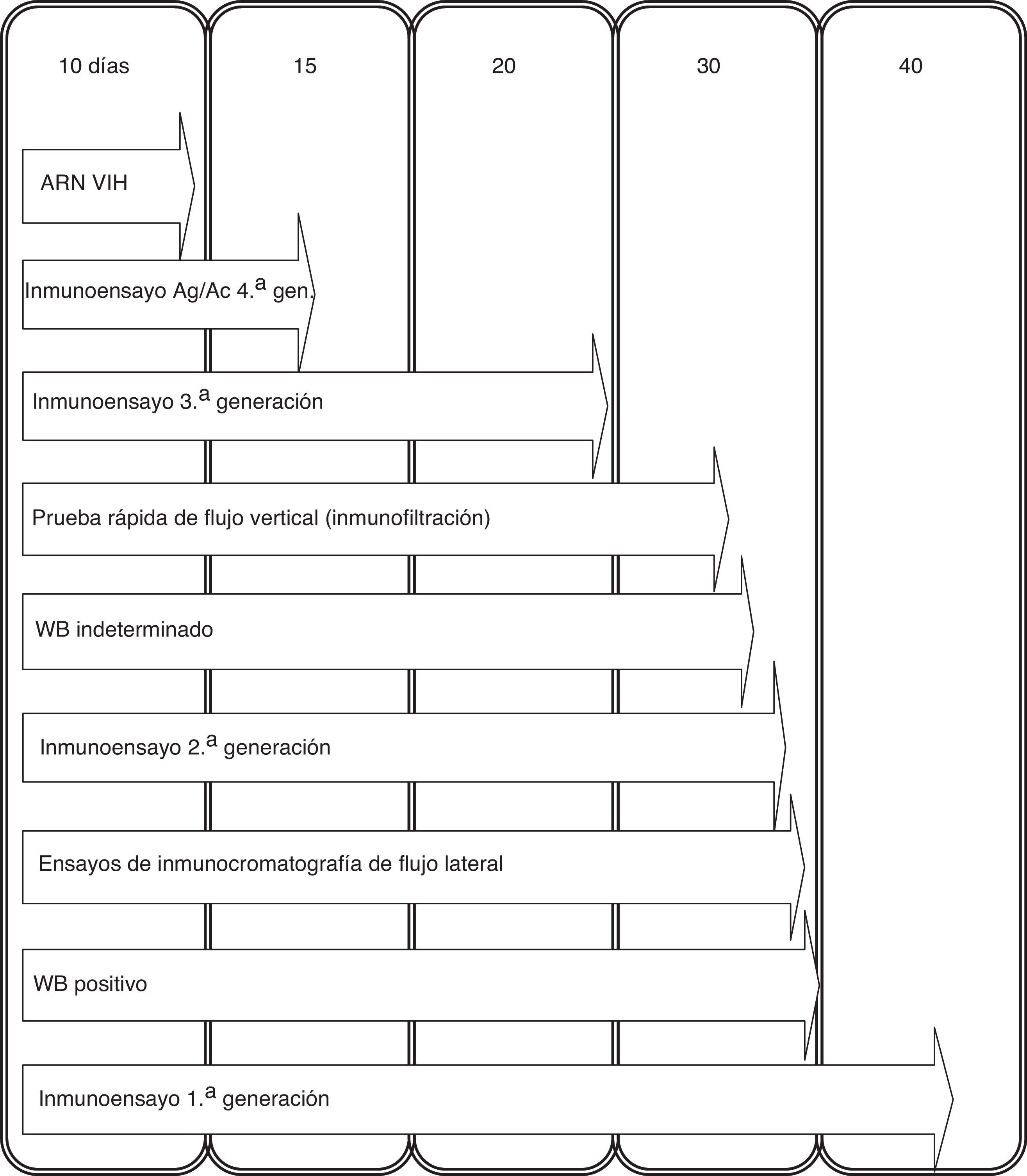
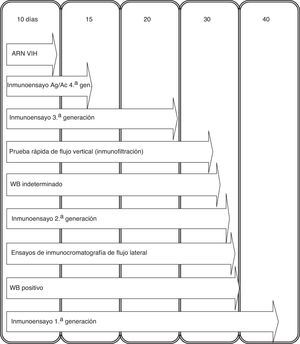
Como prueba de screening el inmunoanálisis es el más coste-efectivo para llevar a cabo en un entorno de laboratorio con un volumen importante de muestras. Debido a la diversificación de la terminología en cuanto a la denominación de los inmunoensayos (EIA, CLIA, CMIA, QIA, etc.) los englobamos a todos con dicho término. Los inmunoensayos han ido evolucionando, y se clasifican en función de la base antigénica utilizada. Los de primera generación, que incorporaban lisado viral como antígeno, eran relativamente sensibles, pero carecían de especificidad, y se detectaban los anticuerpos 40 días después de la infección (fig. 1). Posteriormente, se desarrollaron ensayos de segunda generación que utilizaban como antígenos proteínas recombinantes y péptidos sintéticos que detectaban anticuerpos frente a los subtipos del grupo M, los grupos N y O, y también frente al VIH-2. Se acortó el tiempo de detección de anticuerpos a 33-35 días. Los inmunoensayos de tercera generación o tipo sándwich detectan anticuerpos de clase IgG e IgM acortando el tiempo de detección de 20 a 25 días. Por último, se han introducido las técnicas de cuarta generación que detectan simultáneamente anticuerpos y antígeno p24, reduciéndose el tiempo de detección a 13-15 días. Con estas técnicas de cuarta generación la sensibilidad aumenta y se reduce la posibilidad de un falso negativo, aunque hay que tener en cuenta que esta circunstancia se puede dar, sobre todo, en la primera fase de la infección hasta que se produce la seroconversión (periodo ventana), y en menor medida en estadios finales de la infección, en pacientes con tratamiento inmunosupresor, trasplantados de médula ósea y en personas con alteraciones de linfocitos B. Se está evaluando un nuevo inmunoanálisis de cuarta generación que permite la detección e identificación de forma individual de los anticuerpos del VIH-1, anticuerpos del VIH-2 y del antígeno p24 del VIH-11.
Infección agudaEs la primera etapa de la infección por el VIH, inmediatamente después de esta y antes del desarrollo de los anticuerpos. El 50% de las personas pueden desarrollar manifestaciones clínicas sugestivas de un síndrome retroviral agudo. En esta fase existen niveles elevados del virus y por tanto los pacientes presentan altas tasas de transmisión, por lo que el cribado en esta fase es especialmente importante para prevenir la transmisión. Al comienzo de la infección hay un aumento de la CVP que coincide con un pico en el nivel de antígeno p24. Más tarde, la disminución de la carga viral y los niveles de antígeno p24 coinciden con el incremento de los niveles de anticuerpos. Las pruebas convencionales del VIH a veces no detectan la infección aguda, por lo que puede ser diagnosticada mediante la detección del ARN-VIH en una muestra con un inmunoensayo negativo, o cuando se detecta un inmunoensayo positivo con un test confirmatorio negativo o indeterminado y una prueba virológica positiva (ARN-VIH o antígeno p24)2.
Pruebas rápidas de diagnósticoSon técnicas de rápida ejecución, no necesitan aparataje, su lectura es visual y subjetiva y generan un resultado en menos de 30min; cuando este es reactivo se considera preliminar y se debe enviar una muestra del paciente al laboratorio de microbiología para confirmar con técnicas suplementarias. Debido a su simplicidad, coste y rápida respuesta, la OMS recomienda el uso de pruebas rápidas en entornos de recursos limitados, más que para el diagnóstico en un laboratorio convencional. Las pruebas rápidas se pueden realizar a partir de fluido oral, sangre total recogida mediante punción digital, suero y plasma. En los 2 primeros casos disponen de licencia CLIA, lo que las habilita para su uso en entornos fuera del laboratorio. Estas técnicas emplean antígenos similares a los inmunoensayos, detectando la presencia de anticuerpos VIH-1, VIH-2 y/o el antígeno p24 del VIH-1. Pueden ser de inmunoadherencia inmunocromatográfica (flujo lateral) y de inmunoadherencia por inmunofiltración o inmunoconcentración. Estas pruebas tienen una alta sensibilidad y especificidad, pero inferior a la de los inmunoensayos utilizados actualmente en el diagnóstico convencional3, incluso las de cuarta generación, que detectan anticuerpos y antígeno p244. Por tanto, las pruebas rápidas no son la mejor herramienta para identificar las infecciones agudas, donde aún no se han desarrollado anticuerpos específicos del VIH (falsos negativos)5.
Detección de antígeno p24 del virus de la inmunodeficiencia humanaEl antígeno p24 es una proteína del núcleo viral que aparece en sangre después de la infección por el VIH, coincidiendo con el aumento del nivel del ARN viral. El ensayo de detección de Ag p24 detecta un nivel de antígeno que corresponde aproximadamente a un nivel de ARN del VIH de 30.000 a 50.000copias/ml y se convierte en positivo, aproximadamente, de 5 a 7 días después de la detección del ARN viral. Esta prueba apenas es utilizada hoy en día, su uso queda prácticamente restringido a los ensayos combo de anticuerpo/antígeno, y en algunos laboratorios se utiliza para el diagnóstico de la infección vertical.
Pruebas de confirmaciónTradicionalmente las pruebas más usadas son el Western Blot (WB) y el inmunoanálisis en línea (LIA). Ambas técnicas pueden llevar antígenos de envoltura del VIH-2 para poder diagnosticarlo. Uno de sus principales inconvenientes es la lectura de las bandas para hacer la interpretación, la ausencia de las mismas se interpreta como un resultado negativo, y para valorar los resultados positivos se pueden aplicar varios criterios establecidos por distintas organizaciones: la OMS considera un resultado positivo cuando hay al menos 2 bandas de glucoproteínas, el Center for Disease Control (CDC) 2 bandas de p24, gp41 y gp160/gp120, la Cruz Roja Americana (ARC) 3 bandas, una de cada gen estructural y el Consorcio de Estandarización de la Serología de Retrovirus (CRSS) establece que tiene que existir una banda de glucoproteína y otra que no sea de envoltura. También se pueden seguir los criterios de interpretación suministrados por el fabricante del ensayo confirmatorio. Se considera un WB indeterminado cuando se observan reactividades distintas a las del criterio de positividad, y puede deberse a una infección aguda o estadio muy avanzado de la enfermedad, recién nacido de madre seropositiva, sueros inactivados por el calor, paciente con factor reumatoide, reacciones cruzadas con otros retrovirus, entre otras. En estos casos es conveniente realizar una carga viral para evaluar una posible infección aguda y al mismo tiempo solicitar una nueva muestra.
El algoritmo actual con inmunoanálisis más WB o LIA es lento, costoso y requiere un equipo sofisticado que hace que sea difícil llevarlo a cabo en entornos con pocos recursos. Por todo ello, surge la necesidad de una prueba de confirmación más sencilla, más barata y rápida6. Actualmente se dispone de un sistema de confirmación y diferenciación individualizada del VIH-1 y del VIH-2. Se trata de una prueba inmunocromatográfica que contiene péptidos recombinantes o sintéticos específicos para el VIH-1 (gp160, gp41, p31, p24) y para el VIH-2 (gp140, gp36) su lectura e interpretación están automatizadas, lo que implica una gran objetividad, dando un resultado en menos de 30min.
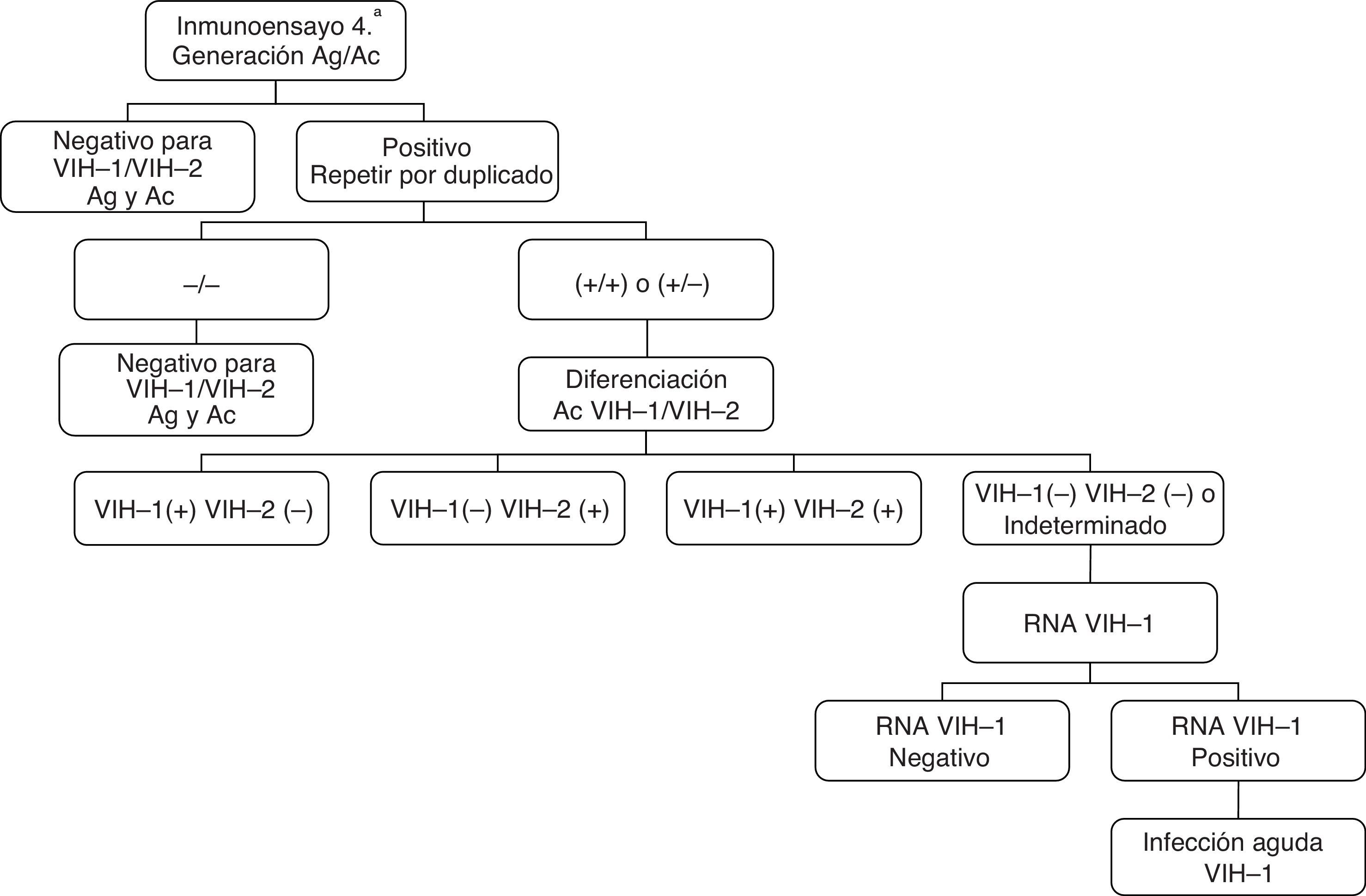
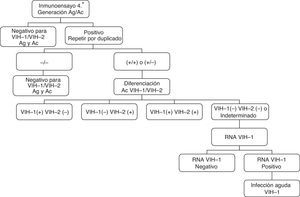
Necesidad de un nuevo algoritmo diagnósticoDistintos organismos internacionales abogan por la búsqueda de nuevos algoritmos para agilizar el diagnóstico de la infección por VIH y así reducir la transmisión, intentar que no se pierdan oportunidades de diagnóstico y resolver resultados de inmunoanálisis positivo con WB negativo o indeterminado7,8. La propuesta de un nuevo algoritmo para los laboratorios asistenciales es la siguiente (fig. 2): en primer lugar, realizar un inmunoensayo combo de cuarta generación, si resulta no reactivo, se interpreta como negativo. Si el inmunoensayo es reactivo se repite por duplicado inmediatamente, y las muestras repetidamente positivas se analizan mediante un ensayo de confirmación del VIH-1 y VIH-2, asegurando tiempos de respuesta lo más cortos posibles. Si este es concluyente se informa como positivo para el VIH-1 y/o positivo para el VIH-2, y la recomendación de enviar una segunda muestra solo para verificar que no han existido errores en la fase preanalítica, pero no hay que esperar al resultado de esta para emitir el diagnóstico. Si el inmunoensayo combo y el de confirmación son discordantes se realiza una determinación del ARN VIH-1, y si este resulta positivo, siempre que se obtengan valores altos (ver apartado de carga viral), se puede informar como una infección aguda por el VIH-1; si el ARN VIH-1 es negativo estaremos ante la ausencia de infección por el VIH-1 (falso positivo) o una posible infección aguda por el VIH-2, situación muy poco probable dada su baja prevalencia en España. El algoritmo también se puede comenzar con un inmunoensayo de tercera generación, pero se puede perder alguna infección aguda al no tener la capacidad de detectar el antígeno p24 del VIH-1.
Interpretación de resultadosLos inmunoensayos actuales tienen una sensibilidad mayor del 99%. Sin embargo no hay que olvidar la importancia de los valores predictivos positivo (VPP) y negativo (VPN), que están condicionados por la prevalencia de la infección por el VIH entre la población estudiada. A menor prevalencia el VPP disminuye y mayor es la probabilidad de que se produzcan falsos positivos. Las causas de los resultados falsos positivos son muy variadas: errores en la identificación de la muestra, hemólisis, contaminación microbiana del suero o inactivación de la muestra por calor. También se han notificado en pacientes con enfermedades autoinmunes, en multíparas, multitransfundidos, hemodializados, en personas con infección por otros virus (VPH, VHB), en individuos recién vacunados frente al VHB y gripe o en pacientes con anticuerpos frente a distintos antígenos HLA.
Finalmente, es necesario resaltar que los resultados de las pruebas de cribado deben expresarse de forma clara y precisa para evitar situaciones diagnósticas confusas y de ansiedad innecesaria en el paciente. En el caso que no se llegue a un diagnóstico definitivo se darán las pautas específicas a llevar a cabo para poder llegar a un diagnóstico concluyente.
Infección vertical y profilaxis postexposiciónEl papel de la serología en el diagnóstico del VIH en el recién nacido es limitado. En este caso el diagnóstico requiere de técnicas de biología molecular (ADN proviral o carga viral), ya que la determinación de anticuerpos carece de valor al detectarse los anticuerpos maternos. En caso de serología positiva en el recién nacido se requiere un seguimiento serológico de hasta 6 meses para verificar la desaparición de anticuerpos. Para la profilaxis postexposición la serología se utiliza para el diagnóstico de la infección por el VIH en el caso fuente de la exposición, siguiendo las recomendaciones que se describen en los apartados anteriores. Además, se emplea para el seguimiento del personal que se ha visto expuesto.
- •
La detección de anticuerpos frente al VIH en plasma o suero se debe realizar con un inmunoanálisis de screening o cribado que poseen una sensibilidad elevada y permiten detectar la infección por los distintos grupos de VIH-1 y por VIH-2.
- •
Las muestras reactivas se deben confirmar siempre con un ensayo de especificidad superior, que realizará la diferenciación entre VIH-1 y VIH-2.
- •
Se recomienda emplear inmunoensayos de cuarta generación que detectan anticuerpos del paciente y antígeno p24 viral, que reduce el periodo ventana a 13-15 días.
- •
Ante la sospecha clínica de infección aguda o ante evidencia serológica es aconsejable realizar la determinación de la CVP del VIH.
- •
El uso de pruebas rápidas está muy extendido, pero hay que poner especial atención a su lectura. Un resultado reactivo siempre hay que confirmarlo en un laboratorio de microbiología.
- •
El algoritmo diagnóstico de la infección por el VIH debe permitir emitir un resultado positivo confirmado de forma rápida para contribuir a reducir la transmisión y no perder oportunidades de diagnóstico.
La carga viral del VIH o viremia plasmática representa la concentración de virus presente en plasma y se expresa como número de copias de ARN por mililitro o en log10. Su determinación, junto con el recuento de linfocitos CD4, son los parámetros que se utilizan en la monitorización del tratamiento antirretroviral9.
Técnicas disponiblesEn la actualidad existen diversas técnicas para la cuantificación de la carga viral que tan solo difieren en sus formatos, tiempos y capacidad de procesamiento (tabla 1)10. Dichas plataformas emplean la PCR a tiempo real, que consigue altos niveles de sensibilidad analítica, reproducibilidad y linealidad. Todas ellas incorporan métodos automatizados de extracción de ARN, reduciendo el tiempo de procesamiento, la variabilidad y el riesgo de contaminación. Realizan una retrotranscripción previa del ARN, posteriormente se amplifica la diana con una PCR a tiempo real empleando sondas fluorescentes. El límite inferior de detección varía entre las 20-100 copias/ml dependiendo de la técnica, empleando un volumen de partida que varía entre 0,5-1,2ml de plasma. Detectan la CVP en todos los subtipos B y no-B del VIH-1 grupo M, algunas incluso el VIH-1 grupo O, sin embargo no detectan la CVP de VIH-2. Se ha descrito variabilidad entre dichas técnicas, sobre todo en la cuantificación de los subtipos no-B, por lo que se recomienda la monitorización de la CVP de un paciente siempre con el mismo sistema.
Técnicas disponibles para la detección de la carga viral plasmática
| Abbott RealTime HIV-1 (m2000rt) | COBAS® AmpliPrep/COBAS® TaqMan HIV-1, v2.0 (Roche) | NucliSens® EasyQ HIV-1 v1.2 (bioMérieux) | VERSANT® HIV-1 RNA 1.0 (kPCR) (Siemens) | Artus HI Virus-1 QS-RGQ (Qiagen) | |
|---|---|---|---|---|---|
| Amplificación | PCR a tiempo real | PCR a tiempo real (TaqMan) | NASBA y detección en tiempo real con molecular beacons | PCR a tiempo real (TaqMan) | PCR a tiempo real |
| Rango de carga viral | 40copias/ml10millones copias/ml | 20copias/ml10millones copias/ml | 10copias/ml10millones copias/ml | 37copias/ml11millones copias/ml | 34copias/ml44 millones copias/ml |
| Subtipos | Grupo M-subtipos A-D, F-H, J; varios CRF, incluidos CRF01_AE y CRF02_AG; grupo N y ONo detecta VIH-2 | Grupo M-subtipos A-D, F-H, varios CRF, incluido CRF01_AE y CRF02_AG; grupo ONo detecta VIH-2 | Grupo M-subtipos A-D, F-H, J; CRF01_AE y CRF02_AGNo detecta VIH-2 | Grupo M-subtipos A-D, F-H, CRF01_AE y CRF02_AG; grupo ONo detecta VIH-2 | Grupo M-subtipos A-D, F-HNo detecta VIH-2 |
| Grado de automatización | Equipo de extracción automatizada separado del equipo de amplificación/detección | Equipo integrado en uma plataforma | Equipo de extracción automatizada separado del equipo de amplificación/detección | Equipo de extracción automatizada separado del equipo de amplificación/detección | Equipo de extracción automatizada separado del equipo de amplificación/detección |
| Tiempo de procesamiento | 6h | 6h | 4h | 5,15h | 5h |
| Número de test/run | 96 | 72 | 72 | 96 | 72 |
La CVP es el principal factor para la monitorización del tratamiento antirretroviral (TAR). Esta desciende de forma acusada tras la instauración del TAR, su objetivo según la guía de GESIDA es reducir la carga viral rápidamente por debajo de los límites de detección de la técnica (20-75copias/ml) en 16-24 semanas y mantenerla suprimida el mayor tiempo posible, ya que con este nivel de carga viral se ha demostrado que no se seleccionan mutaciones de resistencia. Cuando la carga viral basal sea muy elevada el objetivo del tratamiento sería un descenso superior a 1log10 tras 4 semanas9. Se recomienda medir la CVP al comienzo del TAR y a las 4 semanas, y realizar el seguimiento cada 3-4 meses, pudiendo espaciarse su determinación hasta los 12 meses en pacientes con cargas repetidamente suprimidas y estables clínica e inmunológicamente. Se considera que hay un fracaso virológico cuando la carga viral es detectable pasadas 24 semanas desde el comienzo del TAR, o si tras alcanzar la indetectabilidad esta vuelve a ser >50copias/ml en 2 determinaciones consecutivas separadas 2-4 semanas.
La CVP se correlaciona con el riesgo de transmisión del VIH, de tal manera que cuanto mayor es la CVP mayor es el riesgo de transmisión. Aunque no puede excluirse completamente, la transmisión es poco probable por debajo de 1.000copias/ml. Esta cifra junto a las características de la exposición condicionan las diferentes pautas de profilaxis postexposición. En este sentido, hay estudios en los que se ha demostrado que el descenso de la viremia en la población general (carga viral comunitaria) se ha correlacionado con un menor evento de transmisión11.
En el diagnóstico de la infección aguda por VIH, cuando los anticuerpos específicos anti-VIH no son detectables, o cuando los inmunoensayos son positivos pero el confirmatorio es negativo o indeterminado, se puede emplear la CVP para realizar el diagnóstico siempre y cuando se obtenga un nivel de carga viral elevado. Las pruebas de carga viral no han sido desarrolladas con una especificidad suficiente y pueden causar falsos positivos, por tanto, cuando en estas situaciones de obtenga una carga viral baja es preferible utilizar otras pruebas genéticas de tipo cualitativo con sensibilidad y especificidad ampliamente demostrada, como el ADN proviral. Existen situaciones especiales en donde la determinación de la CVP se puede emplear como diagnóstico de la infección VIH, como es el caso del diagnóstico en recién nacidos de madres infectadas, ya que en estos casos los anticuerpos maternos se transfieren al niño y el diagnóstico serológico podría dar un falso positivo. Se considerarán válidos solo aquellos resultados en que el nivel de viremia sea elevado (>5.000copias/ml), si no es preferible descartar la infección mediante el uso del ADN proviral.
En países con pocos recursos se ha propuesto la utilización de gotas de sangre seca (dried blood spots [DBS]) para determinar la carga viral del VIH de un paciente, ya que este método facilita la recogida, transporte y conservación de las muestras12. Una vez seco, el DBS puede ser almacenado con desecante y enviado a los laboratorios centrales para realizar cuantificación o caracterización del VIH. La carga viral en DBS, sin embargo, puede también generar falsos positivos debido a la detección de ADN proviral o ARN viral asociado a células. La especificidad de la técnica aumenta cuando las cuantificaciones son superiores a 3.000copias/ml, cifra muy cercana al cut off propuesto por la OMS para determinar el fracaso virológico que establece en 1.000copias/ml, por tanto esta metodología se puede emplear para estas situaciones. Por otro lado, se están desarrollando otras estrategias para poder realizar CVP en pacientes de zonas alejadas o rurales como la realización de sencillos tests point-of-care (pruebas a pie de paciente) en microchip tras concentración de las muestras, o la estabilización y extracción del ARN en el centro asistencial antes de su envío al laboratorio central13.
Además se ha aplicado la determinación de carga viral sobre muestras como LCR, semen, muestras cervicovaginales o tejidos.
Interpretación de los resultadosPara expresar los resultados cuantitativos de la CVP se emplean habitualmente escalas logarítmicas. Estas permiten manejar más fácilmente las cuantificaciones en valor absoluto que presentan los virus en replicación, y que comprenden varios órdenes de magnitud. La mayoría de los pacientes infectados que no reciben tratamiento tienen una CVP detectable en un rango muy amplio. Hay que considerar también que para confirmar un fracaso virológico se debe confirmar este con una segunda determinación.
Los criterios de respuesta y fracaso virológicos siguiendo la guía de GESIDA son:
- -
Respuesta virológica: reducción de la CVP superior a 1log10 tras 4 semanas desde el inicio del TAR y CVP menor de 50copias/ml tras 16-24 semanas de iniciado el TAR.
- -
Fracaso virológico: corresponde a cualquiera de las siguientes situaciones: a) CVP detectable tras 24 semanas del inicio del TAR; b) si tras alcanzar la CVP indetectable esta vuelve a ser superior a 50copias/ml en 2 determinaciones consecutivas (separadas por 2-4 semanas). En pacientes con CVP muy elevadas (más de 300.000copias/ml) pueden requerirse más de 24 semanas para obtener una CVP indetectable.
El fracaso virológico obliga a considerar distintos factores: falta de adherencia al tratamiento, errores en la dosificación, interacciones medicamentosas o alimentarias, problemas de malabsorción o desarrollo de mutaciones de resistencias a los fármacos antirretrovirales.
Factores que aumentan la carga viralExisten diferentes factores que pueden aumentar la CVP, aparte de la progresión natural de la infección, como cuando se produce un estímulo inmunológico que aumenta la producción de partículas víricas por los linfocitos infectados. Si la medida de la CVP se efectúa tras un proceso infeccioso intercurrente o vacunación puede haber elevaciones transitorias. El tratamiento de algunas coinfecciones como tuberculosis, malaria, herpes, sífilis o gonorrea podrían reducir los niveles de CVP.
En el 15-30% de pacientes con CVP indetectable se pueden producir ocasionalmente episodios aislados y transitorios de viremia de bajo nivel (inferior a 500copias/ml) denominados blips. Esta viremia vuelve espontáneamente a ser indetectable sin ningún cambio en el tratamiento antirretroviral. Los blips podrían estar causados por la replicación del VIH-1 en reservorios, fluctuaciones biológicas o errores en el procesamiento, y no parecen tener especial relevancia clínica. Las nuevas técnicas moleculares para determinar la CVP permiten detectar una proporción importante de pacientes con niveles comprendidos entre 20 y 50copias/ml, además de otros con viremia detectable pero no cuantificable (<20copias/ml). Distintos autores han indicado que esta viremia de bajo nivel cuando se detecta de forma persistente se asocia con un mayor riesgo de fracaso virológico. Los autores de estos estudios concluyen, de forma unánime, sobre la necesidad de estudios de intervención que confirmen sus resultados14–17.
Algunos pacientes infectados (5-7%), después del pico de viremia intensa correspondiente a la infección aguda, pueden mantener la replicación del virus controlada por periodos prolongados en ausencia de tratamiento antirretroviral. En estos casos, la CVP puede ser indetectable durante meses. Esta situación suele ocurrir en pacientes con diagnóstico de certeza de infección, que se encuentren asintomáticos y con cifras de linfocitos CD4 normales. También darían indetectable la CVP cuando el VIH pertenece a una variante no detectada por la técnica que empleemos (VIH-1 grupo O, grupo N, variantes genéticas del grupo M o VIH-2); esta situación se debe resolver por técnicas serológicas y moleculares específicas.
- •
La mayoría de las plataformas actuales emplean la PCR en tiempo real para cuantificar la CVP.
- •
La CVP es el principal factor para la monitorización del tratamiento.
- •
Otras indicaciones son el diagnóstico de la infección aguda, diagnóstico de la transmisión materno-fetal y la valoración del riesgo de transmisión.
- •
Se suele realizar en plasma, si bien también se puede emplear DBS, LCR, semen, muestras cervicovaginales o tejidos.
- •
La comunicación del resultado de CVP se debe realizar en valor absoluto y escala logarítmica para facilitar su interpretación.
- •
Para países en vías de desarrollo se están desarrollando sistemas point of care para la determinación de CVP.
- •
La CVP comunitaria sirve como herramienta epidemiológica para valorar el riesgo de transmisión de VIH.
- •
Cualquier nivel de CVP supone un riesgo para el posterior fracaso virológico.
Las pruebas para la detección de resistencias a antirretrovirales pueden clasificarse en 2 tipos: genotípicas o fenotípicas18. Los métodos genotípicos determinan las mutaciones en la secuencia primaria de nucleótidos del gen del VIH objeto de estudio, comparándola con la secuencia de una cepa salvaje. La resistencia fenotípica se expresa en términos de concentración de fármaco necesario para inhibir la replicación vírica en cultivo celular. Generalmente se expresa en términos de CI50, indicando la concentración de fármaco requerida para reducir al 50% la producción viral in vitro. La interpretación de estos valores es diferente dependiendo del fármaco implicado.
Técnicas genotípicasLa secuenciación de ácidos nucleicos es el método genotípico de referencia para la determinación de la resistencia del VIH a los fármacos antirretrovirales. Se obtiene información completa de la secuencia de la retrotranscriptasa (RT), proteasa e integrasa, en busca de mutaciones que confieran resistencia al tratamiento. Los ensayos de secuenciación comerciales más utilizados han sido TRUGENE™ HIV-1 Genotyping Test (Siemens NAD) y ViroSeq™ HIV genotyping system (Abbott Diagnostics). El primero utiliza la secuenciación bidireccional y la electroforesis en gel, y permite investigar mutaciones en la RT y la proteasa; el segundo emplea el método unidireccional y la electroforesis capilar, y permite investigar mutaciones en la RT, proteasa e integrasa. Ambos sistemas ofrecen un software para la edición de las secuencias y un algoritmo propio de interpretación.
Debido a que es difícil tener un profundo conocimiento de la significación clínica de todas las mutaciones, los ensayos genotípicos se suelen acompañar de una interpretación de las mutaciones encontradas. Es en este punto, en la interpretación, cuando se acaba la reproducibilidad interlaboratorio19. El elevado número de mutaciones de resistencia y la existencia de interacciones entre ellas son algunas de las causas de los problemas en la interpretación. La interpretación de las mutaciones de resistencia se hace principalmente sobre la base de algoritmos informáticos que reconoce las mutaciones de resistencia. De hecho, el genotipo del VIH-1 interpretado por un conjunto de normas del software, predice la evolución virológica cuando se complementa con la información clínica como base para realizar un cambio de tratamiento. Existen más de 25 sistemas de interpretación; algunos están disponibles en Internet (http://www.iasusa.org/) (http://hivdb.stanford.edu/). En España disponemos del algoritmo de la Red de investigación en sida, que se actualiza periódicamente y está disponible en: www.retic-ris.net. Todos ellos son fácilmente accesibles y presentan sistemas de interpretación sencillos para todos los fármacos disponibles, sin embargo cuando se comparan los algoritmos entre sí existen serias discrepancias20.
Finalmente, hay que destacar que es importante interpretar el resultado de las pruebas de resistencia teniendo en cuenta la historia previa de tratamiento antirretroviral y, en su caso, estudios previos de resistencias que se hayan realizado al mismo paciente, intentando disponer del genotipo acumulado21.
Técnicas fenotípicasLos estudios fenotípicos tienen la ventaja de valorar con mayor fidelidad la sensibilidad del virus frente a cada fármaco, y según este grado de sensibilidad se podría valorar en un futuro la posibilidad de sobrepasar la resistencia aumentando los niveles plasmáticos del fármaco. Sin embargo, los estudios fenotípicos deben considerarse por el momento como poco viables, principalmente por la complejidad, laboriosidad de la técnica y el tiempo necesario para la emisión de resultados. Esto ha provocado que queden limitados a métodos que utilizan virus recombinantes, y que solo se realizan en laboratorios especializados de investigación y/o de casas comerciales. Los 3 ensayos de virus recombinantes que se comercializan son: Antivirogram (Tibotec-Virco), Phenosense™ (Virologic) y Phenoscript™ (Viralliance-SAS).
Técnicas para la determinación del tropismo viralEl tropismo viral en el VIH es la capacidad que tiene el virus de utilizar un determinado correceptor, como los receptores de quimiocinas CCR5 y CXCR4, para poder entrar en la célula diana, lo cual ha permitido clasificar a los aislados virales en variantes R5-trópicas, X4-trópicas y dual mixtas (D/M) en función del correceptor utilizado. La aprobación de los antagonistas de CCR5, que como el maraviroc (MVC) presentan actividad específica frente a las variantes R5-trópicas, ha generado la necesidad de disponer de ensayos para la determinación del tropismo viral en aquellos pacientes candidatos a iniciar un tratamiento con este fármaco, ya que la detección de variantes X4-trópicas se ha asociado con el fracaso al tratamiento.
Desde un principio los ensayos utilizados para la determinación del tropismo viral han sido de índole fenotípica y están basados principalmente en la generación de virus recombinantes a partir de la envuelta del virus (ESTA-Trofile, Tropitest, Toulouse Tropism Test, etc.), en la combinación de métodos genotípicos y fenotípicos (Virco type HIV-1, PhenoX-R, HIV-1 Phenoscript Env, etc.) y finalmente en el cocultivo a partir de PBMC del paciente con distintas líneas celulares como U87 (Trocai) o MT2 para determinar el efecto citopático viral. Estos ensayos constituyen todavía el método de referencia para caracterizar el tropismo viral por su gran sensibilidad para detectar las variantes minoritarias CXCR4 y/o CCR5 en los subtipos B y no-B del VIH-1, y además representan el único método para medir la resistencia a los antagonistas de CCR5. Sin embargo, este tipo de ensayos presentan además de un precio elevado complicaciones técnicas y en especial logísticas que dificultan su utilización en la práctica clínica. En este contexto, los métodos genotípicos, basados en el análisis por secuenciación de la tercera región variable (V3) de la gp120 del VIH-1 se presentan como una alternativa más económica, rápida y factible de desarrollar en cualquier laboratorio de virología que cuente con equipo de secuenciación. Se han descrito desde un principio diferentes reglas y algoritmos bioinformáticos para la predicción del tropismo viral (WetCat, WebPSSM, FortinbrasPSSM y Geno2pheno) que se fundamentan en las características de la secuencia de aminoácidos de dicha región.
Actualmente las herramientas genotípicas son comparables en la predicción de la respuesta a MVC y en la capacidad de identificar pacientes respondedores y no respondedores, a pesar de su menor sensibilidad para detectar variantes X4-trópicas respecto de los ensayos fenotípicos. No obstante, hay que considerar, en el panorama terapéutico actual, la contribución de los fármacos administrados junto a MVC para conseguir la supresión viral. Por todo esto, las diversas guías de manejo de la infección por VIH9,22 incluyen entre sus recomendaciones la posibilidad de determinar el tropismo del VIH-1 mediante métodos genotípicos para guiar el uso terapéutico de los antagonistas de CCR5.
Los métodos para la determinación genotípica del tropismo viral basados en la secuenciación directa se presentan hoy en día como la principal alternativa rápida, fiable y factible de realizar en laboratorios de microbiología molecular asistenciales23.
Limitaciones de los test de resistenciaLas técnicas de las que disponemos aseguran un 98-100% de éxito en la amplificación en muestras con una carga viral superior a 500-1.000copias/ml. No obstante, con las adecuadas modificaciones es posible amplificar prácticamente cualquier muestra con carga viral detectable, es decir, con más de 50copias/ml, y estudios recientes han demostrado la utilidad clínica de la determinación de resistencias en pacientes con viremia de bajo nivel, incluso por debajo de 200copias/ml24,25. Por otra parte, ninguno de los métodos genotípicos o fenotípicos asegura la detección de todas las variantes o cuasiespecies virales, sino que solo detectan una variante si esta representa más del 20% del total de la población. Para asegurar la detección de las variantes minoritarias es necesario recurrir a técnicas especiales (clonación de los productos de amplificación, PCR mediante diluciones al límite o PCR a tiempo real) que son de difícil implantación en la rutina del laboratorio. Este hecho explica por qué los métodos que utilizamos predicen el fracaso de un fármaco, pero no aseguran el éxito ya que las variantes minoritarias pueden ser portadoras de mutaciones de resistencia y ser seleccionadas mediante el empleo de ese fármaco en concreto. Sin embargo, la reciente introducción de métodos de secuenciación masiva (UDS), que permiten la detección de mutaciones de resistencia presentes en hasta el 0,5% de la población total, abren una posibilidad real a su implantación en la rutina asistencial.
IndicacionesLas indicaciones para la utilización de las pruebas de resistencia recomendadas a nivel nacional9 e internacional22,26 por diversos grupos de expertos incluyen:
- a)
Pacientes sin tratamiento previo: se debe realizar un test de resistencias en todos los pacientes en el momento del diagnóstico (AII), además de repetirlo en el momento de iniciar tratamiento si este se difiere (GESIDA, AII; DHHS, CIII). Se han de investigar mutaciones trasmitidas en la integrasa viral solo en aquellos casos en los que exista una alta sospecha (DHHS, CIII). Se debe realizar un test genotípico para estimar el tropismo viral en aquellos casos en los que se vaya a iniciar tratamiento con un antagonista de CCR5 (DHHS, AI).
- b)
Paciente con tratamiento antirretroviral: recomendar en todo fracaso al TAR (GESIDA, DHHS, AI). Se han de investigar mutaciones de resistencia en la integrasa solo en aquellos casos en los que se fracasa a un inhibidor de la integrasa (DHHS, CIII). Se debe realizar un test genotípico para estimar el tropismo viral en aquellos casos en los que se vaya a iniciar tratamiento con un antagonista de CCR5 (DHHS, AI). La DHHS recomienda realizar estudios de resistencia por encima de 500copias/ml (BII).
Las guías de la DHHS recomiendan además la realización de los test de resistencias en los casos en los que la supresión virológica tras iniciar tratamiento es subóptima (BIII) y en todas las mujeres embarazadas antes de iniciar tratamiento (AIII). Finalmente desaconsejan su empleo después de suspender un tratamiento (DIIII) por la reversión de la mayoría de las mutaciones que no se detectarían al ser subpoblaciones minoritarias.
Los ensayos de resistencia como herramienta para epidemiología molecularComo consecuencia de su alta diversidad genética, el VIH-1ha sido clasificado en 4 grupos (M, O, N y P) en función de la homología de secuencias genéticas completas o parciales. Mientras que los grupos O, N y P están restringidos principalmente a África central, el grupo M es el responsable de la pandemia del VIH-1. Por su parte, la alta tasa de recombinación observada dentro del grupo M contribuye a incrementar su diversidad genética, dando lugar a una gran variedad de variantes que se clasifican en subtipos (A-D, F-H, J y K), subsubtipos (A1, A2, F1 y F2) e intersubtipos como las formas recombinantes circulantes (CRF, al menos 61 CRF, http://www.hiv.lanl.gov/content/sequence/HIV/CRFs/CRFs.htmL) y únicas (URF). La distribución geográfica de los subtipos del VIH-1 es desigual, así el subtipo B solo representa un 10% de las infecciones, sin embargo es el más prevalente en los países desarrollados, de ahí su interés. El resto de subtipos y formas recombinantes que suelen agruparse como «subtipos no-B» han experimentado estos últimos años un incremento en su prevalencia en los países desarrollados; en España su prevalencia se estima en torno a un 15%. El análisis filogenético es la técnica de referencia para el subtipado y discriminación entre subtipos y CRF, sin embargo debido a su complejidad han sido desarrolladas varias herramientas de subtipado automático que usan las secuencias del gen pol obtenidas de los estudios genotípicos de resistencia y que se encuentran disponibles en la Web (REGA, Standford, geno2pheno, NCBI, STAR, EuResist, TherapyEdge y jpHMM). Por último, y por lo que respecta al VIH-2, se han descrito 8 grupos (A-H) siendo los más frecuentes a nivel mundial el A y el B; hasta el momento solo se ha descrito una única forma de CRF (http://www.hiv.lanl.gov/content/sequence/HelpDocs/subtypes-more.htmL).
- •
Los métodos genotípicos son los preferidos para la determinación de resistencias y para la estimación del tropismo.
- •
Se pueden determinar resistencias en la transcriptasa reversa (RT), proteasa (Pro) e integrasa (INT).
- •
En los estudios de resistencias del VIH la mayor variabilidad se produce a la hora de la interpretación.
- •
Las guías clínicas recomiendan la determinación de resistencias tanto en pacientes naïve (solo RT y Pro) como en los fracasos.
- •
Se debe considerar la determinación del tropismo viral en aquellos pacientes en fracaso con indicación de tratamiento con una antagonista de CCR5.
- •
A pesar de su complejidad, las técnicas de variantes minoritarias han demostrado su utilidad clínica, aunque por el momento son poco accesibles a los laboratorios de rutina.
- •
Los estudios de resistencias aportan una información fundamental para conocer la epidemiología molecular del VIH.
Los autores declaran no tener ningún conflicto de intereses.