La infección respiratoria aguda es la enfermedad más frecuente a lo largo de toda la vida del ser humano, con una variación en cuanto a su etiología condicionada, fundamentalmente, por la edad, las circunstancias medioambientales, el ámbito asistencial y la enfermedad de base. Se han identificado más de 200 virus diferentes distribuidos en seis familias implicados en la patogenia de las infecciones del tracto respiratorio. Estos hechos generan una demanda diagnóstica, cuya incorporación al ámbito asistencial no debe ser retrasada. Consciente de ello la Sociedad Española de Enfermedades Infecciosas y Microbiología Clínica ha redactado un procedimiento sobre diagnóstico microbiológico de las infecciones virales respiratorias. En éste se efectúa una puesta al día de las infecciones debidas, en primer término, a los virus de la gripe y, en segundo lugar, a otros virus respiratorios convencionales y emergentes. En todos los casos se revisan los métodos de diagnóstico virológico directo (cultivo celular, detección de antígenos y de ácido nucleico), con particular referencia a las técnicas de detección molecular y de caracterización genética.
Acute respiratory infection is the most common disease occurring over a person's lifetime, with etiological variations determined mainly by age, environmental circumstances, the healthcare setting, and the underlying pathology. More than 200 different viruses distributed in six viral families have been implicated in the pathogenesis of respiratory tract infection. These facts are generating an increasing diagnostic demand that should be incorporated into the healthcare setting without delay. To meet this demand, the Spanish Society of Infectious Diseases and Clinical Microbiology has updated its Standard Procedure for the microbiological diagnosis of viral respiratory infection. This document contains an update primarily of infections caused by influenza viruses, and secondarily, infections due to other conventional and emerging respiratory viruses. In all cases, the methods for direct virological diagnosis (cell culture, and detection of antigens and nucleic acid) are reviewed, with special reference to techniques for molecular detection and genetic characterization.
La infección respiratoria aguda es la enfermedad más frecuente a lo largo de toda la vida del ser humano, con variación en cuanto a su etiología condicionada, fundamentalmente, por la edad, las circunstancias medioambientales, el ámbito asistencial y la enfermedad de base. En nuestro entorno las enfermedades infecciosas son el motivo más frecuente de las consultas en pediatría extrahospitalaria; de ellas, aproximadamente el 70% corresponde a una infección respiratoria y más de la mitad son de origen viral. La mayoría de las infecciones respiratorias sólo afectan al tracto respiratorio superior y pueden ser consideradas leves, de curso benigno y autolimitado (catarro común, rinitis y faringoamigdalitis). Se estima que alrededor del 5% puede implicar al tracto respiratorio inferior (bronquitis, bronquiolitis y neumonía), son potencialmente más graves y, en muchos casos, requieren el ingreso hospitalario. En edad adulta las infecciones respiratorias de origen viral son una causa importante de morbilidad; sin embargo, los cuadros clínicos que precisan atención médica prácticamente se limitan a ancianos, pacientes con inmunodeficiencia grave o a aquellos con enfermedad pulmonar subyacente.
Se han identificado más de 200 virus diferentes, distribuidos en seis familias (tabla 1), implicados en la patogenia de las infecciones del tracto respiratorio.
Diferentes virus distribuidos por familias y géneros relacionados con síndromes respiratorios en el ser humano
| Familia | Género | Virus | Tipos y subtipos |
| Orthomyxoviridae | Influenzavirus A | Virus de la gripe A | H1N1, H3N2, H5N1, H7N7, H7N3, H9N2 |
| Influenzavirus B | Virus de la gripe B | ||
| Influenzavirus C | Virus de la gripe C | ||
| Paramyxoviridae | Respirovirus | Virus parainfluenza 1, virus parainfluenza 3 | |
| Rubulavirus | Virus parainfluenza 2, virus parainfluenza 4 | VPI-4A, VPI-4B | |
| Metapneumovirus | Metapneumovirus humano | ||
| Pneumovirus | Virus respiratorio sincitial | VRS-A, VRS-B | |
| Picornaviridae | Enterovirus, Rhinovirus | ||
| Coronaviridae | Coronavirus | Coronavirus 229E, Coronavirus OC43, Coronavirus SARS, Coronavirus NL63, Coronavirus HKU1 | |
| Adenoviridae | Mastadenovirus | Adenovirus | |
| Parvoviridae | Bocavirus | Bocavirus humano |
Como característica general, cada uno de los virus puede ser el agente etiológico de más de un síndrome diferente (tablas 2 y 3). Sin embargo, cada uno de ellos se relaciona mayoritariamente con un tipo particular de enfermedad, según el área geográfica, la estacionalidad y la edad del paciente. En este sentido, en nuestro entorno los rinovirus son la causa más frecuente de catarro común en los adultos, el virus respiratorio sincitial (VRS) es el más prevalente en las bronquiolitis de niños de corta edad y el virus parainfluenza tipo 1 es el agente causal del mayor número de casos de laringotraqueobronquitis aguda. Las características epidemiológicas de algunos de estos virus les confieren propiedades que también pueden ser identificativas. Los virus parainfluenza 1 y 2, virus de la gripe y VRS suelen aparecer de forma epidémica, con variabilidad estacional entre ellos.
Etiología viral de los síndromes respiratorios más frecuentemente documentados en adultos
| Virus | Catarro común | Faringitis | Traqueobronquitis | Neumonía |
| Virus respiratorio sincitial | + | + | + | – |
| Virus parainfluenza 1 | + | + | + | – |
| Virus parainfluenza 2 | + | + | + | – |
| Virus parainfluenza 3 | + | + | + | – |
| Virus parainfluenza 4 | + | + | + | – |
| Metapneumovirus humano | + | + | + | – |
| Virus influenza A | + | 2+ | 3+ | 2+ |
| Virus influenza B | + | 2+ | 2+ | + |
| Rinovirus | 4+ | 2+ | + | + |
| Coronavirus | + | + | + | + |
| Enterovirus | + | + | + | + |
| Adenovirus | + | + | + | + |
Símbolos de frecuencia relativa: + (caso aislado), 2+ (pequeña proporción de casos), 3+ (proporción considerable de casos), 4+ (mayoría de los casos).
Etiología viral de los síndromes respiratorios más frecuentemente documentados en niños
| Virus | Catarro común | Faringitis | Laringotraqueobronquitis (crup) | Neumonía | Bronquiolitis |
| Virus respiratorio sincitial | 3+ | 2+ | 2+ | 4+ | 4+ |
| Virus parainfluenza 1 | 3+ | 2+ | 4+ | 2+ | 2+ |
| Virus parainfluenza 2 | 2+ | + | + | + | + |
| Virus parainfluenza 3 | 3+ | 2+ | 2+ | 3+ | 3+ |
| Virus parainfluenza 4 | 2+ | + | + | + | + |
| Metapneumovirus humano | 2+ | 2+ | + | + | 3+ |
| Virus influenza A | 2+ | 2+ | 2+ | 3+ | 3+ |
| Virus influenza B | 2+ | 2+ | + | + | + |
| Rinovirus | 2+ | 2+ | + | + | + |
| Coronavirus | + | + | + | + | + |
| Enterovirus | + | + | + | + | + |
| Adenovirus | 3+ | 2+ | + | + | + |
| Bocavirus humano | 2+ | 2+ | + | 2+ | 3+ |
Símbolos de frecuencia relativa:+(caso aislado), 2+ (pequeña proporción de casos), 3+ (proporción considerable de casos), 4+ (mayoría de los casos).
Las focalidades infecciosas, consideradas con un criterio didáctico, son las siguientes: resfriado común, faringitis aguda, laringitis aguda, laringotraqueobronquitis aguda, otitis media aguda, sinusitis, epiglotitis, bronquitis aguda, exacerbación de la enfermedad pulmonar obstructiva crónica, bronquiolitis y neumonía.
Diagnóstico virológico de los virus de la gripeEn el diagnóstico de la gripe resulta fundamental su documentación virológica, que representa una ayuda primordial en el manejo del paciente y el control de los brotes epidémicos anuales1. Desde un punto de vista teórico, este diagnóstico puede adoptar una doble estrategia. De una parte, la que se fundamenta en métodos directos, como los que son capaces de recuperar el virus mediante su aislamiento en cultivo celular y aquellos que permiten detectar el virus en las secreciones respiratorias del paciente (detección de antígenos y de ácidos nucleicos). De otra parte, el diagnóstico indirecto que valora, por el momento, la inducción de una respuesta inmunitaria de tipo humoral mediante la detección de anticuerpos específicos en suero2.
La detección de antígenos y ácidos nucleicos permite la realización de un diagnóstico rápido, que ayuda a la toma de decisiones terapéuticas. Por el contrario, el aislamiento en cultivo celular es un diagnóstico lento y tardío en la historia natural de la infección gripal, pero de extraordinaria importancia en la caracterización epidemiológica, antigénica y filogenética de estos virus. En la actualidad, el interés de la serología se encuentra principalmente en la realización de estudios poblacionales de cobertura vacunal.
Recogida y transporte de muestrasEl requisito principal a la hora de valorar las muestras del tracto respiratorio es que éstas deben contener el mayor número posible de células epiteliales, que son en las que fundamentalmente se replica el virus. En este sentido, resultan apropiadas para la investigación de virus gripales las muestras respiratorias tales como los frotis de faringe o nasofaringe y los lavados o aspirados nasales o bronquiales, obtenidos durante los primeros días de la enfermedad.
El transporte de las muestras debe realizarse a 4°C (o en su defecto congeladas a −70°C) con objeto de asegurar la infectividad de las partículas virales. La recuperación de los virus gripales se favorece con un medio de transporte adecuado, que consiste en una solución salina a pH neutro con estabilizadores de proteínas, como el suero de albúmina bovina, y antibióticos para reducir el crecimiento de bacterias que integran la microbiota acompañante.
Aislamiento mediante cultivoLos virus de la gripe son capaces de replicarse en diferentes líneas celulares primarias, diploides o continuas, aunque la susceptibilidad a la infección es baja en la mayoría de ellas. La línea celular más comúnmente utilizada son las células Madin Darby de riñón de perro (MDCK). Las líneas celulares inoculadas con las muestras respiratorias de los pacientes se incuban a 33–35°C en presencia de tripsina para asegurar la activación proteolítica de los virus. La identificación de crecimiento del virus sobre la monocapa de células se realiza de modo convencional mediante la observación del efecto citopático causado en ellas, que consiste en la aparición de células degenerativas y redondeadas que se desprenden de la monocapa. La caracterización del virus aislado se efectúa por inmunofluorescenia mediante la utilización de anticuerpos monoclonales. En ocasiones, el efecto citopático es difícil de apreciar por lo que es necesario disponer de otros métodos para identificar el crecimiento viral en los cultivos celulares, como la hemaglutinación, la hemadsorción o la detección de antígenos virales con técnicas de inmunofluorescencia.
Una de las principales limitaciones del aislamiento de los virus de la gripe es el tiempo necesario de crecimiento e identificación en cultivo celular (4–7 días). Hay varios métodos capaces de detectar los virus de la gripe de modo más precoz, entre 1 y 3 días después de la inoculación de la línea celular y antes de la aparición del efecto citopático. El más comúnmente utilizado es el Shell vial, en el que las muestras son directamente centrifugadas sobre la monocapa celular para facilitar la adherencia y la penetración viral. Posteriormente, a las 24–48h se detecta la presencia de proteínas virales mediante inmunofluorescencia.
Los virus de la gripe también pueden aislarse tras inocular la muestra en la cavidad alantoidea de huevos de gallina embrionados, ya que estos virus se replican profusamente en las células tras su incubación a 33–35°C durante 3 días para los aislados de virus de la gripe procedentes de mamíferos, y a 37°C para los aislados aviares de virus de la gripe A. Una vez finalizada la incubación, se debe analizar por hemaglutinación los fluidos amniótico y alantoideo en busca de actividad viral. Los virus de la gripe C, sin embargo, solamente crecen en la cavidad amniótica de los embriones de pollo.
Detección de antígenos viralesLa principal ventaja de los métodos basados en la detección de los antígenos virales es su independencia de la infectividad del virus, aunque la calidad de la muestra es muy importante. Entre sus ventajas destaca el hecho de que permite una rápida obtención de resultados, generalmente en unas pocas horas después de la recepción de la muestra. Como limitación reseñable cabe apuntar que los resultados a menudo son difíciles de interpretar, la especificidad dependerá de la experiencia del personal que los realice y la sensibilidad suele ser baja. Los métodos de inmunofluorescencia y enzimoinmunoanálisis (EIA) se emplean habitualmente para la detección de los antígenos virales directamente en la muestra clínica o bien en las células del cultivo en las que previamente se ha inoculado la muestra.
Los antígenos virales utilizados generalmente para el diagnóstico son, en principio, las moléculas que se sitúan en la superficie del virus, la hemaglutinina y la neuraminidasa, y que también pueden encontrarse frecuentemente en la superficie de las células infectadas. No obstante, puesto que estas moléculas están sometidas a una continua variación evolutiva, también es posible emplear otras proteínas menos accesibles del virus, como la nucleoproteína, y por ello menos variables. Un aspecto adicional derivado de nuestra actividad en laboratorios de referencia de gripe es que, con objeto de realizar vigilancia y estudios epidemiológicos, es importante realizar la subtipificación de los virus de la gripe A. La diferenciación del virus de la gripe A del tipo B es tan importante como la diferenciación de los subtipos H1 y H3 dentro de los virus de la gripe A.
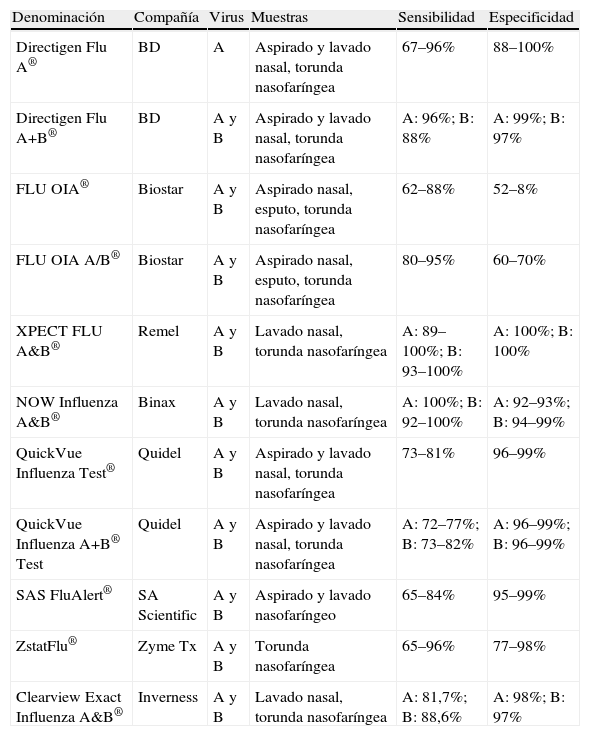
Adicionalmente, se han comercializado técnicas de inmunocromatografía capilar y de enzimoinmunoanálisis de membrana que posibilitan detectar virus gripales o sus antígenos en pocos minutos y cuya lectura es visual. No obstante, su utilidad se ve limitada debido a su coste y bajas sensibilidad y especificidad. Por su amplia implantación en la asistencia urgente y en el ámbito pediátrico recogemos en la tabla 4 diferentes pruebas de detección rápida disponibles en el momento actual y se alude a sus características operacionales intrínsecas.
Diferentes pruebas de detección rápida de antígenos disponibles para virus gripales, con alusión a sus características operacionales intrínsecas
| Denominación | Compañía | Virus | Muestras | Sensibilidad | Especificidad |
| Directigen Flu A® | BD | A | Aspirado y lavado nasal, torunda nasofaríngea | 67–96% | 88–100% |
| Directigen Flu A+B® | BD | A y B | Aspirado y lavado nasal, torunda nasofaríngea | A: 96%; B: 88% | A: 99%; B: 97% |
| FLU OIA® | Biostar | A y B | Aspirado nasal, esputo, torunda nasofaríngea | 62–88% | 52–8% |
| FLU OIA A/B® | Biostar | A y B | Aspirado nasal, esputo, torunda nasofaríngea | 80–95% | 60–70% |
| XPECT FLU A&B® | Remel | A y B | Lavado nasal, torunda nasofaríngea | A: 89–100%; B: 93–100% | A: 100%; B: 100% |
| NOW Influenza A&B® | Binax | A y B | Lavado nasal, torunda nasofaríngea | A: 100%; B: 92–100% | A: 92–93%; B: 94–99% |
| QuickVue Influenza Test® | Quidel | A y B | Aspirado y lavado nasal, torunda nasofaríngea | 73–81% | 96–99% |
| QuickVue Influenza A+B® Test | Quidel | A y B | Aspirado y lavado nasal, torunda nasofaríngea | A: 72–77%; B: 73–82% | A: 96–99%; B: 96–99% |
| SAS FluAlert® | SA Scientific | A y B | Aspirado y lavado nasofaríngeo | 65–84% | 95–99% |
| ZstatFlu® | Zyme Tx | A y B | Torunda nasofaríngea | 65–96% | 77–98% |
| Clearview Exact Influenza A&B® | Inverness | A y B | Lavado nasal, torunda nasofaríngea | A: 81,7%; B: 88,6% | A: 98%; B: 97% |
Los métodos moleculares de diagnóstico que permiten la detección de ácidos nucleicos están basados en la búsqueda y el reconocimiento del genoma viral en la muestra clínica o en el cultivo viral y en la tabla 5 están resumidos los métodos moleculares que pueden ser utilizados en la detección y/o la caracterización de virus gripales.
Características de los métodos moleculares disponibles para la detección de virus gripales
| Método | Ventajas | Desventajas |
| PCR simple | Sensible y específico. Permite análisis posteriores del producto amplificado | Una diana por ensayo |
| PCR múltiple | Sensible y específico. Permite detectar más de una diana por ensayo. Permite análisis posteriores | Los métodos no comerciales requieren una optimización exhaustiva para asegurar la ausencia de falsos negativos y la competición entre iniciadores |
| PCR-EIA | Sensible y específico. Elevado rendimiento. Puede ser múltiple | No permite el análisis posterior de los productos. Requiere evaluación y validación minuciosa antes de la integración sistemática |
| PCR a tiempo real | Sensible y específico. Rápido. Permite cuantificar. Puede ser múltiple | Requiere equipamiento específico. No siempre es posible el análisis del producto. La capacidad de analizar varios genes o patógenos en formato múltiple está limitada por las características del termociclador |
| NASBA | Sensible y específico. Permite cuantificar | Utiliza tres enzimas. Procedimiento largo y costoso. No siempre es posible el análisis del producto |
| Microarrays | Sensible y específico. Detección de muchas dianas en un solo ensayo | Requiere equipamiento específico y amplio desarrollo. No permite el análisis posterior de los productos |
La reacción en cadena de la polimerasa (PCR) es la técnica más empleada, tanto en su vertiente clásica o a tiempo final como en la de tiempo real. En ambas la reacción de amplificación tiene que ir precedida de una transcripción inversa para transformar en ADN cualquiera de los 8 segmentos de ARN que contiene el genoma de los virus de la gripe A y B o de los 7 segmentos del genoma del virus de la gripe C.
Normalmente estas técnicas de PCR se encuentran diseñadas en genes muy conservados, como los que codifican para la proteína matriz (M), la nucleoproteína (NP) o el segmento génico NS, que nos permitirán diferenciar entre los tres géneros: Influenzavirus A, B o C. Otros genes diana en la detección de Influenzavirus A, son la hemaglutinina (HA) y la neuraminidasa (NA), los cuales nos permitirán conocer el subtipo del virus gripal. Los subtipos más importantes desde el punto de vista clínico-epidemiológico son H3N2 y H1N1, porque circulan en el ser humano durante las temporadas de gripe. Desde 1997, cuando se produjo el salto interespecífico del virus gripal A del subtipo H5N1, su detección supone un gran interés por su potencial pandémico y porque su instauración con una transmisión eficiente entre personas implicaría una urgencia sanitaria de repercusión mundial3.
Con respecto a la detección mediante técnicas de PCR convencional a tiempo final, además de las técnicas «artesanales» («caseras» o in house), hay kits comerciales, en formato múltiple, como el Seeplex RV Detection 16 Kit (Seegene, Seoul, Korea), basado en tecnología DPO (dual priming oligonucleotide) que consiste en el diseño de cebadores formados por dos segmentos con el objeto de aumentar la especificidad, uno más largo que el otro y separados entre sí por un espaciador de polideoxiinosinas. Este kit, además de permitir el diagnóstico de la infección gripal por virus A y B, proporciona el diagnóstico diferencial con adenovirus, metapneumovirus, coronavirus humano 229E, OC43 y NL63, virus parainfluenza 1, 2 y 3, VRS tipos A y B y rinovirus. Uno de los principales inconvenientes de la PCR convencional o a tiempo final es que se trata de un método cualitativo, que muchas veces requiere la aplicación de una PCR secuencial (PCR anidada o nested-PCR) para obtener una sensibilidad similar a la que se alcanza con una PCR a tiempo real, lo que comporta más carga de trabajo, un incremento del tiempo necesario hasta la obtención de los resultados y un mayor riesgo de contaminaciones.
El empleo de diferentes sondas marcadas fluorogénicamente (TaqMan, sondas de hibridación, molecular beacon), cebadores marcados que dan lugar a un amplicón autofluorescente (primers scorpions, primers sunrise) o de agentes intercalantes, como el Sybr-Green, en un sistema de PCR a tiempo real, están desplazando el uso de la PCR convencional. Estos métodos de PCR a tiempo real permiten la cuantificación, además de reducir el riesgo de contaminaciones y el tiempo requerido en la emisión de los resultados, con lo que se minimiza la necesidad de un análisis posterior de los amplicones obtenidos. Si a esto se añade la posible utilización de una PCR múltiple que además de detectar los tres géneros de virus gripales con interés humano permitan la subtipificación de los virus gripales A y/o la detección simultánea de otros virus implicados en la infección respiratoria, el valor de esta metodología se ve incrementado notablemente. No obstante, se debe tener en cuenta que un paso limitante para el desarrollo de técnicas basadas en PCR a tiempo real con formato múltiple es el número de canales de detección que posea el termociclador.
Otra técnica poco utilizada en la detección de virus gripales en humanos, pero que está adquiriendo mayor protagonismo en veterinaria para la detección de virus gripales del subtipo H5 en aves de granja, es el método NASBA (nucleic acid sequence-based amplification), que se fundamenta en una retrotranscripción-amplificación y posterior detección basada en un método de electroquimioluminiscencia.
De cara a garantizar la necesidad de efectuar un diagnóstico rápido están disponibles diversos ensayos comerciales de PCR a tiempo real; algunos permiten sólo detectar Influenzavirus A y B, mientras que otros posibilitan diagnosticar la infección por H5N1 (tabla 6). La FDA ha aprobado un ensayo de PCR a tiempo real para la identificación del sublinaje asiático de virus gripales A del subtipo H5 desarrollado en los CDC. La OMS también ha publicado procedimientos para la detección de infección por H5N1 en humanos mediante RT-PCR convencional y PCR a tiempo real (disponible en: http://www.who.int/csr/disease/avian_influenza/guidelines/labtests/en/index.html).
Ensayos comerciales para la detección de virus gripales mediante amplificación genómica a tiempo real
| Ensayo | Proveedor | Termociclador | Dianas | Características |
| Artus® Influenza RT-PCR Kit | Qiagen, Bonsai | Lightcycler® | Influenzavirus A+B | Detecta ambos, pero no permite diferenciar entre virus gripales A y B |
| Artus® Influenza/H5 LC RT-PCR Kit | Qiagen, Bonsai | Lightcycler® | Influenzavirus A+B y H5 | Detecta ambos, pero no permite diferenciar entre virus gripales A y B distintos de H5 |
| LightMix for Influenza A H5N1 detection (TIB Molbiol) | Roche | Lightcycler® | Influenzavirus A, H5N1 | Debemos transformar el ARN viral en ADNc en un ensayo independiente. Único ensayo que detecta N1 |
| LightMix® for the detection of Avian Influenza A Virus (subtipo Asia) H5 (TIB Molbiol) | Roche | Lightcycler® | H5 | Debemos transformar el ARN viral en ADNc en un ensayo independiente |
| Flu A/B Analyte Specific Reagent (Cepheid) | IZASA | SmartCycler® | Influenzavirus A y B | Diferencia entre ambos tipos de virus |
| TaqMan® Influenza A/H5 Detection Kit | Applied Biosystems | ABI PRISM® | Influenzavirus A, H5 | Detección de Influenzavirus A y H5 pero en ensayos separados |
Son técnicas que permiten el procesamiento de una elevada cantidad de muestras por cada ensayo y, entre ellas, cabe destacar las basadas en PCR acoplada a enzimoinmunoanálisis (PCR-EIA) y los microarrays o biochips que posibilitan el análisis simultáneo de varios genes pertenecientes a un mismo organismo.
La PCR-EIA consiste en una amplificación cuyos productos se detectan mediante sondas en solución marcadas. La unión de la sonda se detecta mediante un enzimoinmunoanálisis cuyo anticuerpo es reactivo frente al marcador. En la actualidad hay una técnica comercial basada en la PCR-EIA: Hexaplex® (Prodesse, Milwaukee, Estados Unidos) capaz de diferenciar Influenzavirus A y B, además de detectar virus parainfluenza 1, 2, 3 y VRS4.
El crecimiento en número de infecciones en humanos por virus gripales pertenecientes a subtipos distintos de los habituales y la diseminación de la epizootia por virus del subtipo H5N1 han acrecentado el interés por la caracterización detallada de los virus gripales circulantes, tanto en el ámbito de la salud humana como en el terreno veterinario. Estos acontecimientos han derivado en el desarrollo de una serie de microarrays o biochips para la tipificación y subtipificación de virus gripales que utilizan la hibridación con sondas, en este caso inmovilizadas en una superficie, y que se incuban con el material genético a analizar marcado. En la actualidad hay publicados diversos sistemas «artesanales» no comerciales y específicos para tipificación y subtipificación de virus gripales basados en esta tecnología; cabe destacar entre ellos: el Mchip que es un microarray de baja densidad (15 oligonucleótidos marcados) dirigido frente al gen que codifica para la proteína M de los virus gripales A, que al coevolucionar con los antígenos de superficie (hemaglutinina y neuraminidasa) permite obtener información sobre el subtipo5, y el FluChip, compuesto por 55 sondas capaces de detectar virus gripales B y diferenciar todos los subtipos de Influenzavirus A6.
El descubrimiento, en los últimos años, de nuevos virus implicados en afecciones respiratorias ha estimulado el interés por el conocimiento de la infección viral respiratoria y la disponibilidad de técnicas comerciales basadas en la amplificación múltiple con posterior detección mediante sondas de hibridación. El NGEN RVA ASR (Nanogen, San Diego, Estados Unidos) combina la amplificación múltiple con un array electrónico (NanoChip 400) que utiliza sondas fluorescentes marcadas, capaz de detectar virus gripales A y B, además de virus parainfluenza 1, 2, 3 y virus respiratorio sincitial.
También están disponibles métodos comerciales para la aplicación exclusiva en la tipificación y subtipificación de virus gripales, como el equipo CombiMatrix Influenza A Detection System (CombiMatrix) que contiene un microarray capaz de subtipificar todas las hemaglutininas y las neuraminidasas de los virus gripales A, junto con un analizador y el software para interpretar los resultados.
El kit ResPlex™ III (Genaco Biomedical Products, Huntsville) está basado en un tipo de tecnología muy reciente que permite la detección y la identificación simultáneas de los genes H1, H2, H3, H5, H7, H9, N1 y N2 de los virus gripales A, y los genes NS de Influenzavirus A y B, lo que proporciona a la técnica una gran versatilidad, no sólo a la hora de genotipificar virus gripales que circulan habitualmente durante las temporadas de gripe (H3N2 y H1N1), sino también para aquellos que puedan infectar de forma esporádica a humanos (H5, H7, H9). El ensayo utiliza una tecnología denominada XMAP®, fundamentada en una PCR múltiple que origina productos de amplificación marcados con biotina que posteriormente son detectados mediante microesferas, en este caso marcadas con estreptavidina, y analizados con un instrumento Luminex (Luminex Corporation) cuya tecnología está basada en los principios de la citometría de flujo. El planteamiento es similar al de un microarray, pero con la ventaja de utilizar sondas impresas sobre partículas en suspensión, en lugar de fijas sobre una superficie, con objeto de aumentar la sensibilidad7. Emparentado con este equipo y desarrollado por la misma compañía está disponible el denominado ResPlex™ II, el cual utiliza la misma tecnología para la detección de Influenzavirus A y B, virus parainfluenza 1, 2, 3 y 4, VRS A y B, metapneumovirus, rinovirus, enterovirus y SARS-CoV; al igual que el ID-Tag™ Respiratory Viral Panel (Tm Bioscience 17, Toronto, Canadá) que permite el diagnóstico de infecciones por virus de la gripe A, subtipificación de los virus gripales A H1, H3 y H5, virus de la gripe B, adenovirus, metapneumovirus, coronavirus humano 229E, OC43, HKU1 y NL63, SARS-CoV, virus parainfluenza 1, 2, 3 y 4, VRS A y B y rinovirus8–10.
Caracterización genética de los virus gripalesEl material genético fragmentado de los virus gripales está compuesto de ARN monocatenario de polaridad negativa, la replicación y la transcripción de estos segmentos a cargo de la ARN polimerasa ocasionan errores por carecer de actividad reparadora, originando mutaciones que se conocen como variaciones menores, deriva genética o drift. La frecuencia con la que ocurren espontáneamente estas mutaciones es más elevada para las proteínas de superficie, hemaglutinina y neuraminidasa, que son los principales determinantes antigénicos; las tasas de mutación son menores para el resto de los segmentos génicos.
Otra particularidad genética que presentan los virus gripales A es su capacidad de recombinación, que puede tener lugar entre los segmentos génicos de virus de distintos orígenes (mamíferos, aves). Si en este reordenamiento están implicados los segmentos que codifican para las glucoproteínas de superficie, puede originarse un nuevo subtipo de virus que nunca haya circulado entre la población humana, que daría lugar a un virus con potencial pandémico.
Estos fenómenos de cambio e intercambio genético son los que determinan la alta variabilidad de los virus gripales y la importancia y la necesidad de realizar una caracterización genética en el contexto de la vigilancia de la infección gripal.
La amplificación de los ácidos nucleicos por PCR puede llevarse a cabo directamente en las muestras respiratorias, lo que proporciona un método diagnóstico que puede acoplarse a técnicas de tipificación rápida y caracterización, como la determinación del polimorfismo del virus en función de los fragmentos obtenidos tras su digestión con enzimas de restricción (RFLP: restriction fragment length polymorphism) y, especialmente, la secuenciación de fragmentos de los genes.
La caracterización genética ha experimentado un gran avance en los últimos años gracias al desarrollo de técnicas rápidas de PCR y a la introducción de secuenciadores automáticos que permiten la caracterización genética de los linajes de virus circulantes, lo que ha mejorado el seguimiento de la evolución de los virus de la gripe y la calidad de los datos de vigilancia de su circulación. Ésta se centra en el estudio de los virus gripales A y B debido a que los Influenzavirus C apenas sufren cambios genéticos, en general carecen de estacionalidad y habitualmente producen casos esporádicos.
SecuenciaciónUna caracterización genética concisa se realiza mediante la secuenciación de la hemaglutinina y la neuraminidasa, seguida de su análisis filogenético y del estudio de posiciones (aminoácidos) clave implicadas en alguna actividad biológica: unión a receptores, capacidad antigénica, actividad enzimática o unión de antivirales y cuyo cambio pueda tener repercusión clínica y patogénica de la cepa estudiada. No hay una correlación absoluta entre los perfiles de caracterización antigénica mediante inhibición de la hemaglutinación de las cepas variantes dentro de un determinado subtipo y el árbol filogenético originado al comparar sus secuencias de hemaglutinina. Se ha podido comprobar que la evolución genética avanza de forma gradual, mientras la antigénica lo hace de forma discontinua debido a que no siempre los cambios genéticos reflejan variaciones antigénicas en las proteínas1.
En el caso del análisis de los virus gripales actuales, la disponibilidad de amplias bases de datos y de proyectos nuevos para el depósito de secuencias (http://www.niaid.nih.gov/dmid/genomes/mscs/influenza.htm) ha facilitado la comparación de un elevado número de virus aislados a lo largo de un período de hasta 90 años. En estas bases incluso podemos encontrar secuencias del virus gripal causante de la pandemia de 1918. En el caso de la emergencia del virus gripal H5N1, los estudios filogenéticos realizados han permitido situarlo entre sus ancestros, en el linaje euroasiático, e identificar mutaciones relacionadas con su virulencia y su posible transmisibilidad, y ayudan a comprender el brote y sus posibles consecuencias.
La hemaglutinina es el principal determinante antigénico de los virus gripales. Forma espículas en la envoltura mediante la unión de tres moléculas o monómeros idénticos. El monómero de hemaglutinina contiene dos subunidades: HA1 y HA2, en la subunidad HA1 es donde se encuentran los aminoácidos causales de la antigenicidad y la unión al receptor celular, por lo tanto, la secuenciación de esta región aporta información referente a las características antigénicas y de afinidad por el receptor que se pueden comparar con las de virus originarios de un brote, virus vacunales o cepas de referencia.
La neuraminidasa es una glucoproteína de superficie con actividad enzimática que, al igual que la hemaglutinina, presenta capacidad antigénica, aunque en menor grado que ésta. Desde el desarrollo y la comercialización en 1999 de los antivirales inhibidores de la neuraminidasa (zanamivir y oseltamivir), una de las principales aplicaciones de la secuenciación de este gen es la monitorización de las resistencias genotípicas a este grupo de antivirales. El conocimiento de la sensibilidad a los inhibidores de la neuraminidasa de los virus circulantes tiene particular interés en niños e inmunodeficientes que reciban o vayan a recibir tratamiento. En este grupo de pacientes la probabilidad de seleccionar virus resistentes está incrementada por presentar mayor tasa de replicación viral y una excreción más prolongada de virus en el tiempo11.
De igual manera, la secuenciación del segmento M y, en concreto, la zona que codifica para la proteína M2 permite detectar los cambios que confieren resistencia a los adamantanos (amantadina y rimantadina). La adquisición de resistencia a este grupo de antivirales puede ser independiente de la presión selectiva ejercida por el fármaco. Los cambios genotípicos que causan la resistencia pueden originarse de forma espontánea, y dan lugar a virus genéticamente estables que conservan su capacidad de replicación y transmisibilidad12. Mediante tecnología basada en la pirosecuenciación (Pirosequencing®) la compañía Biotage ha desarrollado un método para la detección de resistencias a adamantanos. Este método de secuenciación consiste en una cascada de reacciones enzimáticas para detectar la incorporación de nucleótidos durante la síntesis de ADN, de forma que cuando un nucleótido es incorporado, se produce una reacción lumínica. Las ventajas que ofrece este sistema de secuenciación son su rapidez y relativo bajo costo. En comparación con otras técnicas de secuenciación automática, este método tiene la desventaja de permitir solamente la secuenciación de un máximo de 100 pares de bases.
Polimorfismos de longitud de fragmentos de restricciónLa técnica de RFLP (restriction fragment length polymorphism) o polimorfismos de longitud de fragmentos de restricción consiste en una primera amplificación mediante RT-PCR de el (los) gen (es) diana, seguido de un tratamiento mediante enzimas de restricción que origina fragmentos y, por lo tanto, patrones de banda característicos de la cepa estudiada y que se comparan con una cepa control. Dependiendo del abordaje, este método de genotipificación es capaz de caracterizar genes que codifican para la hemaglutinina y la neuraminidasa (subtipificación) y a la vez genes internos, lo que facilita la detección de recombinantes y la determinación del origen de los distintos segmentos (humano o zoonótico)13. La PCR-RFLP también se ha utilizado para la detección de resistencias a adamantanos. Tiene los inconvenientes de no permitir la automatización ni la diferenciación entre genes con bajo grado de polimorfismo.
La secuenciación, al proporcionar una caracterización genética más precisa, ha desplazado a este método que puede resultar útil en los laboratorios que no dispongan de un secuenciador o de un servicio centralizado de secuenciación automática.
Detección de la respuesta inmunitaria humoral específicaLa documentación indirecta de la infección por los virus de la gripe se realiza mediante ensayos serológicos, en los cuales se detecta la presencia de anticuerpos frente al antígeno hemaglutinina del virus gripal en el suero de un paciente. Esta aproximación al diagnóstico de la gripe raramente se utiliza en la práctica clínica habitual, aunque puede ser una herramienta básica en la vigilancia epidemiológica de la circulación de los virus gripales. No obstante, un análisis retrospectivo puede establecer un diagnóstico clínico en ausencia de aislamiento de virus a partir de la muestra o de detección de sus antígenos o ácidos nucleicos2.
Una de las principales dificultades del diagnóstico serológico es la necesidad de evaluar muestras de sueros en pares. Esto se requiere debido a que la infección por los virus de la gripe es frecuentemente una reinfección. Así, debe constatarse un incremento significativo del título de anticuerpos entre dos muestras consecutivas separadas entre 2 y 4 semanas. Los análisis más frecuentemente utilizados son la reacción de fijación del complemento, la inhibición de la hemaglutinación y la neutralización. No obstante, los dos últimos constituyen la base del análisis de la respuesta serológica frente a la infección gripal y permiten determinar la concentración de anticuerpos frente a los antígenos específicos de subtipo y de variante.
La reacción de fijación del complemento se utiliza para determinar anticuerpos frente a la nucleoproteína de los virus de la gripe, que es altamente conservada, a diferencia de la hemaglutinina, se emplea básicamente para diferenciar en el suero de pacientes la presencia de anticuerpos dirigidos contra los virus de la gripe tipos A y B. Estos ensayos tienen un valor importante en el caso de la circulación de nuevas variantes antigénicas, frente a las que no haya disponibilidad de anticuerpos monoclonales frente a la hemaglutinina, altamente variable en función de la deriva genética de los virus gripales. También tiene utilidad en el caso de transmisión de los subtipos del virus de la gripe A entre distintas especies. No obstante, el aumento en el título de fijación del complemento después de una infección gripal es lento y el ensayo, por sí solo, tiene una sensibilidad relativamente baja. Por otro lado, este ensayo solamente mide las clases de anticuerpos que están implicadas en la fijación del complemento.
La inhibición de la hemaglutinación se usa habitualmente para el diagnóstico y la tipificación de los virus de la gripe A y B y para la subtipificación de los virus de la gripe A (H1 y H3). El título de anticuerpos presentes en una muestra de suero, calculado mediante esta técnica, se relaciona de manera fidedigna con la protección o la susceptibilidad del paciente frente a la infección gripal. Por ello, se utiliza ampliamente para medir la respuesta del paciente a la administración de la vacuna de la gripe. Sin embargo, la realización y la interpretación de esta técnica son complejas y, frecuentemente, subjetivas, por lo que precisa para su realización de personal cualificado. Es por ello muy habitual que esta técnica carezca de reproducibilidad cuando es realizada por distintos laboratorios. Por otro lado, aunque su ventaja más evidente es su elevada especificidad, se contrarresta por la baja sensibilidad.
El principio básico del ensayo de neutralización del suero es la inhibición de la replicación viral mediante anticuerpos específicos. Los títulos de anticuerpos neutralizantes presentes en el suero del paciente, obtenidos mediante esta técnica, se correlacionan perfectamente con los obtenidos en los ensayos de inhibición de hemaglutinación. El mayor inconveniente que presenta esta técnica es que los anticuerpos monoclonales utilizados tienen que ser validados a menudo con objeto de comprobar que son capaces de reaccionar frente a las nuevas variantes de virus de la gripe.
Diagnóstico de otros virus respiratoriosHabida cuenta de que todos los virus respiratorios pueden estar implicados en la mayor parte de los cuadros de infección respiratoria, y que éstos suelen ocasionar síntomas bastante inespecíficos, al menos en sus comienzos, la realización de un diagnóstico etiológico es de suma importancia; éste puede estar encaminado: a) al tratamiento específico del paciente con el antiviral adecuado, si procede; b) a la toma de las medidas oportunas de aislamiento, o c) a la obtención de información epidemiológica que permita establecer la incidencia de un determinado virus en los diferentes procesos de infección respiratoria, en función de la edad de los pacientes o de las variaciones observadas en su distribución geográfica y estacional.
El diagnóstico virológico de la infección respiratoria dependerá de la calidad de la muestra, su transporte y las condiciones de conservación o almacenamiento hasta que es procesada. La muestra de elección es el aspirado nasofaríngeo, preferiblemente obtenido en los primeros 3 días después del inicio de los síntomas, dado que contiene abundantes células epiteliales y secreciones infectadas. Otras muestras del tracto respiratorio superior que también son adecuadas, pero en las que cabe esperar un menor rendimiento, son los exudados nasales o faríngeos, lavados nasales y gargarismos. Para obtener los exudados es recomendable que los hisopos utilizados sean de poliéster o cualquier otro material sintético, nunca de algodón, alginato de calcio o con vástagos de madera.
En el caso de que el punto de recogida de las muestras y el laboratorio encargado de su procesamiento no estén próximos, éstas se enviarán refrigeradas y en medio de transporte de virus. Los mejores rendimientos se obtienen si las muestras son procesadas 2–3h después de ser obtenidas para la detección de antígenos virales mediante ensayos de inmunofluorescencia y antes de 48h para aislamiento de virus en cultivo celular. Cuando las muestras no puedan ser procesadas en el periodo señalado, deberán conservarse congeladas a −80°C.
El elevado número de virus que pueden estar involucrados en las enfermedades respiratorias, y su enorme heterogeneidad, hace del diagnóstico etiológico una labor ardua y condiciona a la utilización de técnicas de amplio espectro, que abarquen la detección de un número considerable de virus. Los métodos de detección directos, que permiten el aislamiento del virus en cultivos celulares o determinan la presencia de alguno de sus componentes, ya sean antígenos o genes, son los más extendidos en los laboratorios de microbiología clínica. Los métodos indirectos, basados en la detección de anticuerpos específicos en suero, presentan menor utilidad y están siendo relegados como herramientas de estudio epidemiológico.
Aislamiento mediante cultivoDesde hace más de 50 años se ha utilizado el aislamiento viral en cultivos celulares para establecer un diagnóstico virológico. Tras la inoculación de las muestras en diferentes líneas celulares, se puede observar un efecto citopático que demuestre el crecimiento del virus en dichas células. Una vez que éste ha crecido, su identificación se realiza por diferentes métodos. En general, el aislamiento viral depende de diferentes factores, entre los que destacan la calidad de la muestra clínica y que ésta sea adecuada, la viabilidad de las líneas celulares elegidas y los reactivos requeridos en el proceso y la capacitación técnica del personal que realiza los diferentes procedimientos. Una vez aislados en células susceptibles, facilitan estudios posteriores, centrados en la caracterización de cepas circulantes, el descubrimiento de nuevos virus o serotipos no esperados y los estudios fenotípicos de resistencias a antivirales.
El aislamiento viral se ha considerado el estándar para la detección de virus y es el método de referencia. Sin embargo, desde hace más de una década los avances tecnológicos en el diagnóstico han permitido utilizar otras técnicas que incluyen los anticuerpos monoclonales o métodos moleculares, que han resultado ser herramientas poderosas a la hora de establecer un diagnóstico de la infección viral.
La ventaja fundamental del cultivo celular es la confirmación de la viabilidad y la infectividad del virus y la diferenciación entre virus capaces de infectar e incapaces de hacerlo. Esta información no es posible obtenerla con los métodos de amplificación molecular y los métodos antigénicos de detección viral. Además, se puede estudiar los virus aislados para conocer la sensibilidad a determinados antivirales y la realización de estudios fenotípicos14.
La selección, la recogida, el transporte y la preparación de las muestras clínicas son factores muy importantes que considerar en el aislamiento de los virus en cultivos celulares y, especialmente, en los respiratorios ya que son particularmente frágiles.
Factores que intervienen en el aislamiento viralAdemás de la carga viral de la muestra, otros factores son el tiempo transcurrido desde la toma de muestra hasta su procesamiento, la temperatura de conservación y el medio de transporte. El período de excreción viral de los virus respiratorios es, en general, muy corto, por lo que la toma de la muestra debe ser entre las 48 y las 72h del comienzo de los síntomas para evitar resultados falsos negativos. Si se sospecha infección hospitalaria, la muestra del paciente hospitalizado debe ser tomada durante las primeras 24h del ingreso. La temperatura de conservación incide en la calidad de la muestra para el aislamiento viral, ya que si la muestra es conservada y transportada a una temperatura mayor de 4°C, es posible obtener también falsos negativos. Cuando la temperatura disminuye a 0°C o incluso −20°C, la infectividad viral se pierde. Hay diferentes medios de transporte que pueden afectar a la integridad de la partícula viral por lo que pueden ser recomendados o no para realizar al aislamiento viral. El medio esencial mínimo (MEM) se considera la mejor alternativa como medio de transporte, al que se añaden estabilizadores proteínicos cuya función es la protección de la partícula viral; el suero bovino fetal, la albúmina bovina y la lactoalbúmina son los más comunes. Además, la adición de antibióticos es obligatoria, para prevenir contaminaciones bacterianas (penicilina y estreptomicina) y fungicidas, para prevenir la contaminación por hongos, el más comúnmente usado es anfotericina B. El uso de concentraciones altas de estas sustancias puede producir un efecto tóxico en el cultivo celular.
Principales líneas celulares para aislamiento de virus respiratoriosLas líneas celulares comúnmente empleadas para el aislamiento de virus respiratorios se detallan a continuación:
- •
NCI-H292: obtenida a partir de un carcinoma mucoepidermoide de pulmón. Puede sustituir a los cultivos primarios de riñón de mono para el aislamiento de los Paramyxovirus humanos. Además es sensible a adenovirus y Enterovirus. Crece en medio RPMI-1640 suplementado con 20μmol de glutamina, Hepes 25mmol y el 10% de suero bovino fetal. El medio de inoculación es RPMI-1640 con 20μmol de glutamina y 1,5μg/ml de tripsina y antibiótico (penicilina 100U/ml-estreptomicina 100mg/ml).
- •
HeLa: línea celular continua, obtenida de un carcinoma de cérvix humano. Es sensible a poliovirus tipo 1 y adenovirus tipo 3. Crece en MEM con aminoácidos no esenciales y glutamina suplementado con el 10% de suero bovino fetal. El medio tras la inoculación es MEM con el 2% de suero bovino fetal y antibiótico del 0,1% (penicilina 100U/ml-estreptomicina 100mg/ml).
- •
HEp-2: línea continua, derivada de un carcinoma epidermoide de laringe humano. Es sensible a poliovirus tipo 1, adenovirus tipo 3. Crece en Eagle MEM suplementado con el 10% de suero bovino fetal. El medio tras la inoculación es Eagle MEM con el 2% de suero bovino fetal y antibiótico del 0,1% (penicilina 100U/ml-estreptomicina 100mg/ml).
- •
Vero: obtenida en 1952 a partir de un riñón de un mono verde africano adulto normal. Es sensible a numerosos virus respiratorios. El medio de cultivo empleado es MEM con el 5% de suero bovino fetal y el medio tras la inoculación es MEM con el 2% de suero bovino fetal y antibiótico del 0,1% (penicilina 100U/ml-estreptomicina 100mg/ml).
- •
MDCK: línea continua obtenida a partir de riñón de Cocker Spaniel adulto aparentemente normal. Es sensible a los virus de la gripe tipos A y B, ya comentados, y a los adenovirus tipos 4 y 5, Coxsackievirus B5. El medio de cultivo empleado es Eagle MEM suplementado con glutamina 2,5g/l, Hepes 25 mmol y suero bovino fetal al 5%.
- •
MRC-5: obtenida de pulmón normal de un feto masculino de 14 semanas. Se ha descrito que es sensible a numerosos virus respiratorios. Se emplea como medio de cultivo, medio Eagle MEM con aminoácidos no esenciales y glutamina suplementado con el 10% de suero bovino fetal. El medio tras la inoculación es Eagle MEM con el 2% de suero bovino fetal y antibiótico del 0,1% (penicilina 100U/ml-estreptomicina 100mg/ml).
En la tabla 7 se exponen, para diferentes virus respiratorios, las líneas celulares y el tiempo necesario para la observación del efecto citopático en los cultivos convencionales.
Virus respiratorios, líneas celulares y tiempo de observación de efectos citopáticos
| Virus respiratorio | Tipo de línea celular | Observación de efecto citopático |
| VRS | Hep-2 Células primarias de riñón de mono. Fibroblastos humanos | 3–7 días |
| Metapneumovirus humano | Células terciarias de riñón de mono. LLC-MK2. VERO. Hep-2 | 3–7 días |
| Parainfluenza humano | LLC-MK2, VERO, NCI-H292 medio con tripsina PIV1 y 2 no para PIV3 | Más de 10 días |
| Adenovirus | Hep-2, HeLa | |
| Rinovirus | Fibroblastos, He-La |
La utilización generalizada de métodos de amplificación molecular en la detección de virus respiratorios ha permitido incrementar de manera considerable el número de muestras respiratorias en las que se detecta alguno de los virus respiratorios, y ha contribuido a mejorar el conocimiento de las enfermedades en que están implicados estos virus15,16. Este hecho es particularmente evidente en virus de difícil aislamiento como, por ejemplo, VPI-4, que ha pasado de ser considerado un patógeno poco frecuente y causante de infecciones respiratorias leves de vías superiores a tener una repercusión clínica al menos tan importante como la que tienen otros virus incluidos en el diagnóstico sistemático de la infección respiratoria de vías bajas.
Además de mejorar el rendimiento en el diagnóstico, la aplicación de técnicas moleculares ha permitido identificar nuevos virus relacionados con la afección respiratoria en seres humanos. En los últimos años se ha identificado el metapneumovirus humano, el bocavirus humano y tres nuevos coronavirus: CoV-SARS, CoV-NL63 y CoV-HKU117–19.
Por otro lado, la secuenciación de los productos obtenidos en la amplificación posibilita la realización de estudios adicionales de genotipificación, epidemiología molecular y sensibilidad a determinados antivirales. Sin embargo, la elevada sensibilidad de los ensayos de amplificación de ácidos nucleicos también ha aportado algunos inconvenientes, como la frecuente detección de virus en personas asintomáticas, así como la detección prolongada de virus en pacientes que ya están recuperados de una infección respiratoria pasada. Sin duda, el gran reto que plantean las infecciones virales respiratorias no es otro que poder determinar si un virus detectado en el tracto respiratorio es el causante de la afección respiratoria.
Un hallazgo particularmente común en las infecciones virales respiratorias es la relativa frecuencia con la que se observan las denominadas infecciones dobles, coinfecciones o infecciones por múltiples virus. En efecto, la detección de dos (o más) agentes virales en un mismo proceso respiratorio puede interpretarse como infección doble (múltiple) de las células del tracto respiratorio; sin embargo, también debería contemplarse la posibilidad de que sólo uno de los virus sea el verdadero causante del síndrome y el segundo (o el resto) refleje una colonización asintomática del tracto respiratorio. Por lo tanto, sería más preciso hablar de codetección o detección múltiple de virus en una misma muestra respiratoria. Las infecciones respiratorias atribuidas a más de un agente viral no es un fenómeno que haya surgido con la generalización de las técnicas moleculares en los laboratorios de microbiología diagnóstica. Se admite que, con independencia de la utilización de la PCR, el porcentaje de estas infecciones aumenta proporcionalmente con el número de métodos empleados para realizar el diagnóstico20. Una opción más que plausible para tratar de determinar la verdadera implicación de un virus en la afección respiratoria podría ser su cuantificación en las muestras respiratorias mediante técnicas de PCR en tiempo real. No obstante, debemos ser conscientes de los problemas que plantea este tipo de muestras, muy variables en lo que respecta a localización, cantidad de muestra obtenida y homogeneidad.
En la PCR en tiempo real, los procesos de amplificación y detección se producen de manera simultánea en el propio tubo de reacción. La detección de los productos amplificados se produce mediante detección de fluorescencia, que es proporcional a la cantidad de ADN que se vaya sintetizando. La fluorescencia en una reacción puede ser debida al empleo de agentes intercalantes, que aumentan notablemente la emisión de fluorescencia cuando se unen al ADN bicatenario, o bien sondas marcadas con fluorocromos. Además de la rapidez y la disminución del riesgo de contaminación por productos de amplificación, los sistemas de PCR en tiempo real permiten cuantificar, de manera sencilla, la concentración inicial de ADN o ARN presente en una muestra clínica. Para ello, sólo se necesita incluir unos controles externos con concentraciones conocidas y crecientes del ADN diana para generar una curva patrón.
Aunque la comercialización de sistemas de PCR en tiempo real para el diagnóstico de virus respiratorios se limita, en el mejor de los casos, como ya se ha expuesto, al virus de la gripe A, particularmente el subtipo H5N1, y CoV-SARS, comienzan a aparecer en el mercado kits para los demás virus respiratorios que, con respecto a los métodos «caseros», y de manera generalizada, representan una garantía en lo que concierne a correctas evaluación, estandarización y actualización de los oligonucleótidos y sondas utilizados. Estos kits suelen incorporar, además, un control interno de reacción para evitar los falsos negativos por sustancias inhibidoras de la ADN polimerasa en las muestras respiratorias21,22.
Una limitación importante de los métodos de PCR en tiempo real es la detección múltiple. En este sentido los biochips de ADN ofrecen unas posibilidades sin precedentes; sin embargo, la sensibilidad de estas técnicas aún no es comparable con los métodos tradicionales de amplificación múltiple. La aportación más novedosa al diagnóstico de infección respiratoria está llegando de la mano de los denominados chips de ADN en fase líquida. En estos sistemas la accesibilidad de las sondas nucleotídicas se ha mejorado de manera que se ha incrementado la sensibilidad de la técnica, como demuestran varios estudios publicados recientemente.