Sr. Editor: El aislamiento del agente causal a partir de los hemocultivos proporciona un diagnóstico de certeza en la mayoría de los casos de endocarditis infecciosa (EI). Sin embargo, la antibioterapia previa, la implicación de microorganismos que no crecen en los medios de cultivo convencionales o que tienen requerimientos nutricionales especiales y/o necesitan períodos prolongados de incubación, pueden dificultar e incluso imposibilitar la filiación microbiológica de la EI. La ausencia de diagnóstico etiológico dificulta el tratamiento dirigido y puede provocar retrasos terapéuticos de esta grave entidad clínica1. Por su parte, Streptococcus agalactiae es un agente etiológico infrecuente de EI, aunque de una marcada virulencia y que puede llevar a la rápida destrucción de las válvulas2,3. Presentamos un caso (caso 2, tabla 1 de la carta científica de Hurtado-Carrillo et al) de EI producido por S. agalactiae, que cursó con hemocultivos negativos y cuyo diagnóstico etiológico de certeza se pudo realizar mediante la amplificación y posterior secuenciación del gen que codifica el ARNr 16S bacteriano (ARN ribosómico 16S).
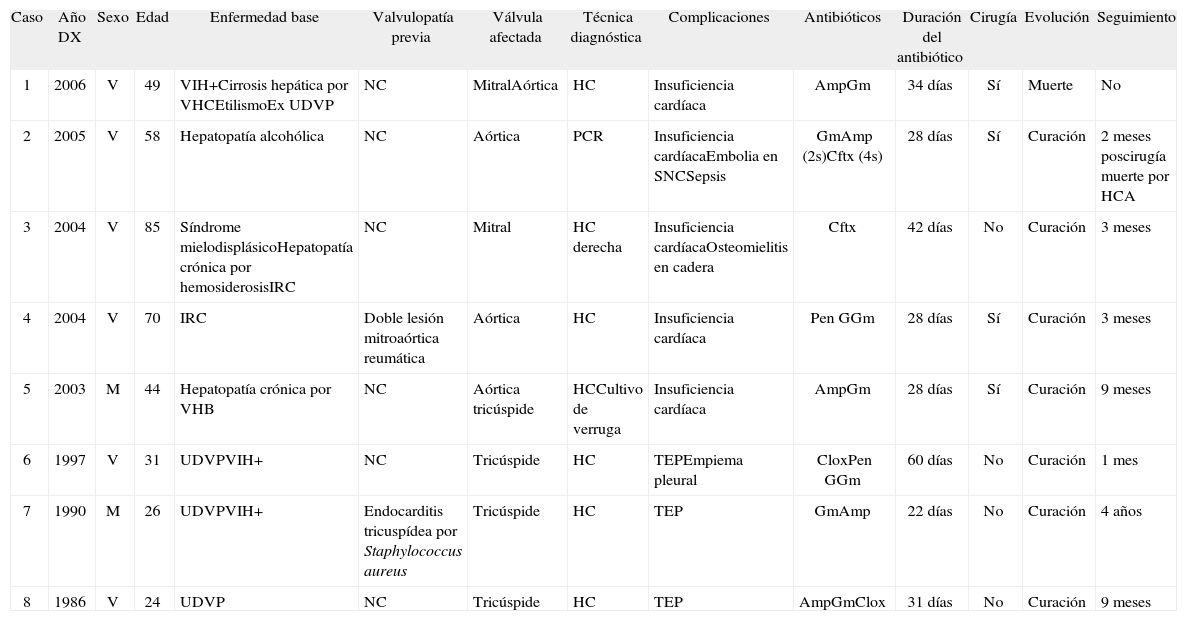
Características clínicas y demográficas de los 8 pacientes diagnosticados de endocarditis por Streptococcus agalactiae
| Caso | Año DX | Sexo | Edad | Enfermedad base | Valvulopatía previa | Válvula afectada | Técnica diagnóstica | Complicaciones | Antibióticos | Duración del antibiótico | Cirugía | Evolución | Seguimiento |
| 1 | 2006 | V | 49 | VIH+Cirrosis hepática por VHCEtilismoEx UDVP | NC | MitralAórtica | HC | Insuficiencia cardíaca | AmpGm | 34 días | Sí | Muerte | No |
| 2 | 2005 | V | 58 | Hepatopatía alcohólica | NC | Aórtica | PCR | Insuficiencia cardíacaEmbolia en SNCSepsis | GmAmp (2s)Cftx (4s) | 28 días | Sí | Curación | 2 meses poscirugía muerte por HCA |
| 3 | 2004 | V | 85 | Síndrome mielodisplásicoHepatopatía crónica por hemosiderosisIRC | NC | Mitral | HC derecha | Insuficiencia cardíacaOsteomielitis en cadera | Cftx | 42 días | No | Curación | 3 meses |
| 4 | 2004 | V | 70 | IRC | Doble lesión mitroaórtica reumática | Aórtica | HC | Insuficiencia cardíaca | Pen GGm | 28 días | Sí | Curación | 3 meses |
| 5 | 2003 | M | 44 | Hepatopatía crónica por VHB | NC | Aórtica tricúspide | HCCultivo de verruga | Insuficiencia cardíaca | AmpGm | 28 días | Sí | Curación | 9 meses |
| 6 | 1997 | V | 31 | UDVPVIH+ | NC | Tricúspide | HC | TEPEmpiema pleural | CloxPen GGm | 60 días | No | Curación | 1 mes |
| 7 | 1990 | M | 26 | UDVPVIH+ | Endocarditis tricuspídea por Staphylococcus aureus | Tricúspide | HC | TEP | GmAmp | 22 días | No | Curación | 4 años |
| 8 | 1986 | V | 24 | UDVP | NC | Tricúspide | HC | TEP | AmpGmClox | 31 días | No | Curación | 9 meses |
Amp: ampicilina; Cftx: ceftriaxona: Clox: cloxacilina: DX: diagnóstico; Gm: gentamicina; HC: hemocultivos; HCA: hemorragia cerebral aguda; IRC: insuficiencia renal crónica; NC: no conocida; PCR: reacción en cadena de la polimerasa; Pen G: penicilina G; SNC: sistema nervioso central; ST: estreptomicina; TEP: tromboembolismo pulmonar; UDVP: usuario de drogas por vía parenteral; Van: vancomicina; VHB: virus de la hepatitis B; VHC: virus hepatitis C; VIH: virus de la inmunodeficiencia humana.
El paciente era un varón de 58 años que ingresó en abril de 2005 con fiebre y disnea de mínimo esfuerzo. Había sido diagnosticado de hepatopatía crónica alcohólica, no se conocía que presentara valvulopatía previa y no se encontraba bajo tratamiento antibiótico. En la exploración presentaba signos compatibles con sepsis e insuficiencia cardíaca congestiva con soplo de insuficiencia aórtica IV/VI. Se inició antibioterapia empírica con ampicilina (2 g/4 h) y gentamicina (80 mg/8 h) y se realizó un ecocardiograma transesofágico que mostró una vegetación aórtica de 0,6 cm e insuficiencia aórtica grave. Los cuatro hemocultivos extraídos al ingreso fueron estériles. El paciente, que al sexto día presentó un cuadro compatible con embolia cerebral en el territorio de la arteria cerebral media izquierda, fue intervenido quirúrgicamente con sustitución valvular aórtica mediante prótesis biológica. El paciente completó 6 semanas de terapia antibiótica intravenosa con ceftriaxona (2 g/24 h). Fue dado de alta en situación clínica estable pero falleció 2 meses después por un cuadro de hemorragia cerebral no relacionada con la EI.
La válvula y la vegetación fueron enviadas al laboratorio de microbiología para su estudio microbiológico y los cultivos de ambas muestras resultaron negativos. Ante esta situación, se realizó la amplificación mediante reacción en cadena de la polimerasa (PCR) del ADN extraído del material valvular (DNeasy Blood & Tissue Kit, QIAGEN). Se amplificó el gen que codifica el ARNr 16S bacteriano (PCR universal) utilizando iniciadores previamente descritos4, y el producto obtenido (aproximadamente 1.500 pb) se envió para la secuenciación automática de ambas cadenas con los iniciadores empleados en la amplificación y dos iniciadores internos4. Las secuencias obtenidas se compararon con las existentes en la base de datos Gen- Bank NCBI (National Center for Biotechnology Information) y la alineación correspondió, con un 100% de identidad, con S. agalactiae.
La identificación bacteriana mediante la PCR universal a partir de muestras clínicas consiste en la amplificación del gen rrs (o los genes rrs, ya que la mayoría de las bacterias tienen varias copias de dicho gen) que codifica el ARNr 16S que forma parte de la subunidad menor (30 S) del ribosoma bacteriano5,6. El ADN que se utiliza como molde en la reacción de amplificación es extraído directamente de la muestra, utilizando, en la mayoría de los casos, equipos comerciales. En la PCR pueden utilizarse diferentes iniciadores (primers) que por tanto permitirán, según el caso, la amplificación de diferentes regiones del gen rrs y la obtención de segmentos génicos (amplicones) de diferente tamaño. Generalmente se utilizan iniciadores que permiten la amplificación de un fragmento de unos 500 pb del extremo 5′ del gen en el que se encuentra la mayor variabilidad y cuya secuencia aporta una adecuada información filogenética6. Una vez purificado el correspondiente amplicón, se determina su secuencia genética. La secuencia obtenida se compara, utilizando un programa informático, con aquellas depositadas en las bases de datos (bancos de secuencias) que existen en internet (EMBL, GenBank, RIDOM). Según el grado de homología entre la secuencia incógnita y aquella de la base de datos se puede establecer la identidad del microorganismo en estudio. En casos de EI, la identificación bacteriana empleando la PCR universal presenta una sensibilidad igual a la del hemocultivo en caso de microorganismos sin problemas de crecimiento y superior al cultivo valvular, particularmente si el paciente está recibiendo o ha sido sometido a tratamiento antibiótico5,7,8. De hecho, se ha propuesto que la PCR universal sea incluida como criterio mayor en los criterios diagnósticos de Duke para la EI9,10.
De acuerdo con los resultados obtenidos en este caso, S. agalactiae, un agente etiológico poco frecuente de EI, podría ser identificado mediante PCR universal en aquellos casos que cursan con cultivos convencionales negativos.