Introducción
La necesidad de diagnosticar y tratar adecuadamente las enfermedades infecciosas ha hecho imprescindible la presencia en los hospitales de los servicios de microbiología. Cada vez es más ineludible la necesidad de acreditar su competencia, lo que lleva consigo la validación y el control de todos los procesos diagnósticos allí desarrollados (preanalíticos, analíticos y postanalíticos), así como la cualificación del personal que forma parte de dichos servicios.
Una de las herramientas con que se cuenta en el laboratorio de microbiología para llevar a cabo estos objetivos es mediante el control de calidad interno y externo 1. El primero va destinado fundamentalmente a la detección de errores aleatorios o sistemáticos, basándose en la inclusión de muestras de resultado conocido en los ensayos realizados en el laboratorio. En el control de calidad externo las muestras o productos a caracterizar, medir o comparar proceden de otros centros o programas, y se usan para ejercicios de intercomparación entre los laboratorios, lo que permite detectar esencialmente errores aleatorios o evidenciar que determinados reactivos o equipos diagnósticos no son adecuados para el fin para el que han sido comercializados. Además, los programas de control externo brindan una excelente oportunidad para actividades de formación continuada del personal que faciliten establecer medidas correctoras por parte del propio laboratorio 2. Dadas las peculiaridades del área de conocimiento de la microbiología, son características deseables de un Programa de Control Externo, además de la independencia, la objetividad, la confidencialidad, la estabilidad del material de referencia, el procesamiento rápido e individualizado de los datos, la fiabilidad de los resultados de referencia, comunes a otras áreas analíticas, que sea de aplicación a todos los campos de la microbiología y que plantee problemas de auténtica relevancia clínica.
El Programa de Control Externo de la Calidad de la Sociedad Española de Enfermedades Infecciosas y Microbiología Clínica (SEIMC) comenzó hace más de 15 años. En líneas generales, está basado en el envío de una muestra o cepa problema a los participantes, junto con una historia clínica (caso clínico-microbiológico) compatible; se les solicita el procesamiento de la muestra (identificación, estudio de sensibilidad, detección de distintos marcadores serológicos/moleculares), así como la formulación de los comentarios o las recomendaciones de tipo técnico o clínico. Por tanto, como es habitual con los controles de calidad externos, el Programa SEIMC cubre esencialmente las etapas analíticas y, en menor medida, las postanalíticas. Cada participante procesa la muestra de acuerdo con sus medios y su procedimiento, y se remite el resultado al centro coordinador del programa, tal como se haría al médico peticionario. También tiene la posibilidad de remitir las determinaciones analíticas solicitadas, de forma parcial o completa, a un "laboratorio externo", definido como el laboratorio, público o privado, que utiliza el centro participante cuando no dispone de los medios necesarios para realizar las determinaciones solicitadas, lo que puede servirle para controlar la calidad de estos laboratorios de apoyo y referencia.
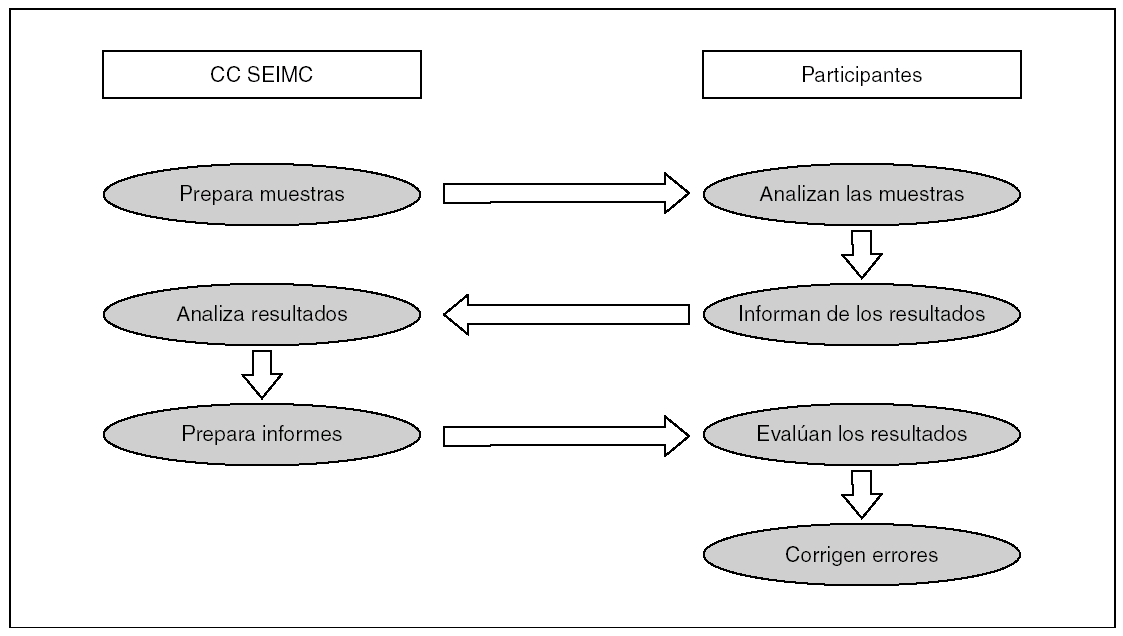
El Programa de Control Externo de Calidad SEIMC analiza los resultados enviados y emite un informe comparado de éstos a cada uno de los participantes (certificado de calidad), donde figuran los resultados emitidos por dicho laboratorio, los aportados por expertos en el tema (resultados de referencia) y los de la media o valor modal de todos los participantes en el Control SEIMC (fig. 1). Este informe también incluye recomendaciones de algunos expertos en lo referente a la selección apropiada de las pruebas de sensibilidad antibiótica. ç
Figura 1. Etapas del Control Externo de la Calidad.
En resumen, el Programa de Control Externo de Calidad SEIMC es una herramienta más al servicio de los laboratorios de microbiología clínica que permite asegurar que sus resultados son fiables y clínicamente útiles, buscando una mejora continua de la calidad que se apoya en 3 elementos: el informe comparado individual, el análisis general de los resultados aportados por los participantes que se envía tras cada control y las revisiones temáticas relacionadas con el objeto de los diversos controles 3. En este artículo se resumen los datos y las conclusiones más importantes extraídos de los diferentes análisis generales.
Análisis de datos de los controles de serología
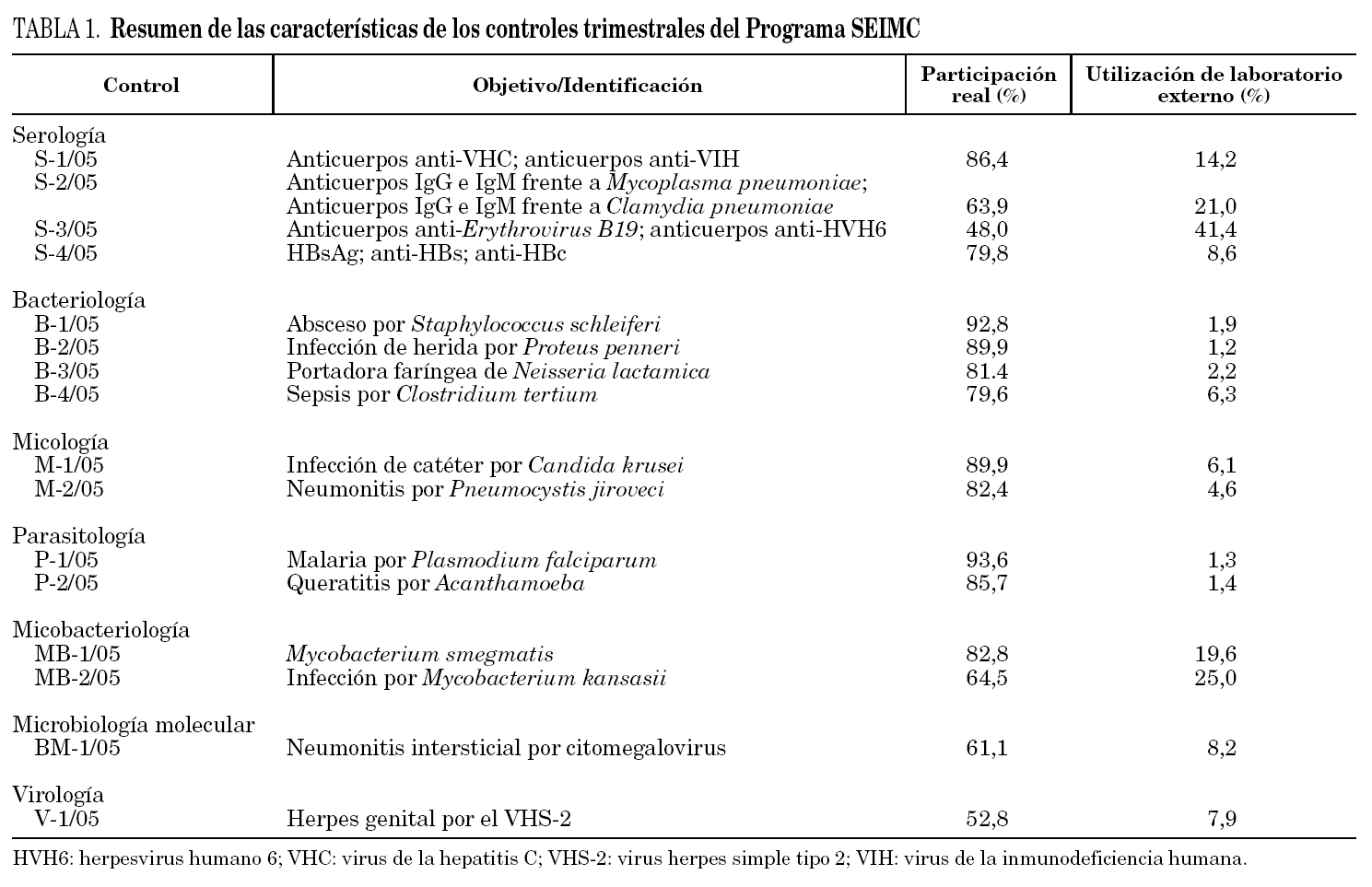
Durante el año 2005 se realizaron 4 envíos de serología (S-1/05, S-2/05, S-3/05 y S-4/05) a una media de 238 centros inscritos en este control. En todas las ocasiones se remitió, junto a la hoja de respuesta y la historia clínica, una muestra de suero liofilizado. Los resultados aportados por los expertos fueron los valores utilizados como patrón para el análisis comparativo de resultados y para la emisión de los certificados. Algunas de las características de dichos controles quedan resumidas en la tabla 1.
En el control S-1/05, que versaba sobre un cuadro viral con adenopatías y afectación hepática en una mujer dedicada a la prostitución, y con antecedentes de numerosas infecciones de transmisión sexual (ITS), el centro de referencia confirmó la presencia de anticuerpos frente al virus de la hepatitis C y frente al virus de la inmunodeficiencia humana. El porcentaje de participación real fue del 86,4% (el más elevado de todos los controles de serología del 2005). En cuanto a los resultados obtenidos en las determinaciones solicitadas, los participantes aportaron mayoritariamente datos concordantes con los del centro que actuó como referencia, y las discrepancias fueron casi anecdóticas. Además, este control puso de manifiesto también la amplia utilización que se hace de los métodos de enzimoinmunoensayo de micropartículas (MEIA) para llevar a cabo estas determinaciones, relacionado probablemente con la oferta y la estructura comercial existente en España.
En el control S-2/05 la muestra presentaba anticuerpos de tipo IgG e IgM frente a Mycoplasma pneumoniae y ambas inmunoglobulinas eran negativas frente a Chlamydophila pneumoniae. El caso clínico planteado revelaba un cuadro de neumonía atípica en un joven de 25 años, con patrón intersticial bilateral y exantema cutáneo. El porcentaje de participación real fue del 63,9% y el de uso de un laboratorio externo fue del 21%. En los resultados de la detección de IgG frente a C. pneumoniae llama la atención el considerable número de participantes que obtuvieron un resultado positivo discrepante con el resultado de referencia, y se observó que un número importante de éstos emplearon una técnica de enzimoinmunoensayo (EIA) de Vircell®, mientras que la mayoría de los que usaron una inmunofluorescencia (IF) obtuvieron un resultado negativo. En la detección de IgM frente a esta misma bacteria C. pneumoniae no se detectaron discrepancias reseñables. En la detección de IgG frente a M. pneumoniae, el método de detección más empleado fue el EIA, y obtuvo un 60,5% de resultados positivos concordantes con el de referencia, mientras que con la IF el porcentaje de resultados discrepantes fue del 69,2%. Por último, en la detección de IgM frente a M. pneumoniae, el EIA obtuvo un elevado índice de aciertos, mientras que la IF ofreció la mayoría de los resultados negativos discordantes. Mediante una técnica de aglutinación (FujireBio®) que detecta IgG e IgM se obtuvieron resultados positivos.
En el control S-3/05, una infección viral exantemática en una niña de 4 años vacunada frente a la rubéola y el sarampión, el suero presentaba anticuerpos de tipo IgG e IgM frente al Erythrovirus B19 y ambas inmunoglobulinas eran negativas para el virus herpes humano tipo 6 (HVH6). El porcentaje de participación real fue bajo (48,0%), y el de uso de un laboratorio de apoyo, elevado (41%), lo que se corresponde con la menor demanda de este tipo de determinaciones en los laboratorios de microbiología generales. En el análisis de resultados, el 96,1% de los participantes coincide en el resultado tomado como referencia en la detección de anticuerpos IgG frente al Erythrovirus B19. En la detección de IgM, el EIA aporta un 93,7% de resultados coincidentes mientras que la IF el 67,7%, siendo Biotrin® la marca comercial más usada en ambos casos. Así, con el EIA de esta marca se informaron el 10,3% de los resultados discrepantes y con la IF de la misma marca el 37,5%. En la detección de IgG frente al VHH-6, el 16,2% de los participantes informaron un resultado discordante con el del centro de referencia, en todos los casos mediante IF, que a su vez fue el método más usado por los participantes. En la detección de IgM frente al mismo virus todos los participantes aportaron un resultado negativo, coincidente con el de referencia.
En el control S-4/05 la muestra remitida, procedente de un paciente con el que una enfermera había tenido una punción accidental, presentaba un patrón serológico frente al virus de la hepatitis B (VHB) que, según el resultado obtenido en la detección del HBsAg (resultados discrepantes según método usado), podía interpretarse como una hepatitis B pasada y curada, o como una hepatitis B crónica con baja replicación. Dado que la mayoría de las técnicas usadas en serología para la determinación del HBsAg dieron un resultado claramente negativo y que, en contraposición, se detectaba ADN de VHB mediante reacción en cadena de la polimerasa (PCR) se realizó una secuenciación completa de la región codificante del HBsAg y se comprobó que la población mayoritaria del VHB era del genotipo A2 (o Ae), sin presencia de mutantes de escape; también se observó una deleción que afectaba a 20 aminoácidos (del 118 al 138) en la región preS, y que ésta producía una disminución de la expresión del HBsAg; ésta pudo ser la causa de que la mayoría de las técnicas serológicas empleadas por los participantes obtuvieran resultados negativos. En cuanto al análisis de resultados, la detección de HBsAg fue negativa en el 67,7% de las ocasiones y en el 29,8% resultó positiva/positiva débil (el 96,6% mediante MEIA del equipo Axsym®, Abbott). De los participantes que obtuvieron un resultado positivo/positivo débil mediante esta técnica, sólo el 33% intentó confirmarlo por distintos medios (repetición de la prueba, realizando otra técnica distinta de la usada inicialmente, mediante neutralización de antígeno o por PCR). La mayoría concluye que se trataba de un falso positivo, con la excepción de aquellos que realizaron PCR. En cuanto a la detección de anticuerpos anti-HBs, todos los participantes coincidieron con el resultado aportado por el laboratorio de referencia, y en la de anticuerpos anti-HBc el porcentaje de concordancia fue del 95,5%; en ambas ocasiones el resultado fue positivo. En general, se puede concluir que la mayoría de los centros participantes realizaron las pruebas fundamentales para este control (detección del HBsAg, anti-HBs y anti-HBc) y, aunque la muestra remitida era muy atípica, sería aconsejable identificar la frecuencia real de este tipo de deleciones del VHB que pueden escapar a los equipos de diagnóstico serológico comercializados.
En resumen, los menores porcentajes de participación se refieren a las serologías más específicas y que no suelen estar en la cartera de servicios de muchos de los centros, sobre todo de aquellos que se ubican en los hospitales comarcales y los porcentajes de menor utilización de un laboratorio externo se corresponden con las serologías de hepatitis y VIH (primer y último control), dato que se correlaciona con un mayor porcentaje real de participación en estos controles. En términos generales, salvo las excepciones ya comentadas, los laboratorios participantes demuestran una buena capacitación, competencia y fiabilidad.
Análisis de datos de los controles de bacteriología
Durante el año 2005, fueron 279 los laboratorios inscritos en el área de bacteriología. Las cepas remitidas fueron estudiadas por distintos expertos, que emitieron los siguientes resultados (tabla 1): Staphylococcus schleiferi (B-1/05), Proteus penneri (B-2/05); Neisseria lactamica (B-3/05) y Clostridium tertium (B-4/05).
El control B-1/05 versaba sobre un paciente neoplásico que desarrolló un absceso paravertebral y posterior bacteriemia. El porcentaje de participación real fue del 92,8%, siguiendo la línea de anteriores controles. Cerca de las 3 cuartas partes (73,9%) de los participantes llegaron a la identificación correcta de la especie (S. schleiferi) sin necesitar mayoritariamente de laboratorios externos, porcentaje superior al de otros controles en los que se remitieron otras especies de estafilococos coagulasa negativos. En el estudio de sensibilidad, cabe destacar que la cepa era sensible a todos los antibióticos ensayados, interpretándose los resultados según los criterios del Clinical Laboratory Standards Institute (CLSI) de 2005 para el género Staphylococcus. En general, el número de antibióticos informados por los participantes se ajustó bastante a las necesidades terapéuticas y al "patrón ideal" que se desprende de la opinión de los expertos. La cepa objeto del control fue considerada, por la gran mayoría de los participantes, como "sensible" a todos los antibióticos estudiados, y existió una gran uniformidad en la interpretación de resultados. Los informes de "resistencia" o "intermedio" se correspondieron con las desviaciones propias de todos los controles.
En el segundo control del año (P. penneri B-2/05) se remitió una cepa aislada en un caso de infección de herida traumática en una paciente diabética. El porcentaje de participación fue del 89,9%, similar al del control anterior, casi sin necesidad de un laboratorio de apoyo externo para completar su estudio. Del total de centros que remitieron hoja de respuesta analizable, la mayoría identificó correctamente el género (95,6%), aunque sólo se aceptó como respuesta válida la identificación correcta de género y especie (P. penneri), por lo que fueron correctas el 84% de las respuestas. Por lo que respecta al estudio de sensibilidad, destaca la uniformidad de los resultados y la notable coincidencia con los resultados de referencia. Las mayores discrepancias las encontramos a la hora de interpretar la sensibilidad a algunos antibióticos β -lactámicos, debido a que la cepa presentaba una β -lactamasa cromosómica de la clase A (HugA), intrínseca de P. penneri que, en condiciones normales de expresión, hace que la cepa sea sensible a la cefotaxima, como así determinó la mayoría de participantes y el laboratorio de referencia. Sin embargo, puede seleccionarse con facilidad mutantes desreprimidas de HugA que hiperexpresan dicha enzima y, en consecuencia, conducen a valores de CMI que deben ser consideradas como "no sensibles", por lo que se decidió considerar válidos los resultados de aquellos participantes que informaron la cepa como "resistente" o "intermedia" a la cefotaxima, manteniendo como resultado de referencia la "sensibilidad", igualmente válido.
El control B-3/05 (N. lactamica) se refería a una cepa aislada en la faringe de una mujer que estaba al cuidado de un niño que había sufrido una meningitis bacteriana aguda. La participación fue del 81,4%, inferior a la de controles anteriores, lo que puede traducir un mayor nivel de dificultad. Así, aunque la mayoría de los participantes identificó correctamente el género (97,2%), hubo un porcentaje considerable que identificó la cepa como Neisseria meningitidis (14,7%), e incluso llegaron a tipificarla dentro de los serogrupos A y B. Puesto que el objetivo fue la identificación de especie, sólo fue correcto el 81,2% de las respuestas. El análisis de los resultados del estudio de sensibilidad muestra una gran concordancia y uniformidad con los resultados de referencia, lo que era de esperar dado el perfil de sensibilidad (sensible a todos los antibióticos probados) de la cepa objeto de este control. En resumen, este control resalta la importancia, ya conocida, de llevar a cabo los procesos analíticos microbiológicos en función de los datos clínicos existentes, y cuando se analizan las muestras al margen de este contexto puede haber errores importantes, de ahí la necesidad de una buena capacitación de los profesionales.
Finalmente, el último control del año (C. tertium B-4/05) hacía referencia a un cuadro séptico de origen hepático en una paciente neoplásica y con neutropenia. El porcentaje de participación fue del 79,6%, claramente inferior a los primeros 2 controles. También la necesidad de utilización de un laboratorio externo fue mayor, del 6,3%. Un porcentaje considerable de participantes (74,8%) identificó correctamente el género, pero sólo se aceptó como respuesta válida la identificación exacta de género y especie (C. tertium), por lo que fueron correctas tan sólo el 61,7% de las respuestas. Este porcentaje de acierto es inferior al de otros controles, posiblemente por el mayor nivel de dificultad de este control. Por lo que respecta al estudio de sensibilidad, se observó una notable coincidencia con los resultados de referencia con respecto a los siguientes antibióticos: amoxicilina-clavulanato, cefoxitina, imipenem y vancomicina. No ocurrió lo mismo en la interpretación de la sensibilidad frente a clindamicina, metronidazol y penicilina, en donde se observó cierta diversidad de resultados.
Análisis de datos de los controles de micología
Durante el año 2005, se realizaron dos envíos a los 238 centros inscritos. En el primero de ellos (M-1/05), se remitió una cepa de Candida krusei, con una participación real del 89,9%. El caso clínico hacía referencia a una paciente de Oncología que sufrió un episodio de fungemia asociada a un catéter para la administración de quimioterapia, que fue tratada con una combinación de antibióticos y fluconazol. El porcentaje de identificaciones correctas fue elevado (92,1%), para lo que se utilizaron habitualmente pruebas bioquímicas incorporadas en sistemas comerciales (el más frecuente, API® bioMérieux, 57,7%). Sin embargo, el objetivo principal fue determinar la sensibilidad de la cepa a los azoles, para los que esta levadura presenta una sensibilidad disminuida y, en el caso del fluconazol, resistencia intrínseca. El 64,5% de los participantes realizó pruebas de sensibilidad, por lo general mediante la determinación de la CMI por microdilución (Sensititre®). La CMI obtenida por el centro de referencia en el caso de fluconazol fue de 32 μg/ml que, según los puntos de corte CLSI, debería interpretarse como sensible dependiente de la dosis (SDD), pero que, dada la resistencia intrínseca de esta levadura, la interpretación final fue "resistente". Aunque el intervalo de CMI aportado por los participantes fue amplio (entre 0,12 y 128 μg/ml), la mayoría de ellos realizó una correcta interpretación basándose en la identificación de especie, y el porcentaje de resultados discrepantes con la interpretación del laboratorio de referencia fue del 23,5%. Estos resultados difieren de otros del año 1998, en que se envió la misma cepa y en esta ocasión hubo un 47,7% de interpretaciones discrepantes. Es de suponer que esta mejoría obedece tanto a la mayor disponibilidad de medios como a los esfuerzos de formación continuada de los participantes.
El segundo envío (M-2/05) se basaba en un caso de neumonitis en un paciente con sida que no realizaba correctamente el tratamiento antirretroviral ni la profilaxis para las infecciones oportunistas. Se remitieron 2 extensiones fijadas de una impronta pulmonar para la detección de Pneumocystis jiroveci. La participación fue, en este caso, del 82,4%, ligeramente inferior a la del control anterior, y también fue mayor la necesidad de apoyo de un laboratorio externo. El porcentaje de acierto en la identificación fue del 88,8%, algo inferior al control anterior, debido, entre otras causas, a que algunos de los participantes realizaron una inmunofluorescencia directa de la muestra en vez de las tinciones clásicas, que eran las que el programa de control solicitó para detectar Pneumocystis en esta muestra (posibles falsos negativos por tratarse de una muestra de pulmón de animal de experimentación). Los métodos más empleados fueron la tinción de Giemsa y las tinciones argénticas, con buena correlación de resultados.
Análisis de datos de los controles de parasitología
Durante el año 2005 se realizaron dos envíos a los 251 laboratorios inscritos en esta área. En el primero de ellos (P-1/05), se remitió a los participantes una extensión de sangre teñida con Giemsa que correspondía a una misionera que debutó, a los siete días tras su regreso de África, con un cuadro febril compatible con malaria. El centro de referencia informó una parasitación por Plasmodium falciparum mayor al 50% con presencia de trofozoítos jóvenes, algunos de ellos binucleados, y hematíes multiparasitados; en este envío, el porcentaje de participación real fue del 93,6%, bastante superior al de otros controles. Se obtuvo un elevado porcentaje de identificaciones correctas (97,0%), lo que era esperable por la baja dificultad, ya que el objetivo perseguido en este control fue, esencialmente, de tipo formativo (presencia de abundantes estructuras parasitarias).
En el segundo control (P-2/05) se remitió una alícuota de medio de conservación para amebas en donde el laboratorio de referencia observó la presencia de quistes y trofozoítos del género Acanthamoeba. La muestra procedía de un joven, portador de lentes de contacto blandas, que desarrolló una queratitis epitelial puntiforme en ambos ojos. Se alcanzó una participación real del 85,7%, ligeramente inferior a la del control anterior, y fueron muy pocos los que requirieron el apoyo de un laboratorio externo (tabla 1). El porcentaje de identificaciones correctas fue incluso superior al del anterior control (99,1%), lo que habla a favor de la pericia y experiencia del microbiólogo que realiza la observación. En ambos controles de parasitología la opción básica diagnóstica fue la observación microscópica, aunque uno de los participantes empleó una técnica sofisticada de PCR para amplificar genoma de P. falciparum en el material del frotis teñido por Giemsa.
En general, podemos concluir que los participantes presentan una buena capacitación en la identificación parasitológica, situación que está avalada por la escasa utilización de un laboratorio externo y por el alto porcentaje de diagnósticos correctos.
Análisis de datos de los controles de micobacterias
Durante el año 2005 fueron 93 los laboratorios adscritos al área de micobacteriología. Se realizaron dos envíos en el primer y el segundo trimestres del año. En el primero de ellos (MB-1/05), se remitió a los participantes una cepa aislada en el líquido peritoneal de un paciente, de significado clínico dudoso, y que había sido identificada por el centro de referencia como Mycobacterium smegmatis. La participación fue del 82,8%, razonablemente elevada, aunque en torno a una quinta parte de los participantes requirió la ayuda de un laboratorio externo. El porcentaje de acierto en la identificación fue aceptable si consideramos que el 58,4% de los participantes identificó correctamente género y especie (M. smegmatis), que fue la única respuesta considerada válida por parte del Programa de Control de Calidad, teniendo en cuenta el nivel de dificultad del presente control para diferenciar esta especie de otras micobacterias de crecimiento rápido más habituales en la práctica diaria. Cabe destacar que una proporción considerable de laboratorios (15,6%) identificó la cepa como Mycobacterium fortuitum. La mayor parte de ellos emplearon para la identificación pruebas bioquímicas convencionales, que les pudieron conducir a error, dada la gran similitud entre las dos especies. Finalmente, en relación con el estudio de sensibilidad, el porcentaje de centros que lo informan es del 51,9%, existiendo cierta variabilidad en cuanto a los antituberculosos informados, lo que parece lógico pues no existe un antibiograma estandarizado.
En el control MB-2/05, el centro que actuó como referencia identificó la cepa como Mycobacterium kansasii subespecie VI. Había sido aislada de un paciente con una infección pulmonar de patrón nodular. El porcentaje de participación fue del 64,5%, claramente inferior a los porcentajes de participación de anteriores controles y un 25,0% empleó un laboratorio externo para poder realizar/completar el estudio solicitado. Como en el anterior control, se alcanzó un porcentaje de aciertos aceptable, si tenemos en cuenta que el 69,9% de los participantes realizó una correcta identificación de especie (M. kansasii), que se consideró suficiente por parte del Programa de Control de Calidad SEIMC, aunque la identificación óptima del genotipo sólo fue alcanzada por el 18,3% de los centros. Parece existir una correlación entre el tipo de método empleado y el grado de identificación de la cepa, de modo que los centros que llegan al diagnóstico más preciso (especie, genotipo), emplean técnicas de biología molecular (PCR-RFLP, hibridación, secuenciación), muchas veces combinados con pruebas bioquímicas convencionales. En este caso, el porcentaje de centros que realizaron estudio de sensibilidad fue tan sólo del 36,7%, siendo un porcentaje mucho más bajo que el conseguido en otros controles en que se enviaron otras micobacterias de crecimiento lento; también en este control existe cierta variabilidad en cuanto a los antituberculosos informados, aunque en todos los casos tan sólo se consideraron aquellos antibióticos para los que existían criterios de interpretación según el CLSI.
Análisis de datos del control de microbiología molecular
En el año 2005 se realizó un único envío de microbiología molecular (BM-1/05) a los participantes. Se les remitió una muestra de plasma liofilizado, procedente de un caso de neumonitis intersticial en un trasplantado de precursores hematopoyéticos, para la detección de citomegalovirus (CMV) mediante PCR y que el centro de referencia había informado positiva mediante PCR real time (no se solicitó detección cuantitativa puesto que la muestra había sido liofilizada). En total se enviaron 72 muestras, aportando hoja de respuesta con resultados valorables (porcentaje real de participación) el 61,1%, porcentaje similar al de controles de otros años. El porcentaje que declaró utilizar un laboratorio externo fue del 8,2%. En todas las ocasiones la detección resultó positiva, por lo que la concordancia con el centro de referencia fue total. La técnica de PCR-EIA de Herplex® (Genomica) fue la más usada (el 19,1% de los centros), siguiéndole en frecuencia la PCR cuantitativa de Cobas-Amplicor® Roche (17,0%). Un porcentaje notable (17,0%) utilizó métodos de desarrollo propio. En resumen, aunque el número de laboratorios capacitados para llevar a cabo esta técnica no es muy grande, todos ellos mostraron un adecuado nivel de competencia.
Análisis de datos del control de virología
En 2005 se realizó un único envío a los participantes (V-1/05), consistente en un portaobjetos preparado para la detección de virus herpes simple (VHS-2) mediante inmunofluorescencia (IF), aunque algunos de los participantes, al carecer de esta opción diagnóstica y a pesar de no ser una muestra adecuada, realizaron una PCR del material obtenido tras el raspado de los pocillos. En total se enviaron 72 portaobjetos, aportando hoja de respuesta con resultados valorables el 52,8%, porcentaje este último muy inferior al de los controles de virología de otros años, posiblemente debido a que muchos de los laboratorios realizan otras técnicas diferentes de la IF para la detección de VHS. La mayor parte de los que respondieron llevó a cabo la detección sin necesidad de recurrir a un laboratorio externo.
El caso clínico que acompañaba a la muestra correspondía al de un herpes genital en una paciente joven, con antecedentes de otras ITS. Tan sólo en una ocasión se detectaron resultados discrepantes con los aportados por el centro de referencia (un falso negativo), lo que supone que el 97,4% informó la detección de VHS como positiva, siendo este porcentaje de concordancia con el laboratorio de referencia muy alto. Las marcas de los anticuerpos que se usaron principalmente para realizar la IF fueron MicroTrack® y Dako®. En resumen, el nivel de competencia mostrado ha sido bueno, si bien el número de laboratorios con capacidad para diagnóstico virológico no es muy alto.
Análisis de datos de los controles de bacteriología mensual
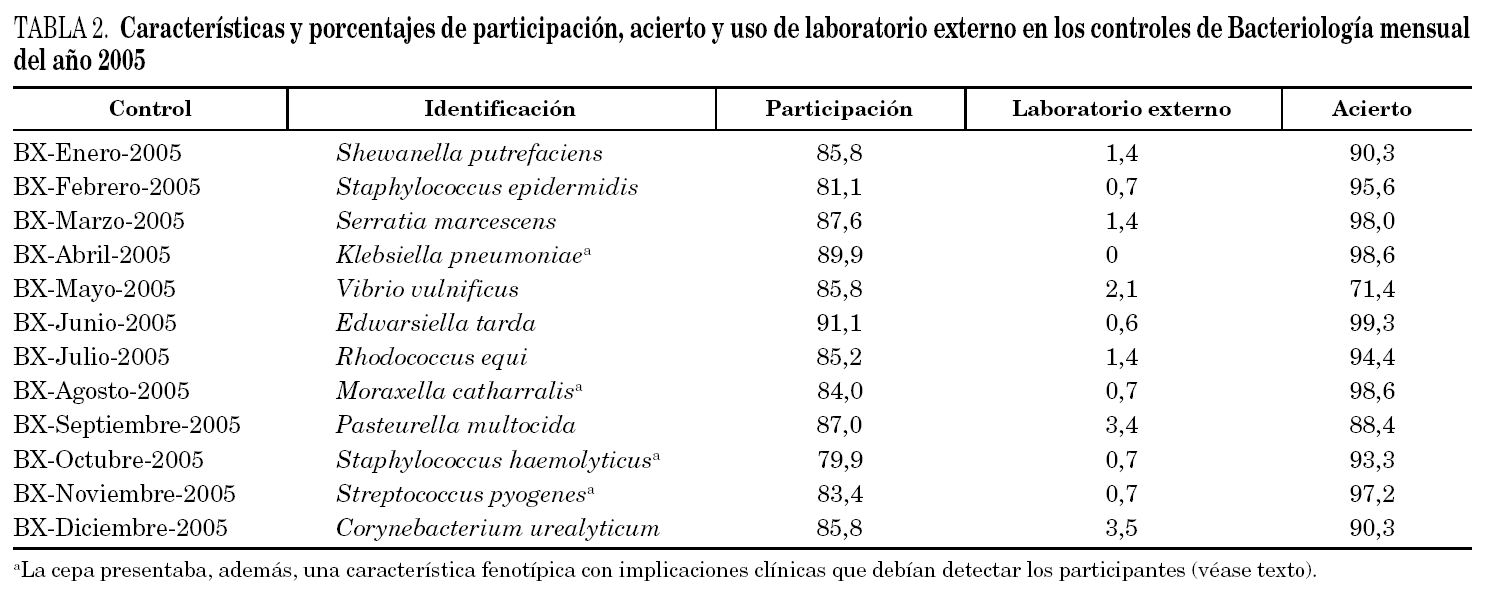
Durante el año 2005, se enviaron 12 controles mensuales de bacteriología, con un rango de participación que osciló entre el 79,9% y el 91,1%. El porcentaje de participantes que emplearon el laboratorio externo fue, en términos generales, bajo, entre un 0 (Klebsiella pneumoniae) y un 3,5% (Corynebacterium urealyticum); las variaciones fueron reflejo de la dificultad intrínseca de cada control. Los porcentajes de identificaciones correctas conseguidos por los participantes fueron elevados, alcanzándose un máximo en el control de junio, en que se remitió una cepa de Edwarsiella tarda. El porcentaje más bajo se correspondió con el control de Vibrio vulnificus. Todos estos datos se exponen en la tabla 2.
Cabe comentar que, en cuatro ocasiones, la cepa presentaba una característica especial, lo que constituía el verdadero objetivo perseguido. En el control BX-Abril-05 (Klebsiella pneumoniae), un 89,4% de los participantes informaron correctamente dicha característica especial, lo que indica que se trataba de una cepa productora de una β -lactamasa de espectro extendido (BLEE). En el control BX-Agosto-2005 (Moraxella catharralis), sólo un 53,5% de los centros informó que se trataba de una M. catharralis productora de b -lactamasa. En los restantes controles, los porcentajes de participantes que indicaron la característica especial todavía fueron menores, de modo que en el control BX-Octubre-2005 (Staphylococcus haemolyticus), un 48,1% de los centros indicaron que se trataba de un S. haemolyticus sensible a la vancomicina y resistente/intermedio a la teicoplanina. Por último, en el control BX-Noviembre-2005 (Streptococcus pyogenes) sólo un 36,5% de los participantes informó la cepa enviada como S. pyogenes con fenotipo M. En resumen, mientras que la capacidad para llevar a cabo la identificación correcta de las cepas es elevada, un porcentaje no despreciable de participantes no detecta la presencia de una característica fenotípica con implicaciones desde el punto de vista clínico, o no lo declara a pesar de que se le solicita.
Correspondencia: Dr. J.L. Pérez.
Servicio de Microbiología. Hospital Son Dureta.
Andrea Doria, 55. 07014 Palma de Mallorca. España.
Correo electrónico: jlperez@hsd.es