Pese a que la tuberculosis (TB) viene acompañando a la humanidad desde el neolítico, ésta se mantiene como un serio problema de salud pública, agravado en los últimos años por el fenómeno de la resistencia a los antibióticos.
La OMS establece que el diagnóstico de la tuberculosis drogorresistente (TB-DR) es fundamental para su control y al mismo tiempo considera que los procedimientos diagnósticos tradicionales han llegado a un límite resolutivo. Ante este crítico escenario, el entendimiento de los mecanismos moleculares que explican el fenómeno de la TB-DR, combinado con novedosas técnicas moleculares, están permitiendo desarrollar de toda una nueva generación de procedimientos diagnósticos de TB-DR.
Sin embargo, ¿cuáles son estos mecanismos genéticos generadores de TB-DR? y ¿cuáles son las características, ventajas y limitaciones de estas nuevas metodologías diagnósticas? son las preguntas que se pretenden responder con el presente trabajo.
Drug resistant tuberculosis: Molecular mechanisms and diagnostic methods.
Despite the fact that Tuberculosis (TB) has been found in humans since the Neolithic Age, it still remains serious public health problem, increased in the last few years due to the phenomenon of drug resistance (DR).
The World Health Organization (WHO) established that the diagnosis of tuberculosis drug resistance (DR) is essential for its control, and at the same time considers that the traditional diagnostic procedures have reached their limit. In view of this critical scenario, the understanding of the molecular mechanisms that explain the phenomenon of the TB-DR, in combination with novel techniques in molecular biology, are allowing a new generation of diagnostic procedures to be developed for TB-DR.
However, this work sets out to answer the questions of what these molecular mechanisms TB-DR are,as well as their characteristics, advantages and limitations of these new diagnostic methodologies.
La TB es una enfermedad remergente causada por bacterias del género Mycobacterium. De acuerdo a la OMS en 2007, se reportaron 9,27 millones de casos nuevos y 1,78 millones de defunciones. Se estiman 13,7 millones de casos prevalentes1,2.
Uno de los factores que dificultan el manejo y control clínico de la TB a nivel mundial es la drogoresistencia, la OMS estima que el 20% de los casos de TB son resistentes a un antibiótico (TB-DR) y el 5,3% son resistentes a isoniacida y rifampicina (TB-MDR)3.
De manera general, los bacilos de Mycobacterium desarrollan mutaciones espontáneas en genes específicos. Estos mutantes son seleccionados tras la exposición a un tratamiento farmacológico mal aplicado, pasan a constituir la población microbiana predominante y son causa de fracaso clínico4,5. Por sus implicaciones terapéuticas y pronósticas, resultan especialmente relevantes las cepas TB multidrogorresistente y las denominadas TB extensamente resistentes, es decir, con resistencia a isoniacida, rifampicina, alguna fluoroquinolona y un antibiótico inyectable de segunda línea1,3.
Tradicionalmente, el diagnóstico de la TB-DR requiere de 4–9 semanas para ser confirmatorio, lo cual permite el contagio de los contactos del paciente con TB-DR y favorece su dispersión5–7.
En función de lo anterior, los últimos años han evidenciado la aparición nuevos métodos diagnósticos fundamentados en el cultivo de Mycobacterium en medio líquido, con una marcada reducción de los tiempos diagnósticos, alta especificidad y economía. Por otra parte, la identificación de los mecanismos moleculares y las mutaciones en genes asociados al fenómeno de resistencia, así como el empleo de nuevas técnicas en biología molecular, han permitido desarrollar novedosos métodos diagnósticos de TB-DR, específicos y rápidos. Sin embargo, poseen limitaciones que requieren de un adecuado análisis, si es que se desean incorporar en los programas de lucha contra TB.
El objetivo de este trabajo es hacer una descripción de los mecanismos moleculares responsables de resistencia a fármacos de primera línea en Mycobacterium tuberculosis y de los métodos diagnósticos desarrollados para su detección.
Mecanismos moleculares generadores de drogorresistencia a fármacos de primera líneaLa «isoniacida» es un potente fármaco antituberculoso y bactericida que se utiliza contra la TB desde 19525, actúa específicamente contra bacterias que se encuentran en fase de replicación activa8. Su mecanismo de acción aún no está del todo aclarado, pero se sugiere que la isoniacida se transforma en su principio activo gracias a la enzima catalasaperoxidasa, inhibiendo la síntesis de ácido micólico de la pared bacteriana, lo cual permite que el microorganismo sea susceptible a la acción de radicales de oxígeno reactivo y a otros elementos externos de respuesta del huésped5,8 (tabla 1).
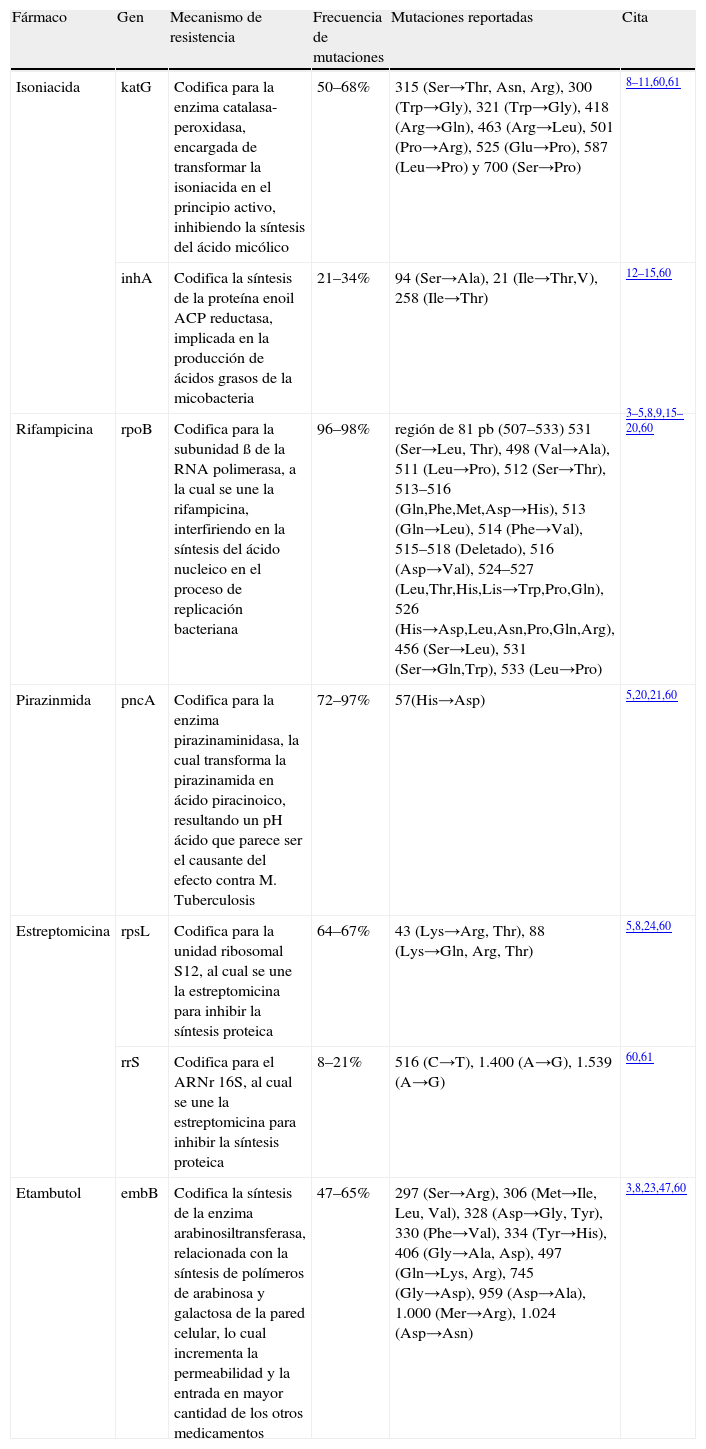
Genes asociados a TB DR
| Fármaco | Gen | Mecanismo de resistencia | Frecuencia de mutaciones | Mutaciones reportadas | Cita |
| Isoniacida | katG | Codifica para la enzima catalasa-peroxidasa, encargada de transformar la isoniacida en el principio activo, inhibiendo la síntesis del ácido micólico | 50–68% | 315 (Ser→Thr, Asn, Arg), 300 (Trp→Gly), 321 (Trp→Gly), 418 (Arg→Gln), 463 (Arg→Leu), 501 (Pro→Arg), 525 (Glu→Pro), 587 (Leu→Pro) y 700 (Ser→Pro) | 8–11,60,61 |
| inhA | Codifica la síntesis de la proteína enoil ACP reductasa, implicada en la producción de ácidos grasos de la micobacteria | 21–34% | 94 (Ser→Ala), 21 (Ile→Thr,V), 258 (Ile→Thr) | 12–15,60 | |
| Rifampicina | rpoB | Codifica para la subunidad ß de la RNA polimerasa, a la cual se une la rifampicina, interfiriendo en la síntesis del ácido nucleico en el proceso de replicación bacteriana | 96–98% | región de 81pb (507–533) 531 (Ser→Leu, Thr), 498 (Val→Ala), 511 (Leu→Pro), 512 (Ser→Thr), 513–516 (Gln,Phe,Met,Asp→His), 513 (Gln→Leu), 514 (Phe→Val), 515–518 (Deletado), 516 (Asp→Val), 524–527 (Leu,Thr,His,Lis→Trp,Pro,Gln), 526 (His→Asp,Leu,Asn,Pro,Gln,Arg), 456 (Ser→Leu), 531 (Ser→Gln,Trp), 533 (Leu→Pro) | 3–5,8,9,15–20,60 |
| Pirazinmida | pncA | Codifica para la enzima pirazinaminidasa, la cual transforma la pirazinamida en ácido piracinoico, resultando un pH ácido que parece ser el causante del efecto contra M. Tuberculosis | 72–97% | 57(His→Asp) | 5,20,21,60 |
| Estreptomicina | rpsL | Codifica para la unidad ribosomal S12, al cual se une la estreptomicina para inhibir la síntesis proteica | 64–67% | 43 (Lys→Arg, Thr), 88 (Lys→Gln, Arg, Thr) | 5,8,24,60 |
| rrS | Codifica para el ARNr 16S, al cual se une la estreptomicina para inhibir la síntesis proteica | 8–21% | 516 (C→T), 1.400 (A→G), 1.539 (A→G) | 60,61 | |
| Etambutol | embB | Codifica la síntesis de la enzima arabinosiltransferasa, relacionada con la síntesis de polímeros de arabinosa y galactosa de la pared celular, lo cual incrementa la permeabilidad y la entrada en mayor cantidad de los otros medicamentos | 47–65% | 297 (Ser→Arg), 306 (Met→Ile, Leu, Val), 328 (Asp→Gly, Tyr), 330 (Phe→Val), 334 (Tyr→His), 406 (Gly→Ala, Asp), 497 (Gln→Lys, Arg), 745 (Gly→Asp), 959 (Asp→Ala), 1.000 (Mer→Arg), 1.024 (Asp→Asn) | 3,8,23,47,60 |
ACP: proteína transportadora del acilo.
El gen katG codifica para la enzima catalasa-peroxidasa. La presencia de mutaciones o deleciones en este gen se ha relacionado con el 60% de la cepas de M. tuberculosis resistentes a isoniacida5,9. El katG tiene un tamaño de 1.771 pares de bases (pb); no obstante, del 30–65% de las mutaciones se localizan en el codón 315 Ser8, el cual cambia a Thr, Asn o Arg10; otras mutaciones de menor frecuencia se han reportado en los codones 300, 321, 418, 463, 501, 525, 587 y 70011,12 (tabla 1).
La 2.a causa de resistencia a isoniacida se explica por mutaciones que afectan al gen inhA y con mayor frecuencia a su regulador13,14, el cual codifica para la proteína inhA, responsable de la producción de ácidos grasos5,9; recientemente, se ha propuesto a este gen como el responsable de la corresistencia a isoniacida y etionamida8. Las mutaciones en los genes katG e inhA están asociadas al 70–80% de los aislados resistentes a isoniacida8; pero alrededor del 15–25% de cepas de M. tuberculosis resistentes a este fármaco poseen el genotipo silvestre tanto en el gen katG como inhA5, por lo cual se piensa debe existir otro mecanismo de resistencia.
En este sentido, se ha observado que un 15% de cepas resistentes a isoniacida presentan mutaciones en el locus kasA15, lo cual implica que otro posible objetivo de isoniacida es la proteína kasA, involucrada en el mecanismo de elongación de los ácidos grasos5. También se han reportado con menor frecuencia mutaciones involucradas en la adquisición de resistencia a isoniacida en el gen oxyR-ahpC4,8,9.
La «rifampicina» es otro fármaco importante, que debido a su fuerte actividad bactericida ha sido empleado desde 19705,8; desafortunadamente, su mala administración ha generado un incremento considerable en cepas resistentes8.
Respecto a su mecanismo de acción, la rifampicina se une a la ARN polimerasa de la micobacteria e interfiere durante el proceso de replicación y síntesis de ácido nucleico5,8,16. La ARN polimerasa es un complejo oligomérico compuesto por cuatro subunidades: α, ß, ß’ y σ; codificadas por los genes rpoA, rpoB, rpoC y rpoD, respectivamente. Se ha demostrado que mutaciones en rpoB producen cambios conformacionales en la subunidad ß de la ARN polimerasa, disminuyendo la afinidad por rifampicina y otorgando resistencia al fármaco8, (tabla 1).
Pese a que el gen rpoB tiene un tamaño de 3.534pb, el 96 o 97% de las mutaciones que causan resistencia a rifampicina están localizadas en una región de 81pb4,8,10,15,16 entre los codones 507–5339,16 y consisten por lo general en mutaciones puntuales no sinónimas17. De acuerdo con los resultados de diversos estudios, en el 40–70% de los aislados, se observaron mutaciones puntuales en el codón 531 Ser5,8,16 por Leu (TCG→ TTG) o por Thr (AGC→ACA)17. Del 32–36% de las cepas estudiadas mostraron cambios en el codón 526 His; y del 7–9% en el codón 516 Asp16,17. También se han reportado mutaciones o deleciones de menor frecuencia en otros codones como: 498, 511–518, 524–527, 456, 531, 53318 (tabla 1). En los últimos años se han observado algunos aislados resistentes cuya mutación no se localiza en la región de 81pb19, por lo cual se propone la existencia de mecanismos adicionales generadores de resistencia5,8.
Las cepas resistentes a rifampicina presentan resistencia cruzada a drogas químicamente relacionadas o con sitios de acción similares dentro de la célula, como rifapentina y parcialmente a rifabutina y rifalacina20.
Finalmente, el diagnóstico de resistencia a rifampicina es especialmente importante debido a su fuerte asociación con la resistencia a isoniacida, considerándose como un marcador importante de TB-MDR5.
El empleo de la «pirazinamida» se inició en el año de 1952, funciona específicamente contra bacilos semilatentes que no son afectados por ningún otro medicamento antituberculoso. Una de sus principales ventajas es que disminuye el tiempo de tratamiento, debido a su sinergia con isoniacida y rifampicina; sin embargo, su principal desventaja radica en su alta especificidad en contra de M. tuberculosis, de manera que si la cepa infectante es diferente, como pudiera ser Mycobacterium bovis, el tratamiento no es efectivo5,8.
Respecto a su mecanismo de acción, la pirazinamida es transformada por la enzima pirazinamidasa a su principio activo, ácido pirazinoico, el cual genera un pH ácido intrabacteriano, al parecer causante del efecto contra M. tuberculosis5,21 (tabla 1). La pirazinamidasa es codificada por el gen pncA, mutaciones en este gen explican el 80% de las cepas resistentes a este fármaco5. Para el caso particular de M. bovis en el codón 57 del gen pncA se ha identificado una mutación puntual (C→G), que resulta en la sustitución de His por Asp, suficiente para inactivar la enzima pirazinamidasa8,21 (tabla 1).
La «estreptomicina» se descubrió en 1943, fue el primer fármaco con actividad antituberculosa probada, actúa específicamente en la forma extracelular de la micobacteria5. El mecanismo de acción de este fármaco se basa en su unión al ARN ribosomal (ARNr) con lo cual inhibe la síntesis proteica de la bacteria. La resistencia a este fármaco se asocia con mutaciones en los genes rpsL y rrs (o rrnS), los cuales codifican respectivamente la síntesis de la subunidad proteica 12S y el ARNr 16S8,9. EL gen rpsL presenta mutaciones con mayor frecuencia, encontradas en los codones 43 (Lys→Arg y/o Thr) y 88 (Lys→Gln, Arg y/o Thr)9 (tabla 1). Al igual que ocurre con otros fármacos, en estreptomicina, se cree que existen mecanismos adicionales de resistencia, en este caso probablemente relacionados con la permeabilidad de la pared y membrana bacteriana, ya que el 30% de las cepas resistentes no muestran mutaciones en los genes rrs o rpsL5.
El «etambutol» es otro fármaco empleado en contra de TB, se utilizó por 1.a vez en 1961, tiene actividad bacteriostática, es un buen antimicobacteriano y solo actúa contra bacterias en fase de multiplicación activa5. Se recomienda para tratar infecciones diseminadas con bacterias pertenecientes al complejo Mycobacterium avium, especialmente en personas infectadas con VIH, que cursan con DM, o con antecedentes de abandono o recaída8,22. La probabilidad de resistencia es más baja que con otras drogas, por lo cual se incluye en la pauta básica de tratamiento primario en los países con una tasa elevada de resistencia primaria a otro fármaco de primera línea22.
El mecanismo de acción de este medicamento no ha sido claramente definido, se cree que está relacionado con la interferencia en el metabolismo del ARNr, la síntesis de fosfolípidos, la síntesis de ácido micólico y la síntesis de polisacáridos de la pared celular bacteriana5. Evidencias experimentales y clínicas indican que el etambutol ejerce un efecto sinérgico con otros fármacos antituberculosos como consecuencia del incremento en la permeabilidad bacteriana, permitiendo el ingreso en mayor cantidad de otros medicamentos8 (tabla 1).
La resistencia a etambutol se encuentra asociada a mutaciones en tres genes: embA, embB, y embC localizados en un locus de 10.000pb (embABC), que codifican para la enzima arabinosiltransferasa, relacionada con la síntesis de polímeros de arabinosa y galactosa de la pared celular5,8,23. Cerca del 70% de las cepas resistentes a etambutol presentan una mutación puntual en el codón 306 Met del gen embB, causando la sustitución por Val, Leu o Ile4,23. Otras mutaciones reportadas se encuentran en los codones 297, 306, 328, 330, 334, 406, 497, 745, 959, 1.000 y 1.02424 (tabla 1).
Métodos diagnósticos de TB-DRDada la importancia de actuar de forma rápida y efectiva ante la sospecha de estar frente a una cepa de TB-DR y gracias al conocimiento de los mecanismos genéticos generadores de resistencias, así como al desarrollo de nuevas técnicas en biología molecular, ha sido posible desarrollar en los últimos años una nueva serie de técnicas enfocadas al diagnóstico de drogorresistencia a fármacos antituberculosos, buscando aquellas con alta sensibilidad y especificidad, que al mismo tiempo sean rápidas y de bajo costo (tablas 2A,B). En términos generales, se ha establecido una clasificación de estos métodos, en fenotípicos, considerando el cultivo microbiológico de la muestra a diagnosticar, y genotípicos, basados en el empleo de ADN de micobacterias provenientes de la muestra.
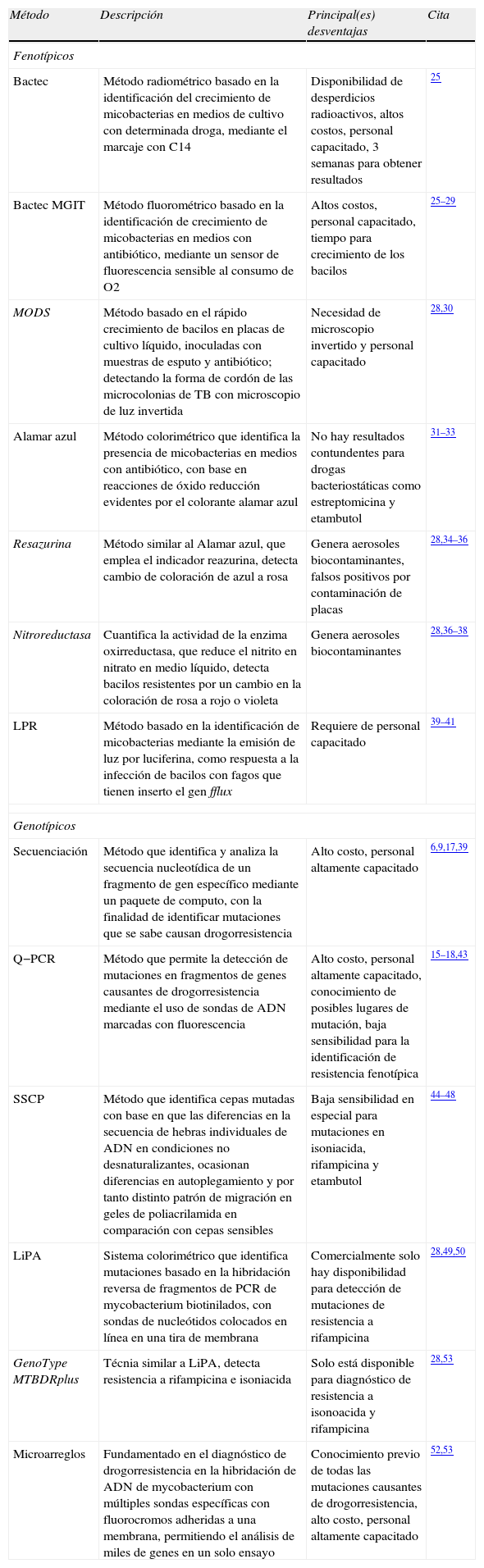
Métodos de diagnóstico de TB-DR
| Método | Descripción | Principal(es) desventajas | Cita |
| Fenotípicos | |||
| Bactec | Método radiométrico basado en la identificación del crecimiento de micobacterias en medios de cultivo con determinada droga, mediante el marcaje con C14 | Disponibilidad de desperdicios radioactivos, altos costos, personal capacitado, 3 semanas para obtener resultados | 25 |
| Bactec MGIT | Método fluorométrico basado en la identificación de crecimiento de micobacterias en medios con antibiótico, mediante un sensor de fluorescencia sensible al consumo de O2 | Altos costos, personal capacitado, tiempo para crecimiento de los bacilos | 25–29 |
| MODS | Método basado en el rápido crecimiento de bacilos en placas de cultivo líquido, inoculadas con muestras de esputo y antibiótico; detectando la forma de cordón de las microcolonias de TB con microscopio de luz invertida | Necesidad de microscopio invertido y personal capacitado | 28,30 |
| Alamar azul | Método colorimétrico que identifica la presencia de micobacterias en medios con antibiótico, con base en reacciones de óxido reducción evidentes por el colorante alamar azul | No hay resultados contundentes para drogas bacteriostáticas como estreptomicina y etambutol | 31–33 |
| Resazurina | Método similar al Alamar azul, que emplea el indicador reazurina, detecta cambio de coloración de azul a rosa | Genera aerosoles biocontaminantes, falsos positivos por contaminación de placas | 28,34–36 |
| Nitroreductasa | Cuantifica la actividad de la enzima oxirreductasa, que reduce el nitrito en nitrato en medio líquido, detecta bacilos resistentes por un cambio en la coloración de rosa a rojo o violeta | Genera aerosoles biocontaminantes | 28,36–38 |
| LPR | Método basado en la identificación de micobacterias mediante la emisión de luz por luciferina, como respuesta a la infección de bacilos con fagos que tienen inserto el gen fflux | Requiere de personal capacitado | 39–41 |
| Genotípicos | |||
| Secuenciación | Método que identifica y analiza la secuencia nucleotídica de un fragmento de gen específico mediante un paquete de computo, con la finalidad de identificar mutaciones que se sabe causan drogorresistencia | Alto costo, personal altamente capacitado | 6,9,17,39 |
| Q−PCR | Método que permite la detección de mutaciones en fragmentos de genes causantes de drogorresistencia mediante el uso de sondas de ADN marcadas con fluorescencia | Alto costo, personal altamente capacitado, conocimiento de posibles lugares de mutación, baja sensibilidad para la identificación de resistencia fenotípica | 15–18,43 |
| SSCP | Método que identifica cepas mutadas con base en que las diferencias en la secuencia de hebras individuales de ADN en condiciones no desnaturalizantes, ocasionan diferencias en autoplegamiento y por tanto distinto patrón de migración en geles de poliacrilamida en comparación con cepas sensibles | Baja sensibilidad en especial para mutaciones en isoniacida, rifampicina y etambutol | 44–48 |
| LiPA | Sistema colorimétrico que identifica mutaciones basado en la hibridación reversa de fragmentos de PCR de mycobacterium biotinilados, con sondas de nucleótidos colocados en línea en una tira de membrana | Comercialmente solo hay disponibilidad para detección de mutaciones de resistencia a rifampicina | 28,49,50 |
| GenoType MTBDRplus | Técnia similar a LiPA, detecta resistencia a rifampicina e isoniacida | Solo está disponible para diagnóstico de resistencia a isonoacida y rifampicina | 28,53 |
| Microarreglos | Fundamentado en el diagnóstico de drogorresistencia en la hibridación de ADN de mycobacterium con múltiples sondas específicas con fluorocromos adheridas a una membrana, permitiendo el análisis de miles de genes en un solo ensayo | Conocimiento previo de todas las mutaciones causantes de drogorresistencia, alto costo, personal altamente capacitado | 52,53 |
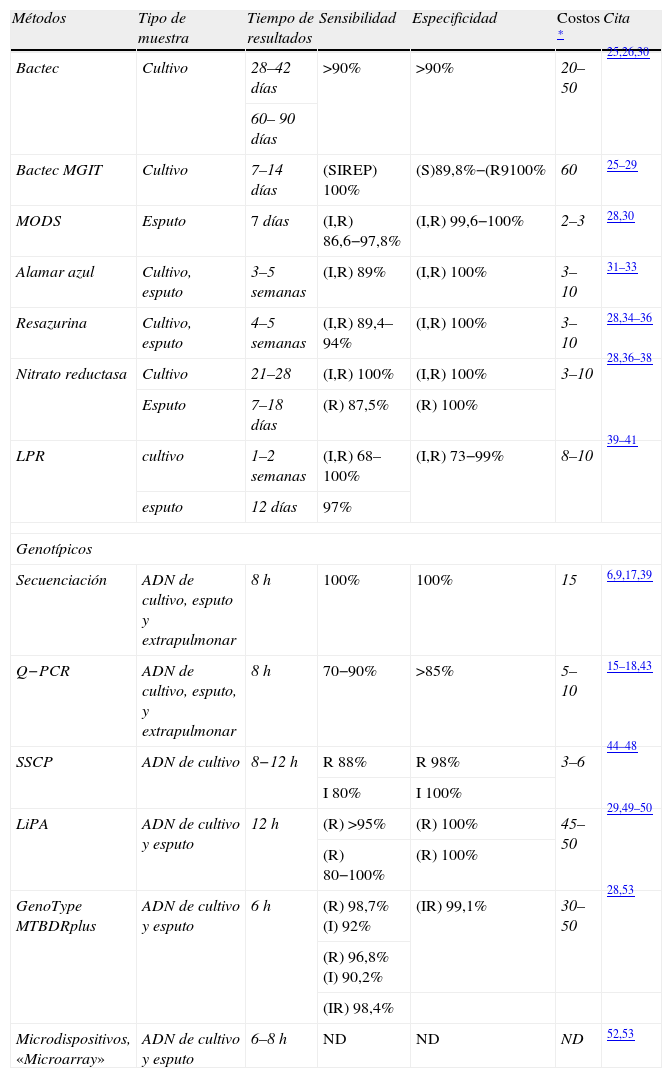
Métodos de diagnóstico de TB-DR
| Métodos | Tipo de muestra | Tiempo de resultados | Sensibilidad | Especificidad | Costos * | Cita |
| Bactec | Cultivo | 28–42días | >90% | >90% | 20–50 | 25,26,30 |
| 60– 90días | ||||||
| Bactec MGIT | Cultivo | 7–14días | (SIREP) 100% | (S)89,8%−(R9100% | 60 | 25–29 |
| MODS | Esputo | 7días | (I,R) 86,6−97,8% | (I,R) 99,6−100% | 2–3 | 28,30 |
| Alamar azul | Cultivo, esputo | 3–5 semanas | (I,R) 89% | (I,R) 100% | 3–10 | 31–33 |
| Resazurina | Cultivo, esputo | 4–5 semanas | (I,R) 89,4–94% | (I,R) 100% | 3–10 | 28,34–36 |
| Nitrato reductasa | Cultivo | 21–28 | (I,R) 100% | (I,R) 100% | 3–10 | 28,36–38 |
| Esputo | 7–18días | (R) 87,5% | (R) 100% | |||
| LPR | cultivo | 1–2 semanas | (I,R) 68–100% | (I,R) 73−99% | 8–10 | 39–41 |
| esputo | 12días | 97% | ||||
| Genotípicos | ||||||
| Secuenciación | ADN de cultivo, esputo y extrapulmonar | 8h | 100% | 100% | 15 | 6,9,17,39 |
| Q−PCR | ADN de cultivo, esputo, y extrapulmonar | 8h | 70−90% | >85% | 5–10 | 15–18,43 |
| SSCP | ADN de cultivo | 8−12h | R 88% | R 98% | 3–6 | 44–48 |
| I 80% | I 100% | |||||
| LiPA | ADN de cultivo y esputo | 12h | (R) >95% | (R) 100% | 45–50 | 29,49–50 |
| (R) 80−100% | (R) 100% | |||||
| GenoType MTBDRplus | ADN de cultivo y esputo | 6h | (R) 98,7% (I) 92% | (IR) 99,1% | 30–50 | 28,53 |
| (R) 96,8% (I) 90,2% | ||||||
| (IR) 98,4% | ||||||
| Microdispositivos, «Microarray» | ADN de cultivo y esputo | 6–8h | ND | ND | ND | 52,53 |
E: etambutol; I: isoniacida; LiPA: ensayo de prueba en línea; LPR: Fagos reporteros de luciferasa; MODS: microscopic observation drug susceptibility assay; P: pirazinamida; Q−PCR: reacción en cadena de la polimerasa en tiempo real; R: rifampicina; S: estreptomicina; SSCP: polimorfismos conformacionales de hebra sencilla.
Este método descrito por Canneti y Groset, mide la proporción de colonias que han crecido en medios con concentraciones críticas de antibióticos, en relación con el crecimiento observado en un medio sin antibiótico. Dicho método es la base del sistema automatizado BACTEC, el cual consiste en el sembrado de los bacilos previamente cultivados en un medio líquido Middlebrook, rico en ácido palmítico con carbono 14 (C14). El C14 es un isótopo radioactivo natural que emite radiaciones beta, de manera que cuando las micobacterias metabolizan el ácido palmítico liberan al medio ambiente dicho isótopo en forma de CO2, marcado con C14. Este CO2 es aspirado y llevado a una cámara de ionización, en donde se transforma en una corriente eléctrica que se cuantifica y es proporcional a la cantidad de bacilos que se encuentran en crecimiento, es decir la señal eléctrica se expresa como «índice de crecimiento»25. En cuanto al diagnóstico de resistencia, se emplean frascos con medio de cultivo, en los cuales se inocula la cepa a estudiar y se agrega el fármaco antituberculoso a evaluar, de modo que la emisión de radioactividad en un medio de cultivo con determinada droga, significa que el bacilo se está multiplicando y por tanto es resistente25. Este es un sistema completamente automatizado y presenta sensibilidad y especificidad superiores al 90%. Entre sus inconvenientes se encuentran el generar desperdicios radioactivos, la importante inversión inicial en la compra de equipo, reactivos y materiales, y la necesidad de personal altamente capacitado. Quizá su principal desventaja es el tiempo requerido para confirmar una negatividad, pues incluye por lo menos de 3–4 semanas para el cultivo y una semana más para obtener resultados de farmacorresistencia, contribuyendo a la transmisión de la TB-MDR y poniendo en entredicho su pertinencia futura en programas de salud específicos contra TB-DR y MDR5–6,25.
Bactec MGIT 960El método del tubo indicador de crecimiento micobacteriano o Bactec MGIT 960® (Becton Dickinson Diagnostic Instrument System, Inc). Es un sistema para crecimiento y detección de Mycobacterium completamente automatizado; funciona mediante un sensor especial de fluorescencia sensible al consumo de O2, que permite monitorear el crecimiento microbiano, presenta una sensibilidad del 100% y una especificidad del 89,8%, para estreptomicina. Al compararlo con el sistema BACTEC tiene la ventaja de detectar el crecimiento micobacteriano en forma más rápida26–28 y evita la producción de desechos radioactivos; no obstante, mantiene varias de sus desventajas como requerir personal capacitado y altos costos de inversión en equipo, materiales y reactivos26,29.
Microscopic Observation Drug Susceptibility assay (MODS)Este método detecta la resistencia de M. tuberculosis a isoniacida y rifampicina, directamente de las muestras de esputo. Tomando como base que el crecimiento del bacilo es más rápido en medio líquido que en medio sólido, esta técnica consiste en examinar mediante un microscopio de luz invertida, placas de cultivo con medio Middlebrook 7H9 y el antibiótico a evaluar, inoculadas con muestras de esputo; detectando en un promedio de 7días la forma de cordón de las microcolonias características de TB28,30.
Estos últimos 2 factores, además de su bajo costo, son sus principales ventajas, ya que reduce considerablemente el tiempo de diagnóstico y posee una mayor sensibilidad y especificidad que el medio sólido, al evidenciar el crecimiento característico de M. tuberculosis28,30 (tablas 2A,B).
Métodos de óxido reducción: alamar azul, resazurina y actividad nitroreductasaEl alamar azul es un método colorimétrico utilizado desde 1995 para medir cuantitativamente la susceptibilidad de M. tuberculosis a agentes antimicrobianos. Se basa en la utilización del colorante alamar azul como indicador de óxido reducción; cuando hay presencia de células viables, dicha reacción se lleva a cabo y el medio de cultivo cambia de una coloración azul a rosa31,32.
Estudios realizados con este método concluyen que, mediante el empleo de determinados puntos de corte, es posible generar una buena detección de cepas sensibles y resistentes a isoniacida y rifampicina (con una sensibilidad de 89% y especificidad del 100%) (tabla 2B); sin embargo, aún no hay resultados concluyentes para los casos particulares de estreptomicina y etambutol, ya que estas drogas son bacteriostáticas y no bactericidas. Quizá la principal ventaja de esta técnica es su bajo costo y la fácil disponibilidad de los reactivos, por lo que bien podría apoyar programas de control de TB-DR en países con escasos recursos económicos32,33 (tabla 2A).
El ensayo de micro valoración con resazurina (o REMA por sus siglas en inglés), es un método colorimétrico que permite, a partir de bacilos aislados de esputo, determinar drogorresistencia en un periodo de una semana. Consiste en la incorporación del indicador resazurina a medio de cultivo líquido con diferentes concentraciones de antibiótico, si el bacilo se mantiene vivo, se detecta un cambio de color azul a rosa, como resultado de la reducción del indicador. Es una técnica barata, sencilla y con buenos resultados de sensibilidad y especificidad; entre sus inconvenientes está la producción de aerosoles y la posibilidad de transferir bacilos de un pozo a otro de la microplaca34–36 (tablas 2A,B).
Otra técnica similar es nitroreductasa o «prueba de Griess». Se fundamentada en la actividad de la enzima nitroreductasa, que le confiere a Mycobacterium la capacidad para reducir el nitrito en nitrato al emplear NaNO3 en el medio de cultivo, detectando la resistencia mediante un cambio de color, que puede ir del rosa al violeta en función de la cantidad de bacilos. Ofrece resultados de 10–14 días a partir de un cultivo positivo o de muestras de esputo con baciloscopia positiva. Entre sus ventajas se encuentran que es de bajo costo, requiere equipamiento microbiológico básico y posee buenos niveles de sensibilidad y especificidad28,37,38, (tablas 2A,B).
MicobacteriófagosEl uso de micobacteriófagos para el diagnóstico de TB-DR ofrece resultados fenotípicos en poco tiempo y a bajo costo. Las 2 técnicas de mayor aceptación basadas en micobacteriófagos son la phage-amplified biological assay (phaB, ‘amplificación biológica de fagos’) y la luciferase reporter phages (LRP, ‘identificación de fagos reporteros de luciferasa’)39.
Amplificación biológica de fagosEsta técnica se basa en la amplificación de fagos en micobacterias sospechosas previamente tratadas con antibiótico, de manera que después del proceso fagoinfeccioso, los fagos extracelulares son retirados del medio, mientras los fagos que lograron ingresar a las bacterias que sobrevivieron aún con los fármacos, se replican; así nuevas partículas de fago serán liberadas dentro del medio y se podrá observar y cuantificar las placas de lisis en las células infectadas39. De esta manera las placas aparecerán sólo en las células de micobacterias resistentes.
Con esta base, los laboratorios Biotec (Biotec Laboratories, Ltd. Reino Unido) desarrollaron recientemente un sistema comercial para la detección de M. tuberculosis con resistencia a rifampicina llamado prueba rápida de placa de TB-Rif (FASTplaqueTB-Rif assay), con la ventaja de requerir 48h para proporcionar resultados confirmatorios, pero con el inconveniente de necesitar de un cultivo previo de la bacteria39.
Fagos reporteros de luciferasaEsta técnica permite determinar la susceptibilidad a fármacos antituberculosos mediante fagos reporteros de luciferasa. Se fundamenta en la infección de micobacterias con fagos que contienen inserto en su genoma el gen reportero fflux, el cual codifica para la luciferasa de luciérnaga. Estos fagos son capaces de replicar y expresar el gen fflux solo en células viables de Mycobacterium, de forma que la proteína luciferasa, en presencia de ATP, cataliza una reacción que libera O2 y luciferina, emitiendo florescencia39–41.
En lo que se refiere a muestras clínicas con sospecha de contener TB-DR, estas se cocultivan con el panel de antibióticos y finalmente se infectan con LRP, de modo que solo aquellas cepas resistentes al fármaco sobrevivirán y serán detectadas fácil y rápidamente por su emisión de fluorescencia mediante un luminómetro o una película radiográfica. La primera opción ofrece una alta sensibilidad y resultados cuantitativos en aproximadamente 54h (con una sensibilidad de 86–100% y especificidad del 73–99%, para rifampicina); mientras que la película radiográfica tiene similares valores de eficacia, pero su principal atributo es la disminución en costos; no obstante, requiere mayor tiempo (94h) para proporcionar resultados confirmatorios39–41.
Esta técnica también se ha aplicado sobre muestras de esputo, donde aporta resultados en aproximadamente 12 días, y su utilización resulta atractiva en los países en vías de desarrollo con alta prevalencia de TB-DR41.
Métodos diagnósticos genotípicos o molecularesLa aplicación de métodos de diagnóstico genotípicos ha sido posible, entre otras cosas, gracias a la secuenciación del genoma de M. tuberculosis, y a la identificación y caracterización de genes asociados a drogoresistencia. Estos métodos ofrecen la posibilidad de identificar mutaciones asociadas a TB-DR de forma precisa y generar resultados a partir de 100 bacilos o menos por muestra. Quizá lo más importante es que permiten realizar el diagnóstico en cuestión de horas42, ya que al utilizar la muestra clínica directamente como material biológico, se elimina el tiempo relacionado con el crecimiento del bacilo.
En todas las técnicas moleculares el primer paso es la extracción del material genético y la amplificación, mediante reacción en cadena de la polimerasa o PCR (de acuerdo a sus siglas en inglés), de la región que contiene la(s) mutación(es) responsable(s) de la resistencia, para posteriormente identificarse mediante el empleo de alguno de los siguientes métodos:
Secuenciación genómicaEsta técnica es la más exacta y está considerada como el estándar molecular para definir genotípicamente a una cepa drogorresistente6,9,39. Permite la ubicación concreta de la(s) mutación(es) y consiste en identificar y analizar la secuencia nucleotídica de un fragmento de ADN específico6,9,39. Solo se requiere obtener el ADN de la cepa a estudiar, amplificar por PCR la región del gen de interés y este producto es analizado por un secuenciador automático, el cual finalmente presenta la sucesión de ácidos nucleicos6,39.
Para el caso del diagnóstico de resistencia a fármacos en micobacterias, es posible secuenciar el gen involucrado en dicho mecanismo, e identificar la mutación o mutaciones específicas mediante comparación con el gen de una cepa sensible6,39. Sin embargo, las limitaciones de esta técnica son los altos costos del equipo, los materiales y reactivos que se requieren, y la necesidad de contar con personal altamente capacitado. Pese a estas desventajas, varios estudios demuestran que la secuenciación es considerada la técnica de elección para la detección genotípica de resistencias en cepas de M. tuberculosis17.
Sistemas de detección por fluorescencia mediante PCR en tiempo realLa técnica de PCR en tiempo real permite la detección de mutaciones con ayuda de sondas de ADN marcadas con diferentes tipos de fluorescencia. Para ello, se amplifica la región en donde se ubica la mutación causante de resistencia al fármaco y se analiza la fluorescencia del producto generado15–18,43. De esta forma se han identificado mutaciones en los genes rpoB, katG, inhA y embB (tabla 2A–2B).
Este sistema detecta el ADN de micobacterias provenientes de esputo, lavado bronco alveolar, fluido cerebro espinal, fluido pleural o muestras de tejido, lo que amplía su utilidad como técnica diagnóstica. Además, pueden emplearse en un mismo ensayo diversas sondas marcadas con diferentes fluorógenos, evidenciando, distintas mutaciones en varias muestras clínicas, inclusive si difieren por un solo nucleótido (single nucleotide polimorphism [SNP, ‘polimorfismo mononucleótido’]), proporcionándole una versatilidad y rapidez importante15–18,43,44.
Para este procedimiento se han desarrollado diversos tipos de sondas, con diferentes características y niveles de eficiencia. Destacan las sondas «Molecular Beacons»y las sondas «FRET», ambas permiten detectar SNP de manera eficiente; sin embargo, comparten el inconveniente de ser costosas, requerir equipo sofisticado y personal altamente especializado. Las ventajas de esta técnica radican en el corto tiempo para obtener resultados y los valores de especificidad superior al 85%, con sensibilidad entre 70–90% en su aplicación al diagnóstico de resistencia a rifampicina e isoniacida15–8,39,43,44 (tablas 2A y B).
Polimorfismo conformacional de hebra sencilla por PCR (SSCP, Polymerase Chain reaction single stranded conformational polymorphism)Este método ha sido utilizado en investigaciones con la finalidad de identificar de mutaciones relacionadas a enfermedades genéticas y recientemente se ha extendido su empleo al estudio de mutaciones asociadas con la resistencia a antibióticos en tuberculosis45. Se fundamenta en que bajo condiciones no desnaturalizantes, un fragmento de una hebra de ADN adopta una conformación espacial, en función de su secuencia de nucleótidos; sin embargo, ante la existencia de una mutación, la secuencia nucleotídica y el plegamiento serán distintos, generando un patrón de migración electroforético diferente, detectable en una matriz de poliacrilamida39.
Esta técnica se ha aplicado al estudio de TB-DR44–47. Si bien es de bajo costo y sencilla de realizar, existen estudios que cuestionan su sensibilidad y especificidad para la detección de mutaciones en los genes responsables de generar resistencia a isoniacida, rifampicina y etambutol44,46–48 (tabla 1).
Ensayo de prueba en línea (LiPA, Line probe assay)Este método se utiliza para identificar aislados de micobacteria con diagnóstico previo por cultivo en BACTEC. Se basa en la hibridación de fragmentos biotinilados de ADN de una muestra sospechosa de ser TB-DR, sobre una tira de membrana en la cual se encuentran adheridos sondas de oligonucleótidos en línea, detectando dicho acoplamiento mediante un sistema colorimétrico vía biotina-streptavidina49. El kit comercial para identificar resistencia a rifampicina contiene diez sondas con las cuales se puede determinar tanto la pertenencia al complejo M. tuberculosis, como la detección de cuatro mutaciones específicas en el gen rpoB50. Estudios realizados mediante el empleo de esta técnica concluyen que es altamente sensible (95% o más) y específica (100%) para detectar tuberculosis resistente a rifampicina tanto de medios de cultivo como aislados y ligeramente menor para especímenes clínicos50.
GenoType MTBDRplusEs una técnica disponible comercialmente que permite la detección rápida de cepas del complejo M. tuberculosis resistentes a rifampicina e isoniacida, detectando mediante hibridación con sondas específicas, una variedad de mutaciones en el fragmento de 81pb del gen rpoB, así como la mutación del codón 315 del gen katG y mutaciones en la región promotora del gen inhA28,51.
Entre las ventajas que presenta se encuentra el poder aplicarse tanto en cultivo como directamente en esputo; además en cuanto a validez de la prueba ofrece una sensibilidad para detectar resistencia a rifampicina de 98,1% y para isoniacida de 90,2%, con una especificidad de 97,8% y 100% para cada droga respectivamente28,51.
Microdispositivos de ADN (DNA Microarray)Esta es una de las técnicas genotípicas más recientes que emplea la hibridación de sondas específicas marcadas con fluorocromos, con el ADN derivado de las muestras clínicas con sospecha de drogoresistencia. El sistema requiere el empleo de un microdispositivo, el cual consta de un cristal recubierto con una película de oro, sílice o algún otro material al que se adhieren genes o grupos de genes, silvestres y mutados, de modo que al colocar el material genético proveniente de una muestra, esta hibrida con la sonda complementaria y emite una señal fluorescente, cuyo análisis posterior permite identificar los genes portadores o carentes de las mutaciones, identificando así la resistencia o susceptibilidad por el antibiótico52,53.
Quizá el mejor atributo de esta técnica es su capacidad de analizar de manera global, automatizada y simultánea cientos de mutaciones de un mismo o diferentes genes, en un sólo ensayo y por lo tanto generan un ahorro de tiempo considerable. Esta técnica actualmente esta abriendo un nuevo panorama en el diagnóstico de la drogoresistencia de TB52,53; sin embargo, no se cuentan con estudios que evalúen su sensibilidad y especificidad. Por otro lado, los altos costos del equipo para el análisis, así como los relacionados con el diseño y construcción de los microdispositivos y la necesidad de contar con personal altamente calificado, limitan su utilidad en países con escasos recursos.
Discusión y conclusionesEs importante resaltar las ventajas de las técnicas de diagnóstico fenotípico de drogoresistencia en tuberculosis, tales como su alta sensibilidad, especificidad y bajo costo, motivos por los que se ubican como las técnicas de referencia global. Sin embargo, su gran desventaja es que requieren de una gran cantidad de tiempo para generar resultados confirmatorios, factor considerado como el más importante a atender dentro de la dinámica de atención oportuna en contra de la TB-DR. El desarrollo de nuevas técnicas fenotípicas para diagnóstico de drogorresistencia, fundamentadas en el crecimiento de M. tuberculosis en medio líquido reducen los tiempos considerablemente respecto al medio sólido, además son más sensibles, especificas y económicas.
Por otro lado, las nuevas técnicas genotípicas poseen una alta especificidad y mayor disminución en los tiempos para obtención del diagnóstico; pero poseen importantes limitaciones que impiden su aplicación generalizada en el campo clínico, tales como elevado costo y necesidad de un laboratorio y personal altamente especializado.
Aunado a lo anterior, 2 temas están replanteando el futuro papel de los métodos genotípicos. En primer lugar, la resistencia a un fármaco está mediada por la participación de una o más mutaciones en uno o varios genes (tabla 1), inclusive varios de ellos aún no identificados. Esto hace que la especificidad de la técnica para la búsqueda de una mutación en un gen sea alta, pero la sensibilidad para la identificación fenotípica de resistencia sea baja. Es decir, el hecho de no encontrar ciertas mutaciones en un gen o genes no necesariamente se traduce en un diagnóstico confiable de sensibilidad. Se estarían diagnosticando falsos negativos; por lo menos hasta que se logre la descripción de todas las mutaciones causantes de resistencia en todos los genes participantes.
Como segundo punto, en los últimos años el número de publicaciones relacionadas con la identificación de mutaciones en genes asociados con resistencia a TB se ha incrementado considerablemente19,54–59. En ellas se aprecia que las frecuencias en las mutaciones poseen un amplio rango de variación dependiendo de la procedencia, por ejemplo la mutación 315 para katG, explica la resistencia para isoniacida en el 52% de las cepas provenientes de México19, en el 62% de cepas procedentes de España54 y en el 100% de cepas originarias de Kazajistán57.
Lo anterior obliga a desarrollar mayor número de estudios que identifiquen las mutaciones más frecuentes en cada país o región geográfica, con la finalidad de desarrollar o adaptar procedimientos diagnósticos moleculares específicos, incrementando los niveles de eficiencia y logrando un mayor impacto en los programas de estas regiones o países.
En este sentido, quizá la técnica de microdispositivos de ADN sea la única que permitirá analizar todas las mutaciones de un sólo gen o de combinaciones de genes, por lo que se coloca como una de las más viables de subsistir, siempre y cuando se disminuyan sus costos y se evalúe adecuadamente su sensibilidad y especificidad.
En conclusión, podríamos decir que las condiciones económicas y el escaso conocimiento sobre la frecuencia de las mutaciones en los genes asociados a la DR de varios de los países afectados por la nueva dinámica de la TB-DR, son tan solo 2 de los factores que proyectan un futuro incierto para las técnicas genotípicas y más aún si se comparan con las nuevas técnicas fenotípicas (MOD, alamar azul, reazurina, nitrato reductasa y LPR), cuyos atributos las situarían como aquellas con las mejores perspectivas.
Es por esto que resulta urgente desarrollar estudios clínicos amplios que confirmen la utilidad de dichas técnicas fenotípicas para finalmente establecer marcos normativos o reglas generales de operación que permitan su inclusión en los programas contra TB-DR, con énfasis en países con escasos recursos económicos.
Conflicto de interesesLos autores declaran no tener conflicto de intereses.
Este trabajo fue apoyado parcialmente por un financiamiento del Consejo Nacional de Ciencia y Tecnología (Fondos CONACYT-SALUD, 2005-02-14380) y Universidad Veracruzana POA-UV 2007–2008. B. Cuevas-Córdoba es becaria de doctorado, CONACYT N.o 171183.