Los hallazgos acerca de la relevancia biológica de los espermatozoides, más allá de la mera aportación del genoma paterno al embrión, han abierto las puertas al estudio del RNA espermático bajo nuevos enfoques. Lejos de constituir una mera reserva residual, esta fracción ha demostrado ser una población compleja y multifuncional, con una amplia gama de implicaciones en procesos biológicos tanto espermatogénicos como de desarrollo embrionario tras la fecundación del ovocito. Más allá de la población de RNA codificante, el papel ejercido por los RNA no codificantes comienza a desentramarse e integrarse con el funcionamiento celular global, demostrando su relevancia sobre la regulación celular e incluso su influencia epigenética. Los estudios comparativos de expresión a gran escala permiten obtener información global sobre los cambios en el transcriptoma espermático en diferentes poblaciones, lo que aporta al RNA un nuevo valor diagnóstico. En esta revisión se ofrece una visión general sobre la biosíntesis, las funciones y los mecanismos biológicos relacionados con los diferentes subtipos de RNA espermáticos, así como sobre su importancia durante la espermatogénesis, su implicación tras la fecundación y su valor como biomarcadores de fertilidad.
The biological relevance of sperm contribution to the embryo has currently been shown to go beyond a mere transmission of the paternal genome. New findings have revealed that spermatozoa carry a complex and multifunctional population of RNA with a wide range of implications in spermatogenesis, fertilisation, and embryogenesis. Accordingly, the consideration of these molecules as a simple residual pool of earlier processes has been left behind. Besides the coding RNAs, the role of non-coding RNAs has started to be untangled and integrated within the global cell processes, demonstrating their relevance in cellular regulation and epigenetics. Comparative high-throughput expression studies have provided a general impression on the differences in the sperm transcriptome between populations. Hence, a new diagnostic value related to these RNAs has emerged. In this review, an overview of biosynthesis, functions and biological mechanisms of different subtypes of sperm RNAs is presented. The relevance of these molecules during spermatogenesis, their role after fertilisation, and their potential value as fertility biomarkers are also discussed.
Los espermatozoides son células altamente diferenciadas cuya función principal consiste en aportar el genoma haploide paterno al ovocito. Hasta hace relativamente poco tiempo, se consideraba que esta aportación genética constituía la única contribución del espermatozoide tras la fecundación; por ello, el descubrimiento de la existencia de una fracción funcional de RNA espermático dio paso a un amplio marco de nuevos estudios y líneas de investigación (Hosken y Hodgson, 2014). De esa forma, se determinó que la compleja población de RNA del espermatozoide suponía una nueva herramienta potencial para la investigación básica y aplicada en el ámbito de la infertilidad masculina (Hamatani, 2012).
La fracción de transcritos presente en un espermatozoide maduro consta tanto de RNA codificantes (RNA mensajero o mRNA) como no codificantes. Estos últimos se dividen en RNA pequeños no codificantes (small non-coding RNA o sncRNA) y RNA largos no codificantes (long non-coding RNA o lncRNA). A su vez, los sncRNA se clasifican principalmente en microRNA (miRNA), RNA asociados a PIWI (PIWI-interanting RNA o piRNA), RNA pequeños de interferencia (small interfering RNA o siRNA) y siRNA endógenos (endogenous siRNA o endo-siRNA) (Jodar et al., 2013). Se estima que cada espermatozoide contiene aproximadamente unos 50 fg (femtogramos) de lncRNA y mRNA (de tamaños de más de 200 nucleótidos) y 0,3 fg de sncRNA (menos de 200 nucleótidos) (Goodrich et al., 2013). Esto supone una cantidad de RNA 200 veces menor a la de otros tipos celulares (Jodar et al., 2013).
El RNA espermático codificanteComo se ha descrito previamente, los mRNA suponen la fracción codificante del total de moléculas de RNA. Su localización en los espermatozoides se sitúa en la pieza intermedia y en la cabeza espermática, donde se concentran cerca de la envuelta del núcleo y constituyen una parte estructural de la matriz nuclear (Li y Zhou, 2012; Hamatani, 2012). Aunque algunas moléculas de mRNA espermático están intactas, existe una marcada tendencia a una degradación progresiva hacia el extremo 3′ (Jodar et al., 2013).
La caracterización de los transcritos presentes en los espermatozoides humanos se ha llevado a cabo gradualmente en estudios basados en la realización de RT-PCR (reverse transcription polymerase chain reaction) (Kumar et al., 1993), serial analysis of gene expression (Zhao et al., 2006), microarrays (Ostermeier et al., 2002) e hibridación in situ (Wykes et al., 1997). El primer mRNA identificado fue el del gen C-MYC, el cual está involucrado en la funcionalidad espermática, concretamente en la capacitación y en la reacción acrosomal (Naz et al., 1991). En la actualidad conocemos la presencia de numerosos transcritos correspondientes a factores de transcripción, como NFjB (nuclear factor kappa B), HOX2A (homeobox protein Hox-2a) o ICSBP (interferon consensus sequence-binding protein). De la misma forma, también se han descrito transcritos correspondientes a la proteína cinasa JNK2, del factor de crecimiento HBEGF y de los receptores ERBB3 y RXRB (Garrido et al., 2013; Dadoune et al., 2005). Las mismas técnicas desvelaron la presencia de mRNA codificantes para la ciclina B1 (proteína reguladora del ciclo celular), el modulador de la transcripción STAT4, la familia de genes DAZL (deleted in azoospermia-like), el gen SRY (sex-determining region Y) y la proteína PLC-Z (phospholipase C-Z), junto con protaminas, receptores de progesterona y receptores de estrógenos (Dadoune, 2009).
Un serial analysis of gene expression permitió clasificar los mRNA espermáticos en 25 grupos funcionales diferentes. El grupo predominante englobaba principalmente genes de proteínas nucleares involucradas en la transcripción. Analizando los 30 transcritos no fragmentados más frecuentes, se observó que la mayoría presentaba funciones relacionadas con la espermatogénesis, la funcionalidad espermática, la fertilidad masculina, la fecundación y el desarrollo embrionario (Zhao et al., 2006). La presencia de estos transcritos y sus funciones asociadas supone un indicativo de la importancia biológica de estas moléculas en la regulación de la fertilidad masculina (Jodar et al., 2013).
El RNA espermático no codificanteSe estima que solamente un 5-10% del genoma de los mamíferos corresponde a zonas codificantes. Aun así, es sabido que la mayoría del genoma se transcribe, sobrepasando el número de secuencias génicas registradas; esto significa que gran parte del transcriptoma de los mamíferos no es codificante (Luk et al., 2014). Aunque inicialmente estos ncRNA (non-coding RNA) fueron asociados a una fracción «basura» del genoma (junk RNA), más recientemente se han descrito numerosas funciones biológicas en las cuales estos transcritos están involucrados. Si bien, en general, los ncRNA (non-coding RNA) tienen un papel regulador de la expresión génica, pueden ejercer o estar involucrados en otros procesos biológicos. Según su tamaño, su proceso de biogénesis y el tipo de proteínas a las que se encuentren asociados, podemos clasificar a los ncRNA en diferentes familias con funciones específicas (Luk et al., 2014).
Los lncRNADentro de la familia de ncRNA, aquellos con una longitud superior a las 200 pb son clasificados como lncRNA. Aunque no son codificantes, gran parte de ellos se originan por transcripción mediante RNA polimerasa ii como los mRNA; también pueden producirse a partir del procesamiento de otros tipos de transcritos (Luk et al., 2014).
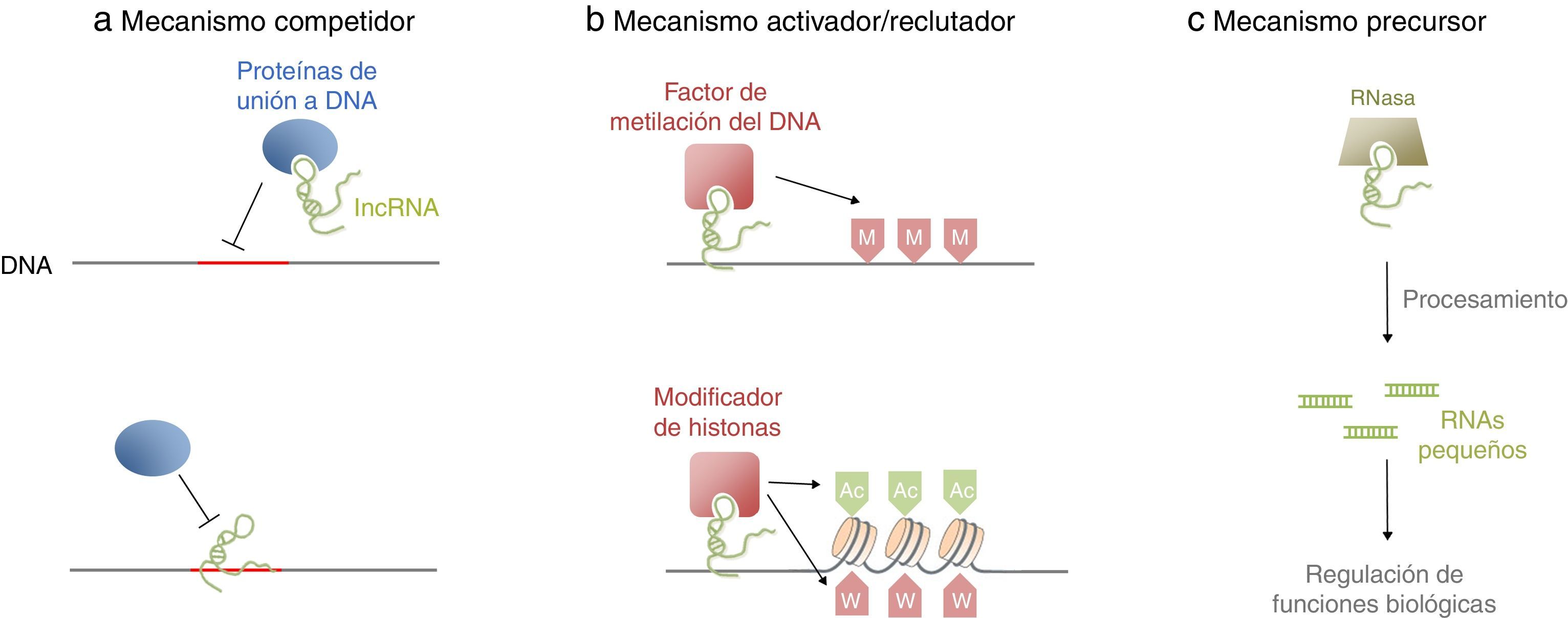
Al contrario de lo que ocurre con algunos sncRNA, los lncRNA no necesitan asociarse a ningún grupo proteico para ser procesados o ejercer su papel biológico (Luk et al., 2014). Su función principal es intervenir en diversos procesos de regulación; varios de ellos están relacionados con modificaciones epigenéticas que modulan el estado transcripcional de determinados mRNA o incluso de cromosomas completos (Bao et al., 2013). Se han propuesto 3mecanismos de regulación ejercida por los lncRNA: el mecanismo competidor, el activador/reclutador y el precursor (Luk et al., 2014) (fig. 1). El mecanismo competidor contempla la capacidad de estos RNA para anclarse a ciertas proteínas que se unen específicamente a determinadas regiones del DNA (como los factores de transcripción), e impedir su funcionamiento; también pueden hibridar con secuencias complementarias del DNA e impedir la unión de esas proteínas específicas a dicha región (Kraus et al., 2013; Luk et al., 2014). El mecanismo activador/reclutador consiste en la activación de modificadores epigenéticos, o bien en aproximar y retener a dichos modificadores en lugares específicos del genoma, promoviendo así la metilación del DNA y la modificación de las histonas (Luk et al., 2014). Por último, el mecanismo precursor contempla la posibilidad de que los lncRNA sean procesados para dar lugar a RNA más cortos que pueden estar implicados en otros procesos de regulación postraduccional (Keniry et al., 2012).
Mecanismos de regulación de la expresión génica ejercida por los lncRNA: a) Mecanismo competidor, en el que el lncRNA inhibe la unión de una proteína a su secuencia complementaria de DNA, ya sea anclándose a la proteína o a la secuencia. b) Mecanismo activador/reclutador, en el que el lncRNA activa modificadores epigenéticos y los retiene en regiones específicas del genoma. c) Mecanismo precursor, en el que el lncRNA puede ser procesado y originar RNA pequeños involucrados en otros procesos de regulación de la expresión génica.
Se han identificado algunos tipos de lncRNA que aparecen especialmente enriquecidos en la fracción de RNA espermáticos: los RNA asociados a cromatina (chromatin-associated RNA o CAR) y algunos RNA pequeños nucleares asociados a ILF3/NF30 (small ILF3/NF90-associated RNA o snaR). Los CAR podrían ejercer influencia sobre la arquitectura genómica o regular la expresión génica (Jodar et al., 2013). Uno de los snaR más abundantes en espermatozoides es el snaR-G1; se sospecha que su papel funcional en estas células es significativo debido a su alta concentración, mayor que la hallada en células germinales testiculares (las cuales ya presentan una concentración de snaR-G1 aproximadamente 100 veces mayor que en tejidos somáticos) (Jodar et al., 2013).
Los sncRNALos sncRNA son moléculas no codificantes de RNA de tamaño menor a 200 nucleótidos. La mayor parte de los sncRNA pertenecen a 3categorías principales: miRNA, piRNA y endo-siRNA (Röther y Meister, 2011). Estos se caracterizan por que su funcionalidad depende de su asociación con las denominadas proteínas argonautas (AGO). Esta familia de proteínas se subdivide en 2grupos: la subfamilia AGO y la subfamilia PIWI (específica de la línea germinal). Tanto los miRNA como los endo-siRNA se unen específicamente a proteínas AGO, mientras que los piRNA se asocian con proteínas PIWI (De Mateo y Sassone-corsi, 2014).
Aparte, también se encuentran otras categorías menos frecuentes como los elementos de repetición, los elementos asociados a sitios de inicio de la transcripción o a los promotores, los snRNA (small nuclear RNA), los snoRNA (small nucleolar RNA), los mse-tsRNA (mature-sperm-enriched tRNA-derived small RNA) y los YRNA (RNA asociados a proteínas Ro, que forman un complejo ribonucleoproteico) (Jodar et al., 2013). También existen ciertas moléculas pequeñas de RNA que constituyen fragmentos de transcritos codificantes o no codificantes provenientes de zonas no descritas del genoma, cuya función específica aún no se conoce (Jodar et al., 2013).
Los miRNALos miRNA suponen la clase de sncRNA mejor caracterizada hasta la fecha. Se trata de moléculas monocatenarias de RNA de unos 22 nucleótidos de longitud. Su función principal es la de regular la expresión génica, generalmente mediante la unión a la región 3′ UTR de sus mRNA diana, provocando principalmente su represión (Luo et al., 2015). Por otro lado, existen estudios que señalan que algunos miRNA se unen específicamente a otras regiones, como a los promotores, y ejercen una función activadora sobre la expresión de dichos genes (Place et al., 2008). Se estima que pueden llegar a regular más del 60% de los genes codificantes de proteínas del genoma; un mismo miRNA puede tener cientos de genes diana, mientras que un gen también puede estar regulado por más de un miRNA (Luo et al., 2015).
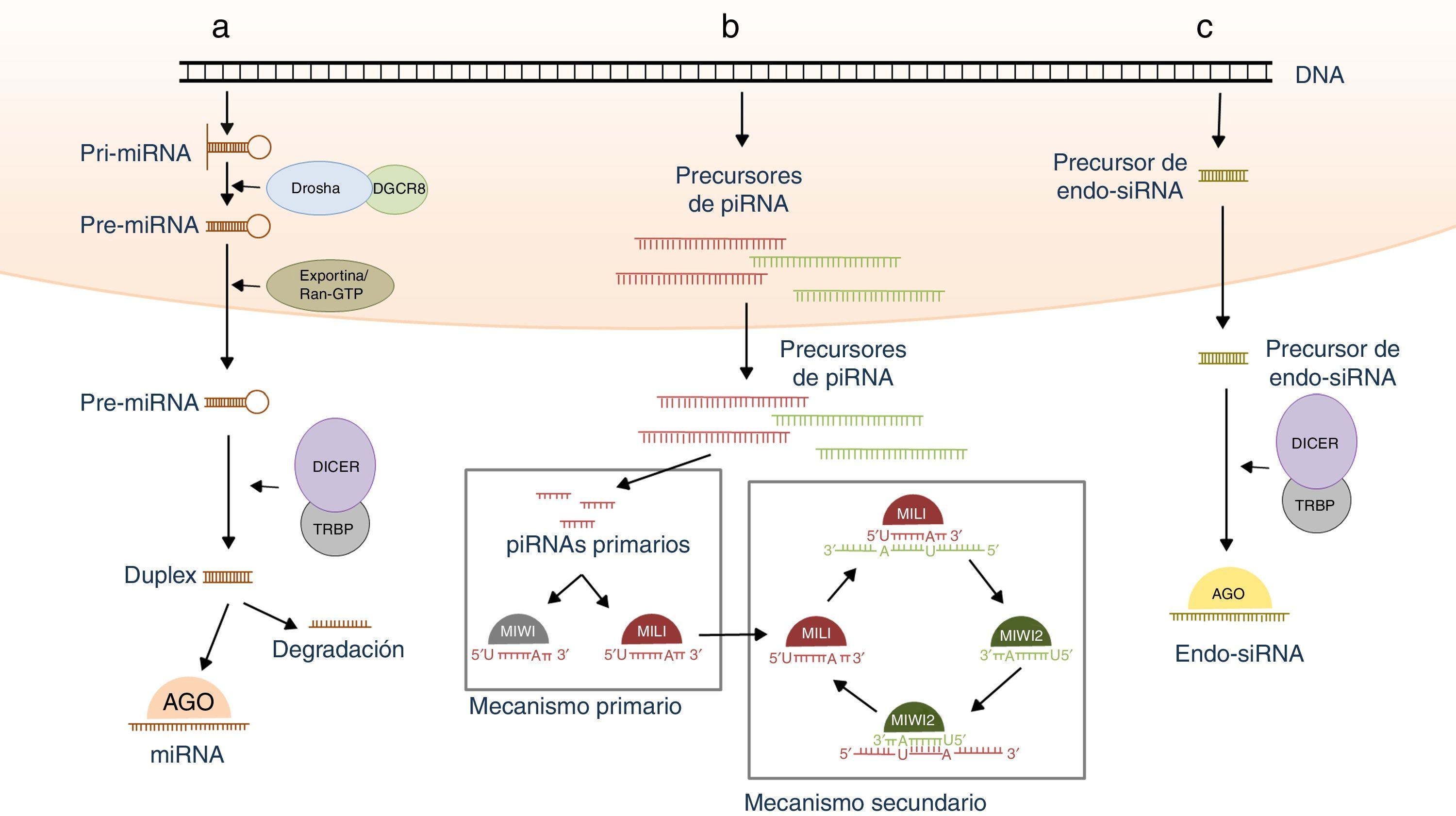
El proceso de biosíntesis de los miRNA comienza en el núcleo con la acción de la polimerasa ii, la cual produce la transcripción de unos precursores de mayor tamaño, los miRNA primarios (pri-miRNA) (Luo et al., 2015). Aunque la mayoría de las secuencias de miRNA se encuentran distribuidas individualmente por el genoma, existe una fracción importante de ellas que se agrupa en clústeres que contienen múltiples miRNA, transcritos en un único precursor que es posteriormente procesado en miRNA individuales (Ambros, 2001). Los pri-miRNA son procesados por el complejo denominado «microprocesador», cuyos componentes principales son DROSHA (ribonucleasa III) y DGCR8 (DiGeorge syndrome critical region 8). Mediante este mecanismo se da lugar a una forma intermedia (pre-miRNA). Independientemente de la ruta de síntesis, una vez generados estos precursores, son transportados hasta el citoplasma mediante exportina/Ran-GTP para ser procesados por DICER (una RNasa III endonucleasa) hasta su forma madura de miRNA monocatenario (Luo et al., 2015). En general, solamente una de ambas cadenas del dúplex inicial suele permanecer funcional, mientras que la otra es degradada, aunque hay casos en los que ambas cadenas pueden ser funcionales por separado (Schwarz et al., 2003). Los miRNA maduros se incorporan a un complejo ribonucleoproteico denominado miRISC (miRNA-induced silencing complex), en el cual también están englobadas las proteínas AGO (Thomas et al., 2010) (fig. 2a).
Proceso de biosíntesis en humanos de los RNA pequeños no codificantes (sncRNA). a) Las moléculas de miRNA primario (pri-miRNA) dan lugar a los precursores pre-miRNA, que son procesados en el citoplasma para dar lugar a los miRNA funcionales. b) Las moléculas precursoras de piRNA se transcriben a partir de diferentes clústeres y son procesadas en el citoplasma mediante el mecanismo primario de biosíntesis o el mecanismo secundario o de amplificación «ping-pong». c) Los endo-siRNA son generados de forma independiente a la acción de la maquinaria DROSHA/DGCR8, por lo cual sus moléculas precursoras son directamente transportadas al citoplasma y procesadas para generar los endo-siRNA maduros.
Se han identificado más de un centenar de moléculas de miRNA en células germinales testiculares; aproximadamente el 40% de ellas se expresa de forma diferencial en el tejido testicular respecto a los tejidos somáticos (Luo et al., 2015). Diversos estudios se han centrado en caracterizar los miRNA presentes en espermatozoides y en determinar cuáles de ellos son más abundantes (algunos ejemplos son los miRNA hsa-miR-34b-3p, hsa-miR-375 y hsa-miR-191-5p) (Ostermeier et al., 2005; Salas-Huetos et al., 2014). Entre ellos, se ha hallado un grupo de 221 miRNA expresado constitutivamente en todos los individuos fértiles estudiados, de forma independiente a factores como la edad o los parámetros seminales básicos (volumen, morfología, concentración y movilidad). Estos miRNA están involucrados en la regulación de una serie de genes asociados a procesos biológicos que intervienen en la espermatogénesis y la embriogénesis (Salas-Huetos et al., 2014). En otro estudio de caracterización se detectaron 35 miRNA espermáticos que ejercían una función reguladora sobre mRNA, mayoritariamente relacionados con procesos de desarrollo embrionario y crecimiento celular (Krawetz et al., 2011).
Los piRNALos piRNA son moléculas monocatenarias de 26-31 nucleótidos con una fuerte tendencia a la presencia de uridinas en el extremo 5′. Estas moléculas se caracterizan por estar asociadas a proteínas PIWI y, por tanto, están presentes exclusivamente en la línea germinal (Röther y Meister, 2011). En ratón se han descrito 3proteínas PIWI expresadas principalmente en células germinales masculinas: PIWIL1 o MIWI, PIWIL2 o MILI y PIWIL4 o MIWI2 (Chuma y Nakano, 2013). Se ha detectado que los piRNA son el tipo de sncRNA más abundante en los espermatozoides humanos (Pantano et al., 2015).
La función principal de estas moléculas es la de silenciar a los retrotransposones mediante control transcripcional y postranscripcional. Este papel silenciador está íntimamente relacionado con el desarrollo de las células germinales: se trata de un mecanismo de protección del genoma frente a las modificaciones producidas por los elementos retrotransponibles (Chuma y Nakano, 2013). El retrotransposón LINE1 (long interspersed nuclear elements 1) constituye la diana más abundante de los piRNA espermáticos (Pantano et al., 2015). Además de este mecanismo de silenciamiento, hay indicios de que los piRNA juegan un papel en el control de la regulación epigenética, concretamente en la metilación de novo en embriones y en proespermatogonias (células germinales masculinas prenatales), aunque su mecanismo aún no está completamente caracterizado (Chuma y Nakano, 2013).
Los piRNA se originan a partir de diferentes clústeres localizados en distintas regiones cromosómicas y cuyo tamaño puede variar desde las pocas unidades hasta cientos de kilobases (Girard et al., 2006). Cada clúster contiene varias secuencias de piRNA que no se solapan entre ellas; la mayoría de estas secuencias pertenecen únicamente a una de las cadenas de cada clúster. A partir de esta información, se han propuesto 2mecanismos paralelos de biosíntesis de los piRNA: la ruta de procesamiento primario y el mecanismo secundario o también llamado amplificación ping-pong (fig. 2b) (Chuma y Nakano, 2013).
Tomando el ratón como modelo de estudio, según el mecanismo primario, se ha señalado que los RNA trascritos a partir de los clústeres son precursores de piRNA, carentes de estructuras hairpin que requieran un procesamiento mediante DICER (como sí sucede con los miRNA). El precursor es procesado por nucleasas que producen un extremo 5′ con un grupo fosfato (el que se unirá a la proteína PIWI) y un extremo 3′ con un grupo OH y una metilación 2′-OH. El tamaño final de ese piRNA primario vendrá determinado por el tamaño de hendidura del tipo de proteína PIWI en cuestión, que es de unos 26 nucleótidos en el caso de MILI, de 28 para MIWI2 y de 29-30 para MIWI (Chuma y Nakano, 2013).
El mecanismo secundario de amplificación ping-pong es un ciclo que genera nuevos piRNA a partir de transcritos con una secuencia complementaria a la de los piRNA primarios. Esta ruta está directamente relacionada con la regulación de los retrotransposones. Una fracción considerable de los piRNA existentes contiene una zona de solapamiento de 10 pb de tamaño, presente en todos ellos (ya sea en la dirección sentido o antisentido), con un uracilo en el extremo 5′ y una adenina situada en la posición número 10; esto sucede como causa y también como motivo del mencionado mecanismo secundario. Los piRNA primarios hibridan con las secuencias complementarias de sus transcritos diana (en su mayoría elementos de repetición o retrotransposones) y la proteína PIWI actúa cortando el transcrito diana a la altura de la adenina situada en el décimo nucleótido del piRNA primario; de esta forma se genera un precursor de piRNA secundario con un uracilo en su extremo 5′, complementario a la adenina. El extremo 3′ del precursor es procesado a través de un mecanismo similar al de los piRNA primarios, y se produce el piRNA secundario maduro al unirse a MIWI2. A través de este ciclo se produce un silenciamiento general de los niveles retrotransposones existentes (Chuma y Nakano, 2013). Es probable que los mecanismos de biosíntesis descritos en ratones también estén presentes en humanos (Röther y Meister, 2011).
Los endo-siRNALos endo-siRNA tienen un tamaño de unos 22 nucleótidos (Luo et al., 2015). Su función biológica de regulación es muy similar a la de los miRNA: sobre todo, difieren de estos sncRNA en su proceso de biosíntesis. Generalmente, sus moléculas precursoras son fragmentos grandes de RNA de doble cadena generados de forma espontánea: puede tratarse de 2fragmentos procedentes de diferentes loci (denominados trans-nat-dsRNA) o de una transcripción bidireccional del mismo locus (los llamados cis-nat-dsRNA) (Song et al., 2011). Este precursor es transportado al citoplasma desde el núcleo, donde es procesado por DICER de forma independiente a la maquinaria DROSHA-DGCR8. De esta forma, una de las cadenas del precursor se asocia a proteínas AGO: forman un complejo ribonucleoproteico denominado RISC (RNA-induced silencing complex) y constituyen el endo-siRNA maduro y funcional (fig. 2c). Principalmente, los endo-siRNA regulan la expresión génica postranscripcional mediante su unión específica a los mRNA diana, mayoritariamente a su región 3′ UTR (Song et al., 2011). De esta forma, pueden inducir su silenciamiento o, si la complementariedad de su secuencia con la del transcrito diana es perfecta, su degradación mediante el complejo RISC (Luo et al., 2015).
En un principio fueron identificados en levaduras, plantas y C. elegans, aunque en la actualidad se han descrito también en mamíferos (Song et al., 2011). Su expresión se produce en todos los tejidos (Luo et al., 2015), aunque en ratones se ha hallado una expresión especialmente alta en células germinales masculinas. Concretamente, se han hallado 73 endo-siRNA por secuenciación de RNA procedente de células testiculares, con dianas principalmente en mRNA (92%), pero también en ncRNA (4%), pseudogenes (3%) y retrotransposones (1%) (Song et al., 2011).
En relación con su función de control postranscripcional, los endo-siRNA intervienen en la regulación de la espermatogénesis (Luo et al., 2015). En células germinales masculinas, se señala que podrían tener dianas en cientos de transcritos expresados a lo largo de todas las fases de este proceso y ejercer un posible papel en el control de la expresión durante esta espermatogénesis. Aparte, se ha descubierto que pueden tener un rol regulador transcripcional al actuar como moléculas guía para dirigir la acción de factores epigenéticos (como son las metiltransferasas o las histonas metiltransferasas) con el objetivo de modificar la conformación de la cromatina, de modo que también intervienen en la actividad transcripcional (Song et al., 2011). En la posfecundación, hay indicios de que los endo-siRNA son necesarios para que se produzca correctamente el desarrollo embrionario preimplantacional (Suh et al., 2010).
Funciones del RNA en la espermatogénesisLa espermatogénesis es el proceso de biogénesis de los espermatozoides a partir de las espermatogonias y se puede dividir en 3 fases principales: la mitótica, la meiótica y la posmeiótica o espermiogénesis (para una revisión, véase Hamatani, 2012). Los cambios más significativos en términos de diferenciación celular se producen durante esta última etapa, que engloba la transformación de las espermátidas redondas en espermatozoides. Estas modificaciones tienen lugar principalmente en la estructura: se elimina gran parte del citoplasma junto con sus componentes ribonucleicos para ser fagocitado por las células de Sertoli y se produce la formación del acrosoma y el flagelo (Dadoune, 2009; Cappallo-Obermann et al., 2011). Por otro lado, también se producen una serie de eventos de transformación nuclear, en los que la mayoría de las histonas son sustituidas por protaminas. Como consecuencia, el DNA sufre un empaquetamiento altamente compacto y el núcleo adopta una estructura muy condensada (Cappallo-Obermann et al., 2011).
Durante la espermatogénesis, la actividad transcripcional es continua desde los primeros estadios hasta la generación de las espermátidas redondas (Jodar et al., 2013). No obstante, aumenta considerablemente en 2periodos: el periodo premeiótico (espermatogonias) y el periodo comprendido entre la fase paquiténica de la primera división meiótica y las espermátidas redondas. Durante estas «olas transcripcionales», altamente reguladas, se producen gran cantidad de transcritos de diferentes clases. Esta síntesis es específica para cada estadio celular en función de los RNA requeridos para continuar con el desarrollo espermático en cada etapa (De Mateo y Sassone-corsi, 2014). Los transcritos sintetizados durante estos periodos pertenecen a diversos tipos de RNA tanto codificante (mRNA) como no codificante (miRNA, siRNA, piRNA y lncRNA) (Jodar et al., 2013).
Finalmente, durante la fase acrosómica de la espermiogénesis, se produce una detención de la actividad transcripcional, coincidiendo con la reorganización de la cromatina (Miller et al., 2005). Los procesos de traducción también sufren una detención: una de las causas es la ausencia de RNA ribosómico (rRNA) intacto y funcional en el citoplasma del espermatozoide (Dadoune, 2009). Se ha propuesto que esta ausencia de rRNA funcional puede suponer un mecanismo para evitar la traducción de transcritos no deseados después de la fecundación, que puedan comprometer el desarrollo del embrión (Cappallo-Obermann et al., 2011). En la mitocondria, sin embargo, la transcripción y traducción siguen produciéndose (Dadoune, 2009).
El RNA codificante en la espermatogénesisHaciendo uso de bases de datos de ontología génica, se han descrito 760 genes implicados en la espermatogénesis humana (Carbon et al., 2009). Como ya se ha mencionado previamente, durante las 2«olas transcripcionales» que se producen en la espermatogénesis se generan una serie de transcritos específicos para cada etapa del proceso. De esta forma, las células mitóticas expresan transcritos necesarios tanto para mantener la pluripotencialidad de las espermatogonias de tipo A como para diferenciar las de tipo B hasta su estadio de espermatocitos (Kotaja, 2014). Por otro lado, los espermatocitos expresan el RNA necesario para llevar a cabo la meiosis mientras que las espermátidas contienen transcritos involucrados en la regulación de la espermiogénesis, producidos previamente a la detención de la transcripción, entre los que se incluyen los que codifican protaminas (Dadoune, 2009; Kotaja, 2014). Tanto estos transcritos como parte de los previamente sintetizados se almacenan en el citoplasma en forma de ribonucleoproteínas (Jodar et al., 2013), se mantienen protegidos y sirven como una fuente de transcritos necesarios tras la detención de la transcripción (Dadoune, 2009; Miller et al., 2005).
En humanos, se ha descrito que la acción conjunta de los factores de transcripción NFAT (nuclear factor of activated T-cells protein family) y JUN (ju-nana, «diecisiete» en japonés por corresponderse al oncogén homólogo al virus del sarcoma aviar 17) es necesaria para los procesos de regulación producidos en las espermatogonias. NFAT tiene un importante papel en la respuesta inmune y la expresión anormal del oncogén JUN suele asociarse a la generación de tumores. Dada su sobreexpresión en espermatogonias, se ha propuesto su gran importancia en la regulación del mantenimiento de la indiferenciación celular (Zhu et al., 2016).
Durante la fase meiótica de la espermatogénesis, se ha revelado la importancia de los genes HOX en la regulación de la organización cromosómica. Por otro lado, el factor de transcripción SP1 (specificity protein 1) es especialmente importante para el proceso de remodelación de la cromatina durante esta etapa, además de intervenir en la regulación de otros eventos como el crecimiento y la diferenciación celular, la apoptosis, las respuestas inmunes y la respuesta al daño del DNA (Zhu et al., 2016).
Respecto a las espermátidas, es importante la acción del factor de transcripción TCF3 (transcriptin factor 3) en el proceso de espermiogénesis, ya que está directamente involucrado en la regulación de la vía de señalización Wnt (wingless-related integration site), vital para el control de la etapa de elongación de las espermátidas (Zhu et al., 2016).
Además de la expresión de estos genes necesarios para la diferenciación de las espermatogonias, también se ha propuesto otro mecanismo por el cual los mRNA espermáticos ejercen un control sobre la espermatogénesis. Se ha sugerido que el mRNA espermático podría actuar durante el proceso de empaquetamiento del DNA, ejerciendo un papel pasivo en la reorganización de la cromatina y, a su vez, una función más activa al determinar las secuencias de DNA que serán empaquetadas con histonas y no por protaminas (Miller et al., 2005). Estudios de ontología génica indican que la cromatina espermática histónica no está distribuida aleatoriamente, sino que se encuentra primordialmente en loci que contienen genes implicados en el desarrollo embrionario, incluyendo factores de transcripción embrionarios y proteínas que intervienen en rutas de señalización (Hammoud et al., 2009). Por tanto, se contempla que los mRNA espermáticos pueden promover el empaquetamiento nucleosómico selectivo de secuencias que son importantes a nivel embrionario, de forma que puedan ser transcritas con más facilidad (Hamatani, 2012).
El miRNA en la espermatogénesisA lo largo de los años, la caracterización de los distintos miRNA que intervienen en la regulación de la espermatogénesis humana se ha ido detallando. Aunque la expresión de miRNA es abundante a lo largo de toda la espermatogénesis, se conoce que en las espermatogonias se produce una ola de síntesis de miRNA cuya acción se ha relacionado con el mantenimiento de la indiferenciación celular y, por tanto, del nicho de espermatogonias. Algunos ejemplos de miRNA de ratones involucrados en esta función son miR-20, miR-21, miR-34c, miR-106a, mir-135a, miR-146a, miR-182, miR-183, el clúster miR-17-92 y los miRNA miR-221 y miR-222 ligados al cromosoma X (De Mateo y Sassone-corsi, 2014; Kotaja, 2014). Por el contrario, se ha descrito que la función ejercida por la familia let-7 promueve la diferenciación de las espermatogonias a espermatocitos (Tong et al., 2011). El miR-383 actúa en los primeros estadios de la espermatogénesis regulando la proliferación y la muerte celular (De Mateo y Sassone-corsi, 2014). Posteriormente, durante la segunda ola de transcripción (paquiteno-espermátidas redondas), participan miRNA relacionados con diversas funciones. Por ejemplo, el clúster miR-449 y los miR-34b/c están involucrados en regular y evitar la apoptosis masiva. Los miR-18 y miR-214 se relacionan con la interacción con factores de transcripción de choque térmico implicados en la correcta reorganización de la cromatina y en la maduración espermática. Además, otros miRNA intervienen en la regulación de la expresión de protaminas y proteínas de transición (TPs); 2ejemplos de ello son el miR-469 (que tiene por dianas a los genes de la protamina PRM2 y de TP2) y el miR-122a (también específico del gen TP2) (De Mateo y Sassone-corsi, 2014; Kotaja, 2014).
El piRNA en la espermatogénesisEl control sobre los retrotransposones de los piRNA en las células germinales es un mecanismo crítico para el desarrollo de la espermatogénesis. En ratones, la interrupción de la ruta biosintética de los piRNA da lugar a individuos estériles sin espermatozoides funcionales (Pillai y Chuma, 2012).
Los piRNA y sus proteínas asociadas actúan dependiendo de la etapa en la que se produzca su síntesis; se clasifican principalmente en prepaquiténicos y paquiténicos (Pillai y Chuma, 2012). Los piRNA prepaquiténicos se sintetizan en las espermatogonias desde el periodo fetal, bien mediante mecanismo primario de biosíntesis o bien a través del mecanismo ping-pong (De Mateo y Sassone-corsi, 2014), y se asocian específicamente a MIWI2 y MILI (Yadav y Kotaja, 2014). Se ha comprobado que la ausencia de estas proteínas desemboca en la activación e hipometilación de elementos transponibles como IAP (intracisternal A-particle) y LINE1. Por ello, se ha señalado que su acción puede provocar una represión de estos elementos tanto transcripcional (mediante el impulso de la metilación de sus promotores) como postranscripcional (a través de su degradación mediante piRNA). A nivel postranscripcional, la proteína MIWI2 mantiene un repositorio de piRNA antisentido para las secuencias de los transposones que se encuentren activos en el genoma, con un funcionamiento comparable al del sistema inmunitario (Pillai y Chuma, 2012).
Por otro lado, los piRNA paquiténicos se sintetizan únicamente mediante el mecanismo de biogénesis primario (De Mateo y Sassone-corsi, 2014). Estos piRNA se unen específicamente a proteínas MIWI2 y silencian elementos transponibles únicamente a nivel postranscripcional. Al contrario de lo que ocurre con los piRNA prepaquiténicos, no provocan alteraciones en los patrones de metilación (Pillai y Chuma, 2012).
El lncRNA en la espermatogénesisSe han identificado lncRNA que pueden estar potencialmente involucrados en el desarrollo de las células germinales masculinas, aunque solo algunos de ellos han sido caracterizados en espermatozoides (Luk et al., 2014). Entre ellos, se ha propuesto que el lncRNA DMR (DMRT1 related gene), expresado en espermatogonias, contribuye a controlar la diferenciación de estas células a espermatocitos primarios, pues su gen diana es DMRT1, que codifica para un factor de transcripción esencial en el desarrollo germinal. También en las espermatogonias se expresan los denominados Spga-lncRNA1 y 2, que podrían ser importantes en el mantenimiento de la pluripotencialidad de las espermatogonias. Por otro lado, entre los lncRNA expresados en los espermatocitos paquiténicos destaca TSX (testis-specific X-linked), que podría estar involucrado en la regulación de la meiosis, y MRHL (meiotic recombination hot spot locus), cuya función dentro del proceso de la espermatogénesis aún está por caracterizar (Luk et al., 2014).
Funciones del RNA espermático en el desarrollo embrionario tempranoCuando se produce la fecundación, además del genoma paterno, el espermatozoide libera otros componentes al interior del ovocito. Entre ellos destacan el centriolo, PLC-Z y otras proteínas perinucleares de la teca, incluyendo moléculas de señalización de rutas de cinasas, factores de transcripción y proteínas estructurales. La aportación de todos estos componentes intervendrá en los procesos de activación y desarrollo embrionario temprano (Boerke et al., 2007).
La liberación del RNA espermático al citoplasma del ovocito y su funcionalidad fue cuestionada durante mucho tiempo. Tras su descubrimiento, se postuló que estas moléculas constituían una fracción de RNA reminiscente sintetizado durante la última fase de la expresión en espermátidas y sin ninguna función posterior (Cummins, 2001). Esto estaba respaldado por el hecho de que la fracción de RNA espermático liberado era muy pequeña (10-20 fg en humanos) (Krawetz, 2005) en comparación con la del ovocito (alrededor de 330.000 fg en humanos) (Kocabas et al., 2006), por lo que su trascendencia en el embrión se ponía en entredicho. Sin embargo, cada vez surgieron más datos que indicaban que el RNA espermático tiene una funcionalidad en el cigoto.
Los mRNA espermáticos liberados al cigoto pueden dividirse en 3grupos principales según su procedencia y función. El primer grupo englobaría los transcritos expresados específicamente en las espermátidas sin una función potencial dentro del embrión que se haya predicho o descubierto hasta el momento. Estas moléculas podrían suponer un mero pool que ha ido permaneciendo de forma residual en las células resultantes, sin función en el embrión, para el que incluso pueden resultar perjudiciales, por lo que deben ser degradadas durante el desarrollo embrionario (Boerke et al., 2007). El ejemplo más importante de este grupo lo constituirían los transcritos de PRM2, expresados específicamente para la condensación de la cromatina espermática. Su expresión resulta potencialmente dañina para el embrión, por lo que son degradados con rapidez (Ziyyat y Lefèvre, 2001). Otro ejemplo de transcrito englobado en esta categoría es el de la proteína fusogénica GA17, uno de los mRNA más abundantes en espermatozoides maduros. Este transcrito se expresa durante el estadio de espermátida y está involucrado en la interacción espermatozoide-ovocito; su función tras la fecundación se desconoce (Miller y Ostermeier, 2006).
En el segundo grupo estarían incluidos los mRNA sintetizados de forma específica en las espermátidas y que sí muestran una función potencial dentro del desarrollo del cigoto. Esta población de mRNA se mantiene estable al menos hasta la activación del genoma embrionario y codifica proteínas involucradas en procesos de respuesta al estrés, embriogénesis, morfogénesis e implantación, aunque sus implicaciones aún necesitan demostrarse experimentalmente con más detalle (Boerke et al., 2007). El mRNA AKAP-4 (A-kinase anchoring protein 4) sería un ejemplo de este grupo: esta proteína de anclaje es necesaria durante la diferenciación de las espermátidas, para la formación del flagelo y su movilidad, pero también está involucrada en diversas cascadas de señalización y es importante para la activación inicial del ovocito después de la fecundación (Appert-collin et al., 2006; Boerke et al., 2007). PLC-Z, cuyo mensajero también es específico de las espermátidas, ejerce un papel en la embriogénesis temprana al desencadenar un incremento de la concentración de Ca2+ en el ovocito fecundado, lo que causa que complete la meiosis y se produzca la activación del cigoto (Saunders et al., 2007). Igualmente, dentro de este grupo también cabe mencionar a los mRNA de FOXG1B (forkhead box G1, importante en el desarrollo embrionario temprano), a la molécula de señalización WNT5A (involucrada en la diferenciación celular asociada al desarrollo morfológico), a la proteína de choque térmico HSBP-1, al factor de transcripción STAT 4 (posible modulador de la transcripción del pronúcleo masculino) y a la ciclina B1 (promueve el avance de la fase G2 a la M en el ciclo celular) (Boerke et al., 2007).
El tercer grupo abarca los mRNA que no son sintetizados dentro de las células espermáticas en sus distintos estadios, sino que son incorporados al interior del espermatozoide (Boerke et al., 2007). El plasma seminal contiene una alta concentración de RNA extracelular proveniente de distintos tejidos y tipos celulares, en comparación con otros fluidos corporales. En su mayoría, este RNA se encuentra almacenado en vesículas extracelulares, ya sean exosomas (originadas de forma endosomal y liberadas al exterior) o microvesículas (originadas por evaginación, por lo que están cubiertas por parte de su membrana plasmática) (Raposo y Stoorvogel, 2013). En el plasma seminal, la mayoría del RNA extracelular permanece en microvesículas de menos de 1μm de tamaño, divididas principalmente en epididimosomas (provenientes del epidídimo) y prostasomas (procedentes de la próstata) (Li et al., 2012). Hay hipótesis que sostienen que tanto las microvesículas como los exosomas intervienen en la transmisión de transcritos extracelulares al interior del espermatozoide (Valadi et al., 2007). El ejemplo más conocido de entre estos transcritos es el de la clusterina o SGP2, expresada sobre todo en el epidídimo y la próstata, que está involucrada con la maduración espermática. En el contexto de su incorporación al ovocito, cabe resaltar su importancia en la interacción entre el espermatozoide y la zona pelúcida, y en otros procesos relacionados con el desarrollo embrionario (Boerke et al., 2007).
Además del conjunto de mRNA transmitidos del espermatozoide al ovocito, diversos estudios indican que los miRNA, los endo-siRNA y los piRNA provenientes del espermatozoide llevan a cabo una regulación de la expresión génica durante el desarrollo del cigoto. Se cree que los miRNA y endo-siRNA espermáticos, al igual que los mRNA, son liberados en el ovocito y permanecen intactos hasta el comienzo de la activación del genoma embrionario (Boerke et al., 2007). Por ejemplo, se ha descubierto que, en ovocitos de ratones en metafase ii, se expresan diversos mRNA diana de miRNA presentes exclusivamente en espermatozoides maduros (Amanai et al., 2006). De hecho, la influencia epigenética ejercida por los miRNA es un foco de creciente interés científico. Uno de estos mecanismos mejor descritos de modificación epigenética en embriones por parte de los miRNA es la introducción de paramutaciones. Estas siguen una herencia no mendeliana y se producen como consecuencia de una alteración epigenética de un alelo (denominado «paramutagénico») que se transfiere a otro alelo (denominado «paramutable») y modifican su expresión génica. El alelo paramutable mantiene las alteraciones epigenéticas en las siguientes generaciones, aun cuando el alelo paramutagénico ya no está presente (Pilu, 2011).
El RNA espermático como biomarcador de fertilidadLa infertilidad es un problema que afecta aproximadamente a un 15% de las parejas en edad reproductiva. Dentro de la población infértil, aproximadamente el 50% de los casos de infertilidad son debidos a factores masculinos. A pesar de los avances experimentados en la última década en el desarrollo e implementación de técnicas diagnósticas, un porcentaje significativo de individuos presentan infertilidad de origen idiopático (Dohle et al., 2005).
Para desenmascarar estos casos, diversos autores han optado por el análisis del transcriptoma de los espermatozoides. La necesidad de identificar biomarcadores de infertilidad dentro del transcriptoma espermático constituye, por tanto, un campo de creciente interés dado que supone una buena oportunidad de investigación y diagnóstico más allá de la comprobación de los parámetros seminales observables (recuento espermático, movilidad, o morfología), cuyo valor pronóstico no siempre es fiable (Anton y Krawetz, 2012).
Ciertos RNA espermáticos han demostrado presentar una expresión diferencial y específica en poblaciones de individuos infértiles, por lo que su posible rol como biomarcadores supone el foco de atención dentro de esta área de investigación (Anton y Krawetz, 2012). Los transcritos de las protaminas 1 y 2 (PRM1 y PRM2) han sido los más estudiados hasta el momento y la mayoría de los resultados han revelado alteraciones en su expresión con una clara asociación con la infertilidad (Aoki et al., 2006; Rogenhofer et al., 2013; Steger et al., 2008). Además de las protaminas, otros transcritos como el del regulador de la apoptosis BCL2 (B cell lymphoma 2) también han mostrado una expresión diferencial (sobreexpresión) en individuos infértiles (Steger et al., 2008), mientras que los de la glicoproteína PSG1 (pregnancy-specific b-1-glycoprotein 1) y el HLA-E (human leukocyte antigen-E) muestran una expresión diferencial menor a la normal (Avendaño et al., 2009). Lo mismo sucede con el mRNA de la proteína de choque térmico HSPA2 (heat-shock protein A2) (Motiei et al., 2013) (tabla 1).
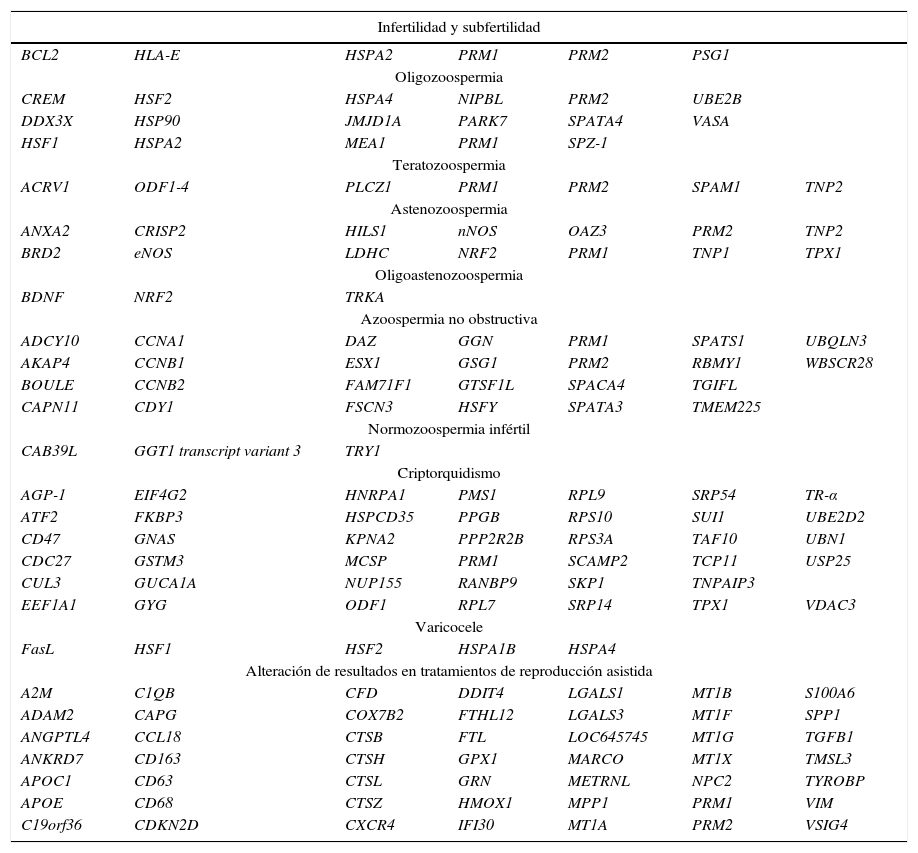
Relación de los mRNA espermáticos humanos descritos hasta la fecha como posibles biomarcadores de diferentes alteraciones relacionadas con la infertilidad
| Infertilidad y subfertilidad | ||||||
|---|---|---|---|---|---|---|
| BCL2 | HLA-E | HSPA2 | PRM1 | PRM2 | PSG1 | |
| Oligozoospermia | ||||||
| CREM | HSF2 | HSPA4 | NIPBL | PRM2 | UBE2B | |
| DDX3X | HSP90 | JMJD1A | PARK7 | SPATA4 | VASA | |
| HSF1 | HSPA2 | MEA1 | PRM1 | SPZ-1 | ||
| Teratozoospermia | ||||||
| ACRV1 | ODF1-4 | PLCZ1 | PRM1 | PRM2 | SPAM1 | TNP2 |
| Astenozoospermia | ||||||
| ANXA2 | CRISP2 | HILS1 | nNOS | OAZ3 | PRM2 | TNP2 |
| BRD2 | eNOS | LDHC | NRF2 | PRM1 | TNP1 | TPX1 |
| Oligoastenozoospermia | ||||||
| BDNF | NRF2 | TRKA | ||||
| Azoospermia no obstructiva | ||||||
| ADCY10 | CCNA1 | DAZ | GGN | PRM1 | SPATS1 | UBQLN3 |
| AKAP4 | CCNB1 | ESX1 | GSG1 | PRM2 | RBMY1 | WBSCR28 |
| BOULE | CCNB2 | FAM71F1 | GTSF1L | SPACA4 | TGIFL | |
| CAPN11 | CDY1 | FSCN3 | HSFY | SPATA3 | TMEM225 | |
| Normozoospermia infértil | ||||||
| CAB39L | GGT1 transcript variant 3 | TRY1 | ||||
| Criptorquidismo | ||||||
| AGP-1 | EIF4G2 | HNRPA1 | PMS1 | RPL9 | SRP54 | TR-α |
| ATF2 | FKBP3 | HSPCD35 | PPGB | RPS10 | SUI1 | UBE2D2 |
| CD47 | GNAS | KPNA2 | PPP2R2B | RPS3A | TAF10 | UBN1 |
| CDC27 | GSTM3 | MCSP | PRM1 | SCAMP2 | TCP11 | USP25 |
| CUL3 | GUCA1A | NUP155 | RANBP9 | SKP1 | TNPAIP3 | |
| EEF1A1 | GYG | ODF1 | RPL7 | SRP14 | TPX1 | VDAC3 |
| Varicocele | ||||||
| FasL | HSF1 | HSF2 | HSPA1B | HSPA4 | ||
| Alteración de resultados en tratamientos de reproducción asistida | ||||||
| A2M | C1QB | CFD | DDIT4 | LGALS1 | MT1B | S100A6 |
| ADAM2 | CAPG | COX7B2 | FTHL12 | LGALS3 | MT1F | SPP1 |
| ANGPTL4 | CCL18 | CTSB | FTL | LOC645745 | MT1G | TGFB1 |
| ANKRD7 | CD163 | CTSH | GPX1 | MARCO | MT1X | TMSL3 |
| APOC1 | CD63 | CTSL | GRN | METRNL | NPC2 | TYROBP |
| APOE | CD68 | CTSZ | HMOX1 | MPP1 | PRM1 | VIM |
| C19orf36 | CDKN2D | CXCR4 | IFI30 | MT1A | PRM2 | VSIG4 |
Otras líneas de investigación persiguen la identificación de biomarcadores de alteraciones seminales específicas. Por ejemplo, en poblaciones de individuos con oligozoospermia se ha hallado una expresión alterada de los mRNA de, entre otros, HSPA2 (Cedenho et al., 2006), del receptor tirosina cinasa TRKA (Li et al., 2010) o de la familia de genes VASA (Guo et al., 2007) (tabla 1).
Respecto a los biomarcadores de teratozoospermia, los principales candidatos hallados han sido los mRNA de PRM1 y PRM2 (Savadi-Shiraz et al., 2015), PLC-Z1, de las proteínas acrosomales ACRV1 (acrosomal vesicle protein 1) y SPAM1 (sperm adhesion molecule 1), del componente de la cola del espermatozoide ODF1-4 (outer dense fiber of sperm tails 1-4) (Platts et al., 2007) y de TP2 (Savadi-Shiraz et al., 2015) (tabla 1).
Contemplando la astenozoospermia, se ha descrito que la mayoría de los mRNA expresados de forma diferencial en individuos con movilidad espermática reducida codifican proteínas involucradas en diferentes procesos del desarrollo o de la funcionalidad de los espermatozoides. Por ejemplo, los mRNA de la anexina A2 (ANXA2), el regulador transcripcional BRD2 (bromodomain-containing protein 2) o la enzima OAZ3 (ornithine decarboxylase antizyme 3) han sido propuestos como biomarcadores en estos casos (Jodar et al., 2012). Una vez más, también se ha hallado una relación directa entre los niveles de expresión de PRM1 y PRM2 y la movilidad espermática (Kempisty et al., 2007; Lambard et al., 2004). Otros ejemplos son los transcritos codificantes de HILS1, proteína específica de espermatozoides, o de las proteínas TP1 y TP2, vitales durante la espermatogénesis (Jedrzejczak et al., 2007). Aparte de estos biomarcadores, otros estudios han hallado una expresión alterada de otros genes relacionada con alteraciones de la movilidad, como TPX-1 (testis-specific protein), LDHC (lactate dehydrogenase C), eNOS (endothelial nitric oxide synthase) y nNOS (neuronal nitric oxide synthase) (Lambard et al., 2004; Wang et al., 2004) (tabla 1).
Los estudios de expresión en espermatozoides y células germinales recuperados de tejido testicular de individuos azoospérmicos han desvelado también una mayor expresión de los mRNA de PRM1 y PRM2 en dichas células (Haraguchi et al., 2009; Song et al., 2000). Otros ejemplos de biomarcadores detectados en biopsias testiculares o en muestras de fluido seminal en casos de azoospermia no obstructiva han sido los transcritos de los homeobox ESX1 (extraembryonic, spermatogenesis, homeobox 1 homolog) y TGIFLX (X-linked TGIF-like protein) (Aarabi et al., 2008; Pansa et al., 2014), así como los mRNA de los genes DAZ (deleted in azoospermia) (Aslani et al., 2011; Kuo et al., 2004). Otros estudios se han centrado en hallar transcritos con valor pronóstico para una buena recuperación de espermatozoides a partir de biopsia testicular en pacientes azoospérmicos, como son los de las ciclinas A1, B1 y B2 (Haraguchi et al., 2009), los mRNA de los genes CDY1 (chromodomain Y1) y BOULE (perteneciente a la familia de genes DAZ) (Kleiman et al., 2011) y el mRNA de HSFY (heat shock factor Y chromosome) (Stahl et al., 2011) (tabla 1).
Por otro lado, aunque de forma menos exhaustiva, también se han descrito algunos candidatos a biomarcadores en los casos de infertilidad idiopática en individuos normozoospérmicos, como son los mRNA de la tripsina TRY1, la variante 3 del transcrito de la glutamiltransferasa GGT1 y el mRNA de la proteína CAB39L (calcium binding protein 39 like) (Garrido et al., 2009) (tabla 1).
Englobando otro tipo de enfermedades relacionadas con la infertilidad, también se han logrado identificar diversos biomarcadores de criptorquidismo (Nguyen et al., 2009) o de varicocele (Del Giudice et al., 2010; Ferlin et al., 2010; Ji et al., 2014; Ni et al., 2014). Aparte de estas alteraciones, otros estudios de expresión se han centrado en identificar biomarcadores específicos de otros parámetros como son el éxito obtenido en los tratamientos de reproducción asistida (Depa-Martynów et al., 2007; Garcia-Herrero et al., 2011) (tabla 1).
Por otro lado, y aunque aún de forma mucho más incipiente, algunos autores también han propuesto la evaluación de los miRNA espermáticos como una nueva fuente de posibles biomarcadores. Este planteamiento se basa en la gran importancia que poseen dichos sncRNA dentro del proceso de regulación de la estabilidad de los mRNA. Abu-Halima et al. realizaron un análisis comparativo mediante microarrays entre controles fértiles e individuos astenozoospérmicos y oligoastenozoospérmicos; como resultado, se encontraron una serie de miRNA sobreexpresados (50 en astenozoospérmicos y 42 en oligoastenozoospérmicos) y otros con expresión reducida (27 en astenozoospérmicos y 44 en oligoastenozoospérmicos) (Abu-Halima et al., 2013). Posteriormente, se realizó la validación de 5 miRNA concretos (miR-34b*, miR-34b, miR-34c-5p, miR-429 y miR-122) mediante qRT-PCR, se confirmó su expresión diferencial en individuos infértiles y se proponen como posibles biomarcadores de las alteraciones diagnosticadas (Abu-Halima et al., 2014). Zhou et al. se centraron en la astenozoospermia, y proponen el miR-27b como biomarcador al regular directamente al mRNA de CRISP2 (cysteine-rich secretory protein 2), poco expresado en individuos astenozoospérmicos (Zhou et al., 2015). Salas-Huetos et al. por otro lado, compararon los perfiles de individuos fértiles normozoospérmicos (Salas-Huetos et al., 2014) con los de pacientes con diferentes anomalías seminales puras, e identificaron variaciones en la expresión de miRNA en individuos astenozoospérmicos, teratozoospérmicos, oligozoospérmicos (Salas-Huetos et al., 2015) y normozoospérmicos infértiles (Salas-Huetos et al., 2016). Además, Ji et al. identificaron una expresión diferencial del miR-15a en pacientes con varicocele (Ji et al., 2014).
Consideraciones finalesDurante el transcurso de las investigaciones acerca del RNA espermático, la idea de que el espermatozoide es un mero vehículo del genoma paterno ha sido sustituida por una visión más profunda y detallada del funcionamiento y papel biológico de su transcriptoma. Se ha demostrado que su función no está limitada únicamente a transmitir la fracción codificante, sino que existe un complejo sector de ncRNA que participa en importantes mecanismos de regulación y procesos celulares. El RNA espermático no solo supone una huella residual del transcriptoma involucrado en la espermatogénesis, sino que también permite prever las características de la futura aportación transcriptómica al cigoto y anticipar en parte cómo este factor paterno afectará al desarrollo embrionario. Esta aportación extragenómica supone un foco de atención para investigaciones futuras: tal vez, haya que hacer especial hincapié en la huella epigenética mediada por RNA que conlleva la incorporación de ciertos transcritos espermáticos al embrión. Las investigaciones en este campo podrían llegar a aplicarse para mejorar el pronóstico de la calidad embrionaria durante tratamientos de reproducción asistida. Por otro lado, los avances en las tecnologías de estudio de la expresión transcriptómica a gran escala están abriendo las puertas a nuevos frentes en el ámbito de los biomarcadores de fertilidad y están permitiendo ahondar en su aplicación de forma más detallada. Los hallazgos en esta área dejan paso a potenciales aplicaciones futuras en el ámbito clínico, para el diagnóstico molecular de la infertilidad masculina. Como visión general, se estima que la investigación en un campo tan complejo como es el RNA espermático generará nuevos descubrimientos teóricos y mejoras en sus posibles aplicaciones prácticas.
Responsabilidades éticasProtección de personas y animalesLos autores declaran que para esta investigación no se han realizado experimentos en seres humanos ni en animales.
Confidencialidad de los datosLos autores declaran que en este artículo no aparecen datos de pacientes.
Derecho a la privacidad y consentimiento informadoLos autores declaran que en este artículo no aparecen datos de pacientes.
Conflicto de interesesTodos los autores declaran no tener ningún tipo de conflicto de intereses.