La epigenética es el estudio de los cambios heredables en el ADN sin afectar a las secuencia de nucleótidos. Entre los mecanismos de regulación epigenética, los más estudiados y conocidos hasta la fecha son la metilación del ADN, la modificación de las histonas y los ARN no codificantes. Mediante estos mecanismos se regula la expresividad génica y la alteración de los mismos puede llevar al desarrollo de patologías.
DesarrolloDescribimos los principales mecanismos de regulación epigenética y realizamos una revisión de la bibliografía reciente sobre los mecanismos de regulación epigenética y su implicación en distintos síndromes epilépticos.
ConclusiónLa identificación de los mecanismos epigenéticos implicados en la epilepsia constituye una prometedora vía de investigación para profundizar en el conocimiento de la fisiopatología y terapéutica de esta enfermedad.
Epigenetics is the study of heritable modifications in gene expression that do not change the DNA nucleotide sequence. Some of the most thoroughly studied epigenetic mechanisms at present are DNA methylation, post-transcriptional modifications of histones, and the effect of non-coding RNA molecules. Gene expression is regulated by means of these mechanisms and disruption of these molecular pathways may elicit development of diseases.
DevelopmentWe describe the main epigenetic regulatory mechanisms and review the most recent literature about epigenetic mechanisms and how those mechanisms are involved in different epileptic syndromes.
ConclusionIdentifying the epigenetic mechanisms involved in epilepsy is a promising line of research that will deliver more in-depth knowledge of epilepsy pathophysiology and treatments.
El término epigenética se atribuye a Conrad Waddington, quien en el año 1942 lo definió como «la rama de la biología que estudia las interacciones causales entre los genes y sus productos que dan lugar al fenotipo»1. Hoy en día es un área en plena expansión, cuyo estudio está dando resultados prometedores en el campo de la medicina.
De un modo sencillo, podemos definir la epigenética como el estudio de los cambios heredables en la expresión génica que se producen sin modificaciones en la secuencia del ADN2. Los procesos epigenéticos, por tanto, podrían ser entendidos como aquellos mecanismos que regulan la expresión de los genes, determinando no solo la expresión o el silenciamiento, sino también, dónde, cuándo y en qué intensidad se expresan.
Existe evidencia de que estos procesos epigenéticos pueden ser modificados por factores físicos, químicos, nutricionales, incluso psicosociales, de modo que el ambiente en el que nos desenvolvemos y nuestros hábitos de vida, son capaces de modificar nuestra expresión génica a través de los mecanismos epigenéticos3,4.
La epigenética puede ayudarnos a dar respuestas a preguntas que todavía están por resolver, como ¿por qué 2 individuos con igual carga genética, como los gemelos univitelinos, tienen apariencia, comportamientos y modo de enfermar, entre otros, distintos?5, o ¿de qué manera influye el ambiente en el genoma?
Mecanismos de regulación epigenética en epilepsiaMecanismos epigenéticos básicosActualmente, son varios los mecanismos epigenéticos conocidos y, aunque están apareciendo algunos nuevos o surgiendo acciones nuevas de los ya conocidos, los más estudiados son 3: la metilación del ADN, la modificación de las histonas y la acción de los ARN no codificantes.
Metilación del ADNEs el mecanismo epigenético más ampliamente estudiado. Consiste en la adición de un grupo metilo al carbono 5 de la citosina y ocurre en las citosinas que son seguidas de guaninas (dinucleótidos CpG). Estos no se encuentran distribuidos de manera uniforme en el genoma, existiendo regiones donde abundan, las denominadas islas CpG, que se localizan preferentemente en las regiones promotoras de los genes. Las islas CpG se encuentran generalmente no metiladas; sin embargo, algunos procesos fisiológicos requieren de la metilación de estas regiones, como, por ejemplo, la impronta genética o la inactivación del cromosoma X en las mujeres. Por su parte, el resto de dinucleótidos CpG no incluidos en las islas CpG se encuentran metilados y se localizan principalmente en secuencias repetitivas o regiones centroméricas. La metilación es llevada a cabo por las ADN metiltransferasas (DNMT) que catalizan la transferencia de un grupo metilo de la S-adenosil-L-metionina al carbono 5 de la citosina, conociéndose en el mamífero 5 tipos, las DNMT1, DNMT2, DNMT3A, DNMT3B y DNMT3L6. Para este proceso resulta de vital importancia el correcto mantenimiento del metabolismo de la vitamina B12, el ácido fólico y la homocisteína.
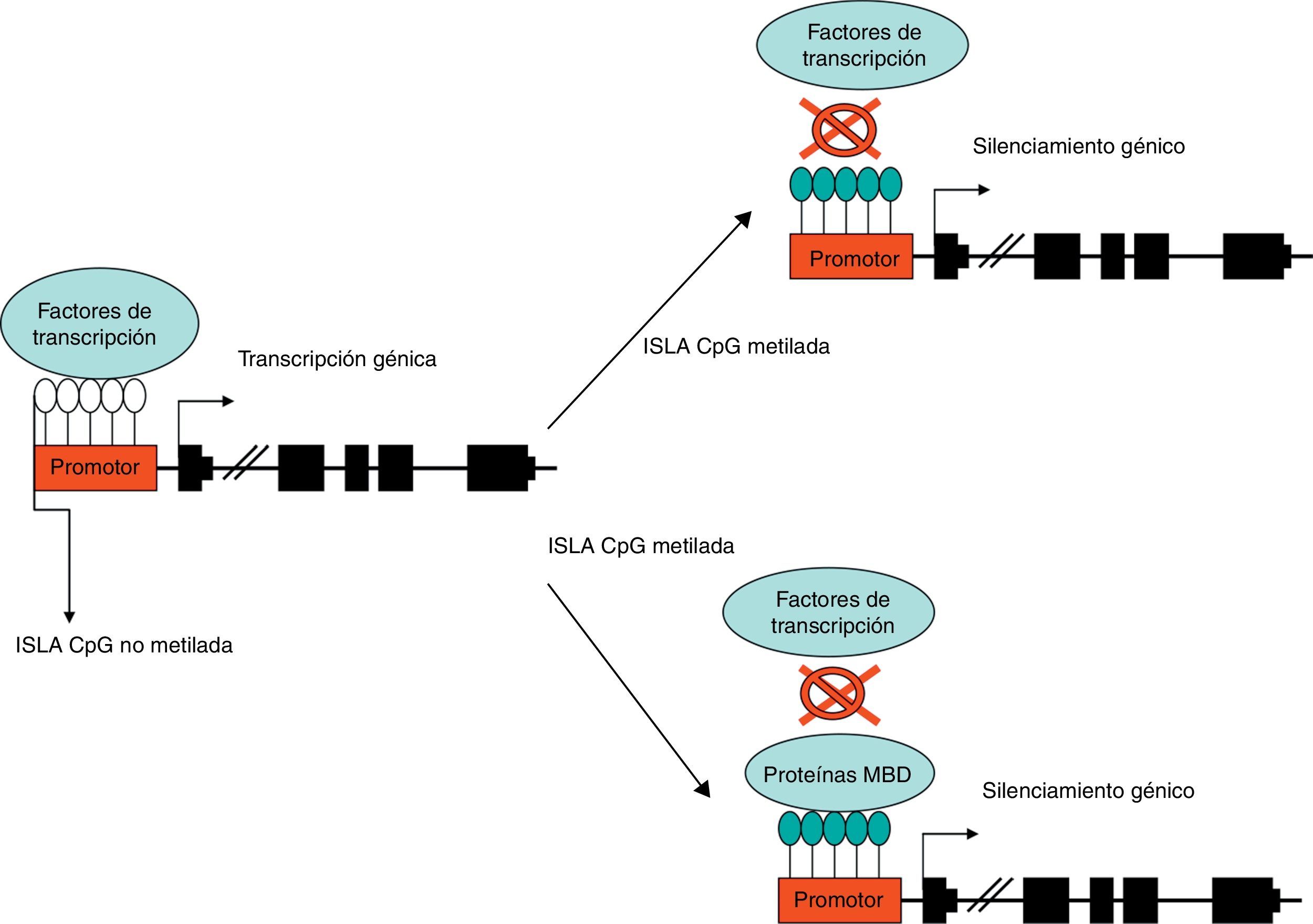
La metilación de la región promotora de los genes se asocia generalmente a la inhibición de la transcripción, es decir, el silenciamiento génico. La metilación bloquea la transcripción básicamente mediante 2 mecanismos. El primero de ellos impide la unión de factores reguladores de la transcripción que contengan CpG en sus sitios de reconocimiento. El segundo involucra a complejos proteicos que se unen específicamente a regiones CpG metiladas, bloqueando indirectamente la unión de factores de transcripción al limitar el acceso de elementos reguladores6. Estos complejos proteicos son las denominadas Methyl-CpG binding domain (MBD) y en los mamíferos se conocen 5 familias: MeCP2, MBD1, MBD2, MBD3 y MBD4. De este modo, la metilación del ADN regula de manera indirecta la estructura de la cromatina y su accesibilidad a los factores de transcripción7 (fig. 1).
Metilación del ADN en isla CpG situada en la región promotora de un gen. La metilación induce el bloqueo de la transcripción bien al impedir la unión de factores de transcripción de manera directa, o de manera indirecta por medio de la unión de proteínas reguladores con methylbinding domain (MBD).
El ADN nuclear está asociado a un complejo multiproteico formando la cromatina. El nucleosoma es la unidad estructural básica de la cromatina y consiste en 146 pares de bases de ADN enrollado en un octámero de proteínas denominadas histonas (un tetrámero H3/H4, 2 dímeros H2A y H2B y una molécula H1). Las histonas, además de ofrecer soporte estructural, regulan la accesibilidad de los factores de transcripción y, por tanto, regulan la expresión génica. Esto se consigue por medio de modificaciones epigenéticas que tienen lugar en cada una de las colas amino-terminal que se extienden fuera del nucleosoma, principalmente en los extremos amino-terminal de H3 y H4. Estas modificaciones incluyen la metilación, la acetilación, la fosforilación, la ubiquitinación y la ADP ribosilación8. De ellos, el mejor estudiado es la acetilación de los residuos de lisina. El estado de acetilación de las histonas H3 y H4 incrementa la expresión génica al promover la estructura abierta de la cromatina; por su parte, el estado de hipoacetilación es característico de zonas del genoma transcripcionalmente inactivas9 (fig. 2). Este proceso es llevado a cabo por la acetilasa de histonas y revertido por la desacetilasa de histonas (HDAC). Otra modificación que también tiene lugar es la metilación, proceso que ocurre por acción de la metiltransferasa y por la desmetiltransferasa de histonas. El efecto de esta modificación depende de la identidad del residuo modificado y del grado de metilación7. Todas estas modificaciones constituyen un verdadero código que se ha dado en llamar el código de las histonas, que media el estado de la cromatina y, por tanto, determina la expresión génica.
Acción de los ARN no codificantesSon pequeñas moléculas de ARN endógeno que no codifican para proteínas. Los micro-ARN (miARN) son un tipo de ARN no codificantes (ARNnc) que participan en la regulación de la expresión génica de manera postranscripcional10. Principalmente, actúan a través de 2 mecanismos, la degradación del ARN mensajero (ARNm) diana o mediante el bloqueo de la traducción (fig. 3). La puesta en marcha de uno u otro mecanismo dependerá del ARNm diana. Se estima que cada miARN puede tener más de una diana y a su vez que cada ARNm puede ser regulado por más de un miARN. Además de los miARN, se han descrito otros tipos de ARNnc sin que actualmente se conozca con certeza su función pero que también pueden estar implicados en mecanismos de regulación epigenética.
Esta separación académica de los principales mecanismos de regulación epigenética hasta ahora conocidos no refleja la verdadera complejidad de la regulación epigenética puesto que, como es de esperar, no son eventos aislados, sino que interactúan entre sí, ejerciendo influencias unos sobre los otros. Así los mecanismos de regulación epigenética, actuando de forma coordinada, desempeñan un papel clave para el normal desarrollo y funcionamiento del organismo.
Interés del estudio de la epigenética de las enfermedadesLa regulación epigenética es de suma importancia para el desarrollo y el correcto funcionamiento del organismo. Las modificaciones en los mecanismos de regulación permiten al organismo adaptarse a las circunstancias de cada momento, de manera que alteraciones en estos patrones de regulación podrían llevar al desarrollo de distintas enfermedades.
Los primeros datos en relacionar las alteraciones en los mecanismos de regulación epigenética y enfermedad provienen del campo de la oncología, concretamente del cáncer colorrectal (CCR). En concreto, se observó una pérdida de metilación global en pacientes con CCR en comparación con controles sanos, de manera que esta hipometilación llevaba a una activación anormal de genes. De igual modo, se observó que existía hipermetilación en varios genes supresores de tumores11. Esta primera incursión ha dado paso a sucesivos estudios en otros campos de la medicina, como las alteraciones del neurodesarrollo y las enfermedades neurodegenerativas12.
Otro aspecto importante de los avances en epigenética es la posibilidad de adentrarse en nuevas terapias, que nos permitan intervenir directamente sobre los cambios epigenéticos asociados a la enfermedad13, encontrándose a día de hoy en ensayo varios de ellos.
Así, la epigenética hoy por hoy constituye un campo en auge sobre el que se depositan esperanzas acerca del mayor conocimiento de múltiples enfermedades y sobre su terapéutica.
Epigenética y enfermedades que cursan epilepsiaLa epilepsia es una de las enfermedades neurológicas crónicas más prevalente donde hasta en el 30% de los pacientes no se consigue una buena respuesta con el tratamiento farmacológico14. Presenta una gran heterogeneidad clínica y fisiopatológica, y, a pesar de los avances realizados en los últimos años con la identificación de varios genes responsables de síndromes epilépticos, la causa de la mayoría de las epilepsias están aun sin aclarar. El estudio de los mecanismos epigenéticos en el campo de la epilepsia está aportando evidencia sobre el papel que las alteraciones de la regulación epigenética tienen en la susceptibilidad, el desarrollo y el mantenimiento de la epilepsia15-18. A continuación, comentaremos algunas de estas evidencias, que a modo didáctico encuadraremos en 3 grupos:
Mutaciones en genes «epigenéticos» que causan epilepsiaCiertas enfermedades neurológicas y defectos del neurodesarrollo están causadas por mutaciones en genes que codifican para proteínas involucradas en la regulación epigenética7,12. Estos desórdenes neurológicos están caracterizados por retraso intelectual, que puede acompañarse de otras características neurológicas, como crisis epilépticas, espectro autista y retraso del desarrollo psicomotor12.
Dentro de este grupo, una de las enfermedades mejor conocidas es el síndrome de Rett, causado por mutaciones en el gen MECP2 que codifica para la subfamilia de MBP, MeCP2. Esta proteína se une a secuencias especificas de ADN metilado llevando a la supresión de la expresión génica. La mutación responsable de este síndrome fue descubierta en 1999 en el gen MECP2 localizado en el cromosoma Xq2819 y, por tanto, presenta un patrón de transmisión genética asociado al sexo, siendo en más del 99% de los casos mutaciones de novo.
La enfermedad afecta a niñas que, tras un desarrollo normal hasta los 6-18 meses, comienzan con una pérdida progresiva de las capacidades adquiridas, con una desaceleración del crecimiento, pérdida del lenguaje y desarrollo de estereotipias (muy características el gesto de lavado de manos). La enfermedad es progresiva y lleva a un periodo de autismo donde aparece deterioro cognitivo. Las crisis epilépticas, la hiperventilación y las apneas son también características comunes7. Se describen cifras de epilepsia en torno al 50-80% y se asocian a mayor severidad clínica20. Los tipos de crisis epilépticas que se pueden presentar son muy variados (crisis tónico-clónicas generalizadas, crisis parciales simples o complejas, crisis atónicas, crisis tónicas, crisis fotosensibles), pudiéndose presentar varios tipos en el mismo paciente21. En cuanto al tratamiento de las crisis, no existen fármacos antiepilépticos específicamente recomendados, siendo la carbamacepina en monoterapia o en combinación con clobazam el tratamiento más eficaz22.
Alteración de los patrones de metilación. Defectos en la impronta genómicaComo comentábamos con anterioridad, los dinucleótidos CpG localizados en regiones promotoras se encuentran de manera general no metilados, pero de forma fisiológica se encuentran metilados en los casos de inactivación del cromosoma X en las mujeres y en los casos de impronta genómica7.
La impronta genómica es un mecanismo de gran importancia, que consiste en que determinadas regiones cromosómicas solo expresan el alelo génico proveniente de uno de los progenitores, mientras que el otro alelo permanece silenciado mediante un mecanismo epigenético. La impronta no afecta a la totalidad del genoma, sino que se limita a ciertas regiones. Los genes improntados tienden a agruparse en grupos controlados por elementos controladores de impronta, constituidos por regiones ricas en dinucleótidos CpG y que son regulados por metilación de uno de los alelos23. Las alteraciones en este proceso llevan a la inapropiada expresión génica que puede ocasionar alteraciones en el desarrollo normal, pudiendo ser el origen de ciertas enfermedades12.
Un ejemplo de trastorno neurodegenerativo que cursa con epilepsia producido por alteraciones de la impronta es el síndrome de Angelman (SA). En 1987, Magendis et al. identificaron una deleción del cromosoma 15q 11-13 en 2 pacientes con SA24. En posteriores trabajos se ha visto que el SA puede ser causado por una variedad de mecanismos genéticos, como deleciones, disomía uniparental paterna o defectos de impronta, afectando todos ellos a la expresión del alelo materno del gen UBE3A. Presenta un espectro clínico amplio que incluye retraso intelectual, afectación del lenguaje que puede llegar a ser severa, ataxia y esterotipias (realizan un aleteo de las manos al andar), crisis epilépticas, dismorfismos faciales y trastornos del comportamiento con un comportamiento feliz inapropiado y ocasionales risas no motivadas o de fácil provocación25. Las primeras evidencias del síndrome surgen entre los 6-12 meses de vida cuando se comienzan apreciar cierto retraso en el desarrollo psicomotor; no obstante, no se objetiva ninguna pérdida evidente en las habilidades del niño, por lo que generalmente se llega al diagnóstico cuando los síntomas se hacen más evidente, en torno a los 3-7 años26. En cuanto a las crisis epilépticas, se presentan hasta en el 80% de los casos25, suelen comenzar entre el año y los 5 años, y suelen hacerlo como crisis febriles. Posteriormente, las crisis observadas son variables, siendo los tipos de crisis más frecuentes las ausencias atípicas, las mioclónicas, las tónico-clónicas generalizadas y las crisis atónicas. El control de las mismas suele ser difícil en la adolescencia y el fármaco más efectivo es el ácido valproico, bien como monoterapia o asociado a clonazepam u otras benzodiacepinas27.
Cambios epigenéticos en epilepsiaAlteraciones en la metilaciónEn los últimos años, han surgido evidencias, estudiando modelos animales y tejidos humanos de pacientes epilépticos, de alteraciones en los patrones de metilación entre los afectados de epilepsia frente a los controles.
Muestra de ello es el trabajo de Kobow et al.28 quienes, estudiando muestras de hipocampo de pacientes con epilepsia del lóbulo temporal y controles, encontraron mayor metilación en el promotor reelin en las muestras de pacientes con epilepsia del lóbulo temporal. La reelin es una proteína extracelular cerebral con importantes funciones en el proceso de migración neuronal y la plasticidad sináptica, y con implicación en el correcto mantenimiento de la estructura laminar de las células granulares del hipocampo. Se sabe que la pérdida de esta estructura en el núcleo dentado del hipocampo (dispersión granular) está presente en hasta un 50% de los pacientes afectados de esclerosis del lóbulo temporal. Estudios previos29 ya habían demostrado la importancia del reelin en la dispersión granular en la epilepsia del lóbulo temporal, pero el trabajo de Kobow et al.28 es el primero que muestra que tras esta se encuentra una alteración de la regulación epigenética.
En esta misma línea de estudio sobre la epilepsia del lóbulo temporal, el grupo de Zhu et al.30 estudió la expresión de las DNMT 1 y 3 a en pacientes con epilepsia del lóbulo temporal frente a controles sanos. Como comentábamos en los primeros apartados, las DNMT son las enzimas encargadas de llevar a cabo la metilación, siendo las DNMT1 encargadas del mantenimiento de los patrones de metilación y las DNMT3 de promover la metilación de novo durante el desarrollo. En el trabajo encuentran que ambas DNMT están incrementadas en los pacientes con epilepsia del lóbulo temporal, por lo que indican un papel de las mismas en la patogénesis de este tipo de epilepsia.
El mismo grupo de Kobow, en un trabajo publicado recientemente31, realiza un análisis de la metilación global del ADN en hipocampo de ratas con epilepsia crónica frente a controles, encontrando un incremento de metilación global en el grupo con epilepsia crónica. Así mismo evalúa el efecto de la administración de una dieta cetogénica y observan, con la misma, una disminución de la frecuencia de crisis y un cambio en el patrón de metilación del ADN. Conviene señalar que este es el primer trabajo que se realiza de manera global sobre la totalidad del ADN.
En otro estudio también realizado en hipocampo (región C3) de ratas, el grupo de Miller-Delaney et al.32 encontró diferente patrón de metilación en el grupo de roedores con estatus epiléptico definido frente a los del grupo con tolerancia al estatus. Este último grupo lo constituían ratones a los que, previamente a inducir el estatus epiléptico, estimulaban la aparición de crisis con quimioconvulsivantes o estímulos eléctricos, de modo que mostraban tolerancia al posterior estatus. Este grupo, además, encuentra que algunas de estas diferencias del perfil de metilación acontecen en genes en los que hasta entonces no estaba descripta su implicación en epilepsia.
Modificación de las histonasEstudios en modelos animales de epilepsia comienzan a mostrar evidencias de cambios en la cromatina, mediado por modificaciones de las histonas, tras las crisis epilépticas.
Una de las primeras evidencias vino del grupo de Huang et al.33, quienes en muestras de hipocampos de ratones analizados tras 3 h de inducción del estatus epiléptico utilizando pilocarpina, encontraron una hipoacetilación H4 (marca de represión génica) en el promotor del receptor de glutamato 2 (GluR2, subunidad del receptor AMPA) y una hiperacetilación (marca de transcripción génica) en el promotor del factor neurotrófico derivado del cerebro). Estos hallazgos ponen de manifiesto que el estatus epiléptico activa de manera rápida la modulación de la acetilación de las histonas. En el mismo estudio observaron que la administración previa de un inhibidor de HDAC prevenía la hiperacetilación del promotor del GluR2.
El mismo autor, en un trabajo reciente34, encuentra una mayor expresión de la HDAC2 en muestras humanas de epilepsia del lóbulo temporal y en modelos animales de estatus epiléptico, en comparación con controles. La HDAC2 es una HDAC expresada en el sistema nervioso central, implicada en el neurodesarrollo. También es esencial para la función cognitiva, dado que interviene en la represión de genes asociados con la plasticidad sináptica y la formación de la memoria35. Los resultados del estudio indican una importante implicación de la HDAC2 en la patogénesis de la epilepsia del lóbulo temporal, así como de la afectación cognitiva que en ocasiones asocia este tipo de epilepsia.
En otro modelo animal de epilepsia, en este caso con crisis electroinducidas, el grupo de Tsankova et al.36 encontró cambios en la acetilación de las histonas H3 y H4 del promotor del CREB, en hipocampo de ratas. El CREB es un importante activador transcripcional implicado, entre otros, en la modulación de la expresión del receptor GABAa en el hipocampo, desempeñando un papel importante en el proceso de epileptogénesis.
Todos estos trabajos ilustran el papel de los mecanismos epigenéticos a través de la modificación de las histonas, en la regulación de genes implicados en el control de la actividad epileptiforme.
Micro-ARN y epilepsiaRecientemente, han surgido varios trabajos sobre el perfil de expresión de los miARN en la epilepsia, con resultados esperanzadores sobre su posible papel como biomarcador.
Ejemplo de esto es el trabajo del grupo de Liu et al.37, quienes estudian el perfil de expresión de miARN en ratas en las que se les induce estatus epiléptico mediante administración de kainato, analizando muestras cerebrales y sanguíneas. Los autores encuentran, para un subconjunto de miARN, un perfil de expresividad similar en sangre y en hipocampo, por lo que apoyan la posibilidad de que sean utilizados como biomarcadores en sangre.
Por otro lado, el estudio de los miARN también está aportando datos acerca del mayor conocimiento sobre el proceso de epileptogénesis. Muestra de ello, en 3 trabajos recientes38-40 también sobre modelos animales, coinciden en el aumento de expresión del miARN-132 en el hipocampo de ratas a las que se les induce estatus epiléptico. Se sabe que el miARN-132 cumple funciones antiinflamatorias y se ha indicado el papel de la inflamación en el desarrollo de la epileptogénesis, por lo que el incremento de miARN-132 podría contribuir al desarrollo de la misma.
ConclusiónLa epigenética y su relación con las enfermedades humanas constituyen una vía de investigación emergente en la que se depositan grandes esperanzas para mejorar el conocimiento de la fisiopatología y la terapéutica de las enfermedades comunes.
En el campo de la epilepsia, el estudio de las modificaciones epigenéticas, tanto en modelos animales como en muestras de tejido humano, está ofreciendo resultados prometedores que profundizan en el conocimiento de la epileptogénesis (tabla 1). Además, la detección de modificaciones epigenéticas compartidas por el compartimento cerebral y sanguíneo abre la puerta a la utilización de estas modificaciones como marcadores epigenéticos que contribuyan a mejorar el diagnóstico, el pronóstico y el tratamiento de los diferentes síndromes epilépticos.
Estudio de las modificaciones epigenéticas en el campo de la epilepsia
| Modelo de epilepsia | Tejido estudiado | Mecanismo epigenético | Referencia |
|---|---|---|---|
| Epilepsia lóbulo temporal en humanos | Hipocampo | Hipermetilación Reelin | Kobow et al.28 |
| Epilepsia lóbulo temporal en humanos | Córtex temporal | Diferente expresión DNMT 1 y 3A | Zhu et al.30 |
| Ratas. Crisis inducidas por pilocarpina | Hipocampo | Diferente perfil de metilación global | Kobow et al.31 |
| Ratas. Status inducido por kainato | Hipocampo | Diferente perfil de metilación global | Miller-Delaney et al.32 |
| Ratas. Crisis inducidas por pilocarpina | Hipocampo | Hipoacetilación H4 GluR2Hiperacetilación H4 BDNF | Huang et al.33 |
| Ratas. Crisis inducidas por pilocarpina. Epilepsia lóbulo temporal humanos | Córtex temporal | Diferente expresión HDAC2 | Huang et al.34 |
| Ratas. Crisis electroinducidas | Hipocampo | Hipoacetilación H4 CREBHiperacetilación H3 CREB | Tsankova et al.36 |
| Ratas. Crisis inducida por kainato | Hipocampo, sangre periférica | miARN | Liu et al.37 |
| Ratas. Status epiléptico | Hipocampo | Aumento expresión miARN 132 | Hu et al.38, Jimenez-Mateo et al.39, Song et al.40 |
Estos avances contribuirán de manera notable a investigar sobre nuevas maneras de afrontar esta patología neurológica que, actualmente, todavía tiene repercusiones sanitarias, familiares y sociales muy elevadas.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.