Anaerobiospirillum thomasii ha sido descrito como causante de diarrea en humanos, pero no se han informado bacteriemias asociadas a este organismo. En esta comunicación describimos el primer aislamiento de A. thomasii como causa de bacteriemia fatal en una paciente alcohólica.
Anaerobiospirillum thomasii has been reported as a causative agent of diarrhea in humans; however no bacteremia associated with this pathogen has been described so far. We present here the first case of fatal A. thomasii bacteremia in an alcoholic patient.
El género Anaerobiospirillum consiste en un grupo de bacilos gram negativos anaerobios espiralados, que comúnmente forma parte de la microbiota habitual intestinal de gatos, perros y humanos3,7. En 1976, Davis et al.1 aíslan por primera vez al microorganismo conocido como Anaerobiospirillum succiniciproducens, hasta ese momento el único miembro del género Anaerobiospirillum. En 1997, Malnick8 propone Anaerobiospirillum thomasii como un nueva especie dentro de este género. Ocasionalmente, estos bacilos pueden infectar a humanos. En una revisión bibliográfica se encontraron 56 casos de infección en humanos5. Ambas especies fueron aisladas en materia fecal de pacientes con diarrea4,7.
A. succiniciproducens ha sido descrito como agente causal de bacteriemias, generalmente en pacientes con algún grado de inmunodepresión5,9-11. No existen casos descritos de bacteriemias por A. thomasii.
Se presenta el caso de una paciente de sexo femenino de 54 años, con antecedentes de enolismo de 30 años de evolución, fumadora, con internaciones previas por síndrome ascítico edematoso y poca adherencia al tratamiento. La paciente refirió haber comenzado 2 semanas antes de la consulta con aumento del perímetro abdominal, astenia, anorexia y somnolencia. Debido a una mala evolución se decidió la internación. A la admisión, la paciente se presentó afebril, con hipotensión, desorientación, abdomen globoso y depresible, y abundante catarsis diarreica. El examen de sangre mostró valores de urea, creatinina y electrolitos normales, hiperbilirrubinemia (6,7mg%), hipoalbuminemia (23g/l) e hiperamonemia (230μg%), hematocrito 27%, hemoglobina 8,7g/dl, recuento de blancos 17 000 por mm3, con 88% de neutrófilos, velocidad de sedimentación globular de 50mm/h. Se tomaron 2 muestras de hemocultivo en frascos BacT/Alert FA (FAN Aerobic) y BacT/Alert FN (FAN Anaerobic) de bioMèrieux antes del inicio del tratamiento con ampicilina sulbactama 1,5g/6h. La paciente continuó con mala evolución y falleció al cuarto día de internación.
Se detectó crecimiento bacteriano en las botellas anaeróbicas al segundo día de incubación usando el sistema de detección de microorganismos BacT/ALERT® 3D60 (bioMèrieux). La coloración de Gram mostró la presencia de bacilos gram negativos con forma espiralada. El organismo se subcultivó en agar sangre y agar chocolate incubados a 35°C en 5% de CO2, y en agar Brucella suplementado con vitamina K y hemina, incubado a 35°C en condiciones anaeróbicas (cámara AnaeroPouch System con generador AnaeroPouch-Anaero, Mitsubishi Gas Chemical Company, Inc.). Todos los subcultivos fueron negativos después de 10 días de incubación, por lo que se decidió realizar la identificación molecular de la bacteria por secuenciación del gen del ARN ribosomal 16S a partir de los frascos originales de FAN Anaerobic.
Se procedió a la extracción de ADN directamente a partir de la botella de hemocultivo. Se tomaron 400μl de cada uno de los frascos anaeróbicos. El ADN se purificó utilizando un sistema automatizado MagnaPure Compact (Roche), con un volumen final de elución de 50μl.
El gen del ARNr 16S se amplificó mediante el empleo de los primers universales 16S forward primer y DG74 reverse primer, previamente descritos2,6. El producto obtenido fue de aproximadamente 1200 pb, cubriendo desde la región variable V3 a la V9. La PCR se realizó en un termociclador Veriti (Applied Biosystems) usando los siguientes parámetros de ciclado: 95°C 15min, 30 ciclos de 94°C 30 s, 55°C 30 s y 72°C 1min, y una extensión final de 72°C por 10min. La amplificación se llevó a cabo usando el HotStarTaq Plus Master Mix Kit (QIAGEN), con una concentración final de la master mix de 1×, 0,5-5ng de ADN y 0,5μM de cada uno de los primers, en un volumen final de 25μl.
Los productos de PCR se corrieron en un gel de agarosa al 1% (Agarosa LE grado analítico, Promega) y las bandas se visualizaron al UV por tinción con bromuro de etidio.
Finalmente, los productos fueron purificados con el Qiaquick PCR purification kit (Qiagen) y cuantificados con un espectrofotómetro NanoDrop 1000 (Thermo Scientific).
El producto de PCR purificado se secuenció en ambos sentidos usando el kit BigDye Terminator V1.1 de Applied Biosystem en un volumen final de 20μl, con una dilución de la master mix de 0,125×, 1μM del primer 16S y DG74 y 15ng de producto de PCR. Los parámetros de ciclado fueron: 96°C 1min, 30 ciclos de 96°C 10 s, 50°C 5 s y 60°C 4min. Los productos de secuenciación se corrieron en un secuenciador automático ABI 3500 de Applied Biosystem y se analizaron mediante el programa Sequencing Analysis v5.4.
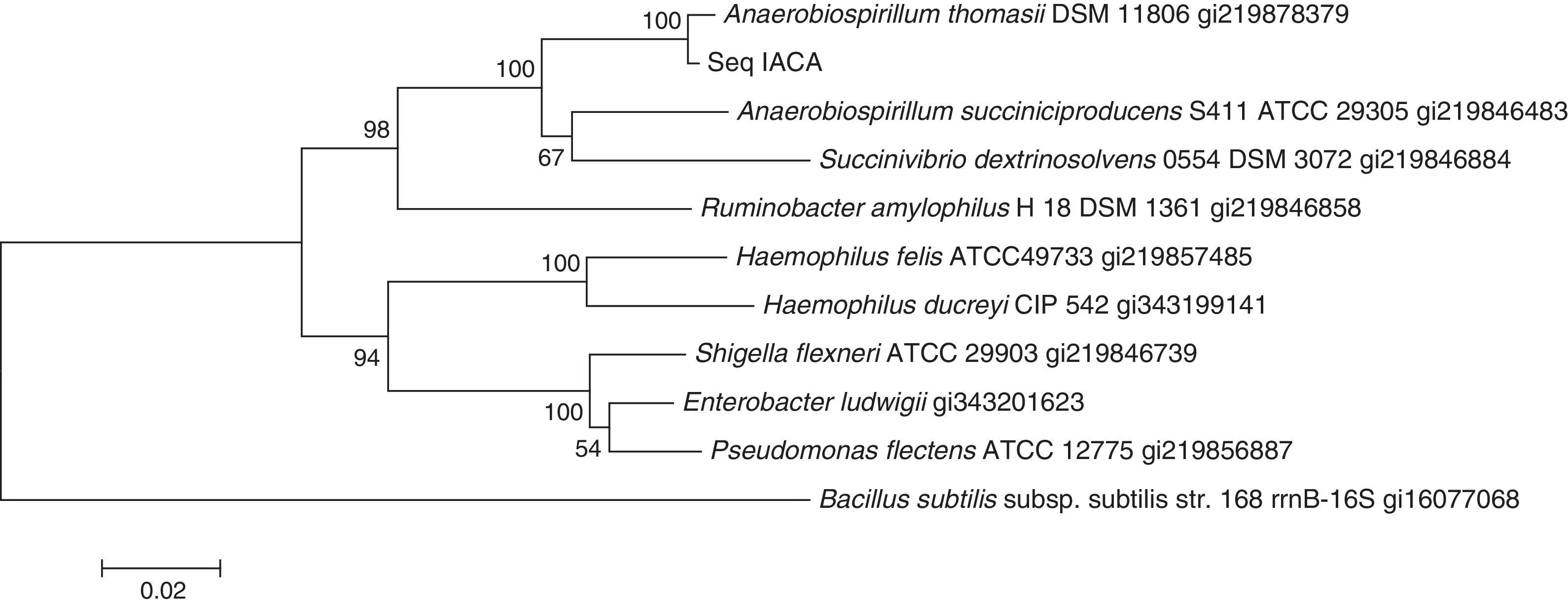
La secuencia obtenida (1130 nucleótidos) se comparó con secuencias conocidas de ARNr 16S contenidas en la base de datos GenBank, con el fin de identificar al microorganismo. Posteriormente, se construyó un árbol filogenético mediante el uso del programa Mega v5.05. Las secuencias se alinearon con Clustal W, se construyó el árbol mediante el algoritmo de Neighbor-Joining con un número de réplicas de 1000 empleando Bacillus subtilis como grupo externo.
La comparación de la secuencia obtenida con secuencias de referencia depositadas en la base de datos GenBank mostró un 99% de identidad (1121 concordancias sobre 1130 bases comparadas) entre el microorganismo hallado y A. thomasii cepa DSM 11806 y un 94% de similitud (1058 de 1130 comparaciones) con A. succiniciproducens cepa S411.
En la figura 1 puede observarse la relación filogenética existente entre la cepa aislada y las 2 especies que forman parte del género Anaerobiospirillum. Como se aprecia, el microorganismo hallado forma un cluster con A. thomasii, con un valor de bootstrap de 100%, mientras que A. succiniciproducens se encuentra en un cluster diferente. De acuerdo con los criterios de interpretación propuestos por el Clinical and Laboratory Standard Institute12, la secuenciación parcial del gen del ARNr 16S es una herramienta efectiva para la identificación de microorganismos anaerobios gram negativos. Estas guías establecen que la obtención de un porcentaje de similitud ≥ 99% con más de un 0,8% de separación entre especies permite informar con seguridad género y especie. La cepa secuenciada mostró un 99% similitud con A. thomasii y un 5% de separación con A. succiniciproducens, la otra especie del género.
Dendograma que muestra la relación filogenética entre la cepa hallada y un grupo de especies relacionadas.Seq IACA (cepa hallada). La línea inferior indica las distancias genéticas. La escala representa un 2% de sustitución nucleotídica esperada por sitio. A la derecha de cada microorganismo se indica el número de acceso GenBank de cada secuencia utilizada para construir este dendograma.
La abundante catarsis diarreica presentada por la paciente y los signos y síntomas de infección gastrointestinal que acompañaron a la bacteriemia indican que el portal primario de entrada de esta bacteria fue el tracto gastrointestinal9. Por otro lado, la condición subyacente de alcoholismo crónico ha sido descrita como un factor que predispone al desarrollo de bacteriemia por este género8. En el paciente estudiado, el pobre estado nutricional, la disrupción de la integridad de la mucosa gastrointestinal y la ascitis podrían haber sido importantes mecanismos que facilitaron la infección. No hay publicados estudios de sensibilidad a antibióticos por tratarse de una infección poco común, por lo que aún no se ha establecido una terapia óptima.
Responsabilidades éticasProtección de personas y animalesLos autores declaran que para esta investigación no se han realizado experimentos en seres humanos ni en animales.
Confidencialidad de los datosLos autores declaran que han seguido los protocolos de su centro de trabajo sobre la publicación de datos de pacientes.
Derecho a la privacidad y consentimiento informadoLos autores declaran que han seguido los protocolos de su centro de trabajo sobre la publicación de datos de pacientes.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.