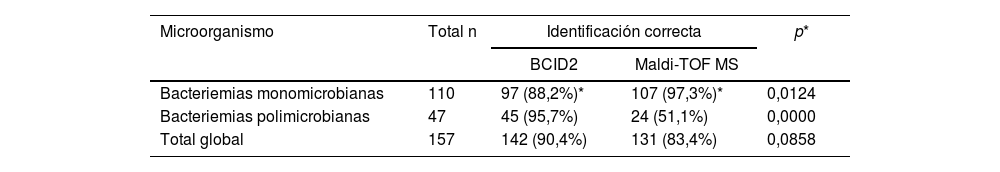
Se analizó la utilidad del uso combinado de MALDI-TOF MS a partir de un subcultivo con 3-5h de incubación y el panel BCID2 (FilmArray) para la identificación de microorganismos provenientes de hemocultivos positivos y su importancia en el ajuste de la terapia antimicrobiana. La identificación global con BCID2 fue del 90,4% (142/157) y con MALDI-TOF MS del 83,4% (131/157) (p=0,0858). Sobre 23 episodios polimicrobianos (47 cepas), el panel BCID2 identificó a 45 (95,7%) cepas y la técnica de MALDI-TOF MS a 24 (51,1%) (p<0,0000). BCID2 detectó la presencia de los genes de resistencia mecA/C (n=16), blaKPC (n=8); blaCTX-M (n=17), blaNDM (n=8), blaOXA-48 (n=1) y vanA/B (n=2). El tiempo promedio para informar un resultado fue de 2h con el uso del BCID2 y de 4h con el de MALDI-TOF MS (p<0,0000). Sobre 124 episodios analizados, el resultado rápido de BCID2 llevó al 82,3% (102/124) de cambios terapéuticos.
The usefulness of the combined use of MALDI-TOF MS from a subculture with 3-5h of incubation and the BCID2 panel (FilmArray) for the identification of microorganisms from positive blood cultures and its importance in the adjustment of antimicrobial therapy was analyzed. Overall identification with BCID2 was 90.4% (142/157) and with Maldi-TOF MS 83.4% (131/157) (p=0.0858); in 23 polymicrobial episodes (47 strains), the BCID2 panel identified 45 (95.7%) and MALDI-TOF MS 24 (51.1%) (p<0.0000). BCID2 detected the presence of the resistance genes mecA/C (n=16), blaKPC (n=8); blaCTX-M (n=17), blaNDM (n=8), blaOXA-48 (n=1), and vanA/B (n=2). The median time to report a result was 2.0h for BCID2 and 4.0h for MALDI-TOF MS (p<0.0000). Of 124 episodes analyzed, the rapid result of BCID2 led to 82.3% (102/124) therapeutic changes.
El tiempo para instaurar un tratamiento adecuado en pacientes con sepsis y, sobre todo, con shock séptico es crítico en lo que hace a la sobrevida de estos5; por tal razón, la identificación de los microorganismos responsables y su sensibilidad a los antibióticos deben establecerse en el menor tiempo posible.
El sistema de PCR múltiple FilmArrayTM (BioFire Diagnostic LLC, EE.UU.) con el panel BCID2 permite la detección simultánea de 43 blancos moleculares y los resultados se obtienen en 1h, con solo 2 a 3min de preparación a partir de hemocultivos positivos.
Existen dos protocolos basados en el empleo de espectrometría de masas con tiempo de vuelo (MALDI-TOF MS) para identificar microorganismos desde un hemocultivo positivo. Uno de ellos es directamente desde la botella, con diversos procedimientos de lavado con agua o saponina, centrifugación y concentrado; el segundo, mucho más simple, parte de una pátina de un subcultivo en medios sólidos con un período de incubación de 3-5h realizado desde el frasco (MTDR)6.
Los objetivos de este trabajo fueron los siguientes: a) determinar la utilidad del uso combinado del panel BCID2 y MTDR para la identificación de microorganismos a partir de hemocultivos positivos, b) establecer el tiempo de informe con cada sistema y determinar el impacto del resultado rápido en el ajuste de la terapia antimicrobiana.
En el período comprendido entre septiembre de 2021 y febrero de 2022, se realizó un estudio prospectivo observacional y colaborativo entre los servicios de Microbiología e Infectología de tres hospitales de la Ciudad de Buenos Aires. Para los hemocultivos, se utilizó el sistema automatizado Bact-Alert (Biomerieux, Marcy, Francia).
Cuando un hemocultivo se detectó como positivo, se realizó coloración de Gram, subcultivo en agar sangre, agar chocolate, CLDE y luego la determinación de BCID2 por medio del sistema de FilmArrayTM con el software 2.0, de acuerdo con las indicaciones del fabricante. Se identificaron los microorganismos por medio de MTDR y se compararon los resultados obtenidos con los arrojados tras una incubación de 24h (método de referencia); en dos hospitales se usó el sistema Vitek-MS (Biomerieux, Marcy, Francia) y en el tercero el sistema Bruker (Bruker, Daltonics, Bremen, Alemania).
Cuando fue necesario, se agregaron métodos convencionales adicionales para ciertos mecanismos de resistencia, como por ejemplo, la evaluación de sinergia entre discos de cefotaxima y ceftazidima con discos de amoxicilina ácido-clavulánico (Oxoid Ltd., Basingstoke, Reino Unido) para β-lactamasas de espectro extendido o por medio de sinergia con discos de ácido borónico y EDTA (Laboratorios Britania, Argentina) con respecto a imipenem y meropenem (Oxoid Ltd., Basingstoke, Reino Unido), métodos cromatográficos (TEST NG CARBA5; NG Biotech, Guipry, Francia) y Carba Blue7 para carbapenemasas. También se determinó la sensibilidad antibiótica por medio de los sistemas automatizados Vitek 2C (Biomerieux, Marcy, l’Etoile, Francia) o Phoenix (Becton Dickinson, Spark EE.UU.).
Los resultados fueron comunicados inmediatamente por el servicio de Microbiología al médico responsable y al servicio de Infectología de cada hospital. Se registró el tratamiento antimicrobiano que el paciente estaba recibiendo antes del informe y los cambios indicados después de tener el informe.
Se consideró que hubo un escalamiento en la terapia antimicrobiana cuando se agregó un antibiótico o antifúngico al esquema que se estaba suministrando o cuando se pasó a uno de mayor espectro; por el contrario, se consideró que se cambió a un esquema de menor espectro cuando se sacó un antimicrobiano o se pasó a uno de menor espectro.
Los procedimientos seguidos se ajustaron a las normas éticas del Comité de Experimentación Humana Responsable de cada institución, así como a las de la Asociación Médica Mundial y la Declaración de Helsinki.
Las variables continuas se informaron como medias con su desvío estándar o medianas con sus rangos intercuartílicos, según correspondiese. Las variables nominales se expresaron con su frecuencia absoluta y relativa. Se determinó la sensibilidad y especificidad de cada prueba por medio de los métodos convencionales, como métodos de referencia. La comparación de variables se realizó a través de la prueba de Wilcoxon o el test de McNemar, según correspondiera. Se consideró para la significación estadística un valor de p<0,05 para dos colas. El análisis estadístico se realizó utilizando el software STATA versión 14.2 (StataCorp, Texas, EE.UU.).
Se documentaron 133 episodios de bacteriemias/fungemias correspondientes a 133 pacientes. La mediana de edad en pacientes adultos fue de 68,5 años (18 a 99 años) y en pediátricos de 2,25 años (recién nacidos a 17 años). El 50,4% (n=67) de ellos fueron de sexo masculino y el 49,6% (n=66) de sexo femenino. Se documentaron 110 (82,7%) episodios monomicrobianos y 23 (17,3%) polimicrobianos, con un total de 157 cepas.
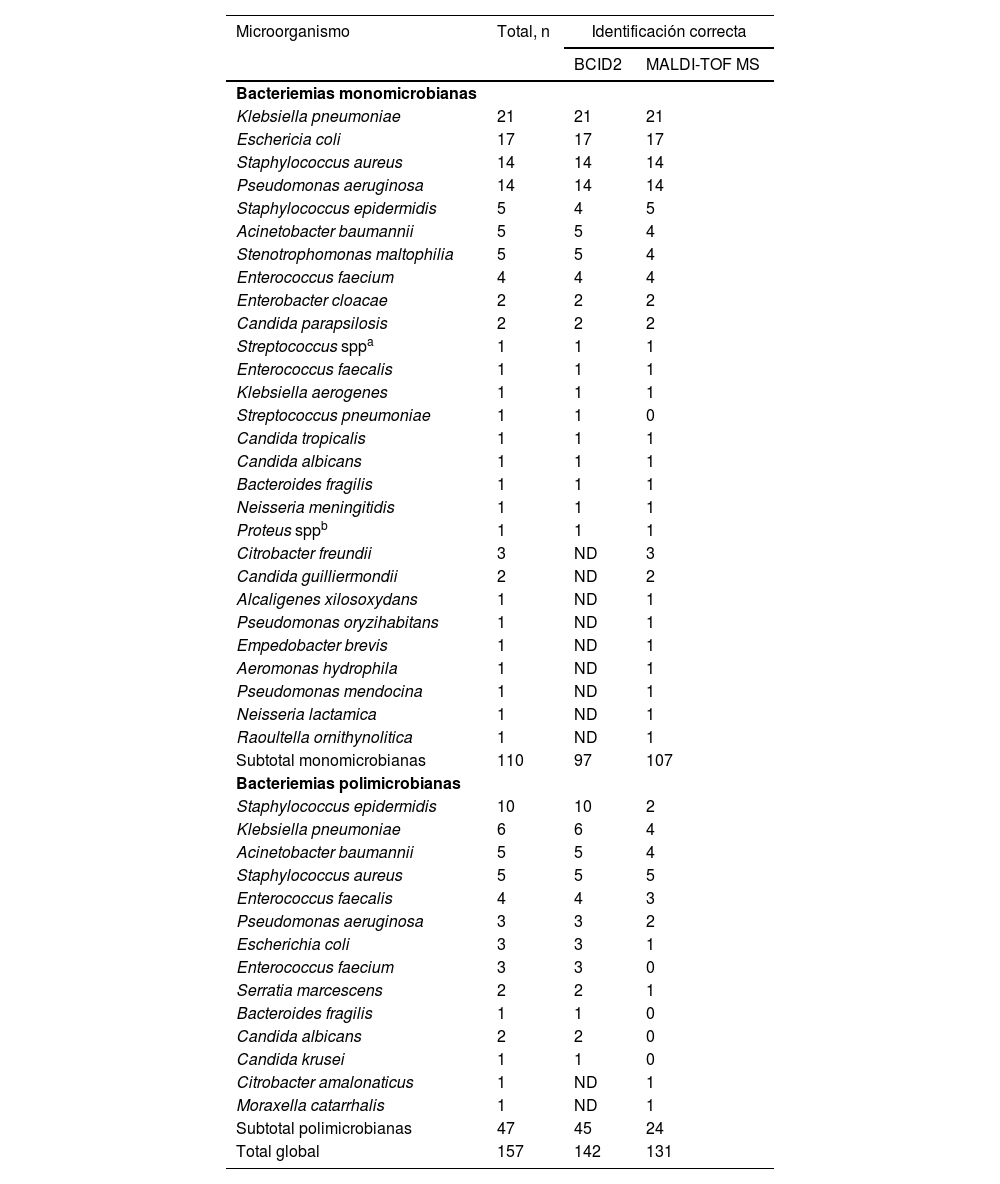
En la tabla 1 se muestra el rendimiento de ambas metodologías para bacteriemias mono- y polimicrobianas. En bacteriemias monomicrobianas, la metodología de MTDR mostró un mejor rendimiento que la de BCID2 (97,3% vs. 88,2%, respectivamente; p=0,0124), pero en bacteriemias polimicrobianas el BCID2 fue superior a MALDI-TOF MS (95.7% vs. 51,1%, respectivamente; p<0,0000) (tabla 1). La identificación global con BCID2 fue del 90,4% (142/157) y con MTDR del 83,4% (131/157) (p=0,0858). Los principales microorganismos no detectados por BCID2 correspondieron a aquellos que no están en la base de datos y representaron en conjunto solo un 8,9% (14/157) (tabla 2). Los microorganismos que se identificaron por BCID2 pero no por MALDI-TOF MS, en todos los casos, correspondieron a fallas en la caracterización, tanto a nivel de género como de especie.
Rendimiento comparativo entre BCID2 y MALDI-TOF MS en bacteriemias
Identificación de los microorganismos a través de BCID2 y MALDI-TOF MS, bacteriemias mono- y polimicrobianas
| Microorganismo | Total, n | Identificación correcta | |
|---|---|---|---|
| BCID2 | MALDI-TOF MS | ||
| Bacteriemias monomicrobianas | |||
| Klebsiella pneumoniae | 21 | 21 | 21 |
| Eschericia coli | 17 | 17 | 17 |
| Staphylococcus aureus | 14 | 14 | 14 |
| Pseudomonas aeruginosa | 14 | 14 | 14 |
| Staphylococcus epidermidis | 5 | 4 | 5 |
| Acinetobacter baumannii | 5 | 5 | 4 |
| Stenotrophomonas maltophilia | 5 | 5 | 4 |
| Enterococcus faecium | 4 | 4 | 4 |
| Enterobacter cloacae | 2 | 2 | 2 |
| Candida parapsilosis | 2 | 2 | 2 |
| Streptococcus sppa | 1 | 1 | 1 |
| Enterococcus faecalis | 1 | 1 | 1 |
| Klebsiella aerogenes | 1 | 1 | 1 |
| Streptococcus pneumoniae | 1 | 1 | 0 |
| Candida tropicalis | 1 | 1 | 1 |
| Candida albicans | 1 | 1 | 1 |
| Bacteroides fragilis | 1 | 1 | 1 |
| Neisseria meningitidis | 1 | 1 | 1 |
| Proteus sppb | 1 | 1 | 1 |
| Citrobacter freundii | 3 | ND | 3 |
| Candida guilliermondii | 2 | ND | 2 |
| Alcaligenes xilosoxydans | 1 | ND | 1 |
| Pseudomonas oryzihabitans | 1 | ND | 1 |
| Empedobacter brevis | 1 | ND | 1 |
| Aeromonas hydrophila | 1 | ND | 1 |
| Pseudomonas mendocina | 1 | ND | 1 |
| Neisseria lactamica | 1 | ND | 1 |
| Raoultella ornithynolitica | 1 | ND | 1 |
| Subtotal monomicrobianas | 110 | 97 | 107 |
| Bacteriemias polimicrobianas | |||
| Staphylococcus epidermidis | 10 | 10 | 2 |
| Klebsiella pneumoniae | 6 | 6 | 4 |
| Acinetobacter baumannii | 5 | 5 | 4 |
| Staphylococcus aureus | 5 | 5 | 5 |
| Enterococcus faecalis | 4 | 4 | 3 |
| Pseudomonas aeruginosa | 3 | 3 | 2 |
| Escherichia coli | 3 | 3 | 1 |
| Enterococcus faecium | 3 | 3 | 0 |
| Serratia marcescens | 2 | 2 | 1 |
| Bacteroides fragilis | 1 | 1 | 0 |
| Candida albicans | 2 | 2 | 0 |
| Candida krusei | 1 | 1 | 0 |
| Citrobacter amalonaticus | 1 | ND | 1 |
| Moraxella catarrhalis | 1 | ND | 1 |
| Subtotal polimicrobianas | 47 | 45 | 24 |
| Total global | 157 | 142 | 131 |
ND: No incluida en base de datos BCID2.
La sensibilidad y especificidad para la detección de genes de resistencia mecA/C, vanA/B, blaCTXM, blaKPC, blaNDM, blaOXA48 fue del 100% (16/16, 2/2; 17/17, 8/8, 8/8 y 1/1, respectivamente). No se aislaron cepas con blaVIM, blaIMP, ni con mcr.
La mediana en horas para la comunicación de resultados con BCID2 y con MTDR fue de 2h (rango intercuartílico: 1,3 a 2h) y 4h (rango intercuartílico: 4 a 5h), respectivamente (p<0,0000).
El tratamiento antimicrobiano se evaluó en 124 casos y la comunicación inmediata del resultado de BCID2 llevó a cambios en el 82,3% (102/124) del total y en el 43,5% (10/23) de los episodios polimicrobianos. En el 30,8% (4/13) de los casos con cepas no detectadas por BCID2, el resultado de MTDR llevó a cambios terapéuticos.
Cortazzo et al.1 analizaron el rendimiento de BCID2 sobre 90 hemocultivos con aislamientos clínicamente relevantes. Sobre un total de 53 genes de resistencia, el sistema BCID2 detectó 50, mientras que con una versión anterior (BCID) se detectaron 22.
La sensibilidad del BCID2 fue del 100% para CTX-M, KPC, NDM, OXA-48, VIM y vanA, así como para mecA en Staphylococcus aureus. Con respecto a la identificación, tanto el panel BCID2 como el BCID detectaron el 100% de los microorganismos incorporados en sus respectivas bases de datos; la especificidad de ambos fue del 100% y también mostraron una excelente correlación entre sí en lo que hace a la detección en bacteriemias polimicrobianas.
Un estudio multicéntrico argentino realizado en 5 hospitales encontró un rendimiento global del panel BCID2 del 94%, del 97% con respecto a los microorganismos incluidos en la base de datos y del 100% en la detección de los genes de resistencia blaCTX-M, blaKPC, blaNDM, blaVIM, blaIMP, mecA/C y vanA/B, con cambios terapéuticos generados por estos resultados en el orden del 58%8.
El rendimiento global encontrado con BCID2 en este trabajo fue del 90,4%, en tanto que si se consideran solo los microorganismos incluidos en la base de datos este fue del 99,3%. Los principales microorganismos que no se detectaron correspondieron a aquellos que no están en la base de datos y representaron en conjunto solo un 8,9% (14/157).
La sensibilidad y la especificidad en la detección de carbapenemasas del tipo KPC y NDM, de CTX-M y de meticilino resistencia (mecA/C) fueron excelentes (100%). Todos estos resultados están de acuerdo con las publicaciones mencionadas previamente. En este trabajo, no fue posible evaluar la detección de los genes de resistencia plasmídica a colistina ni los genes blaVIM y blaIMP por no presentarse ninguna cepa con estas características. Solo se aisló una cepa de Klebsiella pneumoniae con OXA-48 y dos de Enterococcus faecium con vanA, por lo que es necesario confirmar estos hallazgos con un número mayor de cepas.
La media en el tiempo para la comunicación de resultados a partir de BCID2 fue de 2h, mucho más reducida que la correspondiente a MTDR (4h). El alto rendimiento en la detección de genes de resistencia sumado al corto tiempo para obtener y comunicar el resultado al servicio de Infectología de cada hospital por medio de BCID2 y la detección por MTDR de microorganismos no incluidos en la base de datos de BCID2 pueden explicar el gran impacto en el cambio terapéutico observado (82,2%), tanto en lo que hace a escalamiento (39,5%) como a desescalamiento (42,7%) antimicrobiano.
En el caso de emplear MALDI-TOF MS, cuando se hace directo desde el frasco esta técnica tiene un rendimiento promedio del 80%, pero es necesario primero tratar con un agente lítico, como saponina o agua destilada, y luego realizar centrifugación. Muchas veces se espera a que haya un grupo de hemocultivos positivos para hacerlo y esto puede complicar el flujo de trabajo y retrasar los resultados. Además, este procedimiento necesita confirmación a partir de las colonias aisladas. Otra posibilidad es a partir de MTDR, esto acelera el flujo de trabajo y, en general, no es necesario confirmar los resultados. De acuerdo con distintas publicaciones, es mejor para los bacilos gram negativos (82-99%) que para los cocos gram positivos (67-93%). Ambos métodos presentan problemas cuando el cultivo es mixto6.
Ha et al.4 demostraron la utilidad de la identificación con MALDI-TOF MS a partir de la pátina con 3-6h de incubación. Sobre 254 cultivos monomicrobianos de hemocultivos, la identificación fue correcta con Vitek MS en el 79% y 86,6% a nivel de especie y género, respectivamente, en tanto que los valores para el sistema de Bruker fueron del 82% y 87,4%, respectivamente.
Huang et al.3 estudiaron el impacto de la identificación rápida con MALDI-TOF MS y su comunicación al Programa de Uso Racional de Antibióticos (PROA), y demostraron que se disminuía el tiempo de identificación (84 vs. 55,9h; p<0,001) y se mejoraba el tiempo para lograr una terapia efectiva (30,1 vs. 20,4h; p=0,021) u óptima (90,3 vs. 47,3h; p=0,001) con respecto a los métodos convencionales.
En el presente trabajo, MTDR mostró un mejor rendimiento en bacteriemias monomicrobianas que BCID2 (p=0,0124) debido, principalmente, a la detección de microorganismos no incluidos en la base de datos de este último; sin embargo, si se analizan solo los microorganismos que están en la base de datos de BCID2, no hubo diferencias significativas entre ambos métodos (p=0,3173). En bacteriemias polimicrobianas, el BCID2 fue superior al MTDR (p<0,0000), que no identificó 23 cepas, y esto fue relevante porque llevó a cambios terapéuticos en el 43,5% de estos casos. Por otro lado, el MTDR condujo a cambios terapéuticos en el 30,8% (4/13) de los episodios con cepas no identificadas por BCID2.
Fiori et al.2 combinaron ambas metodologías y lograron identificar al 97,7% de los microorganismos. Sin embargo, en las bacteriemias polimicrobianas, mediante MALDI-TOF MS se identificó a un solo microorganismo en el 56,7% de los casos, en tanto que mediante FilmArray se detectó al 89,5%.
Timbrook et al.9 hallaron que solo se lograban beneficios en cuanto a la reducción del tiempo para llegar a una terapia efectiva, del tiempo de internación y de la mortalidad si los resultados rápidos iban a un PROA; sin embargo, la mayoría de los estudios incluidos fueron observacionales, por lo que hace falta más evidencia para demostrar una reducción en la mortalidad. Esto es muy difícil de resolver por cuestiones éticas y porque se necesitaría un número enorme de pacientes en estudios aleatorios para demostrar una diferencia significativa con el uso de las técnicas rápidas, motivos por los que la mortalidad no se incluyó como objetivo en el presente trabajo.
Nuestros datos son coincidentes con la evidencia publicada en lo que hace al alto impacto en cambios terapéuticos logrados con los resultados rápidos cuando estos son comunicados inmediatamente al servicio de Infectología de cada hospital.
Para optimizar el uso de estas metodologías en términos de costo-beneficio, es muy importante la correcta selección de los pacientes en cuanto a la gravedad del cuadro clínico y la posibilidad de cambios terapéuticos, y que estos resultados vayan a un PROA. Por lo tanto, cada hospital debe diseñar algoritmos para su utilización basados en la población de cada uno de ellos.
Una limitación de este estudio es no haber analizado la reducción en la mortalidad dado que esta se debe a múltiples factores y, como ya se indicó, se debe incluir un número muy grande de pacientes para lograr encontrar una diferencia significativa.
En suma, el sistema BCID2 permite identificar con rapidez a los microorganismos causantes de bacteriemia y sus principales genes de resistencia, además, tiene un alto rendimiento diagnóstico en bacteriemias polimicrobianas. Este, sumado a la metodología de MALDI-TOF MS para la detección de microorganismos que no están en la base de datos de dicha PCR múltiple, y los métodos tradicionales para determinar la CIM y confirmar las resistencias representan, sin duda, una sinergia de herramientas diagnósticas que, comunicadas inmediatamente al servicio de Infectología de cada hospital o al PROA, llevan a adecuar el tratamiento antimicrobiano desde el mismo comienzo de la bacteriemia.
Conflicto de interesesRolando Soloaga se desempeña además como Medical Science Liason de Biomerieux Argentina.
No se contó con fuentes de financiamiento externas a cada hospital.