Se presentan 2 casos de bacteriemia por Helicobacter cinaedi. El primero se diagnosticó en un varón de 76 años y resultó secundario a la colocación de un acceso vascular; el segundo correspondió a un lactante febril de 37 días de vida, asociado a un cuadro de gastroenterocolitis aguda. H. cinaedi es un microorganismo que presenta dificultad para desarrollarse en diferentes medios de cultivo y lograr su identificación a nivel de especie. En ambos casos fue fundamental la observación microscópica en fresco de las botellas de hemocultivo, la utilización de la espectrometría de masas y la posterior secuenciación del gen hsp60 para llegar a esa instancia. En los últimos años se han informado infecciones por H. cinaedi con frecuencia creciente en otras partes del mundo. En este trabajo presentamos los primeros casos de bacteriemia por H. cinaedi documentados en Argentina.
Two cases of bacteremia caused by Helicobacter cinaedi are presented. The first case was diagnosed in a 76-year-old male patient, and was secondary to a vascular access device placement; the second case corresponded to a febrile infant of 37 days of life, and was associated with acute gastroenteritis. H. cinaedi is a microorganism difficult to grow in different culture media and also to identify to species level. In both cases, the microscopic observation of blood culture bottles, the use of mass spectrometry and the subsequent sequencing of the hsp60 gene were essential. In the recent literature, H. cinaedi infections are being reported more frequently. In this report we present the first documented cases of bacteremia caused by H. cinaedi in Argentina.
Helicobacter cinaedi es una bacteria gram negativa, móvil, con morfología espirilar. Fue descrita por Fennell en 1984 como un microorganismo Campylobacter-like, aislado de muestras rectales de varones homosexuales con proctocolitis. Estudios de taxonomía molecular mediante secuenciación del ARN ribosómico y de hibridización ADN-rARN demostraron que estas bacterias presentaban una posición filogenética diferente a las especies de Campylobacter, lo que derivó en la creación del género Helicobacter a fines de los ochenta3,7,16. Dicho género actualmente está compuesto por 36 especies (http://www.bacterio.net/helicobacter.html). En el campo de la patología humana, la mayor atención se ha centrado siempre en H. pylori; sin embargo, existen otras especies, denominadas genéricamente «non-H. pylori», con potencial capacidad patogénica. Muchas de ellas han sido aisladas tanto del hombre como de animales, por lo que se las considera probables agentes zoonóticos3,13.
H. cinaedi ha sido reportado en enteritis, bacteriemia, celulitis, artritis, osteomielitis, meningitis e infección asintomática en pacientes con algún factor de inmunocompromiso1,2,4,7,9,12,14. También ha sido asociado a infecciones en personas inmunocompetentes10,14. H. cinaedi coloniza el tracto gastrointestinal de mamíferos. Su principal reservorio natural es el hámster3, el cual podría constituir una fuente de contaminación para el hombre. También se ha reportado su aislamiento a partir de materia fecal de perros y gatos3,15.
En nuestro medio H. cinaedi no es un patógeno aislado con frecuencia. El objetivo de esta comunicación es describir 2 casos de bacteriemia por H. cinaedi y destacar las estrategias empleadas para su identificación a nivel de especie.
Caso 1: varón de 76 años, que se internó en el hospital H1 el 3 de agosto de 2014 para ser sometido a una operación de reemplazo valvular aórtico biológico y cirugía de Maze por insuficiencia aórtica. Como antecedentes presentó hipertensión arterial, dislipidemia y fibrilación auricular crónica. Refirió consumir alcohol en forma moderada y haber fumado 20 cigarrillos/día hasta el año anterior. Sin complicaciones intraoperatorias, en el posoperatorio inmediato postextubación el paciente presentó excitación psicomotriz; evolucionó favorablemente y fue dado de alta a los 10 días del ingreso. A los 20 días de la cirugía consultó por presentar registros febriles con lesión eritematosa en el miembro inferior izquierdo, que se interpretó como secundaria a la colocación de un acceso venoso femoral. Se instauró tratamiento empírico con cefalexina. El paciente fue nuevamente internado el 5 de septiembre de 2104. Se le realizaron 2 tandas de hemocultivos (Hc), totalizando 6 frascos aeróbicos y uno anaeróbico (Sistema Bactec Serie 9000, Becton Dickinson, EE. UU.). Luego de 3 días de incubación de la segunda tanda de Hc, se detectó señal positiva en los 3 frascos aeróbicos. Por examen microscópico en fresco del caldo del Hc se visualizaron bacterias espirilares móviles, por lo que se rotó el tratamiento antibiótico a ceftriaxona (2g/día) parenteral y ciprofloxacina (400mg c/12h). Tras 72h de incubación a 37°C, se visualizó crecimiento en agar sangre ovina al 5%, en atmósfera de CO2 (5-10%) y humedad, lo que se interpretó como posible aislamiento de Campylobacter sp. Se rotó el tratamiento antibiótico a ceftriaxona (2g/día) y doxiciclina por 6 semanas, con evolución favorable. El cuadro se interpretó como una infección asociada al acceso vascular.
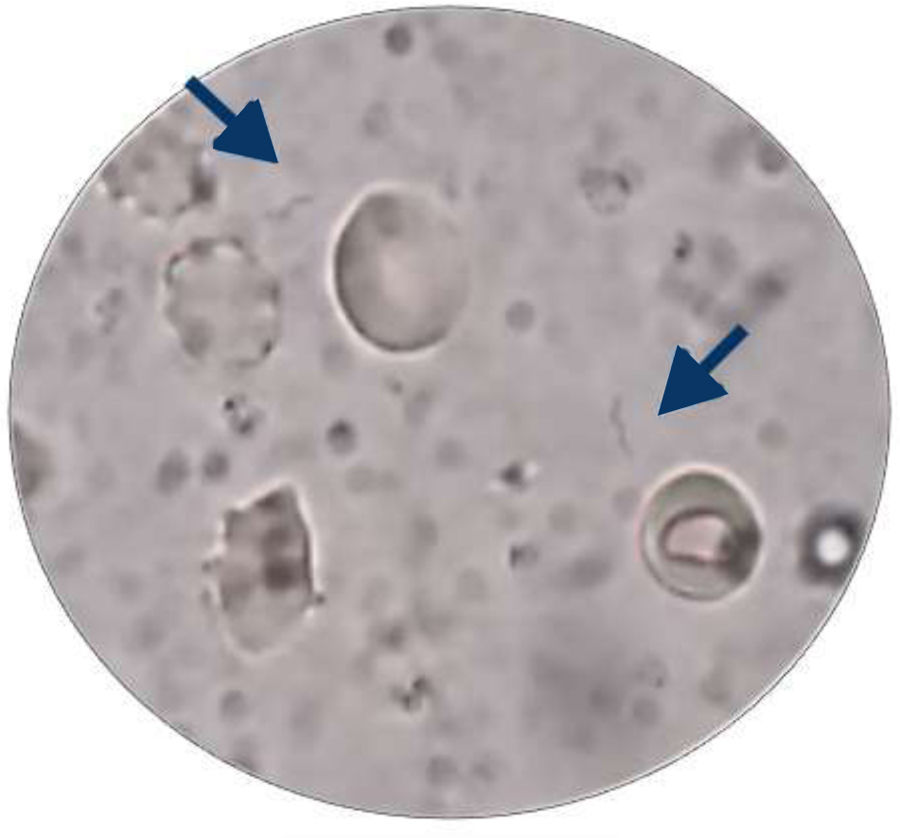
Caso 2: varón de 37 días de vida, sin antecedentes perinatales de importancia ni de enfermedad, que se internó en el hospital H2 el 26 de enero de 2015 por fiebre sin foco aparente, asociada a un episodio de hipertonía en los miembros superiores y el cuello, de 2 minutos de duración. Catalogado por esta razón como lactante febril de alto riesgo, se realizaron cultivos de orina, de líquido cefalorraquídeo y una tanda de 2 frascos de Hc aeróbicos (Sistema Bactec Serie 9000, Becton Dickinson, EE. UU.). Se indicó empíricamente ampicilina y ceftriaxona. Tras 48horas de incubación se detectó señal positiva en los 2 frascos de Hc. En el examen en fresco directo del caldo del Hc se observaron bacterias espirilares móviles (fig. 1), que no crecieron tras diversos subcultivos en agar sangre ovina al 5%, agar chocolate y agar Skirrow, incubados a 37°C y hasta 96horas, en atmósfera de CO2 (5-10%). La madre del lactante refirió que un hermano del niño había presentado gastroenterocolitis; practicaba colecho familiar, no alimentaba al pequeño con leche materna ni convivía con mascotas. Por los antecedentes epidemiológicos disponibles, el caso se interpretó como infección enteral atribuida a Campylobacter sp. Finalmente, dada la buena evolución clínica se decidió el egreso hospitalario, que fue el 5 de febrero de 2015 tras haber cumplido 10 días de tratamiento antibiótico, sin presentar otro foco de infección.
Las flechas señalan la posición de los «espirilos». La imagen se obtuvo por observación en fresco de una gota obtenida del frasco de hemocultivo, colocada entre el portaobjeto y el cubreobjeto, sobre el cual se agregó aceite de inmersión (1.000×). La imagen fue captada con un dispositivo celular.
Ante la dificultad de identificar a nivel de especie las bacterias espirilares visualizadas, el aislado del caso 1 y los frascos de Hc del caso 2 se derivaron al Laboratorio Nacional de Referencia (LNR), Servicio Bacteriología Especial, INEI-ANLIS Dr. Carlos G. Malbrán, donde se realizaron subcultivos en agar BHI con sangre ovina al 5% (figura disponible en el material adicional) y agar chocolate, incubados a 37°C en condiciones de microaerobiosis (5% de O2, 5 a 10% de H2). Los cultivos obtenidos en agar sangre fueron analizados por espectrometría de masas con el equipo Microflex LT (MALDI-TOF MS). Se trabajó a partir de una colonia aislada, que fue depositada por duplicado sobre una placa de titanio (Bruker Daltonics®, Bremen, Alemania) mediante el método de extendido directo, después de hacer la calibración del equipo con el estándar bacteriano (BTS; Bruker Daltonics®).
Luego, cada pocillo fue cubierto con 1ml de matriz (solución saturada de ácido α-ciano-4-hidroxicinámico [HCCA; Bruker Daltonics]) en 50% de acetonitrilo y 2,5% de ácido trifluoroacético. Los parámetros fueron ajustados según las recomendaciones del fabricante15. El análisis se realizó con los programas FlexControl, Maldi Biotyper RTC y Maldi Biotyper OC (v3.1, Bruker Daltonics). Los espectros obtenidos fueron comparados con los que aporta la base de datos, lo cual permite asignar un puntaje o score. Los criterios de identificación recomendados por el fabricante son los siguientes: valor de score>2.000, identificación a nivel de especie; score entre 1.700 y 1.999, identificación confiable solo a nivel de género; y valores<1.700 no permiten identificación. La comparación de los espectros obtenidos con el espectro de referencia correspondiente a la cepa H. cinaedi CCUG 18818 mostró un score de 1.478 para el aislado 1 y de 1.686 para el aislado 2. Dado que en ambos casos el score obtenido fue menor que el establecido como criterio de identificación confiable, la identidad de estos aislamientos fue confirmada mediante amplificación y secuenciación parcial de los genes 16S rRNA y hsp60. La amplificación del gen 16S rRNA fue llevada a cabo con los cebadores universales denominados 8-27F (5’-AGAGTTTGATCCTGGCTCAG-3’) y 1492R (5’-GGTTACCTTGTTACGACTT-3’). La secuenciación parcial del gen hsp60 se realizó con los cebadores HSP60F (5’-ACGGCGACGGTGCTTGCATA-3’) y HSP60R (5’-TTCGCCGAAGCCGGGGGCTT-3’)15. Los productos de PCR fueron secuenciados utilizando el equipo Big Dye Terminator v 3.1 Cycle sequencing kit (Applied Biosystems) y analizados en el ABI 377 Genetic Analyzer (PE Applied Biosystems). Las secuencias obtenidas fueron comparadas con las depositadas en el GenBank (http://www.ncbi.nlm.nih.gov/genbank/), utilizando BLASTN y la base de datos 16S Ribosomal RNA sequences (National Center for Biotechnology Information) (http://blast.ncbi.nlm.nih.gov/Blast.cgi). Los resultados fueron interpretados según lo descrito en el documento CLSI MM18-A4.
La similitud de las secuencias nucleotídicas para el gen 16S rRNA de los aislados 1 y 2 con respecto a la cepa tipo de H. cinaedi CCUG 18818 fue del 97% y 99%, en ese orden. Cuando se analizó el gen hsp60 respecto de la cepa tipo de H. cinaedi PAGU 0611, la similitud de las secuencias nucleotídicas fue de 99% para los 2 aislados. En ambos casos se evidenció una divergencia mayor del 0,8% con las especies más relacionadas. Estos resultados permiten asumir que los aislados 1 y 2 corresponden a H. cinaedi. Las secuencias fueron depositadas en la base de datos GenBank. Los números de acceso asignados fueron KP178537 (gen 16S rRNA) MG592730 (gen hsp60) y MF440359 (gen 16S rRNA) MG586158 (gen hsp60), respectivamente.
En la literatura cada vez son más numerosas las publicaciones donde se pone énfasis en el rol de H. cinaedi como agente causal de bacteriemias2,4,6,8-10,12. En la mayoría de estos trabajos H. cinaedi fue aislado sin dificultad en botellas aeróbicas del Sistema Bactec, aunque algunos autores propusieron la necesidad de incrementar el tiempo de incubación de las botellas con el fin de permitir el desarrollo bacteriano1.
En relación con las características clínicas de los pacientes con bacteriemia por este microorganismo, en adultos se refiere la presencia de algún factor de inmunocompromiso12, y se ha destacado la terapia con corticoides y la quimioterapia de pacientes con cáncer1,5,6,8,12,13. También se lo relacionó con la presencia de enfermedades de base como diabetes, enfermedad renal crónica, tumor sólido, enfermedad pulmonar crónica, artritis reumatoidea, lupus, etc.
La manifestación clínica más frecuente es la celulitis1,4,10,14 y, en segundo lugar, la fiebre sin foco evidente. Incluso se lo reconoce como agente causal de bacteriemias asociadas al cuidado de la salud, situación que puede estar relacionada con la capacidad de translocación de este microorganismo desde la mucosa intestinal. Al respecto, en los pacientes con bacteriemia por H. cinaedi en los que este agente se investigó también en materia fecal, el cultivo resultó positivo en el 50% de los casos1,7.
La identificación de H. cinaedi por métodos fenotípicos ha demostrado grandes limitaciones asociadas a la falta de reproducibilidad de las pruebas, la variabilidad de los resultados entre las especies y al poco poder de discriminación entre especies estrechamente relacionadas, a lo que se agrega la dificultad inicial de obtener el desarrollo del microorganismo7,13,15. Esto plantea la necesidad de recurrir a otras metodologías que no están disponibles en el laboratorio clínico, ya sea por la dificultad de su implementación, los costos agregados o la demora en la obtención del resultado16.
La reciente incorporación de técnicas proteómicas, como la espectometría de masas aplicada a la identificación de microorganismos, permitió resolver parte del problema y acortar sustancialmente el tiempo insumido en la identificación a nivel de especie, tal como se describe en la literatura15.
En los casos relatados el crecimiento de H. cinaedi en los frascos de Hc no presentó dificultad, y la señal de positividad se detectó dentro de las 48-72horas desde el inicio de la incubación.
El primer caso fue el resultante de una probable bacteriemia asociada al cuidado de la salud; el segundo caso se interpretó como un lactante febril con posible foco intestinal, dado el contexto epidemiológico del paciente. En relación con el tratamiento es importante señalar que en la literatura no hay guías clínicas disponibles en cuanto a la elección y duración de la terapia antibiótica; además, la prueba de sensibilidad a los antimicrobianos no siempre está disponible. Estudios in vitro muestran que carbapenemes, aminoglucósidos y tetraciclinas son los antimicrobianos que presentaron valores de CIM más bajos7. Lacruz et al.11 combinaron imipenem y gentamicina durante 2 semanas y doxiciclina durante 5 semanas para tratar la bacteriemia de un paciente infectado por virus de la hepatitis C. Bartels et al.2 trataron un caso de endocarditis combinando ceftriazona más gentamicina. Para el tratamiento de la bacteriemia en un paciente con enfermedad renal Mandai et al.12 asociaron cefotaxima seguida de meropenem y ciprofloxacina, con posterior administración oral de doxiciclina para erradicar la infección. Shimizu et al.14 combinaron el uso de meropenem con minociclina oral para el tratamiento de un caso de bacteriemia con celulitis recurrente en un paciente inmunocompetente. Es por eso que no podemos asegurar que el tratamiento administrado fue el apropiado, en parte por la diversidad de los esquemas usados que refiere la literatura, la falta de casuística a nivel local, la demora en conocer la identificación del agente causal y la limitación que agrega el realizar una evaluación retrospectiva. Además, tal valoración excede los objetivos de este trabajo.
En ninguno de los pacientes se pudo constatar fehacientemente el contacto con mascotas. Al respecto, en la literatura se indica que las cepas de H. cinaedi aisladas del ser humano difieren de las halladas en animales domésticos, lo que pone en controversia su rol como agente zoonótico3,15. Tampoco se investigó si los pacientes eran portadores intestinales de H. cinaedi, pero se debe resaltar que este microorganismo no fue inicialmente sospechado.
En la detección de H. cinaedi fue fundamental la observación microscópica en fresco realizada desde el frasco de Hc, la que permitió detectar bacterias espiriladas de movilidad característica. Si bien en principio fueron interpretadas como posible Campylobacter sp., este hallazgo obligó a implementar subcultivos en medios más enriquecidos, también en otras condiciones de crecimiento. Creemos importante resaltar que en los centros H1 y H2 la observación microscópica directa de las gotas de caldo del Hc con señal positiva se realiza de rutina, y es una guía para aplicar otros procedimientos que permitirán acortar los tiempos para la detección del agente causal. La microscopía es una técnica de bajo costo y fácil implementación, que puede brindar información útil en el diagnóstico en los casos con señal positiva de los sistemas automatizados y subcultivo negativo.
El empleo de la espectrometría de masa fue una herramienta clave para orientar el uso de otra metodología que permitiera la identificación a nivel de especie, como la secuenciación. Si bien actualmente esta metodología queda circunscrita a los laboratorios de referencia o a aquellos con presupuestos que permiten su utilización, la estrategia de idear una red de derivación accesible posibilitará el diagnóstico de infecciones por microorganismos de difícil identificación o que requieren más tiempo.
H. cinaedi es un microorganismo que puede presentar dificultad en el aislamiento y la identificación en el laboratorio clínico. El trabajo coordinado con el LNR permitió realizar el diagnóstico etiológico, además del aporte epidemiológico que implica haber descrito por primera vez en la Argentina H. cinaedi como causante de bacteriemia.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.