En este trabajo se evalúa una prueba rápida in house para la detección de enterobacterias sensibles a cefotaxima, basada en el cambio de pH del rojo fenol debido a la hidrólisis de este antibiótico. Las cepas de enterobacterias procedentes de 1.947 urocultivos se evaluaron mediante los paneles MicroScan y esta prueba in house. Mediante los paneles de MicroScan se estudiaron 499 aislados de enterobacterias, entre los cuales había 27 aislados de Escherichia coli productora de β-lactamasa de espectro extendido (BLEE), 16 de Klebsiella pneumoniae BLEE y una de Klebsiella oxytoca BLEE. La prueba in house mostró una sensibilidad del 98% y una especificidad del 97%, con un valor predictivo negativo del 100% y un valor predictivo positivo del 78%. La prueba in house basada en el cambio de pH es útil en nuestro medio para detectar presuntivamente de forma rápida cepas de enterobacterias con cierta resistencia a cefotaxima.
In this work an “in house” rapid test based on the change in pH that is due to hydrolysis for detecting Enterobacteriaceae susceptible to cefotaxime is evaluated. The strains of Enterobacteriaceae from 1947 urine cultures were assessed using MicroScan panels and the “in house” test. This rapid test includes red phenol solution and cefotaxime. Using MicroScan panels, 499 Enterobacteriaceae isolates were evaluated, which included 27 isolates of Escherichia coli producing extended-spectrum beta-lactamases (ESBL), 16 isolates of Klebsiella pneumoniae ESBL and 1 isolate of Klebsiella oxytoca ESBL. The “in house” test offers the following values: sensitivity 98% and specificity 97%, with negative predictive value 100% and positive predictive value 78%. The “in house” test based on the change of pH is useful in our area for detecting presumptively cefotaxime-resistant Enterobacteriaceae strains.
La infección del tracto urinario (ITU) es una de las infecciones de mayor prevalencia, y en España se estima que hay casi 4 millones de episodios anuales de ITU, solamente en mujeres de entre 20 y 44 años; de hecho, es la población femenina de esta franja etaria aquella en la cual la incidencia de ITU es más alta4,5.
La multirresistencia a los antibióticos en las bacterias gram negativas, principales agentes causales de ITU, es un problema emergente en todo el mundo2. La producción de β-lactamasas, muchas de ellas de tipo espectro extendido, es el mecanismo de resistencia más frecuente de los bacilos gram negativos13,15, sobre todo en las enterobacterias. Cuando las bacterias son productoras de BLEE, son resistentes frente a la mayoría de los β-lactámicos, pero esta resistencia desaparece en presencia de inhibidores de β-lactamasas, como el ácido clavulánico. Este tipo de mecanismo no conlleva resistencia frente a los carbapenémicos ni a la cefoxitina16.
Los métodos de detección fenotípica de BLEE mediante el uso de la técnica de disco-placa, también llamada Kirby-Bauer, tienen un amplio uso y son económicos, pero requieren al menos 48 h desde la llegada de la muestra al laboratorio para dar un resultado. La detección mediante PCR es rápida, pero costosa, y requiere personal especialmente entrenado7,9. Recientemente se han desarrollado pruebas comerciales rápidas y económicas basadas en la detección del cambio de pH debido a la hidrólisis del anillo β-lactámico de estos antibióticos, sobre todo de la cefotaxima, muy sensible a la degradación por las BLEE6. Debido a la limitada experiencia, en este trabajo se evalúa una prueba in house para la detección rápida de enterobacterias resistentes a cefotaxima basada en el cambio de pH que produce su hidrólisis.
Se analizaron de forma prospectiva durante noviembre de 2014 todos los urocultivos solicitados (n: 1.947) al laboratorio de microbiología del Complejo Hospitalario Universitario de Granada (Hospital Virgen de las Nieves, Granada, España); aquellos con un recuento significativo de acuerdo con nuestro protocolo normalizado de trabajo para al menos un uropatógeno de tipo enterobacteria10 fueron sometidos a la prueba in house y a un estudio de identificación a nivel de especie y de sensibilidad fenotípica a los antibióticos.
Se evitó la duplicidad de muestra y paciente consultando el historial de muestras microbiológicas de cada uno de ellos en el sistema informático de nuestro laboratorio. Nuestro hospital es un centro de referencia que asiste a una población de 440.000 personas. Todas las muestras se obtuvieron de pacientes atendidos en Centros de Salud de Atención Primaria o ingresados en las salas de hospitalización de los 3 hospitales que componen el complejo hospitalario (Hospital General de Especialidades, Hospital Materno-Infantil y Hospital de Rehabilitación y Traumatología).
Para la prueba in house, se prepararon alícuotas a partir 50ml de reactivo de extracción de proteínas bacterianas (B-PER, Thermo Scientific, Rockford, EE. UU.), 50ml de una solución de indicador de pH de rojo fenol en agua destilada, a una concentración final de 0,0125%, y 150mg de cefotaxima (Sigma Aldrich Química S.A, Tres Cantos, Madrid, España), con una concentración final 1,5mg/l, ajustando a un pH de 7,8. Se empleó en cada ensayo una solución control sin cefotaxima. Se transfirieron 200μl de las soluciones control y con cefotaxima a tubos Eppendorf. Estos se congelaron a –80°C, a fin de ir descongelando diariamente los tubos necesarios para ensayar los aislados. En esta prueba se mide la actividad degradativa de diferentes enzimas, ya que tras la hidrólisis del anillo β-lactámico, la cefotaxima se transforma en una forma carboxílica que hace descender el pH de la solución, lo que le otorga un color amarillo al rojo fenol.
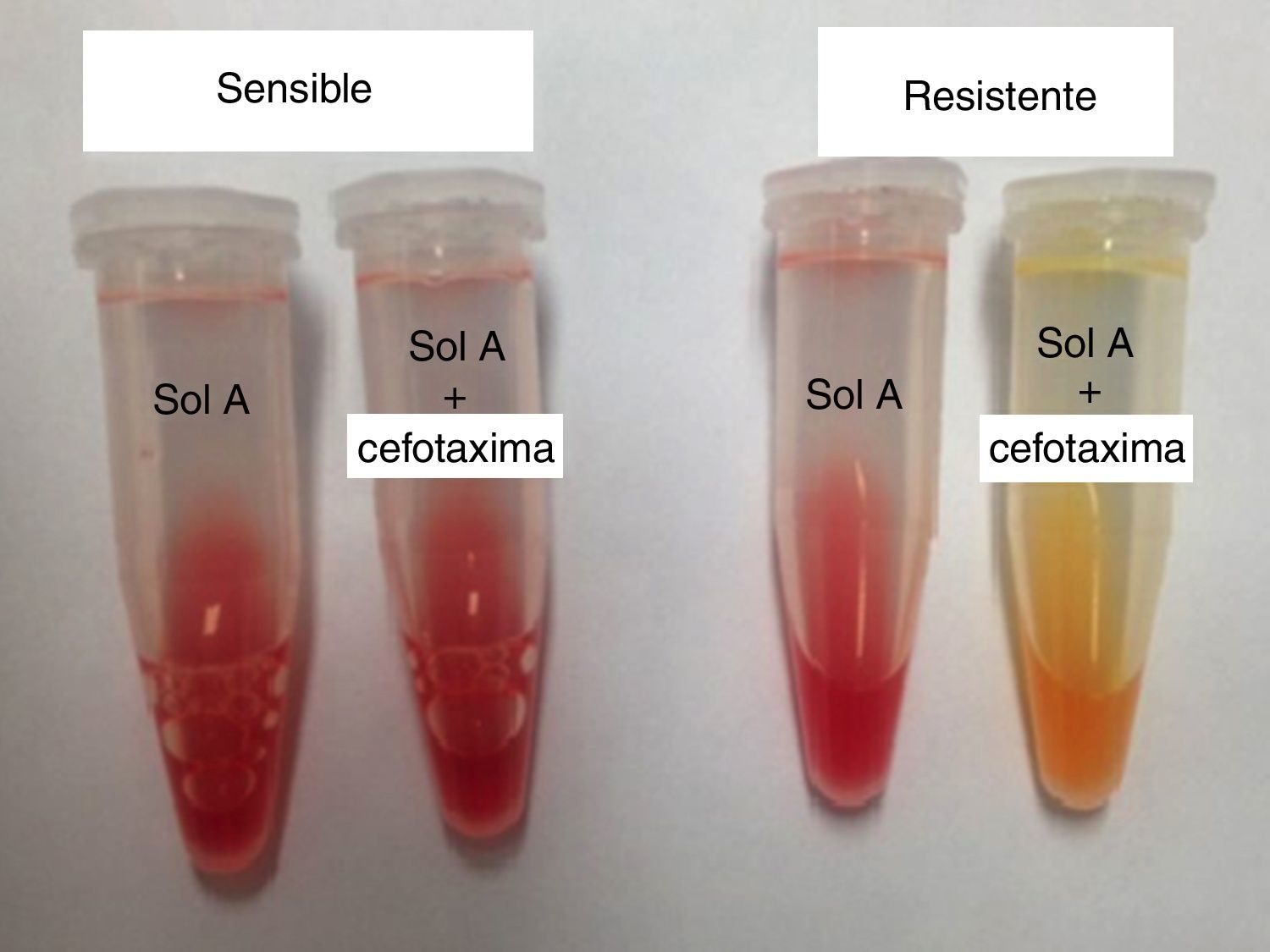
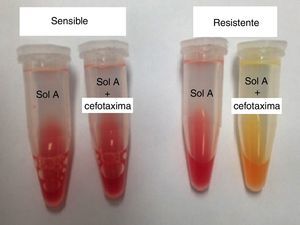
Las enterobacterias ensayadas provenían del cultivo de las muestras de orina en medio cromogénico CHROMagar Orientation, tras incubarlas 18-24 h a 37°C11,17. Para realizar la prueba rápida de cada aislamiento procedente del urocultivo, se tomaron con asa calibrada (Copan Italia Spa, Brescia, Italia) de 1μl al menos 3 colonias aisladas. Estas se suspendieron en la solución in house con cefotaxima y sin este antibiótico, y se agitaron brevemente mediante vórtex a fin de lograr su homogenización. Tras una incubación a 37°C durante 60 min, 2 observadores independientes evaluaron el cambio de color respecto del tubo control y consideraron el viraje de la solución a color amarillo como resultado positivo, la presencia de color rojo como negativo y la aparición de color naranja como indeterminado (fig. 1). Solo se consideraron valorables los resultados si hubo acuerdo entre los observadores.
Simultáneamente a la prueba in house, los aislados fueron identificados a nivel de especie, y se evaluó su sensibilidad antibiótica con el sistema automatizado MicroScan (Beckman-Coulter Inc, Alcobendas, Madrid) empleando los paneles Neg/Urine Combo Type 52 Panel (NC52), cuyos resultados fueron interpretados mediante las normas del CLSI 20153. Adicionalmente, el fabricante indica que existe producción de BLEE cuando la concentración inhibitoria mínima (CIM) de cefotaxima/ceftazidima con ácido clavulánico es 4 veces menor que la CIM de cefotaxima/ceftazidima sola. Las cepas resistentes a cefotaxima o ceftazidima, sin inhibición con el ácido clavulánico, se ensayaron con E-test de cefotaxima/ceftazidima/cefepime con ácido clavulánico en medio de agar Müller-Hinton, en presencia o en ausencia de cloxacilina, para descartar la presencia de BLEE enmascarada por β-lactamasas tipo AmpC3. En caso de discrepancias entre la prueba in house y MicroScan, no se realizó una tercera prueba.
Los datos se analizaron con el paquete del programa estadístico SPSS por MS Windows versión 17.0 (Chicago, EE. UU.). Las variables cuantitativas se describen como la frecuencia de distribución de cada una de las categorías. Se calcularon la sensibilidad (S), la especificidad (E), el valor predictivo positivo (VPP) y el valor predictivo negativo (VPN) para el estudio de la rentabilidad de la prueba in house cuando se produjo el viraje al color amarillo, por separado, con un intervalo de confianza (IC) del 95%.
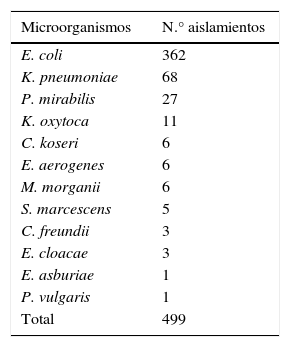
A esta prueba se sometieron 499 aislados de enterobacterias (tabla 1) obtenidos a partir de los urocultivos estudiados. Mediante los paneles de MicroScan se obtuvo que 47 (9,4%) aislados fueron resistentes a cefotaxima; de ellos, 44 (8,8% del total) mostraron fenotipo BLEE (tabla 2) y fueron identificados como Escherichia coli (27), Klebsiella pneumoniae (16) y Klebsiella oxytoca (1). Esto representó el 61,4, el 36,4 y el 2,2% de todos los que mostraron dicho fenotipo y comprendió el 7,4, el 23,5 y 9,2% del total de cada especie, en igual orden. Los 3 aislados restantes correspondieron a E. coli con fenotipo productor de β-lactamasas tipo AmpC.
Distribución por especie de las enterobacterias recuperadas de urocultivos y sometida a la prueba rápida in house
| Microorganismos | N.° aislamientos |
|---|---|
| E. coli | 362 |
| K. pneumoniae | 68 |
| P. mirabilis | 27 |
| K. oxytoca | 11 |
| C. koseri | 6 |
| E. aerogenes | 6 |
| M. morganii | 6 |
| S. marcescens | 5 |
| C. freundii | 3 |
| E. cloacae | 3 |
| E. asburiae | 1 |
| P. vulgaris | 1 |
| Total | 499 |
Distribución de los aislamientos según el resultado de la prueba in house y la presencia de fenotipo BLEE o AmpC
| Prueba in house | Fenotipo BLEE/AmpC | ||
|---|---|---|---|
| BLEE (+) | BLEE (–) | Total | |
| Positiva | 43 0 AmpC | 12 5 AmpC | 55 5 AmpC |
| Negativa | 1 0 AmpC | 443 22 AmpC | 444 22 AmpC |
| Total | 44 0 AmpC | 455 27 AmpC | 499 27 AmpC |
Negrita: número total de cepas con ese fenotipo.
Los resultados de los 2 observadores fueron coincidentes en la prueba in house y en ninguno de los tubos con solución control se produjo viraje a color amarillo o naranja. Se observó que un total de 55 (tabla 2) aislados viraron el medio a color amarillo: 32 fueron E. coli (27 con fenotipo BLEE y 1 con fenotipo de β-lactamasas tipo AmpC); 16 fueron K. pneumoniae (15 con fenotipo BLEE); 3 aislados fueron Enterobacter aerogenes (microorganismo productor de AmpC inducible) y 1 aislado correspondió a K. oxytoca con fenotipo BLEE. También provocó viraje un aislado de cada uno de los siguientes microorganismos: Citrobacter freundii (microorganismo productor de AmpC inducible), Proteus vulgaris y Proteus mirabilis. Por lo tanto, hubo 7 aislados sin fenotipo BLEE o AmpC que hicieron virar el medio a color amarillo, que correspondieron a E. coli (4), K. pneumoniae (1), P. mirabilis (1) y P. vulgaris (1).
Entre los resultados negativos de la prueba in house, se halló que un total de 45 aislados produjeron viraje a color naranja, de los cuales uno fue un aislado de K. pneumoniae con fenotipo BLEE. Con estos datos, la prueba in house mostró una S del 98% (IC del 95%: 93-100) y una E del 97% (IC del 95%: 96-99), con un VPN del 100% (IC del 95%: 99-100) y un VPP del 78% (IC del 95%: 67-89).
Por otro lado, detectamos 27 cepas con fenotipo productor de β-lactamasas tipo AmpC (tabla 2), que incluyeron 3 E. coli, pero solo uno con positividad en la prueba in house. Las restantes cepas fueron de especies con fenotipo de β-lactamasa AmpC inducible, de las que 4 fueron positivas en la prueba, en ausencia de fenotipo BLEE (3 E. aerogenes y 1C. freundii).
Si evaluamos el comportamiento de la prueba in house suprimiendo las cepas con fenotipo productor de β-lactamasas tipo AmpC, el total de aislados ensayados fue de 472, incluyendo 44 con el fenotipo productor de BLEE (9,3%), de los cuales 43 dieron la prueba positiva y solo un aislado de K. pneumoniae viró el medio a color naranja. Con estos datos obtenemos unos valores de S y E del 98% (IC del 95%: 93-100) y 98% (IC del 95%: 97-99), respectivamente, con un VPN del 100% (IC del 95%: 99-100) y un VPP de 84% (IC del 95%: 74-94).
Uno de los principales mecanismos de resistencia de las enterobacterias a los β-lactámicos es la producción de BLEE. La detección fenotípica de producción de BLEE está condicionada por la cantidad de enzima producida y por la concomitancia con otros mecanismos de resistencia, por lo tanto, su evaluación no es sencilla9. Esta detección se basa en la capacidad de esta enzima para hidrolizar cefalosporinas de tercera y de cuarta generación, así como monobactámicos. La disminución de la sensibilidad de la bacteria frente a estos antibióticos se expresa como un aumento de la CIM en el antibiograma mediante microdilución. Además, como estas enzimas son inhibidas por el ácido clavulánico y no poseen capacidad de hidrólisis sobre la cefoxitina, las bacterias productoras serán sensibles a los antibióticos acompañados del inhibidor y a esta cefamicina.
La metodología de detección fenotípica más extendida en los laboratorios se basa en el método de Kirby-Bauer (disco-placa), que emplea una fuente de ácido clavulánico, lo que conlleva al menos 48 h hasta su detección desde la entrada de la muestra en el laboratorio. Igualmente ocurre con la detección mediante microdilución por métodos automatizados, pues es necesario el transcurso de 24 h para obtener el aislado de la enterobacteria y otras 24 h más tras someterlo al antibiograma. En las ITU, donde el tratamiento empírico generalmente tiene una duración también de 48h (cuando son ITU bajas), este tiempo resulta crucial para la instauración de un tratamiento dirigido más correcto, capaz de disminuir las complicaciones, y esto es posible solo si se detecta de forma muy rápida, al menos presuntivamente, la presencia de este mecanismo de resistencia, tan extendido.
En este estudio, hasta el 8,81% de los urocultivos positivos para bacilos gram negativos se asociaron con microorganismos con fenotipo productor de BLEE observado mediante paneles de MicroScan, lo que se corresponde con un 7,4% de prevalencia de dicho fenotipo en E. coli y un 23,5% en K. pneumoniae.
Hay estudios multicéntricos realizados en la última década que arrojan una prevalencia en España de BLEE en las cepas de E. coli y K. pneumoniae del 4 y el 5%, respectivamente1, lo cual demuestra que en los últimos años se ha multiplicado sobre todo la prevalencia en cepas de E. coli en pacientes no hospitalizados14, muchos de ellos asistidos por afecciones del tracto urinario, como lo fue en nuestro caso. Pero lo más sorprendente en nuestro estudio es la gran prevalencia de fenotipo BLEE en K. pneumoniae, que afecta a casi un cuarto de los aislados, a pesar de que no se considera que exista un brote de este patógeno en nuestro entorno. Esto puede deberse a que la gran mayoría de pacientes que asistimos en este hospital son personas de edad avanzada y con múltiples comorbilidades, y, potencialmente, se convierten en reservorios importantes de estos agentes. Por tal motivo, en nuestro hospital se vienen haciendo desde hace un tiempo estudios frecuentes de colonización por este microorganismo en hospitalizados y se procede al aislamiento del paciente si se obtiene un resultado positivo en el estudio de colonización.
Es aconsejable que en los laboratorios se realice con los aislados de enterobacterias una prueba de detección rápida de producción de BLEE, pues de esa manera se puede tener rápidamente una orientación para un tratamiento más adecuado en las primeras 24 h.
La prueba descrita es muy rápida, económica y aplicable a todo tipo de colonias bacterianas capaces de portar genes codificantes de BLEE, como los gram negativos, con la ventaja de que puede ser almacenada en congelador a una temperatura de –80°C durante un período de hasta 3 meses, sin que parezcan alterarse sus propiedades. Por otro lado, la mayoría de las muestras que produjeron el viraje del medio a color amarillo lo hicieron en menos de 15 min, y en ningún caso hubo reversión. De ese modo, el tiempo de orientación diagnóstica sería mucho menor adaptándolo a un protocolo adecuado de práctica asistencial.
Existen estudios que demuestran el buen desempeño de algunas pruebas comerciales rápidas para detectar la producción de BLEE. Estas pruebas también se basan en el cambio de pH y se realizan directamente con muestras de orina, lo cual representaría el siguiente paso en la evaluación de nuestra prueba in house, esto es, aplicarla directamente sobre alícuotas de muestras de orina. En estas muestras se desconoce aún el efecto que tendría sobre la prueba el ácido bórico empleado como conservante, la carga bacteriana requerida para un resultado positivo y la influencia de los cultivos mixtos. El ácido borónico y sus derivados son inhibidores de β-lactamasas tipo AmpC, las que en estado de hiperproducción pueden enmascarar la presencia de BLEE12. Este tipo de betalactamasas, las de tipo AmpC, hidrolizan cefoxitina y cefalosporinas de primera y segunda generación, principalmente, con una menor capacidad de hidrólisis de cefalosporinas de tercera generación, y, sobre todo, de cuarta.
Entre las principales limitaciones de este estudio, cabe señalar que nuestra prueba no fue capaz de diferenciar entre la resistencia a cefalosporinas de tercera generación debida a la producción de BLEE —causa del viraje del medio en la mayoría de los casos— y la resistencia a este tipo de antibióticos por sobreexpresión de β-lactamasas tipo AmpC, ya sea de origen cromosómico inducible o plasmídico, donde el viraje del medio parece tener un patrón inconstante. Pero esto podría solucionarse parcialmente con el uso de medios cromogénicos para enterobacterias (CHROMagar Orientation® o URI select 4®), en los que especies con β-lactamasas tipo AmpC inducible como Enterobacter spp., C. freundii y Serratia marcescens crecen formando colonias de color azul, perfectamente distinguibles de E. coli, cuyas colonias en estos medios son de color rosa. Otras bacterias, como Providencia spp., Morganella spp., Proteus spp., Pseudomonas spp., Acinetobacter spp. o Salmonella spp., también crecen en este tipo de medios, pero sus colonias adquieren un color muy diferente del azul o rosa. En este tipo de medios cromogénicos para enterobacterias, las colonias de las bacterias del género Klebsiella spp. también crecen de color azul, por lo que no son fácilmente distinguibles del grupo de bacterias productoras de β-lactamasas tipo AmpC y el problema reside en la alta prevalencia de producción de BLEE por parte de K. pneumoniae, generalmente dentro de un contexto de brotes intrahospitalarios. Finalmente, solo mediante la prueba in house se detectaron cepas de Enterobacter, Serratia y Citrobacter resistentes a cefotaxima, ya que el sistema MicroScan a veces no detecta cepas de estos géneros con fenotipos de β-lactamasas tipo AmpC inducible.
En nuestro estudio solo 4 especies constituyeron más del 90% de las enterobacterias patógenas, incluyendo todos los aislados con potencial fenotipo productor de BLEE, que son E. coli, K. pneumoniae, K. oxytoca y P. mirabilis5. La aplicación de la prueba sobre aislados de E. coli, fácilmente distinguibles en medio cromogénico por su color rosa, podría ser muy útil, ya que se trata del principal uropatógeno (tanto en la ITU intra como extrahospitalaria8) y de una de las enterobacterias con mayor prevalencia de BLEE, de modo que podríamos obtener una rápida orientación terapéutica en la mayor parte de urocultivos positivos. En nuestro estudio, E. coli fue responsable del 72,5% de las ITU.
No debería afectar significativamente en la aplicación de la prueba rápida la presencia de cepas de E. coli productoras de β-lactamasas tipo AmpC de origen plasmídico, pues en nuestro medio su prevalencia estimada es muy baja12. La sobreexpresión de β-lactamasas tipo AmpC junto con una producción de BLEE de bajo nivel podría dar falsos positivos en este tipo de pruebas basadas en el pH. Desde el punto de vista práctico, el impacto de esta causa de falsos positivos es bajo, ya que todos los resultados de la prueba in house se deben confirmar con los ensayos estandarizados, con la ventaja de poder predecir la falta de actividad clínica de las cefalosporinas.
La prueba in house aquí descrita, basada en el cambio de pH, es útil en nuestro medio para detectar de forma rápida cepas de enterobacterias con disminución de la sensibilidad a cefotaxima, comúnmente aisladas en el laboratorio.
Responsabilidades éticasProtección de personas y animalesLos autores declaran que para esta investigación no se han realizado experimentos en seres humanos ni en animales.
Confidencialidad de los datosLos autores declaran que en este artículo no aparecen datos de pacientes.
Derecho a la privacidad y consentimiento informadoLos autores declaran que en este artículo no aparecen datos de pacientes.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.