Staphylococcus spp. es uno de los patógenos causantes de mastitis bovina y puede presentar multirresistencia a diferentes grupos de antimicrobianos. En este estudio se planteó el objetivo de identificar fenotípicamente aislamientos de Staphylococcus spp. obtenidos de leche bovina y caracterizar el perfil de resistencia antimicrobiana. Se clasificaron 101 cepas mediante pruebas fenotípicas; además, se determinó la resistencia a oxacilina, cefoxitina, penicilina, ampicilina, tetraciclina, kanamicina, sulfametoxazol/trimetoprima, clindamicina y eritromicina por la técnica Kirby Bauer y la presencia de genes de resistencia por PCR. Un total de 65 cepas fueron S. aureus y 36 estafilococos coagulasa negativos (ECN). Se encontraron diferentes patrones de resistencia a los antibióticos evaluados en las cepas de S. aureus y en los ECN; únicamente la resistencia a ampicilina se encontró asociada a la especie (p<0,05). En un total de 101 cepas se detectó el gen mecA en el 27% y se corroboró la portación de aph(3’)-IIIa en el 75,2%; la de aac(6’)/aph(2”)-3 en el 47,4%; la de ant(4’)-Ia en el 32,7%; la de tetM en el 63% y la de tetK en el 43,6%. Sin embargo, no se encontró asociación entre la portación de estos genes y la resistencia a penicilina, ampicilina, cefoxitina, kanamicina y tetraciclina, respectivamente (p>0,05). Por otra parte, se encontró el gen blaZ en el 59,4% de los aislamientos y el gen ermC en el 62,3%, la portación de estos genes sí estuvo asociada a la resistencia a β-lactámicos y macrólidos, respectivamente (p<0,001). En este estudio se encontró multirresistencia antimicrobiana en cepas de S. aureus y de ECN aisladas de leche cruda; este hallazgo impacta en la industria lechera y representa un riesgo para la salud pública.
Staphylococcus spp. is one of the pathogens that cause bovine mastitis and may present multiple resistance to different antimicrobial groups. The aim of this study was to phenotypically identify Staphylococcus spp. isolates obtained from bovine milk and to characterize their antimicrobial resistance profile. The 101 strains were classified by phenotypic tests, their resistance to oxacillin, cefoxitin, penicillin, ampicillin, tetracycline, kanamycin, sulfamethoxazole / trimethoprim, clindamycin and erythromycin was determined by the Kirby-Bauer technique and the presence of resistance genes by PCR. A total of 65 strains was S. aureus and 36 strains were coagulase-negative staphylococci (CoNS). We found different patterns of resistance to antibiotics evaluated in strains of S. aureus and CoNS, only the resistance to ampicillin was found associated with the species (p<0.005). In the 101 strains, the mecA gene was detected in 27%, aph(3’)-IIIa in 75.2%, aac(6’)/aph(2”)-3 in 47.4%, ant(4’)-Ia in 32.7%, tetM in 63% and tetK in 43.6%; however, no association was found with the resistance to penicillin, ampicillin, cefoxitin, kanamycin and tetracycline, respectively (p>0.05). On the other hand, the blaZ gene was found in 59.4% of the 101 strains and the ermCgene in 62.3%, which was associated with resistance to β-lactams and macrolides, respectively (p<0.001). In this study, antimicrobial multiresistance was found in S. aureus and CoNS strains. This finding impacts on the dairy industry, representing a risk to public health.
El género Staphylococcus es causante de enfermedades en humanos y animales32. En sistemas de producción animal es frecuentemente aislado de leche bovina, y su origen puede ser ambiental o relacionarse con infecciones intramamarias11.
La mastitis es una enfermedad de presentación endémica y es considerada una de las más costosas en la industria láctea. Esta afección puede presentarse de forma clínica o subclínica. Las consecuencias económicas de la mastitis están dadas por el tratamiento, las pérdidas en la producción, el descarte temprano de animales, los cambios en la calidad de la leche y el riesgo de otras enfermedades27,54. Como medida de control se usan antibióticos para limitar las infecciones existentes. Si bien la terapia con antibióticos ha ayudado a reducir la incidencia de mastitis, el manejo inadecuado de estos fármacos también ha favorecido la que emerjan especies de Staphylococcus resistentes, lo que puede ocasionar una baja tasa de curación51,58.
Las especies de Staphylococcus poseen diferentes mecanismos de resistencia antimicrobiana: alteración/reemplazo del sitio blanco, impedimento para acceder al blanco, inactivación enzimática del antibiótico, activación de bombas de eflujo transmembrana para bloquear la acción del antimicrobiano59. En Staphylococcus spp. la resistencia a penicilina es atribuida a la presencia del gen blaZ, que codifica la síntesis de β-lactamasas responsables de la hidrólisis del anillo β-lactámico8,20. Otro mecanismo asociado a la resistencia al grupo β-lactámico se adjudica a la presencia del gen mecA, que codifica una proteína PBP2a de baja afinidad por la penicilina. Las cepas de Staphylococcus spp. que poseen el gen mecA se denominan Staphylococcus meticilino-resistentes (SMR). El gen mecA es parte de un elemento genético móvil denominado «casete cromosómico estafilocócico» (SCCmec), el cual puede transmitirse horizontalmente entre poblaciones de Staphylococcus spp.24,33,59.
Otros determinantes genéticos como aac(6′)/aph(2′), aph(3′)-IIIa y ant(4′)-Ia codifican enzimas modificadoras de aminoglucósidos (conocidas como AME, por sus siglas en inglés): acetiltransferasa (AAC), fosfotransferasa (APH) y nucleotidiltransferasa (ANT). Estas enzimas son las responsables de la resistencia a aminoglucósidos en Staphylococcus spp.1,18,26. La resistencia a tetraciclinas y macrólidos en Staphylococcus spp. está asociada a la presencia de los genes tet (M/K) y erm (A/B/C) respectivamente. Los genes erm (A/B/C) codifican metilasas de ARNr implicadas en la modificación del sitio de acción del antibiótico19,40,48,59,61.
En Colombia el uso de antibióticos en el tratamiento de la mastitis bovina no está totalmente regulado y se permite el acceso libre a los antimicrobianos y, en ocasiones, sin supervisión técnica, lo que favorece la aparición de cepas con resistencia a dichos agentes11,47. Algunos autores reportan el uso de antibióticos β lactámicos, aminoglucósidos, macrólidos y tetraciclinas para el tratamiento de la mastitis en sistema de producción bovina en Colombia; asimismo, han mencionado la resistencia a estos antimicrobianos11,42,46,47. La presencia de cepas SRM en Colombia fue reportada por Vanegas et al.55 en muestras de carne molida y de crema de leche, y Herrera et al.29 la documentaron en queso doble crema; estos estudios corresponden a los primeros reportes de SRM en productos de origen bovino.
Para ampliar la información disponible este trabajo se propuso caracterizar 101 aislamientos de Staphylococcus spp. obtenidos de leche bovina mediante la identificación taxonómica y la elaboración de perfiles fenotípicos de resistencia antimicrobiana, evaluando su relación con la presencia de genes de resistencia a diferentes grupos de antibióticos: β-lactámicos, aminoglucósidos, macrólidos y tetraciclinas.
Materiales y métodosAislamientos e identificación microbianaSe utilizaron 101 cepas del género Staphylococcus seleccionadas al azar, que pertenecen al Sistema de bancos de germoplasma de la nación para la alimentación y la agricultura, de la Corporación colombiana de investigación agropecuaria (AGROSAVIA). Ese conjunto de cepas se obtuvo a lo largo del desarrollo de proyectos de investigación en los sistemas de producción de lechería especializada y doble propósito de los departamentos de Cundinamarca, Nariño, Meta y Cesar, a cargo de AGROSAVIA (período 2010-2014). Se chequeó la viabilidad de las cepas y se confirmó el género y la especie mediante cultivo en agar sangre y pruebas de catalasa y coagulasa. Los cultivos se sembraron en medios selectivos y diferenciales (Baird Parker, salado manitol y DNasa)7,30. Se utilizó Staphylococcus aureus ATCC 25923 como cepa de referencia. Adicionalmente, se realizó la identificación hasta el nivel de especie con el kit comercial BD BBL CRYSTAL™ Gram-Positive ID System56.
Pruebas fenotípicas de resistencia a antimicrobianosLa evaluación de la susceptibilidad a los antimicrobianos in vitro fue realizada por incubación en agar Mueller Hinton a 35(±2)°C por la técnica de difusión en disco o Kirby Bauer, con lectura del tamaño de los halos a las 24horas, de acuerdo con lo señalado por el Clinical and Laboratory Standards Institute (CLSI 2012-2018-2019)13–15. Los discos de antibióticos utilizados fueron los siguientes: penicilina G (10U), ampicilina (10U), tetraciclina (30μg), kanamicina (30μg), sulfametoxazol/trimetoprima (25μg), clindamicina (2μg) y eritromicina (15μg). Para la identificación fenotípica de Staphylococcus meticilino-resistentes se emplearon discos de oxacilina (1μg) para Staphylococcus schleiferi y de cefoxitina (30μg) para S. aureus y otros estafilococos coagulasa negativos (ECN), de acuerdo con las instrucciones del CLSI (2019)15.
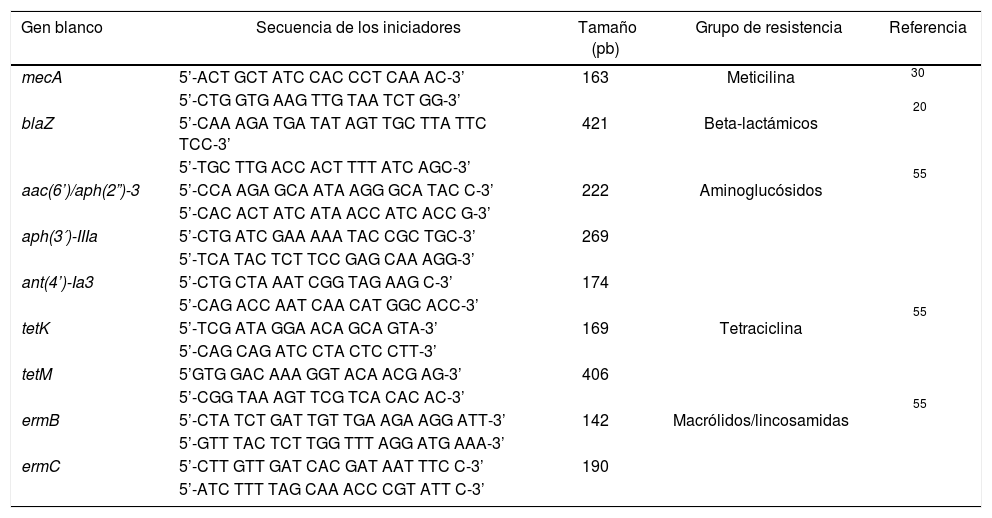
Detección de genes de resistencia a antimicrobianosSe realizó la extracción de ADN por el método fenol-cloroformo-alcohol isoamílico a partir de cultivos puros de Staphylococcus spp. en caldo infusión cerebro-corazón (Oxoid®). Después de un período de incubación de 18h los cultivos se centrifugaron a 8.000rpm×10minutos; la masa celular obtenida fue suspendida en 200μl de buffer TE (Tris 10mM pH 8, EDTA 1mM) con lisozima (2mg/ml) e incubada a 37°C durante 30minutos. Pasado el tiempo de incubación se agregaron 300μl de buffer TE, 4μl proteinasa K (20mg/ml), 30μl SDS al 10%, 80μl bromuro de N,N,N-trimetil,1-hexadecano-amino (CTAB) al 10% y 100μl NaCl 5M. El ADN se extrajo empleando fenol-cloroformo-alcohol isoamílico. El ADN fue precipitado con isopropanol y etanol al 70%, y almacenado a −20°C hasta su procesamiento7,18,31. La amplificación de los genes de resistencia se hizo por PCR empleando iniciadores reportados en otros trabajos5,20,30 (tabla 1).
Iniciadores empleados en la detección de genes de resistencia a antimicrobianos en cepas de Staphylococcus spp
| Gen blanco | Secuencia de los iniciadores | Tamaño (pb) | Grupo de resistencia | Referencia |
|---|---|---|---|---|
| mecA | 5’-ACT GCT ATC CAC CCT CAA AC-3’ | 163 | Meticilina | 30 |
| 5’-CTG GTG AAG TTG TAA TCT GG-3’ | ||||
| blaZ | 5’-CAA AGA TGA TAT AGT TGC TTA TTC TCC-3’ | 421 | Beta-lactámicos | 20 |
| 5’-TGC TTG ACC ACT TTT ATC AGC-3’ | ||||
| aac(6’)/aph(2”)-3 | 5’-CCA AGA GCA ATA AGG GCA TAC C-3’ | 222 | Aminoglucósidos | 55 |
| 5’-CAC ACT ATC ATA ACC ATC ACC G-3’ | ||||
| aph(3′)-IIIa | 5’-CTG ATC GAA AAA TAC CGC TGC-3’ | 269 | ||
| 5’-TCA TAC TCT TCC GAG CAA AGG-3’ | ||||
| ant(4’)-Ia3 | 5’-CTG CTA AAT CGG TAG AAG C-3’ | 174 | ||
| 5’-CAG ACC AAT CAA CAT GGC ACC-3’ | ||||
| tetK | 5’-TCG ATA GGA ACA GCA GTA-3’ | 169 | Tetraciclina | 55 |
| 5’-CAG CAG ATC CTA CTC CTT-3’ | ||||
| tetM | 5’GTG GAC AAA GGT ACA ACG AG-3’ | 406 | ||
| 5’-CGG TAA AGT TCG TCA CAC AC-3’ | ||||
| ermB | 5’-CTA TCT GAT TGT TGA AGA AGG ATT-3’ | 142 | Macrólidos/lincosamidas | 55 |
| 5’-GTT TAC TCT TGG TTT AGG ATG AAA-3’ | ||||
| ermC | 5’-CTT GTT GAT CAC GAT AAT TTC C-3’ | 190 | ||
| 5’-ATC TTT TAG CAA ACC CGT ATT C-3’ |
Se utilizó el análisis de frecuencias por prueba de independencia Chi cuadrado para determinar la asociación entre los perfiles de resistencia o sensibilidad a los antimicrobianos y la presencia o ausencia de genes de resistencia en cepas de S. aureus y en ECN. Se omitieron las respuestas intermedias y solo se consideró la dicotomía resistente/sensible; para este análisis se utilizó el programa Epi Info™ 7.
ResultadosEn función de los cultivos bacteriológicos y las pruebas bioquímicas el 65% (65/101) de las cepas se clasificaron como S. aureus y el 35% (36/101) restante como ECN de las siguientes especies: Staphylococcus haemolyticus (10), Staphylococcus saprophyticus (8), Staphylococcus capitis (6), Staphylococcus xylosus (3), Staphylococcus caprae (2), Staphylococcus simulans (2), Staphylococcus auricularis (1), Staphylococcus sciuri (1), Staphylococcus schleiferi (2) y Staphylococcus warneri (1).
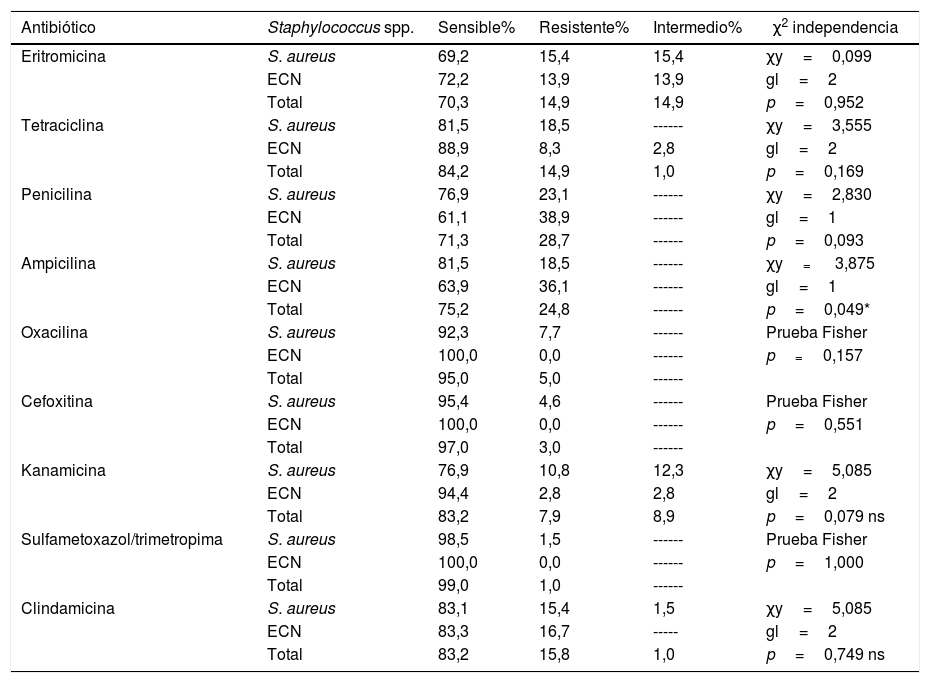
La respuesta de las cepas a los diferentes antibióticos por la técnica de Kirby Bauer fue la siguiente: frente a los 9 antibióticos probados se hallaron cepas de S. aureus resistentes (en un rango de 1,5% a 23,1%, según el caso); en cambio, las cepas de ECN presentaron resistencia a 6 de estos compuestos: eritromicina, tetraciclina, penicilina, ampicilina, kanamicina y clindamicina, en diferentes proporciones (tabla 2). Se investigó la correlación entre la respuesta a 8 de estos antimicrobianos y la especie de Staphylococcus; no se halló correlación (p>0,05), excepto en el caso de la respuesta a la ampicilina, en donde p<0,005 (tabla 2).
Perfil de sensibilidad a diversos antibióticos en las cepas de S. aureus y en estafilococos coagulasa negativos recuperados de muestras de leche bovina, Colombia
| Antibiótico | Staphylococcus spp. | Sensible% | Resistente% | Intermedio% | χ2 independencia |
|---|---|---|---|---|---|
| Eritromicina | S. aureus | 69,2 | 15,4 | 15,4 | χy=0,099 |
| ECN | 72,2 | 13,9 | 13,9 | gl=2 | |
| Total | 70,3 | 14,9 | 14,9 | p=0,952 | |
| Tetraciclina | S. aureus | 81,5 | 18,5 | ------ | χy=3,555 |
| ECN | 88,9 | 8,3 | 2,8 | gl=2 | |
| Total | 84,2 | 14,9 | 1,0 | p=0,169 | |
| Penicilina | S. aureus | 76,9 | 23,1 | ------ | χy=2,830 |
| ECN | 61,1 | 38,9 | ------ | gl=1 | |
| Total | 71,3 | 28,7 | ------ | p=0,093 | |
| Ampicilina | S. aureus | 81,5 | 18,5 | ------ | χy= 3,875 |
| ECN | 63,9 | 36,1 | ------ | gl=1 | |
| Total | 75,2 | 24,8 | ------ | p=0,049* | |
| Oxacilina | S. aureus | 92,3 | 7,7 | ------ | Prueba Fisher |
| ECN | 100,0 | 0,0 | ------ | p=0,157 | |
| Total | 95,0 | 5,0 | ------ | ||
| Cefoxitina | S. aureus | 95,4 | 4,6 | ------ | Prueba Fisher |
| ECN | 100,0 | 0,0 | ------ | p=0,551 | |
| Total | 97,0 | 3,0 | ------ | ||
| Kanamicina | S. aureus | 76,9 | 10,8 | 12,3 | χy=5,085 |
| ECN | 94,4 | 2,8 | 2,8 | gl=2 | |
| Total | 83,2 | 7,9 | 8,9 | p=0,079 ns | |
| Sulfametoxazol/trimetropima | S. aureus | 98,5 | 1,5 | ------ | Prueba Fisher |
| ECN | 100,0 | 0,0 | ------ | p=1,000 | |
| Total | 99,0 | 1,0 | ------ | ||
| Clindamicina | S. aureus | 83,1 | 15,4 | 1,5 | χy=5,085 |
| ECN | 83,3 | 16,7 | ----- | gl=2 | |
| Total | 83,2 | 15,8 | 1,0 | p=0,749 ns |
ECN: estafilococos coagulasa negativos; gl: grados de libertad; ns: no significativo; χ2: Chi-cuadrado.
S. aureus presentó más frecuentemente resistencia o respuesta intermedia a eritromicina que las cepas de ECN (tabla 2). De igual forma, S. aureus presentó más frecuentemente resistencia a tetraciclina que los ECN y una pequeña proporción de ECN expresó respuesta intermedia (tabla 2). A pesar de la diferencia relativa en la respuesta a tetraciclina entre los 2 grupos considerados, esta no fue suficiente para inferir el efecto de la especie de Staphylococcus sobre la respuesta a tetraciclina (p=0,17).
Por otro lado, cuando se analizó la respuesta a los β-lactámicos por la técnica de Kirby Bauer fue más común la resistencia a antibióticos como penicilina y ampicilina en cepas de ECN. En el caso de la respuesta a ampicilina se encontró asociación entre la resistencia y la especie de Staphylococcus con un valor de p=0,049 (tabla 2).
Únicamente se reporta la respuesta a oxacilina de las 2 cepas de S. schleiferi que fueron sensibles; esta misma respuesta se encontró frente a cefoxitina en las 34 cepas de ECN diferentes de S. schleiferi. En cambio, el 4,6% de las cepas de S. aureus mostraron resistencia a cefoxitina; esta respuesta a oxacilina y cefoxitina tampoco se encontró asociada a la especie (p>0,05) (tabla 2).
Fue más frecuente encontrar resistencia o respuesta intermedia a kanamicina entre las cepas de S. aureus que entre las cepas de ECN. A pesar de la mayor proporción de resistencia a kanamicina en S. aureus no se encontró que la especie bacteriana estuviera asociada a la resistencia (p>0,05) (tabla 2). Solo una cepa de S. aureus (1,5%) presentó resistencia a sulfametoxazol-trimetoprima. En contraste, no se observaron ECN resistentes a dicho antimicrobiano.
Hubo mayor resistencia a clindamicina en las cepas de S. aureus que en las cepas de ECN (tabla 2). La respuesta a kanamicina y a sulfametoxazol-trimetoprima no se encontró asociada a la especie de Staphylococcus (p>0,05).
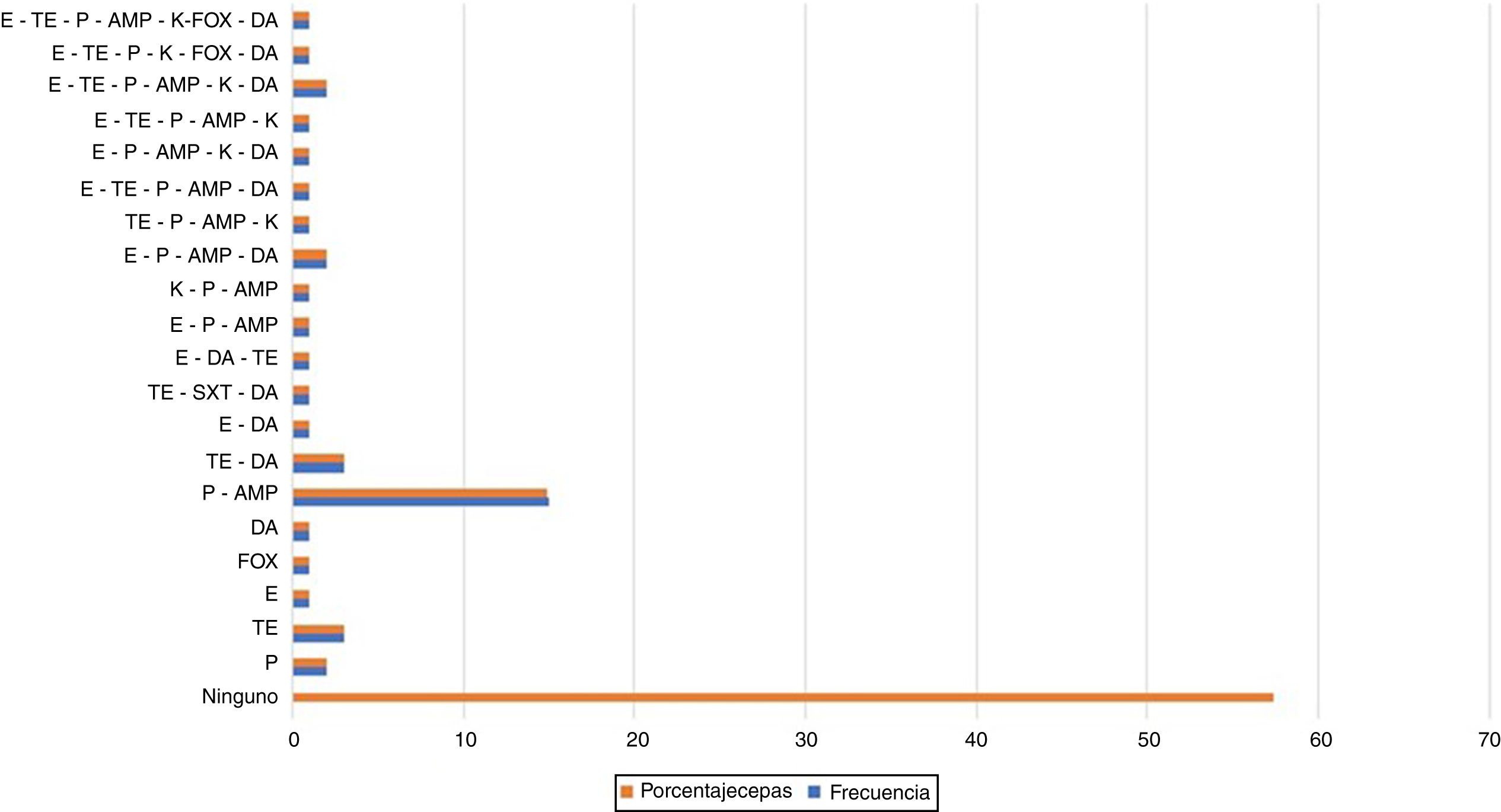
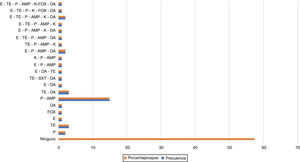
Como se observa en la figura 1, el 57,4% de las cepas evaluadas presentó sensibilidad a todos los antibióticos. En el resto de las cepas se encontraron diversos patrones de resistencia; el más frecuente fue el de resistencia a β lactámicos (un 14,8% de las cepas fueron resistentes a penicilina y ampicilina). Los antibióticos asociados a menor tasa de resistencia fueron sulfametoxazol-trimetoprima y cefoxitina. Se resalta que el número de antibióticos frente a los que estas cepas mostraron resistencia osciló entre 2 y 7 (fig. 1).
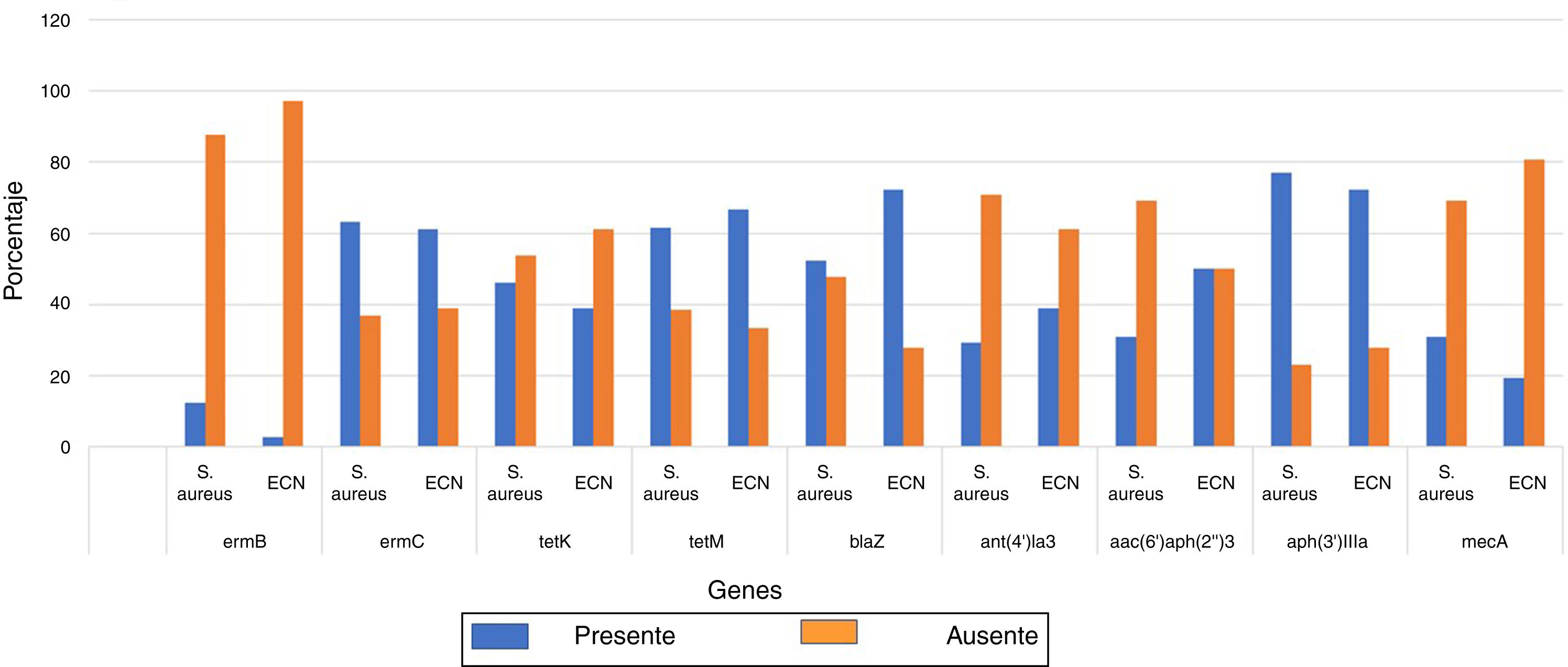
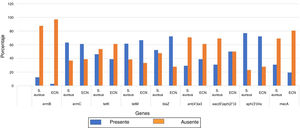
Asimismo, los genes asociados a resistencia más prevalentes fueron aph(3)-IIIa, tetM, ermC y blaZ, asociados a la resistencia a aminoglucósidos, tetraciclinas, macrólidos y ß-lactámicos, respectivamente. Los genes cuya frecuencia de portación fue inferior al 50% fueron aac(6’)/aph(2”)-3 y ant(4’)-Ia, asociados a la resistencia a aminoglucósidos. El gen que se encontró con mayor frecuencia fue el aph(3’)IIIa, y el que se detectó con menor frecuencia fue el gen ermB, relacionados con la resistencia a aminoglucósidos y macrólidos, respectivamente (fig. 2).
En la figura 2 se presenta la distribución de frecuencias de los 9 genes evaluados en S. aureus y en el grupo de ECN; estos genes se encontraron distribuidos indistintamente en estos 2 grupos, por lo tanto, la presencia de los genes no estuvo asociada a la especie de Staphylococcus (p>0,05).
En cuanto al análisis de la relación entre presencia de estos genes y respuesta a los antimicrobianos el fenotipo se tomó como variable ordinal sensible, intermedio o resistente. El gen ermB se encontró en 9 cepas y 6 de estas fueron resistentes a eritromicina. El gen ermC se detectó en 63 cepas y el 14% de estas mostraron resistencia a eritromicina. La presencia de los genes ermB y ermC se encontró asociada a la resistencia a eritromicina (p<0,001, tabla 3).
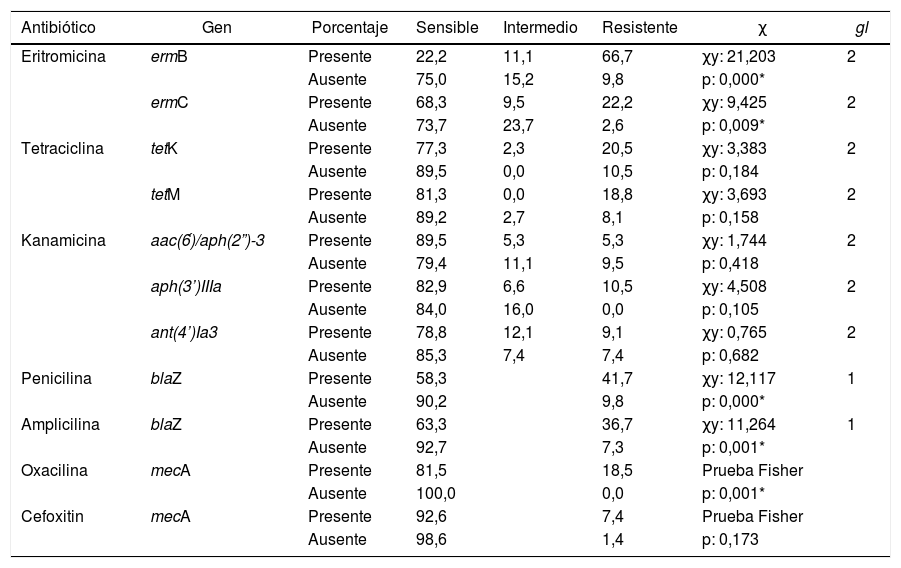
Relación entre portación de genes de resistencia y respuesta a los antimicrobianos en cepas de Staphylococcus spp. recuperadas de leche bovina, Colombia
| Antibiótico | Gen | Porcentaje | Sensible | Intermedio | Resistente | χ | gl |
|---|---|---|---|---|---|---|---|
| Eritromicina | ermB | Presente | 22,2 | 11,1 | 66,7 | χy: 21,203 | 2 |
| Ausente | 75,0 | 15,2 | 9,8 | p: 0,000* | |||
| ermC | Presente | 68,3 | 9,5 | 22,2 | χy: 9,425 | 2 | |
| Ausente | 73,7 | 23,7 | 2,6 | p: 0,009* | |||
| Tetraciclina | tetK | Presente | 77,3 | 2,3 | 20,5 | χy: 3,383 | 2 |
| Ausente | 89,5 | 0,0 | 10,5 | p: 0,184 | |||
| tetM | Presente | 81,3 | 0,0 | 18,8 | χy: 3,693 | 2 | |
| Ausente | 89,2 | 2,7 | 8,1 | p: 0,158 | |||
| Kanamicina | aac(6́)/aph(2”)-3 | Presente | 89,5 | 5,3 | 5,3 | χy: 1,744 | 2 |
| Ausente | 79,4 | 11,1 | 9,5 | p: 0,418 | |||
| aph(3’)IIIa | Presente | 82,9 | 6,6 | 10,5 | χy: 4,508 | 2 | |
| Ausente | 84,0 | 16,0 | 0,0 | p: 0,105 | |||
| ant(4’)Ia3 | Presente | 78,8 | 12,1 | 9,1 | χy: 0,765 | 2 | |
| Ausente | 85,3 | 7,4 | 7,4 | p: 0,682 | |||
| Penicilina | blaZ | Presente | 58,3 | 41,7 | χy: 12,117 | 1 | |
| Ausente | 90,2 | 9,8 | p: 0,000* | ||||
| Amplicilina | blaZ | Presente | 63,3 | 36,7 | χy: 11,264 | 1 | |
| Ausente | 92,7 | 7,3 | p: 0,001* | ||||
| Oxacilina | mecA | Presente | 81,5 | 18,5 | Prueba Fisher | ||
| Ausente | 100,0 | 0,0 | p: 0,001* | ||||
| Cefoxitin | mecA | Presente | 92,6 | 7,4 | Prueba Fisher | ||
| Ausente | 98,6 | 1,4 | p: 0,173 |
gl: grados de libertad; χ2: Chi-cuadrado.
La presencia de los genes tetK y tetM no se encontró asociada a la resistencia a tetraciclina, la característica de sensible o resistente fue independiente de la presencia o ausencia de dichos genes (p>0,05; tabla 3). El grupo de genes aph(3′)-IIIa, aac(6́)/aph(2”)-3 y ant(4’)-Ia3 no presentó relación causal con la resistencia a kanamicina (p>0,05; tabla 3).
El gen mecA, presente en el 26,7% de las cepas evaluadas, no se encontró asociado a la resistencia a ampicilina, penicilina o cefoxitina (p>0,05; tabla 3). De las 27 cepas que portaban el gen mecA el 26% tenían el gen ant(4’)-Ia3, el 59% los genes aac(6’)/aph(2”)-3 y el 93% el gen aph(3’)-IIIa. De igual forma, estas cepas portaban los genes ermB (26%) y ermC (48%) (datos no mostrados).
El gen blaZ se detectó en 60 cepas de Staphylococcus spp. y la resistencia a penicilina y a ampicilina fue superior al 20%. Esta respuesta permitió establecer que la presencia del gen blaZ estuvo asociada a la resistencia a la penicilina y la ampicilina (p<0,05; tabla 3) en este conjunto de aislamientos.
DiscusiónEn la caracterización de un conjunto de 101 aislamientos de Staphylococcus spp., obtenidos de leche cruda bovina en Colombia, la especie predominante fue S. aureus, la cual se asocia a mastitis bovina. La mastitis es considerada una enfermedad de importancia económica, de alto impacto en la salud pública debido al riesgo de infección a través del consumo de leche y derivados lácteos9.
El hallazgo de S. aureus como microorganismo prevalente en muestras de leche cruda ya ha sido reportado en Colombia en sistemas de producción lechera de Montería (Córdoba)11. En el oriente antioqueño, en cambio, la prevalencia de ECN fue de un 23% y la de S. aureus de un 10,3% en 226 muestras tomadas en una finca de lechería especializada53. En un estudio similar, en una microcuenca lechera del norte de Antioquia, de 648 cultivos obtenidos de leche cruda un 10,2% fueron ECN42. En otro estudio realizado en el departamento del Tolima reportaron el aislamiento de ECN y de S. aureus en el 49,3% y 38% de muestras de leche cruda bovina, respectivamente (36/73 y 28/73)46.
La prevalencia fue de 8,3% para S. aureus y 0,46% para ECN en un estudio semejante al aquí descrito. En ese estudio se investigó un total de 5.396 muestras de leche cruda en el altiplano boyacense3. La dinámica epidemiológica de microorganismos patógenos en leche depende del sistema de producción, la región geográfica y los factores relacionados con el manejo de los animales38, lo que podría explicar la discrepancia de resultados al comparar diversos estudios.
La presencia de S. aureus en leche bovina está asociada a la capacidad de este microorganismo de causar infecciones persistentes por expresión de factores de virulencia, los que ayudan a la cronicidad de las infecciones y permiten evadir la respuesta inmunitaria del huésped, con la consecuente supervivencia del patógeno en células de la glándula mamaria bovina. Los animales infectados pueden servir como reservorio y contribuir a la transmisión de este microorganismo a otros animales6,21. En países de Latinoamérica con gran producción lechera, como Brasil y Argentina, el agente etiológico más predominante en animales con mastitis bovina es S. aureus35,39.
En la caracterización de aislamientos de Staphylococcus spp. en leche cruda bovina también se identificaron ECN, considerados patógenos menores y asociados a infecciones intramamarias clínicas o subclínicas44,49,57. Dentro de los ECN la especie que se identificó en mayor número fue S. haemolyticus, en coincidencia con lo reportado previamente33,44,52. Las cepas de S. haemolyticus tienen diversos hábitats y han sido aisladas del ambiente y de sitios extramamarios, incluyendo el ápice de los pezones y la piel de la ubre, lo que explica por qué es común identificar esta especie en la leche bovina54,57.
En lo relacionado con la sensibilidad a diferentes grupos de antimicrobianos, nuestros resultados difieren de los reportados por Calderón et al.11 en sistemas de producción doble propósito en el trópico bajo colombiano, en el cual el 72,4% de las cepas de S. aureus recuperadas mostraron sensibilidad frente a cefoxitina, gentamicina, eritromicina, amoxicilina y ácido clavulánico; la frecuencia de cepas resistentes a tetraciclinas fue del 14,8% y a eritromicina del 0,3%11. De igual forma, Ramírez et al.43 encontraron un 28,6% de aislamientos de S. aureus sensibles a penicilina, un 24,3% tenían sensibilidad intermedia y un 57,1% se mostraron resistentes en un estudio realizado en la cuenca lechera de San Pedro de los Milagros (Antioquia)43.
En cuanto a la respuesta a los diferentes antimicrobianos en las cepas de ECN evaluadas en el presente estudio es de resaltar el hallazgo de resistencia a la penicilina y la eritromicina, en concordancia con lo informado por Sánchez et al.46, quienes reportaron un 61% de cepas de ECN con resistencia a penicilina. Otro estudio del mismo grupo47 muestra en ECN una prevalencia de aislamientos resistentes a penicilina y eritromicina del 58% y 52%, respectivamente. Las cepas de ECN con frecuencia se aíslan de leche procedente de bovinos con mastitis subclínica, y las especies aisladas podrían variar entre localidades y sistemas de producción11,42,45. Así, por ejemplo, en Argentina se ha encontrado resistencia a penicilina en cepas de S. chromogenes y S. haemolyticus44.
Por otro lado, en lo referido a determinantes de resistencia a macrólidos, el gen ermC fue uno de los más frecuentes y estuvo asociado de manera significativa a la resistencia a eritromicina, datos similares a los reportados por varios autores16,18,23,40.
Piatkowska et al.40 y Ding et al.16 reportan que uno de los mecanismos de resistencia a macrólidos utilizados por cepas de Staphylococcus spp. causantes de mastitis bovina es la modificación del sitio blanco, ya sea por metilación o mutación, lo que ocasiona cambios estructurales en el ARN ribosomal para impedir la unión de los macrólidos. Para esto, las cepas de Staphylococcus spp. sintetizan un tipo de enzimas denominadas metilasas ribosomales, codificadas por los genes erm16,40.
Dentro de las cepas de Staphylococcus evaluadas en el presente estudio se encontraron cepas sensibles a la eritromicina que portaban los genes erm, lo cual puede estar relacionado con mutaciones en una región cercana al sitio de metilación en el gen 23S-ARNr16. Sin embargo, existen otros marcadores genéticos como msrA que también se asocian a la resistencia a macrólidos, lo cual ha sido descrito previamente18.
En el presente estudio no se encontró asociación significativa entre la presencia de los genes tetK o tetM y la resistencia a la tetraciclina en las cepas de Staphylococcus spp. provenientes de leche, en concordancia con lo reportado por otros autores14,18. Asimismo, se encontraron cepas sensibles a las tetraciclinas que portaban los genes tet; esta diferencia entre expresión fenotípica y presencia de genes tet ha sido reportada previamente por Duran et al.18y Feng et al.23.
También se encontró una discrepancia en las cepas de Staphylococcus spp. aisladas de leche cruda entre la expresión fenotípica y la presencia de determinantes genéticos de resistencia a aminoglucósidos, datos similares han sido reportados por Choi et al.12. De los genes AME evaluados el que se detectó con mayor frecuencia fue aph(3′)-IIIa. Estos resultados son concordantes con los de otros trabajos que reportan este gen como uno de los más prevalentes en cepas de Staphylococcus17. Sin embargo, en esta investigación, y a diferencia de la citada anteriormente, el gen aph(3′)-IIIa no estuvo asociado a la resistencia a kanamicina. Adicionalmente, la presencia de los otros genes AME, como aac(6’)/aph(2”)-3 y ant(4’)-Ia3 está relacionada con la resistencia a otros antimicrobianos del grupo de aminoglucósidos, como gentamicina, amikacina y neomicina, entre otros12,17,48.
Por otro lado, la resistencia al grupo de antibióticos β-lactámicos en cepas de Staphylococcus spp. ocurre mayormente por producción de β-lactamasas codificadas por el gen blaZ7,22. Los antibióticos β-lactámicos forman parte de los compuestos activos más utilizados para el control de infecciones de glándula mamaria bovina en Colombia11,46,47. Probablemente, el uso indiscriminado de estos antibióticos en medicina veterinaria ocasiona presión de selección y, por tanto, contribuye al desarrollo de resistencia, lo que sugiere que su empleo debe ser controlado7,22.
El frecuente uso de estos antimicrobianos en sistemas de producción bovina podría explicar por qué más del 50% de las cepas de Staphylococcus caracterizadas en este trabajo portaban el gen blaZ, asociado a la resistencia a la penicilina y/o a la ampicilina. Estos resultados son concordantes con los de otros estudios, en los que la resistencia a β-lactámicos se debió a la presencia del gen blaZ18,22,60. No obstante, se encontraron cepas sensibles a penicilina y/o ampicilina que portaban el gen blaZ y, de modo inverso, hubo cepas que fueron blaZ negativas, pero mostraron resistencia a estos antibióticos. Esta discrepancia entre las pruebas fenotípicas y genotípicas ha sido reportada anteriormente y está relacionada con mutaciones puntuales en el lugar de adquisición de los genes22,23.
En este estudio se logró la identificación de S. aureus meticilinorresistentes (SAMR) y ECN meticilinorresistentes aislados de leche bovina mediante la detección del gen mecA, el cual se identificó en el 27% de las cepas evaluadas. En Colombia no se han encontrado SAMR en muestras de leche bovina de acuerdo con lo reportado por varios autores, como Calderón et al.11 y Monistero et al.37. En cambio, se ha reportado el aislamiento de SAMR a partir de productos lácteos y carne bovina29,55. Diversos autores en el mundo han informado la presencia de SAMR y de ECN meticilinorresistentes aislados de leche cruda bovina24,33,36.
Se encontraron en este análisis cepas resistentes a cefoxitina que no portaban el gen mecA; asimismo, cepas mecA positivas fueron sensibles a dicho agente. Este fenómeno de heterorresistencia implicado en la diferencia de expresión fenotípica ya ha sido observado en muestras tomadas de sistemas de producción pecuarios, y puede estar relacionado con mutaciones puntuales en los genes asociados a la resistencia, lo que ocasiona que algunas cepas de un mismo clon no puedan expresar la resistencia antimicrobiana2,7,33,41. Otra razón para la discrepancia de resultados entre pruebas fenotípicas y genotípicas podría vincularse con la presencia de algún otro marcador asociado a la resistencia a meticilina en las cepas SMR, como el gen homólogo mecC4,24,28.
En cepas de este estudio clasificadas como Staphylococcus meticilinorresistentes se observó la presencia de genes AME. Muchos autores han reportado la estrecha relación entre la presencia de dichos genes y la resistencia a la meticilina en cepas de Staphylococcus, lo cual está relacionado con la localización cercana entre el gen mecA y los genes AME12, así como también la presencia de los genes ermB y ermC1,48. La composición del SCCmec permite la incorporación de otros marcadores de resistencia, hecho que explica por qué muchas cepas de SMR pueden ser resistentes a otros antimicrobianos, como aminoglucósidos y macrólidos, entre otros10,24,33.
Debido a la naturaleza móvil del SCCmec de la cual forma parte el gen mecA, la resistencia puede ser transferida de una cepa a otra; asimismo, muchas especies de ECN y de S. aureus pueden encontrarse tanto en humanos como en animales, por lo tanto, la presencia de cepas resistentes puede representar un riesgo en salud pública y medicina veterinaria31,50,52. Otra forma de diseminación de la resistencia entre especies de Staphylococcus es por ECN, que pueden servir como reservorio de determinantes genéticos de resistencia antimicrobiana, los cuales pueden ser transferidos a otras especies como S. aureus52.
La adquisición de determinantes genéticos asociados a la resistencia antimicrobiana en cepas de Staphylococcus y sus mecanismos son tan complejos que la presencia o ausencia de un gen de resistencia no indica que la cepa sea resistente o sensible a un antibiótico en particular23,25. Por tanto, el diagnóstico de mastitis puede complementarse con el aislamiento y la identificación de los microorganismos patógenos causantes de la infección, así como con la determinación de sus perfiles fenotípicos de resistencia a antimicrobianos. Realizar de manera simultánea evaluaciones genotípicas puede ayudar a la caracterización de poblaciones con resistencia antimicrobiana4,34.
ConclusiónLos diferentes patrones fenotípicos y genéticos en relación con la resistencia a los antimicrobianos encontrados en esta investigación sugieren una alta heterogeneidad microbiana interespecies; por lo tanto, las investigaciones podrían estar dirigidas a determinar esas diferencias entre especies, lo que ayudaría a establecer programas de control de mastitis más específicos y efectivos.
La detección de cepas de Staphylococcus de origen animal con resistencia a diversos antimicrobianos cobra particular interés por su posible participación y compromiso epidemiológico en la interacción y distribución de este tipo de cepas en la clínica intrahospitalaria humana, en la comunidad abierta y en la salud animal. Teniendo en cuenta que uno de los factores que contribuyen a que emerja multirresistencia es el uso indiscriminado de antimicrobianos, tanto en la práctica clínica humana como en veterinaria. Se hace necesario prestar atención al uso adecuado de medicamentos en la terapia de enfermedades, además de contemplar otras alternativas al uso de los antibióticos, como el mejoramiento de las condiciones de higiene y la realización de cambios en las prácticas ganaderas que reduzcan la posibilidad de presentación de mastitis y otras enfermedades frecuentes.
FinanciaciónEste estudio fue financiado con recursos del Ministerio de Agricultura y Desarrollo Rural a través del Instituto Colombiano Agropecuario (ICA) y ejecutado por la Corporación Colombiana de Investigación Agropecuaria AGROSAVIA.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.
Los autores desean agradecer al Ministerio de Agricultura y Desarrollo Rural de Colombia (MADR) por financiar este estudio a través del Instituto Colombiano Agropecuario (ICA). Además, queremos expresar nuestro agradecimiento a la Corporación Colombiana de Investigación Agropecuaria (AGROSAVIA) por proporcionar toda la infraestructura necesaria para llevar a cabo la investigación.