Staphylococcus aureus es un patógeno responsable de diversos cuadros clínicos. Los marcadores moleculares son útiles para el estudio de la epidemiología microbiana. Se estudiaron 22 aislamientos de S. aureus resistentes a meticilina (SARM) y 23 sensibles a meticilina (SASM) mediante mecA, cassette SCCmec, leucocidina de Panton Valentine (LPV) y polimorfismo spa; se analizaron datos de los pacientes. SASM predominó en muestras distintas de piel y partes blandas de internados, mientras SARM en partes blandas. Predominó el SCCmec tipo IV seguido del I. Se encontró baja presencia de LPV. En SARM hubo 11 tipos de spa diferentes, t019 fue el más frecuente y en pacientes ambulatorios. En SASM se hallaron 17 tipos con prevalencia del t189. El spa t002 estuvo presente en SASM y SARM. Se hallaron 11 tipos de spa no reportados en nuestro país.
Staphylococcus aureus is a pathogen associated a different kind of infection. Molecular markers are useful tools to study microbial epidemiology. Twenty two methicillin-resistant S. aureus (MRSA) and 23 methicillin-susceptible S. aureus (MSSA) were studied by mecA gene, SCCmec cassette, Panton Valentine leucocidin (PVL) and spa polymorphism. The clinical data patients were analyzed. MSSA was prevalent in samples different from skin and soft tissue (SST) and in hospitalized patients, whereas MRSA in SST. SCCmec type IV was predominant, followed by type I. Low presence of PVL was found. In MRSA 11 different types of spa were detected, t019 was the most frequent and associated with outpatient, 17 types were found in MSSA and t189 was prevalent. spa t002 was present in MSSA and MRSA. We found 11 types of spa not reported in our country.
Staphylococcus aureus coloniza la piel y las mucosas de humanos y animales, y es causa frecuente de una amplia gama de infecciones. Su patogénesis ha sido atribuida a la capacidad de generar resistencia (R) y a sus factores de virulencia10.
S. aureus resistente a meticilina (SARM) se asoció al ámbito hospitalario (SARM-AH) pero desde los años 90ha emergido como agente de infecciones adquiridas en la comunidad (SARM-AC). SARM-AH suele portar cassettes de R de mayor tamaño (I, II y III) e incluir R a múltiples antimicrobianos (AM). SARM-AC suele llevar cassettes chicos (IV o V) sin R asociada y con frecuencia porta la leucocidina de Panton Valentine (LPV)10.
La proteína A protege a la bacteria de los mecanismos de defensa del huésped y está codificada por el gen polimórfico spa. Su secuenciación emerge como un marcador prometedor para la tipificación de cepas y presenta buena correlación con las metodologías de referencia MLST y PFGE5,11.
En Argentina hasta el año 2008 el 50% de los aislamientos nosocomiales eran SARM, siendo prevalente el clon epidémico chileno/cordobés (ST5-SCCmec I)13. En el año 2009 hubo una diseminación de 2 clones SARM-AC: ST5-SCCmecIV-LPV(+)-spa t31112 y ST30-SCCmecIVc-LPV(+)-spa t0193, con importantes diferencias regionales y según los grupos etarios. Entre 2010-2011 predominó este último en pacientes adultos sin factores de riesgo con infecciones invasivas de inicio en la comunidad4.
La importancia de conocer la diversidad genómica de los clones circulantes a nivel local radica en la necesidad de implementar terapias empíricas y medidas de control adecuadas.
El objetivo del presente trabajo fue caracterizar una población de S. aureus. Para ello se propuso: a) el estudio genotípico de mecA, cassette SCCmec, LPV y polimorfismo spa; y b) relacionar datos de los pacientes (procedencia y tipo de muestra) con los resultados feno y genotípicos de los aislamientos.
Se estudiaron 45 aislamientos consecutivos de importancia clínica de pacientes adultos (16-65 años) que concurrieron al hospital Dr. José María Cullen nivel II de la ciudad de Santa Fe, Argentina, durante los meses de enero-abril de 2013. Se estudió un aislamiento por paciente. Fueron caracterizados fenotípicamente en la sección de microbiología del laboratorio central del hospital resultando 22 SARM y 23 S. aureus sensibles a meticilina (SASM). Se registraron los datos de los pacientes (procedencia y tipo de muestra) y el perfil de R de los aislamientos a los AM no betalactámicos obtenido mediante método automatizado Phoenix (BD). El estudio genotípico se realizó en la cátedra de bacteriología clínica de la FBCB-UNL.
Se realizó la extracción de ADN genómico mediante lisis celular utilizando proteinasa K (Promega-Biodynamics SRL, BA, Argentina) y lisozima (Promega-Biodynamics SRL, BA, Argentina) y posterior purificación con solventes orgánicos.
Por técnicas de PCR se corroboró la R a meticilina detectando el gen mecA15, se caracterizó el SCCmec9, se determinó la portación de los genes lukS/F-PV que codifican a la LPV7 y se amplificó el gen spa6. Se cuantificaron los productos spa por espectrofotometría y se enviaron a secuenciar (Unidad de Genómica del Instituto de Biotecnología-CICVyA, INTA Hurlingham, BA, Argentina). Se tipificaron las secuencias con el sitio de referencia http://www.spaserver.ridom.de/. Se analizaron los datos accesorios del mismo: asociación al tipo MLST, frecuencia de aparición, pertenencia a clones, países de procedencia, presencia en SASM o SARM.
Se determinó la frecuencia relativa porcentual de cada variable categórica. Se establecieron las relaciones entre dichas variables con la prueba exacta de Fisher. Se consideró estadísticamente significativo un valor de p≤0,05. Para dicho análisis las variables nominales fueron divididas en 2 grupos: el tipo de muestra de donde provienen los aislamientos: «PPB» y «muestras distintas a PPB», los tipos spa en «spa X» (siendo X los distintos tipos spa más frecuentes) y «otros tipos spa» y los tipos de cassettes en «SCCmec IV/V» y “demás SCCmec”.
En las tablas 1 y 2 se exponen los datos recolectados y los hallazgos del trabajo.
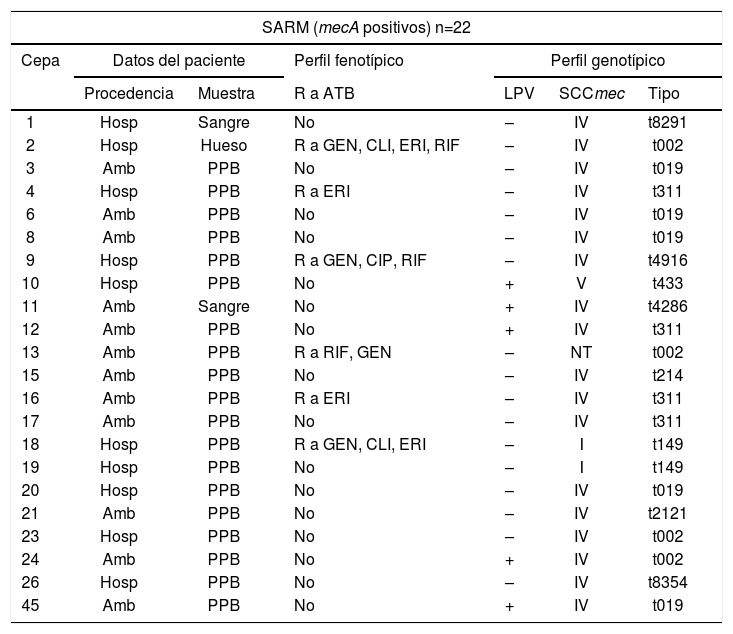
Datos de los pacientes con infecciones por SARM, sensibilidad antimicrobiana y caracterización genotípica de los aislamientos
| SARM (mecA positivos) n=22 | ||||||
|---|---|---|---|---|---|---|
| Cepa | Datos del paciente | Perfil fenotípico | Perfil genotípico | |||
| Procedencia | Muestra | R a ATB | LPV | SCCmec | Tipo | |
| 1 | Hosp | Sangre | No | – | IV | t8291 |
| 2 | Hosp | Hueso | R a GEN, CLI, ERI, RIF | – | IV | t002 |
| 3 | Amb | PPB | No | – | IV | t019 |
| 4 | Hosp | PPB | R a ERI | – | IV | t311 |
| 6 | Amb | PPB | No | – | IV | t019 |
| 8 | Amb | PPB | No | – | IV | t019 |
| 9 | Hosp | PPB | R a GEN, CIP, RIF | – | IV | t4916 |
| 10 | Hosp | PPB | No | + | V | t433 |
| 11 | Amb | Sangre | No | + | IV | t4286 |
| 12 | Amb | PPB | No | + | IV | t311 |
| 13 | Amb | PPB | R a RIF, GEN | – | NT | t002 |
| 15 | Amb | PPB | No | – | IV | t214 |
| 16 | Amb | PPB | R a ERI | – | IV | t311 |
| 17 | Amb | PPB | No | – | IV | t311 |
| 18 | Hosp | PPB | R a GEN, CLI, ERI | – | I | t149 |
| 19 | Hosp | PPB | No | – | I | t149 |
| 20 | Hosp | PPB | No | – | IV | t019 |
| 21 | Amb | PPB | No | – | IV | t2121 |
| 23 | Hosp | PPB | No | – | IV | t002 |
| 24 | Amb | PPB | No | + | IV | t002 |
| 26 | Hosp | PPB | No | – | IV | t8354 |
| 45 | Amb | PPB | No | + | IV | t019 |
R a ATB: resistencia a antibiótico no beta-lactámicos; LPV: leucocidina de Panton-Valentine; SCCmec: staphylococcal cassete chromosome mec; Hosp: procedencia hospitalaria; No: no presenta resistencia conjunta; GEN: gentamicina; CLI: clindamicina; ERI: eritromicina; RIF: rifampicina; Amb: procedencia ambulatoria; PPB: infecciones de piel y partes blandas; CIP: ciprofloxacina; +/–: presencia/ausencia de LPV; NT: no tipificable.
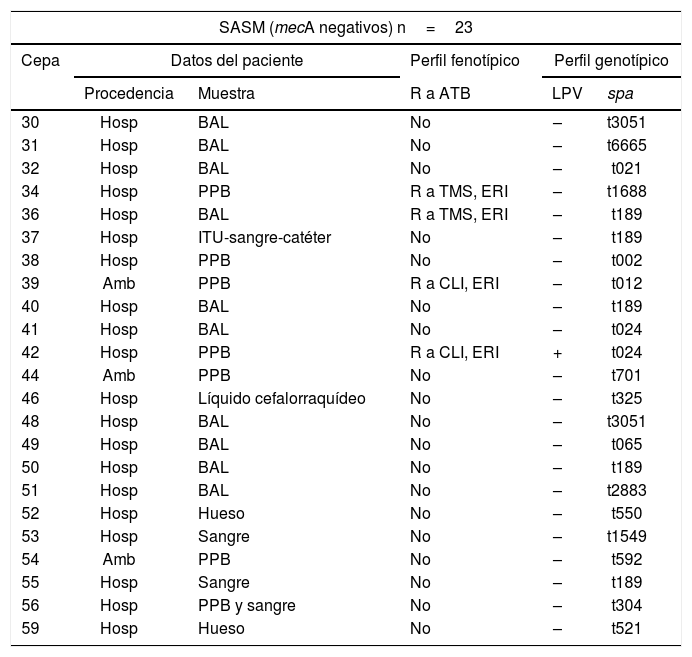
Datos de los pacientes con infecciones por SASM, sensibilidad antimicrobiana y caracterización genotípica de los aislamientos
| SASM (mecA negativos) n=23 | |||||
|---|---|---|---|---|---|
| Cepa | Datos del paciente | Perfil fenotípico | Perfil genotípico | ||
| Procedencia | Muestra | R a ATB | LPV | spa | |
| 30 | Hosp | BAL | No | – | t3051 |
| 31 | Hosp | BAL | No | – | t6665 |
| 32 | Hosp | BAL | No | – | t021 |
| 34 | Hosp | PPB | R a TMS, ERI | – | t1688 |
| 36 | Hosp | BAL | R a TMS, ERI | – | t189 |
| 37 | Hosp | ITU-sangre-catéter | No | – | t189 |
| 38 | Hosp | PPB | No | – | t002 |
| 39 | Amb | PPB | R a CLI, ERI | – | t012 |
| 40 | Hosp | BAL | No | – | t189 |
| 41 | Hosp | BAL | No | – | t024 |
| 42 | Hosp | PPB | R a CLI, ERI | + | t024 |
| 44 | Amb | PPB | No | – | t701 |
| 46 | Hosp | Líquido cefalorraquídeo | No | – | t325 |
| 48 | Hosp | BAL | No | – | t3051 |
| 49 | Hosp | BAL | No | – | t065 |
| 50 | Hosp | BAL | No | – | t189 |
| 51 | Hosp | BAL | No | – | t2883 |
| 52 | Hosp | Hueso | No | – | t550 |
| 53 | Hosp | Sangre | No | – | t1549 |
| 54 | Amb | PPB | No | – | t592 |
| 55 | Hosp | Sangre | No | – | t189 |
| 56 | Hosp | PPB y sangre | No | – | t304 |
| 59 | Hosp | Hueso | No | – | t521 |
R a ATB: resistencia a antibiótico no beta-lactámico; LPV: leucocidina de Panton-Valentine; SCCmec: staphylococcal cassete chromosome mec; Hosp: procedencia hospitalaria; No: no presenta resistencia conjunta; CLI: clindamicina; ERI: eritromicina; Amb: procedencia ambulatoria; PPB: infecciones de piel y partes blandas; +/–: presencia/ausencia de LPV; BAL: lavado broncoalveolar; TMS: trimetoprima/sulfametoxazol; ITU: infección del tracto urinario.
Los SASM se aislaron principalmente de muestras distintas a PPB (73,91%) y fueron de procedencia hospitalaria (p=0,0113); la muestra prevalente fue BAL y todas de pacientes de terapia intensiva sometidos a ventilación mecánica y con infección precoz. La mayoría de los SARM se aislaron de muestras de PPB (86,36%) y fueron de ambas procedencias. Los pacientes ambulatorios, cuyos aislamientos de SASM y SARM provenían de PPB y un único de sangre, no residían en geriátricos, se movilizaban por sus propios medios y no habían tenido contacto con el sistema sanitario. Es necesario aclarar que cuando se expresa procedencia hospitalaria el aislamiento se obtuvo luego de las 48horas de haber ingresado al hospital. En el conjunto de SARM y SASM se detectó una asociación estadística altamente significativa entre el tipo de la muestra y la procedencia del paciente (p=0,0003) acorde con la bibliografía que reporta a SARM-AC como agente etiológico frecuente de infecciones cutáneas.
Se encontró con mayor frecuencia el SCCmec IV (81,82%) seguido del SCCmec I (9,09%), al igual que en la investigación previa realizada en el laboratorio de nuestra cátedra2. Un único aislamiento fue tipo V (4,55%) y una cepa resultó no tipificable.
En SASM y SARM se halló una baja tasa de aislamientos resistentes a antibióticos no beta-lactámicos: 4 SASM (17,39%), 3 de procedencia hospitalaria y uno ambulatorio, y 4 SARM (18,18%), 3 hospitalarios —uno SCCmec I y 2 SCCmec IV— y el cuarto de procedencia ambulatoria con SCCmec incierto. El hallazgo de cepas SASM y SARM con multirresistencia entre los pacientes ambulatorios podría constituir una alerta sobre la diseminación de estas como causantes de infección en la comunidad.
Cinco SARM portaron LPV (22,73%). En la última década se produjo un incremento significativo de las infecciones por SARM-LPV (+) detectadas en la comunidad debido a la diseminación de ciertos linajes, generalmente asociado a lesiones cutáneas en pacientes jóvenes y niños12. La prevalencia de LPV fue baja, al igual que en el estudio del año 2009-2010 de aislamientos SARM provenientes del mismo hospital8.
En SASM se halló un aislamiento LPV (+) (4,35%). Si bien SASM es estudiado con menor énfasis, existen reportes de cepas SASM portadoras de LPV asociadas a infecciones comunitarias y hospitalarias1.
De los 22 aislamientos SARM 12 pertenecieron a pacientes ambulatorios, 4 LPV (+) y SCCmec IV (33,33%) que cumplen con la condición de no presentar resistencia acompañante o asociada a un AM no beta-lactámicos, en su mayoría aislados de muestras de PPB y de pacientes ambulatorios, particularidades que definen a las cepas SARM-AC10. Una cepa con dichas características fue de origen hospitalario, lo que demuestra el ingreso de las cepas SARM-AC a los nosocomios. Esto podría deberse a la colonización preexistente con aislamientos SARM-AC LPV (+) que encuentran una puerta de entrada durante procedimientos invasivos realizados en el hospital.
En las cepas SARM se encontraron 11 tipos de spa: t019 (22,73%), t311 y t002 (18,18%), t149 (9,09%) y un aislamiento de cada uno de los 7 tipos restantes (4,55%). En los aislamientos de SASM se diferenciaron 17 tipos spa con prevalencia del t189 (21,74%), t3051 y t024 (8,70%). De los 14 tipos restantes se encontró un aislamiento de cada uno (4,35%). El tipo spa t002 fue el único encontrado en SARM y SASM.
Al comparar el número de tipos spa hallados en SARM y SASM se demuestra la mayor diversidad genómica de SASM, al igual que en otros trabajos14.
Del análisis de los tipos de spa hallados se desprenden varias asociaciones estadísticamente significativas. Entre el tipo de spa y la procedencia del paciente (p=0,036) el tipo t019 fue predominante en los ambulatorios. Además, entre los tipos de spa y las poblaciones SARM y SASM se encontró asociación entre t311 (p=0,04) y t019 (p=0,0216) con SARM y t189 (p=0,05) con SASM.
En el análisis de los datos accesorios de los tipos spa provistos por la página Ridom SpaServer se encontraron 11 tipos spa no reportados en nuestro país: t4916, t8291, t214, t2121, t8354, t4286, t1549, t024, t6665, t325 y t592.
La cantidad de tipos spa muestran el alto grado de polimorfismo de la región Xr de la proteína A. Al ser un marcador molecular de locus único resulta indispensable el uso de marcadores adicionales como lo son el cassette de R SCCmec y el factor de virulencia LPV. Su rápida evolución hace a la técnica discriminatoria para la investigación de brotes14. Además, la evolución de la región de spa durante un tiempo prolongado resulta en diferentes tipos de spa consecutivos isogénicos o estrechamente relacionados con perfiles de repetición similares, lo que refleja que la tipificación spa también puede ser utilizada como un marcador epidemiológico a largo plazo5. Se debería validar por técnicas de referencia la asociación entre ellos, ya que se reporta un pequeño número de agrupaciones clonales de S. aureus que circulan en todo el mundo.
Es interesante destacar que las cepas SCCmec IV y V portadoras de LPV arrojaron distintos tipos de spa, mientras que los 2 aislamientos que resultaron SCCmec I, el mismo tipo. En la página se localizó la asociación del spa t019 al MLST ST30; por homología de las repeticiones se intuye que el t433 y el t4286, de los cuales no existen datos de la asociación spa-MLST, también pertenecerían a dicho ST. Por otro lado, los tipos de spa t002 y t311 se asocian al ST5. También se podría inferir que spa t149 se podría asociar al mismo ST.
Las características de los 2 aislamientos hallados cassette I, sin portación de LPV, de procedencia hospitalaria, spa t149, asociado al ST5, coinciden con las características al clon hospitalario chileno/cordobés diseminado en el sur de América Latina13. De los 5 aislamientos hallados SARM-AC LPV (+), 2 resultaron portar los tipos spa que definen a los clones SARM-AC epidémicos predominantes en América del Sur y en nuestro país, el t019 (ST30)3 y el t311 (ST5)12.
En esta caracterización hallamos gran diversidad genómica en los aislamientos recolectados de un hospital en un período corto de tiempo. Las cepas SASM presentaron mayor variabilidad genética que SARM. Se destaca que se identificaron tipos spa no comunicados en nuestro país hasta el momento en el sitio de referencia (http://www.spaserver.ridom.de/).
FinanciaciónEste trabajo fue financiado por la Universidad Nacional del Litoral, Santa Fe, Argentina, a través de los fondos de la programación CAI + D 2011.
Conflicto de interesesLos autores declaramos que no existen conflictos de intereses.