La base molecular de oligozoospermia todavía no se comprende completamente en la actualidad, a pesar de que es un hallazgo común presente en los varones con subfertilidad. En parte, esto se debe a la relativa falta de conocimiento de las proteínas que participan en la fisiología normal de los espermatozoides y a las herramientas de análisis disponibles hasta ahora1-3. Actualmente, la disponibilidad de técnicas de electroforesis bidimensional junto a la identificación de proteínas utilizando espectrometría de masas ofrece la oportunidad de comparar el mapa proteómico de muestras seminales independientes4-14. No obstante, el potencial de las herramientas proteómicas en el estudio de las causas de oligozoospermia aún no se ha explorado plenamente. Por tanto, para contribuir a cubrir este hueco, en el presente estudio se ha realizado una comparación de la abundancia relativa de 75 proteínas identificadas a través de electroforesis bidimensional (2D) extraídas de muestras de espermatozoides de pacientes oligozoospérmicos en comparación con donantes de semen.
MÉTODOSSujetos y recogida de muestrasEl comité de bioética del hospital aprobó este proyecto y se obtuvo el consentimiento informado de los participantes. En este estudio se incluyeron 15 pacientes oligozoospérmicos y 5 muestras de donantes de semen. Las muestras de los pacientes correspondían a pacientes oligozoospérmicos consecutivos, diagnosticados como parte de los análisis rutinarios, en parejas en tratamiento de reproducción asistida durante el período comprendido entre 2006 y 2008. Todos los pacientes y controles eran de Cataluña. Las muestras control correspondían a muestras normozoospérmicas de donantes de semen con fertilidad demostrada requeridas por ley para ser desechadas con fines reproductivos después de haber resultado en 6 embarazos. Las muestras se recogieron por masturbación en envases estériles después de al menos 3 días de abstinencia sexual, pero no más de 7 días. Las muestras se mantuvieron entre 22 y 37 °C y se evaluaron en el laboratorio dentro de 1 h desde su recogida. Después de la licuefacción del semen, los parámetros seminales (volumen, concentración espermática, motilidad) se evaluaron de acuerdo con las recomendaciones publicadas15-18 utilizando un analizador automático (CASA, Photolux, S.L. Barcelona, España). Los parámetros básicos de pacientes y de controles se describen en la tabla 1. En ninguna de las muestras de semen había un número significativo de células redondas o leucocitospermia17,18.
TABLA 1. Parámetros seminales básicos en los pacientes oligospérmicos y en los controles incluidos en el presente estudio
Preparación de las muestras para análisis proteómicosSe realizaron 3 pooles independientes de 5 muestras oligospérmicas cada uno (tabla 1). Las muestras de los controles se procesaron independiente (tabla 1). El plasma seminal y otras posibles células contaminantes se eliminaron, tal como se ha descrito anteriormente, a través de centrifugación en un gradiente de Percoll al 50%7,19.
Los espermatozoides de los sedimentos, limpios de células potencialmente contaminantes (comprobado a través de microscopia de contraste de fase), se resuspendieron en Ham F10 1x, contados usando la cámara de Makler (Sefi instrumentos médicos, Hainfa, Israel; se contaron aproximadamente 200 células por muestra) y, a continuación, se lavaron 2 veces con Ham F10 1x. La solubilización de las células de los espermatozoides se realizó esencialmente tal como se ha descrito4,7. El volumen de buffer de lisis se calculó para obtener una concentración correspondiente a aproximadamente 230 millones de espermatozoides/ml en un volumen de 300 µl por cada tira de isolelectroenfoque (IEF). Las muestras se incubaron 1 h a temperatura ambiente para permitir su solubilización y, por último, se centrifugaron a 3.000 g 5 min a 4 °C para eliminar los restos potencialmente insolubles.
Isoelectroenfoque y electroforesisLas proteínas solubilizadas se colocaron en la bandeja de rehidratación con tiras de 17 cm (pH, 5-8) lineales (Bio-Rad) y rehidratadas durante al menos 12 h. El IEF se realizó a 20 °C con el siguiente programa: 15 min con rampa rápida (0-250 V), 2 h con una rampa lenta (250-10.000 V), 45.000-60.000 V con una rampa rápida (10.000 V) y 10 h con una rampa lenta (50 V).
Una vez terminado el IEF, las tiras se equilibraron en 6 M urea, 0,375 M tris-HCl, pH 8,8, 20% glicerol, 2% SDS y 2% DTT 10 min, seguido por el mismo tampón sin DTT y suplementado con un 2,5% de iodoacetamida durante 10 min. La electroforesis se realizó con una protean II (Bio-Rad) a 300 V durante aproximadamente 3 h hasta que el frente de azul de bromofenol comenzaba a migrar fuera del extremo inferior de los geles.
Tinción de la bidimensional y análisis de las proteínasDespués de la electroforesis, los geles se tiñeron con Pink Flamingo (Bio-Rad), siguiendo las instrucciones del proveedor. Para análisis de la imagen, los geles se escanearon con un scanner fluorescente Typhoon (SG-800, Bio-Rad) y las imágenes se procesaron con el software PDQuest 7.1.1 (Bio-Rad). Las proteínas incluidas en el análisis ya se habían identificado previamente como parte de proyectos proteómicos previos7,9 (fig. 1). Los valores se normalizaron con la opción de “cantidad total válida en los puntos” siguiendo instrucciones comerciales (Bio-Rad). Los puntos proteicos de las bidimensionales correspondientes a 2 o más proteínas diferentes se han descartado de este estudio. Asimismo, sólo se consideraron los puntos proteicos que podrían identificarse en todos los 3 pooles de pacientes oligospérmicos. La proteínas que cumplían estas condiciones (n = 75) se analizaron para identificar cambios de al menos 2 veces un incremento o disminución en su cantidad en las muestras de pacientes oligospérmicos en comparación con los controles (Mann Whitney, p < 0,05; paquete estadístico SPSS).
RESULTADOSEn el análisis se incluyeron 75 proteínas identificadas previamente utilizando la espectrometría de masas (fig. 1)7,9,14. La comparación de las muestras de los pacientes oligozoospérmicos con las de los donantes resultó en la detección de 14 puntos de proteínas, con al menos un aumento o disminución del doble en comparación de los controles (tabla 2; fig. 2). El promedio en la cantidad relativa osciló entre 2,1 y 13,6 (tabla 2).
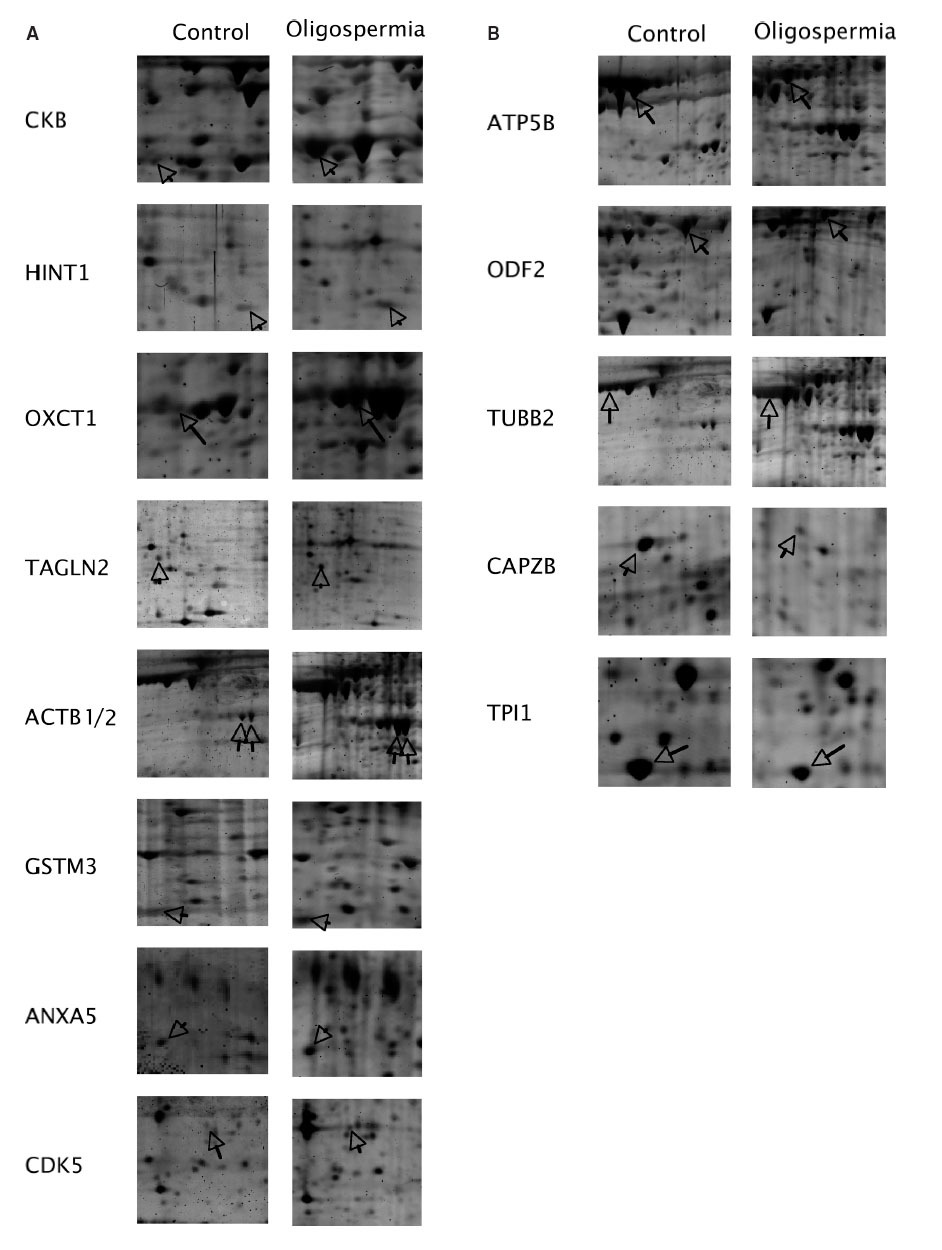
Figura 1. Electroforesis bidimensional de proteínas de espermatozoides identificadas a través de espectrometría de masas y consideras para su análisis en el presente trabajo. La bidimensional corresponde a uno de los controles incluido en el presente trabajo.
TABLA 2. Proteínas detectadas en una cantidad diferente (al menos 2 veces) en muestras de pacientes oligospérmicos en comparación con muestras de donantes normozoospérmicos de fertilidad probada
Nueve de las proteínas se detectaron incrementadas en las muestras oligozoospérmicas en comparación con las muestras control (tabla 2). Estas proteínas son la creatincinasa B (CKB), la proteína HINT1, el precursor de la succinil CoA 3 cetoácido transferasa (OXCT1), la transgelina-2 (TAGLN2), 2 puntos correspondientes a la actina citoesquelética (ActB), la glutation S-transferasa Mu 3 (GSTM3), la annexina A5 (ANXA5) y la cinasa dependiente de ciclina 5 (CDK5). Las proteínas detectadas como disminuidas son el precursor mitocondrial de la cadena beta de la ATP sintasa, (ATP5B), la proteína ODF2 (ODF2), la subunidad beta de la tubulina (TUBB2), la proteína CAPZ beta (CAPZB) y el triosafosfato isomerasa 1 (TPI1). Cabe señalar que muchas de las proteínas no se han analizado, ya que han incluido en el análisis sólo 75 puntos proteicos identificados de los más de 1.000 detectable en los geles bidimensionales (fig. 1). Por lo tanto, cabría esperar que muchas más proteínas diferenciales presentes en pacientes oligozoospérmicos se puedan identificar en futuros estudios.
DISCUSIÓNEn el presente trabajo se ha determinado la abundancia relativa de las principales proteínas extraídas de muestras de espermatozoides de pacientes oligozoospérmicos y de controles normozoospérmicos, y se ha encontrado que 14 puntos proteicos se detectan como diferenciales, ya sea aumentados o disminuidos, con una diferencia de al menos 2 veces en el grupo de pacientes oligozoospérmicos en comparación con los controles (tabla 2; figs. 1 y 2). Todas estas proteínas se describen por primera vez como alteradas en muestras de espermatozoides de pacientes oligozoospérmicos. Es interesante observar que las proteínas detectadas pertenecen a 3 categorías diferentes: 5 relacionadas con la energía, 3 con la señalización y regulación, y 6 proteínas estructurales.
Figura 2. Proteínas detectadas como aumentadas o disminuidas en pacientes oligozoospérmicos en comparación con los espermatozoides de muestras control. Se muestran ejemplos representativos correspondientes a las proteínas detectadas aumentadas o disminuidas en las muestras de pacientes oligozoospérmicos.
Entre las 5 proteínas relacionadas con la producción de energía, se han detectado 3 incrementadas y 2 disminuidas en muestras de pacientes oligospérmicos en comparación con los controles. Proteínas aumentadas son CKB, OXCT1 y GSTM3. Proteínas disminuidas son ATP5B y PTI1.
Entre las 6 proteínas estructurales se han detectado 3 aumentadas y 3 disminuidas en los pacientes oligospérmicos. Proteínas aumentadas son TAGLN2, ActB y ANXA5. Proteínas detectadas como disminuidas en los pacientes son ODF2, TUBB2 y CAPZB. El grupo “señalización celular y regulación” está constituido por CDK5, HINT1 y ANXA5. Podría haber diferentes explicaciones para el aumento o la disminución detectada en diversas proteínas en pacientes oligospérmicos. Una posibilidad podría ser que algunas de las proteínas detectadas estén implicadas en los mecanismos fisiopatogénicos de la oligospermia. Otra posibilidad podría ser que las proteínas alteradas fuesen una consecuencia de una alteración general de la expresión proteica en pacientes oligospérmicos, pero no sean la causa directa de la etiología de la oligospermia. En cualquier caso, las proteínas detectadas abren las puertas a profundizar más en el estudio de los mecanismos patogénicos implicados en la oligospermia y tienen el potencial de ser útiles como biomarcadores en pacientes con oligospermia. Por ejemplo, será interesante confirmar en un futuro las diferencias detectadas en la abundancia relativa de ciertas proteínas utilizando técnicas inmunofluorescentes con anticuerpos específicos. En estudios futuros también será preciso aclarar en qué medida el contacto de los espermatozoides con secreciones del epidídimo, de las glándulas seminales o del líquido prostático puede ser responsable de algunas de las diferencias detectadas en los pacientes oligozoospérmicos.
En estudios proteómicos previos en pacientes astenozoospérmicos se han podido identificar diversas proteínas implicadas en la regulación de la motilidad espermática8,12. Mediante técnicas de separación electroforética convencional también ha sido posible identificar alteración de contenido de protaminas en muestras de pacientes oligospérmicos20-25. No obstante, éste es el primer estudio realizado en pacientes oligozoospérmicos utilizando electroforesis bidimensional y técnicas de análisis proteómico.
CONCLUSIONESEn conclusión, en este trabajo se describe por primera vez la utilización de técnicas de análisis bidimensional proteómico para identificar proteínas alteradas en espermatozoides de pacientes oligozoospérmicos. De relevancia, se han identificado 14 puntos proteicos con un patrón proteómico alterado en pacientes oligozoospérmicos. Este resultado abre ahora las puertas a la realización de estudios adicionales encaminados a la identificación de los mecanismos fisiopatogénicos implicados en la oligozoospermia y al posible desarrollo de las proteínas identificadas como marcadoras en la infertilidad.
AGRADECIMIENTOSLos autores agradecen a Sara de Mateo y a Francisco Fernández por su apoyo técnico.
Trabajo subvencionado con un proyecto del Ministerio de Ciencia y Tecnología BMC2006 03479, fondos FEDER.
Correspondencia:Dr. R. Oliva. Grupo de Genética Humana. Unidad de Genética. Facultad de Medicina. Hospital Clínico y Provincial. Casanova, 143. 08036 Barcelona, España. Correo electrónico: roliva@ub.edu