SARS-CoV-2 es el tercer coronavirus que emerge en las últimas dos décadas y produce la enfermedad denominada COVID-19 (enfermedad infecciosa por coronavirus 2019). Ha demostrado ser fácilmente transmisible entre humanos con una rápida diseminación mundial y declarada como pandemia el 11 de marzo 2020. A la fecha ha causado millones de casos y muertes, disrupción de servicios sanitarios y severas consecuencias sociales, económicas y políticas en todos los países. Los estudios filogenéticos lo relacionan con SARS-CoV presentes en murciélagos. Comparte características de patogenicidad con sus parientes más cercanos, SARS-CoV y MERS-CoV. 15 a 20% de los afectados presentan cuadros graves. A la fecha no se cuenta con antivirales efectivos ni vacunas. Para un adecuado control se hace imprescindible dilucidar aspectos epidemiológicos, moleculares y de patogenicidad. En esta revisión se presenta información básica sobre epidemiología, origen, estructura y patogenia de SARS-CoV-2.
SARS-CoV-2 is the third coronavirus emerging in the last two decades producing a disease denominated COVID-19 (infectious disease by coronavirus 2019). It has demostrated to be easily transmisible between humans with a fast world wide dissemination and declared a pandemia in March 11, 2020. Up to date it has caused millions of cases, deaths, disruptions in medical services and severe social, political and economic consecuences, all around the globe. Phylogenetic studies relate it to SARS-CoV present in bats. It shares pathogenic characteristics with its close relatives, SARS-CoV and MERS-CoV. 15 to 20% of the patients develop a severe clinical course. Up to date there are no effective antiviral drugs or vaccines. For an adecuate control it is essential to learn about epidemiological, pathogenic and molecular aspects. In this review some basic information about epidemiology, origin source, structure and pathogenicity of SARS-CoV-2 is presented.
La emergencia y reemergencia de patógenos constituyen una amenaza constante para la seguridad de la humanidad y las consecuencias sanitarias, sociales y económicas asociadas son impredecibles. En las últimas dos décadas hemos sido testigos del impacto provocado por la emergencia de dos coronavirus, SARS-CoV en 2002 y MERS-CoV en 2012 causando brotes de neumonías graves en humanos y mostrando un claro potencial pandémico. Finalizando el 2019 nos sorprende la emergencia de un tercer nuevo coronavirus inicialmente nominado 2019-nCoV.
Las alarmas se encienden el 31 de diciembre de 2019 cuando las autoridades de salud de China reportan a la Organización Mundial de la Salud (OMS) la ocurrencia de un brote de neumonía de etiología desconocida en 27 pacientes, todos vinculados epidemiológicamente a un mercado de productos del mar en la ciudad de Wuhan, provincia de Hubei1.
Para el 7 de enero de 2020, los investigadores del Centro de Control de Enfermedades (CDC) de China reportaban un nuevo coronavirus como agente responsable del brote de neumonías. El 12 de enero la secuencia genética fue publicada y compartida, lo que permitió el rápido desarrollo de técnicas diagnósticas2. El nuevo agente fue aislado en muestras obtenidas del tracto respiratorio inferior de uno de los pacientes afectados, convirtiéndose en el séptimo de la familia, con transmisión eficiente entre las personas fue presentado como nCoV-20193. Los investigadores además lograron establecer una alta homología entre las secuencias genéticas del nuevo coronavirus y el SARS-CoV que afecta a murciélagos, haciendo altamente posible que estos sean el reservorio natural4,5.
Tempranamente y a tres semanas de la alerta inicial, ya se reportaban casos importados desde Wuhan a Tailandia, Japón y Corea6. El 11 de febrero el Comité Internacional de Taxonomía nomina al nuevo coronavirus como SARS-CoV-27 y la OMS como COVID-19 a la enfermedad infecciosa producida por el nuevo coronavirus8.
Debido a la rápida expansión geográfica y el alarmante incremento en el número de casos, el 30 de enero de 2020 la OMS, de acuerdo con el Reglamento Sanitario Internacional (RSI), declaró a este brote como un evento con características de emergencia de salud pública internacional (ESPII)9 y el 11 de marzo como una nueva pandemia. Para esa fecha el total de casos confirmados en el mundo se elevaba a 118.000 con 4.291 fallecidos10.
En Chile, de acuerdo a las recomendaciones de la OMS, el 30 de enero se decreta alerta sanitaria en todo el país, se activa la búsqueda y notificación de casos inicialmente importados y se implementa la técnica diagnóstica por reacción de polimerasa en cadena (PCR) en el Instituto de Salud Pública (ISP). El primer caso en el país se confirma el 3 de marzo, en un viajero internacional proveniente de Europa y ya el 18 de marzo, el Ministerio del Interior decreta estado de excepción constitucional de catástrofe por la detección de casos secundarios en el país11.
Al 7 de diciembre la OMS reporta alarmantes números de casos a nivel mundial con un total de 65,8 millones de casos y 1,5 millones fallecidos. En los últimos meses los casos se concentran en la región de las Américas, dando cuenta del 50% del total de casos nuevos notificados a nivel mundial12. Al 7 de diciembre en Chile se contabilizan 639.492 casos y 20.902 fallecidos confirmados de SARS-CoV-213.
SARS-CoV-2 ha demostrado transmitirse fácilmente entre las personas, principalmente a través de gotitas respiratorias entre un individuo infectado y uno susceptible, el periodo de incubación es prolongado y se ha descrito entre 5 y hasta 14 días14. La transmisibilidad o período de infectividad comienza 24 a 48 horas antes del inicio de síntomas y dura más o menos hasta el día 7 a 9 desde el inicio de estos y se estima que un caso puede generar 2 o 3 nuevos infectados (número de reproducción de la enfermedad26. Las manifestaciones clínicas más frecuentes descritas a la fecha son cefalea, mialgias, tos, fiebre, dolor abdominal, diarrea, anosmia, pérdida del sentido del gusto, dolor torácico entre otros. La mayoría de los infectados (80%) presentan sintomatología leve o moderada, en cambio un 15% han presentado una evolución grave requiriendo de hospitalización4. Los casos graves se presentan principalmente en adultos mayores e individuos con comorbilidades subyacentes como hipertensión arterial, enfermedad pulmonar obstructiva crónica, diabetes, enfermedad cardiovascular y obesidad. Estos evolucionan con disnea, hipoxemia de rápida progresión a falla ventilatoria, trastornos de la coagulación, shock séptico, y falla multiorgánica4. La letalidad descrita al inicio de la pandemia fue de 2-3%, siendo significativamente mayor en pacientes que requieren manejo en unidad de paciente critico (UPC) y ventilación mecánica invasiva (VMI)4. El diagnóstico de la infección se basa principalmente en la detección del material genético a través de técnicas de biología molecular como reacción en cadena de la polimerasa con transcriptasa inversa (RT-PCR) en muestras de secreción respiratoria, principalmente obtenidas por hisopado nasofaríngeo como también aspirados traqueales y lavado bronquio alveolar14–16. Se ha descrito una sensibilidad para la RT-PCR de 60-70% dependiente de la calidad de la muestra, del momento de la obtención durante la evolución de la enfermedad y de la rapidez de traslado al laboratorio entre otros factores. Por esta razón, se han incorporado exámenes de laboratorio que apoyan el diagnóstico como es la tomografía computada de tórax donde se pueden evidenciar por ejemplo infiltrados pulmonares en vidrio esmerilado4.
ORIGEN Y EVOLUCIÓN DE LOS CORONAVIRUSLos coronavirus son un grupo muy diverso de virus que pueden causar infecciones respiratorias, entéricas, hepáticas y neurológicas de diversa severidad en una gran variedad de animales17,19. Son miembros de la subfamilia Coronavirinae de la familia Coronaviridae y del orden Nidovirales. La subfamilia se subdivide según su estructura genética en cuatro géneros: Alphacoronavirus, Betacoronavirus, Gammacoronavirus y Deltacoronavirus. Los Alphacoronavirus y Betacoronavirus infectan solo a los mamíferos. Los Gammacoronavirus y Deltacoronavirus infectan a las aves, pero algunos de ellos también pueden infectar a los mamíferos17. Los Alphacoronavirus y Betacoronavirus generalmente causan enfermedades respiratorias en humanos y gastroenteritis en animales17.
En 1966 se describen los primeros coronavirus en humanos (HCoV), HCoV-E229 y HCoV-OC43 agentes etiológicos frecuentes de infecciones respiratorias agudas estacionales y que también incluyen a HCoV-NL63 y HKU1 descritos el 2005. Estos HCoV son endémicos, con amplia distribución geográfica, de circulación estacional y responsables del 2 al 18% de todas las infecciones respiratorias altas. Producen enfermedad leve cómo resfrío común en la mayoría de los casos y ocasionalmente cuadros más severos en lactantes, niños pequeños y adultos mayores. Se estima que un 50% de los infectados no presentan síntomas17,20. Estos 4 coronavirus están presentes en las plataformas moleculares múltiples para el diagnóstico de laboratorio de infecciones respiratorias agudas, utilizados frecuentemente en práctica clínica.
En 2002 y 2012 emergen SARS-CoV y MERS-CoV, responsables de síndromes respiratorios agudos severos. SARS-CoV se originó en la provincia de Guangdong, China, provocando un brote de neumonías severas en adultos, inicialmente ligados epidemiológicamente a mercados de animales. El total de casos confirmados alcanzo a 8.098 y 774 fallecidos, con una letalidad de 10%. La severidad y letalidad estuvo fuertemente relacionada con la edad, 20 a 30% de los pacientes mayores de 60 años requirieron de UPC y VMI. La transmisión persona a persona fue documentada principalmente por la transmisión a personal de salud19. Menos del 5% del total de afectados fueron niños. Este brote, el primero del siglo XXI, encendió las alarmas de los científicos por el potencial pandémico de los Coronavirus, demostrando una rápida diseminación geográfica, afectando a más de una docena de países. La respuesta coordinada a nivel internacional permitió un control total del brote para mediados de 2003 a través de implementación de estrictas medidas de salud pública19. En 2012 emerge MERS-CoV en la Arabia Saudita, nuevamente asociado a neumonías graves en pacientes adultos pero con mayor letalidad que su predecesor, siendo de 35 a 45%. Los pacientes que presentaron el mayor riesgo de neumonía grave fueron hombres, presencia de inmunosupresión y comorbilidades asociadas. Desde su debut, los brotes más importantes se han dado en un contexto de transmisión nosocomial19,20. Hasta noviembre de 2019, se habían notificado a la OMS un total de 2.494 casos y 858 fallecidos. A diferencia de SARS-CoV, MERS-CoV no ha logrado diseminarse efectivamente entre distintas áreas geográficas y solo se reportan casos esporádicos fuera de la península arábica (Tabla 1). Dada la severidad y el potencial rol pandémico de los coronavirus, la OMS el año 2017 los incorpora a la lista de agentes prioritarios para ser investigados19.
Center for Evidence Based Medicine, Oxford. Niveles de evidencia
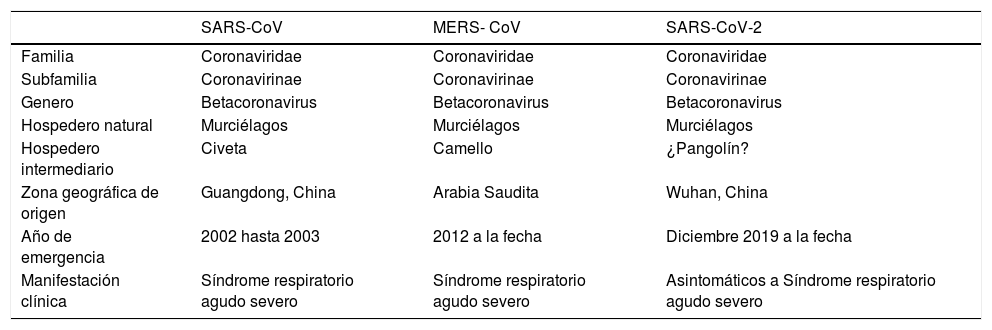
| SARS-CoV | MERS- CoV | SARS-CoV-2 | |
|---|---|---|---|
| Familia | Coronaviridae | Coronaviridae | Coronaviridae |
| Subfamilia | Coronavirinae | Coronavirinae | Coronavirinae |
| Genero | Betacoronavirus | Betacoronavirus | Betacoronavirus |
| Hospedero natural | Murciélagos | Murciélagos | Murciélagos |
| Hospedero intermediario | Civeta | Camello | ¿Pangolín? |
| Zona geográfica de origen | Guangdong, China | Arabia Saudita | Wuhan, China |
| Año de emergencia | 2002 hasta 2003 | 2012 a la fecha | Diciembre 2019 a la fecha |
| Manifestación clínica | Síndrome respiratorio agudo severo | Síndrome respiratorio agudo severo | Asintomáticos a Síndrome respiratorio agudo severo |
Todos los coronavirus humanos, como se ha descrito, tienen un origen zoonótico, para SARS-CoV, MERS-CoV, HCoV-NL63 y HCoV-229E el reservorio natural son los murciélagos y para HCoV-OC43 y HKU1 probablemente el reservorio está en los roedores17. La glicoproteína de superficie (S), presente en la envoltura viral, es fundamental para el reconocimiento y unión a receptores celulares y representa la llave fundamental para determinar la afinidad con un hospedero19.
ORIGEN Y ESTRUCTURA DE SARS-CoV-2SARS-CoV-2 es el séptimo coronavirus que afecta al hombre, perteneciendo al género Betacoronavirus. Estudios filogenéticos han demostrado que está estrechamente relacionado con un 88-90% de homología con dos CoV similares al SARS derivados de murciélagos (bat-SL-CoVZC45 y bat-SL-CoVZXC21) ubicándolo en el subgénero Sarbecovirus. Además, SARS-CoV-2 es genéticamente distinto del SARS-CoV presentando 79% de similitud y de 50% con MERS-CoV4,14.
No se conoce aún con certeza cómo y cuándo ocurrieron los eventos involucrados en la transmisión de SARS-CoV-2 al ser humano y si algún animal actuó como hospedero intermediario como fue descrito para SARS-CoV y MERS-CoV, eventos en los que se demostró la participación como civetas y camellos respectivamente. Estudios experimentales han demostrado que SARS-CoV-2 puede infectar y replicarse también en animales como gatos y hurones4.
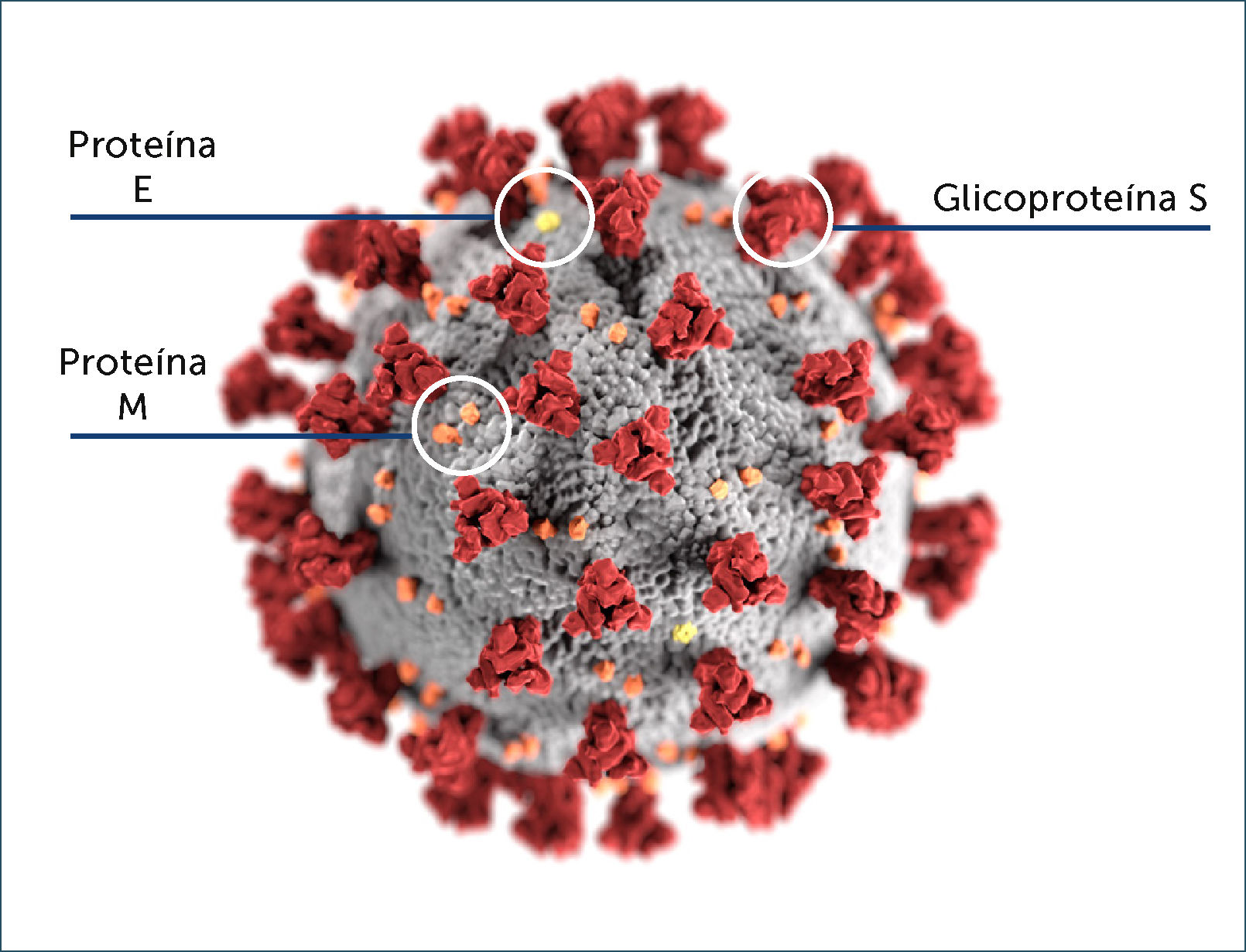
Como todos los coronavirus, posee una envoltura lipoproteica, su genoma está conformado por una hebra no segmentada de ARN de polaridad positiva y de aproximadamente 30.000 pares de nucleótidos20. Codifica para cuatro proteínas estructurales: la glicoproteína de superficie (glicoproteína S o Spike), la proteína de la envoltura (proteína E), la glicoproteína de membrana (M) y la proteína de la nucleocápside (proteína N) (Figura 1). Además, codifica para 8 proteínas funcionales no estructurales, entre estas, la ARN polimerasa, para la replicación viral y dos proteasas encargadas de cortar la poli-proteína formada inicialmente. De la envoltura lipoproteíca emerge la glicoproteína S que le da la apariencia de corona al microscopio electrónico. Es la encargada de actuar como ligando para la adsorción del virus al epitelio respiratorio a través del dominio de unión al receptor celular. La glicoproteína S es determinante para el tropismo del virus, facilita la fusión de la membrana del virus con la de la célula huésped permitiendo la liberación del genoma viral al interior de la célula infectada. Es el blanco principal de los anticuerpos neutralizantes, tiene por lo tanto un papel clave en la inducción de inmunidad humoral y celular durante la infección con SARS-CoV-2. La proteína de membrana (M) es una glicoproteína transmembrana que da la forma al virus y permite la unión a la nucleocápside (N), la proteína E tiene un papel clave en el ensamblaje y liberación del virus y la proteína N forma parte de la nucleocápside al unirse al material genético20.
PATOGÉNESIS DE SARS-CoV-2 Y RESPUESTA INMUNEPara ingresar a la célula huésped, SARS-CoV-2 reconoce a los receptores de la enzima convertidora de angiotensina 2 (ACE2) como puerta de entrada para establecer la infección, mismo receptor utilizado por SARS-CoV pero con una afinidad 10 a 20 veces mayor4,21. Los ACE2 son receptores de membrana ampliamente expresados en casi todos los tejidos como pulmón, corazón, riñón, vía biliar, hígado e intestino18. ACE2 es una proteína transmembrana responsable de la conversión de angiotensina I en angiotensina II, la que tiene un rol importante en la homeostasis y en la respuesta pro-inflamatoria22. La expresión y distribución de los receptores ACE2 regulan el tropismo del virus a los distintos tejidos como también la patogenia22. En el tejido pulmonar los ACE2 están expresados en las células alveolares tipo 2 y también están presentes en monocitos, macrófagos y células endoteliales23. En el proceso de unión al receptor ACE2, SARS-CoV-2 utiliza la glicoproteína S, responsable de mediar dos procesos fundamentales en la infección a través de sus dos subunidades denominadas S1 y S2. La subunidad S1 se une al receptor ACE2 y la subunidad S2 determina la fusión permitiendo la entrada del virus por endocitosis22. En este proceso además participa una proteasa transmembrana de la célula huésped que escinde la glicoproteína S poco después de unirse a ACE222. La menor expresión de receptores ACE2 en epitelio nasofaríngeo en niños menores de 10 años puede explicar la menor susceptibilidad a la infección en este grupo2.
Se estima que un 80% de los pacientes infectados por SARS-CoV-2 son asintomáticos o presentan síntomas leves y un 20% cuadros graves. En pacientes con cuadros severos, SARS-CoV-2 es capaz de evadir el sistema de reconocimiento inmune e interferir en la activación y producción de interferón, que tiene como misión fundamental el limitar la replicación viral e inducir la respuesta inmune adaptativa23. Se ha planteado que la rápida replicación de SARS-CoV-2 en el tejido pulmonar gatilla una potente respuesta inflamatoria con migración de linfocitos, fundamentalmente macrófagos, que inducen el daño intersticial y produciendo como consecuencia la alteración severa del intercambio gaseoso. El descenso relativo de linfocitos en sangre periférica se ha descrito como un marcador indirecto de la inflamación pulmonar subyacente. En estos pacientes además se produce una respuesta desregulada de secreción de citoquinas lo que se manifiesta como un síndrome de hiper inflamación sistémica o también denominada tormenta de citoquinas, induciendo niveles aumentados de IL-2, IL-7, IL-10, G-CSF, IP-10, MCP-1, MIP-1A y TNF-α. La elevación de citoquinas además produce daño microvascular, activación del sistema de coagulación, inhibición de la fibrinólisis con inducción de coagulación intravascular diseminada expresada en el aumento progresivo del dímero D, troponina I y fibrinógeno. Estos fenómenos explicarían el síndrome de dificultad respiratoria aguda, la insuficiencia respiratoria y la falla orgánica múltiple, principales causas de muerte en pacientes con COVID-1923–25.
Respecto a la respuesta serológica inducida por COVID-19, se ha descrito elevación de IgM a partir del día 9 desde el inicio de los síntomas y desde el día 14 para IgG. La magnitud de la respuesta de anticuerpos neutralizantes parece estar correlacionada con la severidad de la enfermedad, siendo menor en individuos asintomáticos, los que además presentan una declinación más rápida. A la fecha los datos son muy limitados para determinar con certeza la duración y calidad de la respuesta inmune26–28.
CONCLUSIÓNLa emergencia de SARS-CoV-2 ha provocado una crisis global con consecuencias sanitarias, económicas y sociales sin precedentes durante la primera ola pandémica y se desconoce el comportamiento en las olas epidémicas sucesivas planteando un escenario complejo para la humanidad.
Los desafíos científicos son enormes en múltiples campos, desde dilucidar el origen y los mecanismos que generaron y facilitaron el salto del virus desde el reservorio animal al hombre, caracterizar aspectos moleculares, patogenia, dinámica de la respuesta del sistema inmune, encontrar moléculas antivirales efectivas y el desarrollo de vacunas entre muchos otros. Mientras tanto las medidas de contención continuarán siendo la detección oportuna de casos y el aislamiento efectivo, la trazabilidad y cuarentena de los contactos y las medidas de autocuidado que incluyen el distanciamiento físico, mascarilla facial y la higiene de manos.
Declaración de conflicto de interés
La autora declara que no tiene conflictos de interés.
Este es un artículo Open Access bajo la licencia CC BY-NC-ND.