A lo largo de los últimos 20 años, el gran avance del desarrollo tecnológico ha traído consigo un importante y profundo conocimiento de las bases genéticas y moleculares del ser humano. Las bases sobre las que asentaban los conocimientos en torno a la genética clínica y las relaciones entre causa genética y el desarrollo de enfermedades hereditarias se han visto modificados de forma trascendental. En este ámbito, los descubrimientos en el campo de la genética clínica se han visto orientados hacia conceptos de heterogeneidad genética y fenotípica, de forma que una misma patología hereditaria actualmente se sabe que podría estar ocasionada por alteraciones en diferentes genes y simultáneamente, variantes en el mismo gen podrían desencadenar fenotipos de gran diversidad.
En este nuevo panorama, las metodologías de análisis masivo de genes así como las implementaciones bioinformáticas necesarias para la interpretación de las alteraciones identificadas, resultan de elevada importancia para poder alcanzar el diagnóstico certero de una enfermedad hereditaria.
Esta misma profundización en el conocimiento genético, ha identificado nuevos ámbitos de estudio que anteriormente se desconocían. La farmacogenética ha emergido como una disciplina de vital relevancia debido a que los perfiles genéticos propios del individuo influyen en la respuesta farmacológica final en términos de seguridad y efectividad. Además ha dejado al descubierto requerimientos adicionales para los estudios genéticos, como parámetros de sensibilidad, límites de detección y tiempos de respuesta, particularmente relevantes en el ámbito oncológico.
Adicionalmente las matrices biológicas sustrato de los estudios genéticos han evolucionado junto con el avance científico. La identificación de microARNs y la existencia de ADN tumoral circulante (ctADN) como biomarcadores plasmáticos, no invasivos que han mostrado en recientes estudios su potencial funcionalidad como indicadores en el screening y monitorización de progresión de enfermedad.
En términos de costo-efectividad, nuestros esfuerzos deberían dirigirse los hacia el diagnóstico precoz de enfermedades de alta heterogeneidad genética y en la rápida detección de recidivas en enfermedad neoplásica. Ambas situaciones incrementan las posibilidades de éxito del tratamiento farmacológico en el paciente y su familia, y por tanto contribuyen a aumentar la supervivencia del paciente y mejorar su calidad de vida.
Over the last 20 years, the great progress of technological development has allowed a profound learning about the genetic and molecular human being knowledge. The criteria on which the clinical genetics and the relationships between genetic cause and the development of hereditary diseases were established have been modified in a transcendental way. In this context, discoveries in the clinical genetics field have been oriented towards genetic and phenotypic heterogeneity concepts, so that the same hereditary pathology is currently known to be caused by alterations in different genes and simultaneously, it has been described that variants in the same gene could trigger so extremely diverse phenotypes.
In this new scenario, massive gene analysis methodologies as well as bioinformatic implementations, needed the identified variant interpretation, are highly important to be able to reach the accurate hereditary disease diagnosis.
This deepening in the genetic knowledge has identified new fields of study that previously were not known. Pharmacogenetics has emerged as a discipline of vital relevance because individual genetic profiles shape the final pharmacological response in terms of safety and effectiveness. It has also revealed additional requirements for genetic studies, such as sensitivity, limit of detection and turn-around time parameters, which are particularly relevant in the oncology field.
In addition, the biological matrices which are the substrate for the genetic studies have evolved along with the scientific advance to. The identification of microRNAs and the existence of cell-free tumour DNA, as non-invasive plasma biomarkers, have shown in recent studies their potential functionality as indicators in the disease screening, monitoring and progression following-up.
In terms of cost-effectiveness, we must direct our efforts towards precocious high heterogeneity genetic disease diagnosis and towards the quickest ability to detect tumoural recurrences. Both situations increase the chances of patient's or his family's treatment effectivity and therefore contribute to the increase of patient survival and improve their quality of life.
Los avances científicos de los últimos 20 años en el conocimiento del genoma humano han permitido determinar las bases genéticas y moleculares de muchas enfermedades. La genética es una de las disciplinas científicas que más ha contribuido a la comprensión de la etiología de las enfermedades, y con ello abrir una puerta a encontrar los medios para su prevención, diagnóstico y tratamiento. Por ello actualmente ya es una herramienta imprescindible para el correcto manejo del paciente en cualquiera de las disciplinas médicas, y la labor del genetista clínico y del bioinformático han pasado a ser definitivas en los diagnósticos diferenciales para la correcta prescripción de tratamiento.
EVOLUCION CIENTÍFICO - TECNOLÓGICALa razón de esta integración cada vez mayor de los Genetistas Clínicos en los equipos multidisciplinares es consecuencia directa de la evolución y el dinamismo que los estudios genéticos han alcanzado:
La genética clínica partió del estudio de las enfermedades monogénicas donde se establece una relación causa-efecto, entre la alteración de un determinado gen y una enfermedad concreta. Existen alrededor de 8000 enfermedades genéticas listadas en OMIM (Online Mendelian inheritance of Man), que presentan un patrón de herencia compatible con una transmisión según las leyes de Mendel o mitocondrial. De más de la mitad (unas 4500 enfermedades), se conoce ya la base molecular y la descripción del fenotipo, pero queda aproximadamente un 50% de enfermedades donde esta relación aún se desconoce1. Su prevalencia es de 1 caso por cada 200 nacimientos. Entre ellas se encuentran: la anemia falciforme, la fibrosis quística, la hemofilia A o la distrofia de Duchenne.
Con posterioridad se han ido conociendo e identificando un número creciente de genes asociados a enfermedades mendelianas de alta heterogeneidad genética y clínica, donde un mismo gen puede estar ligado a distintas enfermedades o bien un mismo fenotipo clínico asociado a una decena o centena de genes, como ocurre en muchos casos en ciertas enfermedades oncológicas, neurológicas, cardiovasculares, donde se ha ampliado el campo de actuación de la Genética. Durante muchos años, el paradigma “un gen-una enfermedad” representó uno de los principios de la genética clínica. Del mismo modo “un gen-una proteína” fue el fundamento para la biología molecular. En el año 2001, y tras publicarse el borrador del genoma humano, ese otro principio de un gen-una proteína también se desmoronó al demostrarse que un gen era capaz de generar diferentes proteínas con diferente funcionalidad dependiendo del órgano o las condiciones que determinan su expresión.
En este sentido la visión del estudio genético clásico también se ha visto modificada debido a que se ha ampliado considerablemente el número de genes candidatos a analizar para un mismo fenotipo. Este nuevo planteamiento en el abordaje de los estudios genéticos conlleva por tanto un impacto a nivel económico en relación al coste y tiempo invertido de los mismos. El estudio de un gen de tamaño medio puede representar un mes de trabajo en cualquier laboratorio de diagnóstico genético y puede representar dependiendo del país, un coste medio de entre 500-800 dólares americanos. Si los genes que se analizan son dos, el tiempo y el coste se incrementan, pero todavía es admisible. Si en lugar de dos genes de tamaño medio hablamos de casos como 40 genes que pueden ser responsables de una sordera autosómica recesiva, 10 genes que pueden provocar una osteogénesis imperfecta, 6 genes asociados a formas de Parkinson de aparición temprana, 30 genes que pueden dar lugar a una miocardiopatía dilatada, 13 genes que explican un síndrome QT largo, o los 6 genes asociados a un cáncer de colon no polipósico hereditario, por indicar diversos ejemplos, tanto el tiempo dedicado por tecnologías tradicionales, como el coste se multiplican “n” veces en función del número de genes y, por lo tanto, se hace inviable en el 99% de los casos ofrecer un diagnóstico óptimo al paciente y a la familia.
Por ello, esta heterogeneidad genética y clínica, tradicionalmente y hasta la aparición de las tecnologías que permitan el estudio múltiple de genes, como las tecnologías de secuenciación masiva, ha sido y es uno de los principales problemas a la hora de realizar un diagnóstico genético.
Los avances del conocimiento tecnológico han propiciado un perfeccionamiento, simplificación y abaratamiento de tecnologías clave, permitiendo que este tipo de enfermedades ya no sea un reto inabordable en la práctica clínica real, y pasen del lado de la investigación al lado de la clínica rutinaria. El tiempo de respuesta ya no es un problema, supone el mismo tiempo analizar dos, 10 ó 500 genes a la vez. Y el coste disminuye drásticamente. No se precisa gastar 500 ó 1000 euros en analizar un gen, o secuencialmente otro gen, si no se ha identificado la causa genética, hasta lograr finalmente el diagnóstico genético. Por el contrario con el coste de analizar un gen se dispone de la información genómica completa del conjunto de genes que se requieren para elaborar un informe diagnóstico y poder dar una solución a cada especialista que facilite el correcto abordaje de sus pacientes.
El problema que se planteó tras disponer de la capacidad sin límite de analizar genes fue el problema del costo de almacenamiento de los datos generados y sobre todo el análisis e interpretación del enorme caudal de datos de secuencia genómica, que aumenta sin cesar a un ritmo vertiginoso y precisa de un tratamiento informático complejo y altamente especializado. Esta nueva situación hace que no sólo en los equipos de diagnóstico están profesionales genetistas, sino un nuevo tipo de profesionales: los bioinformáticos clínicos. Estos nuevos profesionales requieren de un exhaustivo conocimiento del funcionamiento de las nuevas plataformas de secuenciación, así como de lo que hoy llamamos “Biología de Sistemas”2 para avanzar en el conocimiento del genoma humano y progresar hacia un diagnóstico más preciso, un pronóstico más precoz, y un tratamiento personalizado. El desarrollo de algoritmos mediante herramientas bioinformáticas permite procesar ingentes bases de datos genómicos generados con las plataformas de secuenciación masiva, e integrar los datos procedentes de otras plataformas u otras disciplinas del diagnóstico como la bioquímica, la inmunología, la imagenología, etc.; permitiendo de esta forma encontrar las claves para disponer de los datos necesarios para generar protocolos de trabajo, informes con calidad diagnóstica, garantizando unos óptimos estándares de calidad.
Debido a toda la evolución anterior nos encontramos con que, cada vez más, la elección del tratamiento farmacológico idóneo, así como la dosis más adecuada, toma en cuenta el perfil genético de cada paciente. La elección del tratamiento idóneo para cada paciente redunda por tanto, en la mejora en la calidad de la atención, ya que reduce el riesgo de toxicidad así como de reacciones adversas medicamentosas que acaban en fracaso terapéutico.
Si hablamos de cáncer, la detección temprana del tumor, la confirmación del diagnóstico, el pronóstico y predicción de la respuesta terapéutica, así como la monitorización de la enfermedad y recurrencia se sustentan en los avances tecnológicos como la secuenciación masiva y la digital droplet PCR (ddPCR) tanto en tumor como en biopsia líquida, que aportan nuevos beneficios como son la precisión, la eficacia, la seguridad y la rapidez. Esto nos puede permitir intervenir incluso antes que se desarrollen los síntomas y actuar sobre la salud general del paciente, llegando a la llamada Medicina de Precisión3 entendida como el diseño y la aplicación de intervenciones de prevención, diagnóstico y tratamiento más adaptadas al sustrato genético de cada paciente y al perfil molecular de cada enfermedad.
Dada la disminución de precios progresiva en análisis de secuencias y la potencia de cálculo y análisis de cientos de miles de datos Bioinformáticamente, hacen que se piense en análisis completos de Genoma (incluyendo regiones exómicas, intrónicas, reguladoras) por un coste entre 600-1000 dólares americanos. Esta disponibilidad representará un impulso decisivo a la investigación y a la Medicina Genómica, y permitirá incorporar la secuencia del genoma como un dato más en la historia clínica de cada paciente, a un coste “asumible” por los sistemas sanitarios.
Sin embargo, queda de nuevo la pregunta de la salvaguarda de datos de manera eficiente. En la actualidad tenemos variantes de significado desconocido, que han sido reclasificadas como patogénicas cuando las evidencias clínicas publicadas así lo determinan, de manera que si bien un diagnóstico realizado hoy es correcto, sería necesario incorporar mecanismos de análisis y re-análisis en el tiempo de las variantes de los informes. De nuevo volvemos a la importancia de disponer de equipos de bioinformáticos en las unidades clínicas.
Otro factor que definitivamente ha contribuido a explicar el auge del concepto de Medicina de Precisión es el económico.
La industria farmacéutica, en los últimos 12 años ha triplicado su inversión en I+D y sin embargo, el número de nuevos medicamentos aprobados es la tercera parte de lo que se aprobaba anteriormente. Este modelo de “drug discovery” debe cambiar y un mismo tratamiento para todos los pacientes con una patología (concepto de “blockbuster”) está siendo abandonado por la falta de eficiencia en el desarrollo.
La nueva manera de descubrir fármacos se basa en dirigir el uso de los fármacos a subgrupos concretos de pacientes en los que sea posible predecir tanto la eficacia del tratamiento como su seguridad. Ya que es conocido que ni todos los pacientes responden ni toleran igual un mismo tratamiento, ni una misma entidad patológica se presenta o cursa de igual forma en todos los pacientes.
Por ello es necesario desarrollar medicamentos que contribuyan desde el punto de vista del paciente y del ciudadano al uso adecuado de los recursos sanitarios y a la mejora de la salud integral de las personas.
BIOMARCADORESLos recientes avances farmacogenéticos han permitido el enfoque personalizado de la farmacología clínica modificando la forma tradicional de abordar el diagnóstico y el tratamiento de las enfermedades. Ello está permitiendo sustituir el paradigma clásico de prueba genética que diagnóstica una enfermedad genética (por lo general “incurable”), por el de biomarcador genómico (característica mensurable del ADN o del ARN) como indicador de un proceso biológico normal, de un proceso patogénico y/o de predictor de la respuesta a una intervención terapéutica. Por lo tanto, debido al importante esfuerzo promovido por diferentes entidades sociales y estatales, se ha obtenido la descripción y clasificación de los biomarcadores genéticos que proporcionan información útil sobre las diferencias farmacocinéticas y farmacodinámicas en la población, lo cual influye y determina la respuesta farmacológica final4–7. En este contexto, una característica esencial consiste en que la selección de biomarcadores se ha basado en disponibilidad de evidencia científica obtenida a partir de multitud de ensayos clínicos, en los que su utilidad ha sido demostrada y validada. Esta evidencia es la que apoyará la decisión médica, como consecuencia del perfil genético (retirada de fármacos, modificación de dosis o incluso sustitución de fármacos)8,9.
El siguiente paso consiste en la transferencia de la detección de biomarcadores farmacogenéticos a la práctica clínica. Para caracterizar los perfiles genéticos es esencial la selección de la tecnología más adecuada y óptima, con respecto a la sensibilidad y especificidad exigida y respecto al tiempo de respuesta requerido para cada situación médica10,11. La farmacología oncológica requiere un tiempo de respuesta mínimo, una sensibilidad extraordinaria y el menor límite de detección posible que permita detectar el menor porcentaje de células tumorales presentes en la muestra sobre el background de células no portadoras de mutaciones12.
Asimismo, los marcadores constitutivos, principalmente involucrados en el desarrollo de efectos adversos, requieren una detección precoz, más aún si estos efectos resultan en una limitación relevante de la calidad de vida o implican un riesgo potencialmente elevado para la vida del paciente. Además, la tipificación del perfil genético puede llevarse a cabo en diferentes etapas de la atención médica, previa, o simultánea al diagnóstico genético o clínico, o bien durante el seguimiento del paciente con el fin de promover la eficacia y/o reducir al máximo los efectos adversos del fármaco. Todos estos condicionamientos determinarán la selección de la tecnología más eficiente y apropiada para cada paciente y cada situación.
NUEVOS BIOMARCADORESLos microARNs (miARNs) son ARNs no codificantes de 21-25 nucleótidos de longitud que regulan la expresión genética a nivel post-transcripcional de forma negativa, es decir, interaccionan con sus ARNs mensajeros diana produciendo su degradación o inhibiendo la translación. Se ha visto que los niveles de expresión de muchos miARNs (miARNs oncológicos) están alterados cuando hay un cáncer, por lo que presentan gran potencial como marcadores tumorales.
Además, tienen la capacidad para establecer sub- clasificaciones de tumores y monitorizar los efectos de las terapias ya que son reguladores críticos de la expresión global de ARN en procesos biológicos normales y anormales, incluyendo el cáncer. La desregulación de miARNs se asocia a la iniciación del tumor, la resistencia a fármacos y a la aparición de metástasis.
Los miARN son capaces de manipular la expresión genética de múltiples genes, regulando por tanto procesos celulares fundamentales en el cáncer como son la proliferación celular, apoptosis, diferenciación y migración13.
Desde hace unos años se conoce que los miARNs son secretados por exosomas. Los exosomas consisten en nanovesículas formadas por una bicapa lipídica de origen endocítico que se encuentran en la mayoría de los fluidos corporales y son capaces de trasladar diferentes biomoléculas como proteínas, lípidos, ADN, ARNm y ARN no codificante, variando su contenido dependiendo de las condiciones fisiológicas, patológicas y del tipo de célula de origen. Estas vesículas se encuentran implicadas en la comunicación célula-célula transmitiendo la información que contienen. Dado a que estos exosomas son prácticamente ubicuos en los fluidos corporales, los correspondientes miARNs contenidos, estarán presentes en varios fluidos corporales, siendo interesante la diferencia entre las cantidades de miARN circulantes entre pacientes con cáncer e individuos sanos.
Por tanto, esta es una gran ventaja en este tipo de moléculas, la posibilidad de detectarse en circulación además de tejido tumoral, lo que supone que no es necesaria la intervención invasiva para el análisis de estas moléculas. Además, su característica de estabilidad en distintos fluidos corporales, su capacidad de permanecer estables tras varios ciclos de congelación-descongelación y almacenamiento prolongado, así como en tejido fijado con parafina o formol durante varios años, ofrece enormes posibilidades de realizar análisis moleculares restrospectivos en estudios con muestras grandes14. Por ello, la investigación en los procesos que regulan los miARN se ha disparado en los últimos años buscando llegar a poder medir, cuantificar estas moléculas y buscando patrones en función de estas medidas que determinasen progresión de enfermedad.
Por otra parte, el ADN circulante (cADN) en suero y plasma ha tomado protagonismo como biomarcador no invasivo, desde que en la década de los 90s donde se detectaron fragmentos del gen RAS mutado y alteraciones de los microsatélites en el cADN de pacientes con cáncer. Se comprobó que este ADN está presente en la sangre a niveles estables que pueden sufrir incrementos drásticos en caso de daño celular o necrosis. En general, las concentraciones de cADN se encuentran más elevadas en pacientes con cáncer que en individuos sanos y son aún mayores en caso de metástasis.
La necesidad y con esta el interés médico y científico, suscitado por la utilización de estas moléculas como biomarcadores debido a las importantes ventajas que su uso implica en el ámbito oncológico frente a la biopsia estándar, es cada vez mayor, si bien, se ha enfrentado a diversos retos, como son discriminar entre el ADN procedente de tumor y el cADN normal, además en ocasiones este ADN procedente de tumor, ctADN (circulating tumor DNA) se encuentra en concentraciones extremadamente bajas y finalmente conseguir una tecnología que sea capaz de cuantificar el número de fragmentos mutados en la muestra. A pesar de estos retos, las ventajas que ofrece un biomarcador no invasivo a través de la biopsia líquida frente al análisis de tumor en biopsia tisular suponen un gran impacto en la práctica clínica, la facilidad de obtención, sin incomodidad al paciente y sin alto costo, además de que evita la heterogeneidad celular dentro del propio tumor, puesto que la muestra variará sustancialmente según la zona biopsiada.
Por otro lado, la medición del ctADN frente a la medición en marcadores en tejido tumoral o imagen para conocer la dinámica del tumor resulta de mayor precisión ya que tiene una vida media muy corta (de unas 2 horas), lo que permite la evaluación de cambios en el tumor en horas en lugar de semanas o meses y por tanto, la posible toma de decisiones para la modificación del régimen de tratamiento en poco tiempo. Además, es enormemente específico puesto que sólo puede encontrarse en sangre cuando hay un cáncer en el organismo, no viéndose elevado en otras situaciones patológicas como sucedía con otros marcadores serológicos.
Cuestiones como el análisis de la enfermedad mínima residual tras tratamiento quirúrgico o químico, pueden llevarse a cabo gracias a la sensibilidad de las técnicas permite la detección de niveles de ctADN muy bajos liberados de depósitos micrometastásicos no detectables por imagen ni por otras determinaciones15,16.
Finalmente, las terapias oncológicas que antes eran efectivas, están mostrando resistencias clínicas y esto es debido a la adquisición de alteraciones moleculares en genes o rutas determinadas que regulan el mecanismo de acción. El trabajo en biopsia líquida permite ser eficaz en la monitorización de los clones resistentes durante el curso del tratamiento.
TECNOLOGÍASEl objetivo final del desarrollo tecnológico en salud es avanzar hacia una medicina individualizada y de precisión. En este sentido las técnicas cuantitativas, resultan las más apropiadas para la detección de variantes de baja frecuencia con la mayor sensibilidad y exactitud, el menor límite de detección y con el menor tiempo de obtención de resultados. Entre esta nueva generación de tecnologías cuantitativas destacan las metodologías digitales, particularmente la ddPCR, para la que han sido descritos parámetros óptimos de sensibilidad, límite de detección y tiempo de entrega de resultados, que son tan esenciales en el diagnóstico y seguimiento en el campo oncológico.
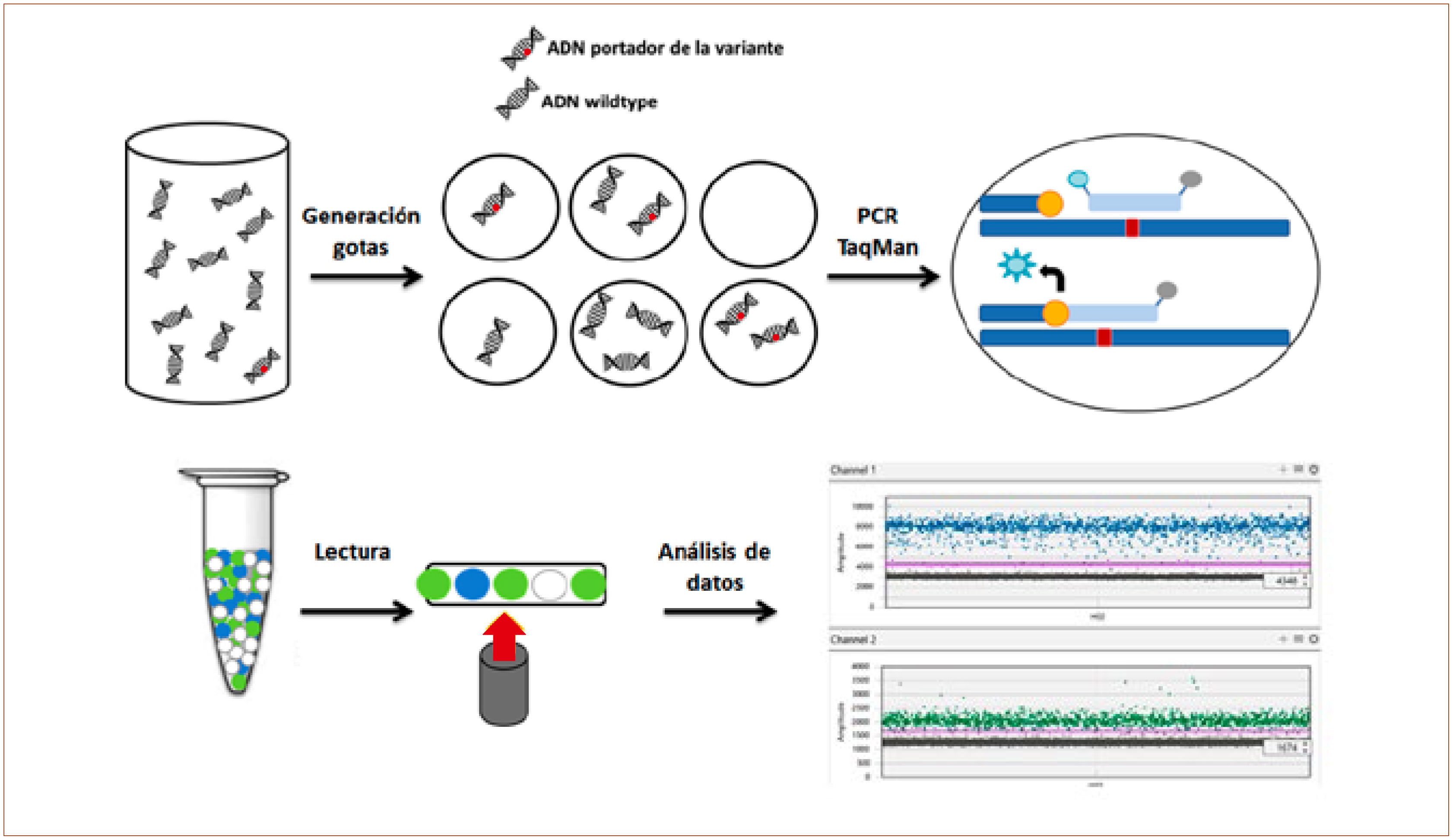
El procedimiento experimental para la metodología ddPCR es de gran sencillez (Figura 1), consistiendo en un primer paso de distribución de los reactivos de PCR junto con la muestra a analizar incluyendo primers y sondas fluorescentes (FAM y VIC/HEX) o bien agentes intercalantes inespecíficos. Este mix de reacción será automáticamente distribuido en hasta 20000 gotas de aceite por medio de un proceso de emulsión. Con este proceso dispondremos en cada gota una molécula de ADN target ya sea portadora de la variante o wildtype, y así de forma individual al introducir las muestras en el correspondiente termociclador se producirá un amplificación clonal independiente del ADN tanto target como wildtype. Por último, la señal analógica se cuantifica mediante un lector debido a la incorporación de sondas/intercalantes fluorescentes y se transforma en una señal digital en forma de presencia o ausencia. Dado que el equipo incorpora un sistema de citometría de flujo, se podrán cuantificar el número de eventos totales y de los eventos positivos y negativos que finalmente se verán transformados en un ratio de porcentaje de ADN para esa muestra portador de la mutación. Al tratarse de una metodología basada en una técnica cuantitativa, el procedimiento experimental sólo implica la preparación de las muestras y el procesamiento propiamente dicho, pudiendo estimarse el proceso completo en aproximadamente 8h incluyendo el análisis de resultados.
ESQUEMA REPRESENTATIVO DE LA METODOLOGÍA DIGITAL DROPLET PCR
El procedimiento se basa un primer paso de generación de gotas en la que se distribuirán las moléculas aleatoriamente. Posteriormente se realizará sobre cada gota la correspondiente amplificación por PCR con una pareja de sondas TaqMan, cada una de ellas reconocerá o bien secuencia mutada o la wildtype, y cada una de ellas estará marcada con diferentes fluoróforos FAM o HEX/VIC (azul y verde respectivamente). Tras la amplificación, mediante el sistema de citometría de flujo, se analizará a través del lector la señal emitida por cada una de las gotas de una en una de ellas. Finalmente el sistema genera el correspondiente gráfico de resultados que permite en este ejemplo la cuantificación correspondiente en valores de copias/μL.
La comparativa entre versiones más clásicas de PCR cuantitativa frente a las nuevas versiones digitales como la ddPCR muestra una serie de ventajas a favor de estas tecnologías emergentes. Entre las características mejoradas se incluye la capacidad de llevar a cabo una cuantificación absoluta sin requerir una curva estándar, debido a los modelos matemáticos incorporados en los diferentes softwares de análisis y que se lleva a cabo conteo total de eventos, y particular de eventos positivos y negativos17. Además permite potenciar la señal debida a la presencia de la variante frente al ruido generador la ausencia de la misma, lo cual resulta de gran utilidad para muestras escasas (tejidos parafinados, tejidos congelados o incluso muestras de ctADN. Esto es así gracias a que el procedimiento experimental se basa en la amplificación clonal de una molécula molde de ADN en cada gota generada, incrementando así el ratio de secuencias de ADN portadoras de la mutación amplificadas frente al ADN wildtype18. Por otro lado, la generación de gotas limita la interferencia de inhibidores como contaminantes o parafinas, gracias al “secuestro” en la misma gota del material de interés quedando fuera los interferentes. Esta ventaja es de particular utilidad cuando tratamos con muestras parafinadas, clásicas del área de oncología19. Finalmente el límite de detección está directamente relacionado con la cantidad de la muestra. De este modo, es posible llevar a cabo diseños experimentales con mayor número de réplicas por cada ensayo a testar. El equipo finalmente lleva a cabo un sumatorio de todos los resultados obtenidos para una misma muestra, para una variante determinada. Con este sistema la metodología permite detectar hasta 1 secuencia portadora de la variante entre 1 millón de secuencias wildtype, simplemente a partir de 10ng de muestra20,21.
Además de su especial utilidad para la determinación de secuencias escasas en el campo de la biomedicina, esta metodología permite el abordaje de la ya comentada cuantificación absoluta para ADN, carga viral entre otras, permite el diseño experimental para la detección de Copy Number Variations (CNVs), así como expresión génica, análisis de microARNs y de transcritos génicos alternativos por splicing diferencial y cuantificación de librerías para secuenciación masiva previo al procedimiento propio de secuenciación22–25.
Por otro lado, las técnicas de secuenciación masiva han modificado el modelo diagnóstico de enfermedades de baja frecuencia así como de enfermedades de alta heterogeneidad genética y clínica. Su uso ha generado más eficiencia, menor coste sanitario y mayor precisión en los resultados frente a técnicas tradicionales tipo Sanger. Por lo tanto, su uso en enfermedades hereditarias es claro y ya está instaurado en la práctica habitual clínica, y su uso en el estudio del ctADN, a partir de biopsias líquidas, como complemento o alternativa a la Next Generation Sequencing (NGS) en los tejidos está siendo ya una realidad en investigación.
La potencia de análisis de ambas tecnologías y la combinación de ellas en el proceso de análisis farmacogenómico y farmacogenético permitirán avanzar en el camino hacia la medicina de precisión.
CONCLUSIONESSi hablamos en términos de costo-efectividad, debemos encaminar los esfuerzos en sanidad hacia el diagnóstico precoz en enfermedades de alta heterogeneidad genética y clínica y en la rápida detección de recidivas en enfermedad neoplásica. Ambas situaciones incrementan enormemente las posibilidades de que los tratamientos del paciente o de sus familiares (ya que en enfermedades genéticas el paciente es “la familia”) resulten eficaces y por tanto contribuyen a aumentar la supervivencia del paciente y mejorar su calidad de vida.
Los avances tecnológicos en la capacidad de obtener datos genómicos procedentes de los pacientes unidos al desarrollo de análisis “big data” hacen que se pueda integrar en la toma de decisiones terapéuticas una cantidad de datos muy valiosa, no solo para el paciente sino para la sociedad, permitiendo la optimización de recursos, el evitar hacer pruebas innecesarias y adelantarse a los procesos de enfermedad en fases más complejas para su manejo.
Esta nueva forma de trabajo está recién iniciándose, y está trayendo un cambio en el enfoque tradicional de la medicina que va a repercutir en las decisiones de las autoridades sanitarias, los planificadores y los gestores sanitarios. Sin embargo, para lograr este nuevo abordaje en la práctica clínica es necesaria una formación continuada y concienciación adicional de los profesionales sanitarios involucrados en estos ámbitos. La utilidad y los beneficios que puede aportar el estudio farmacogenético precoz abarcan la identificación temprana de individuos en riesgo, el establecimiento de un diagnóstico temprano así como la caracterización “genético-molecular” de la enfermedad propiamente establecida, emplear este perfil para predecir el pronóstico y valorar la opción terapéutica más apropiada para el paciente y poder realizar un seguimiento de el mismo a través de este biomarcador y finalmente identificar de forma temprana la posible aparición de resistencias a la terapia o incluso recidivas de la enfermedad. De este modo, evaluando las grandes aportaciones de estos estudios se hace visible la enorme relevancia de incorporar en nuestra práctica clínica diaria este punto de vista, que no sólo beneficiará a los pacientes sino que adicionalmente a la rutina diaria de los clínicos involucrados, facilitando su desempeño diario.
AUTHOR DECLARATIONWe wish to confirm that there are no known conflicts of interest associated with this publication and there has been no significant financial support for this work that could have influenced its outcome.
We confirm that the manuscript has been read and approved by all named authors and that there are no other persons who satisfied the criteria for authorship but are not listed. We further confirm that the order of authors listed in the manuscript has been approved by all of us.
We confirm that we have given due consideration to the protection of intellectual property associated with this work and that there are no impediments to publication, including the timing of publication, with respect to intellectual property. In so doing, we confirm that we have followed the regulations of our institutions concerning intellectual property.
We understand that the Corresponding Author is the sole contact for the Editorial process (including Editorial Manager and direct communications with the office).
She is responsible for communicating with the other authors about progress, submissions of revisions and final approval of proofs. We confirm that we have provided a current, correct email address which is accessible by the Corresponding Author and which has been configured to accept email from mayte.gil@sistemasgenomicos.com