El estadio del cáncer de mama constituye uno de los factores pronósticos más relevantes. Sin embargo, la compleja clasificación TNM, la existencia de diferentes versiones y la variabilidad de la fuente de la información hacen que la recogida de datos sobre texto libre sea compleja. El objetivo de este trabajo es desarrollar una herramienta que permita ayudar a la estadificación de manera automática.
Pacientes y métodosEl trabajo incluyó el estudio de los informes de 100 pacientes con cáncer de mama no metastásico tratadas con cirugía y radioterapia en 2012. La recogida del tamaño tumoral posquirúrgico (séptima edición TNM) se realizó con la herramienta desarrollada y manualmente por un médico en formación especializada de tercer año de oncología radioterápica.
ResultadosLa aplicación fue capaz de detectar el 62% de los casos tras examinar los informes de anatomía patológica, y el 77% al añadir el examen de los informes de oncología radioterápica. Los casos no detectados se debieron a que la información estaba almacenada en otra sección de la estación clínica. Comparando los resultados entre la aplicación y la recogida manual, hubo una diferencia del 13% (10/77). Se observó que en el 50% de los casos (5/10) la aplicación era correcta, mientras que en el otro 50% lo fue la recogida manual.
ConclusionesEsta herramienta innovadora permite recoger automáticamente el tamaño tumoral en el cáncer de mama, ahorrando tiempo en la recogida de datos y evitando errores en la clasificación tumoral, por lo que puede contribuir de modo notable en la decisión terapéutica.
Staging of breast cancer is one of the most important prognostic factors. However, collecting data for staging manually from unstructured free text is variable and imprecise because of the complexity of the TNM classification, the existence of different versions over time, and variability in the source used to obtain data. The aim of this study was to develop an artificial intelligence tool to allow data on tumoral staging to be mined automatically.
Patients and methodsThe study included the reports of the first 100 patients with nonmetastatic breast cancer treated with surgery and radiotherapy in 2012. Data on postoperative tumor size (TNM seventh edition) were collected with a specially designed software tool and manually by a third-year resident physician in radiation oncology.
ResultsThe software application detected 62% of cases when pathology reports were included, and 77% when radiation oncology reports were added. Non-detection was due to the information being stored in another section of the clinical station. When we compared the results of the software application and manual collection, we found a difference of 13% (10/77). In these 10 cases, the application was correct in 50%, while manual collection was correct in the remaining 50%.
ConclusionsThis innovative system allows automatic staging of tumoral size in breast cancer. The use of this tool would save time in data collection and prevent errors in tumoral classification and could also improve therapeutic decisions.
El estadio tumoral constituye uno de los factores pronósticos más relevantes en el cáncer de mama. Sin embargo, la compleja clasificación TNM1, la existencia de diferentes versiones a lo largo del tiempo y la variabilidad de la fuente de la información hacen que la recogida de datos sobre texto libre sea compleja. A pesar del uso cada vez mayor de la historia clínica electrónica, la complejidad del lenguaje natural (texto libre) existente en los informes hace que sea difícil la recuperación sencilla de datos de forma automatizada. Varias técnicas han sido utilizadas para extraer la información de interés o conocimiento de grandes bases de datos2,3.
El objetivo general de este estudio es determinar si la información sobre el estadio tumoral se podría recuperar con precisión a partir de informes de anatomía patológica (AP) y oncología radioterápica (OR), sin estructurar, mediante un simple procesador de lenguaje natural (NLP) y la aplicación de algoritmos de clasificación usados en minería de datos y aprendizaje automático. Este estudio preliminar se encamina a desarrollar una herramienta que permita obtener la clasificación del tamaño tumoral (estadioT) en el cáncer de mama de manera automática, ahorrando tiempo y reduciendo la variabilidad en la recogida de datos.
MétodosSelección de pacientesSe trata de un trabajo de naturaleza retrospectiva que ha seguido los protocolos establecidos por nuestra institución para el acceso a los datos de historias clínicas con fines de investigación y divulgación científica. El estudio incluyó los informes clínicos de las 100 primeras pacientes con cáncer de mama no metastásico tratadas con cirugía y radioterapia en 2012. Diez pacientes recibieron quimioterapia neoayuvante. Estos datos se obtuvieron de la base de datos del Servicio de Oncología Radioterápica. La recogida del tamaño tumoral posquirúrgico (pT; según la séptima edición TNM1) se realizó con la herramienta desarrollada, y paralelamente, de forma manual por un médico en formación especializada de tercer año de oncología radioterápica.
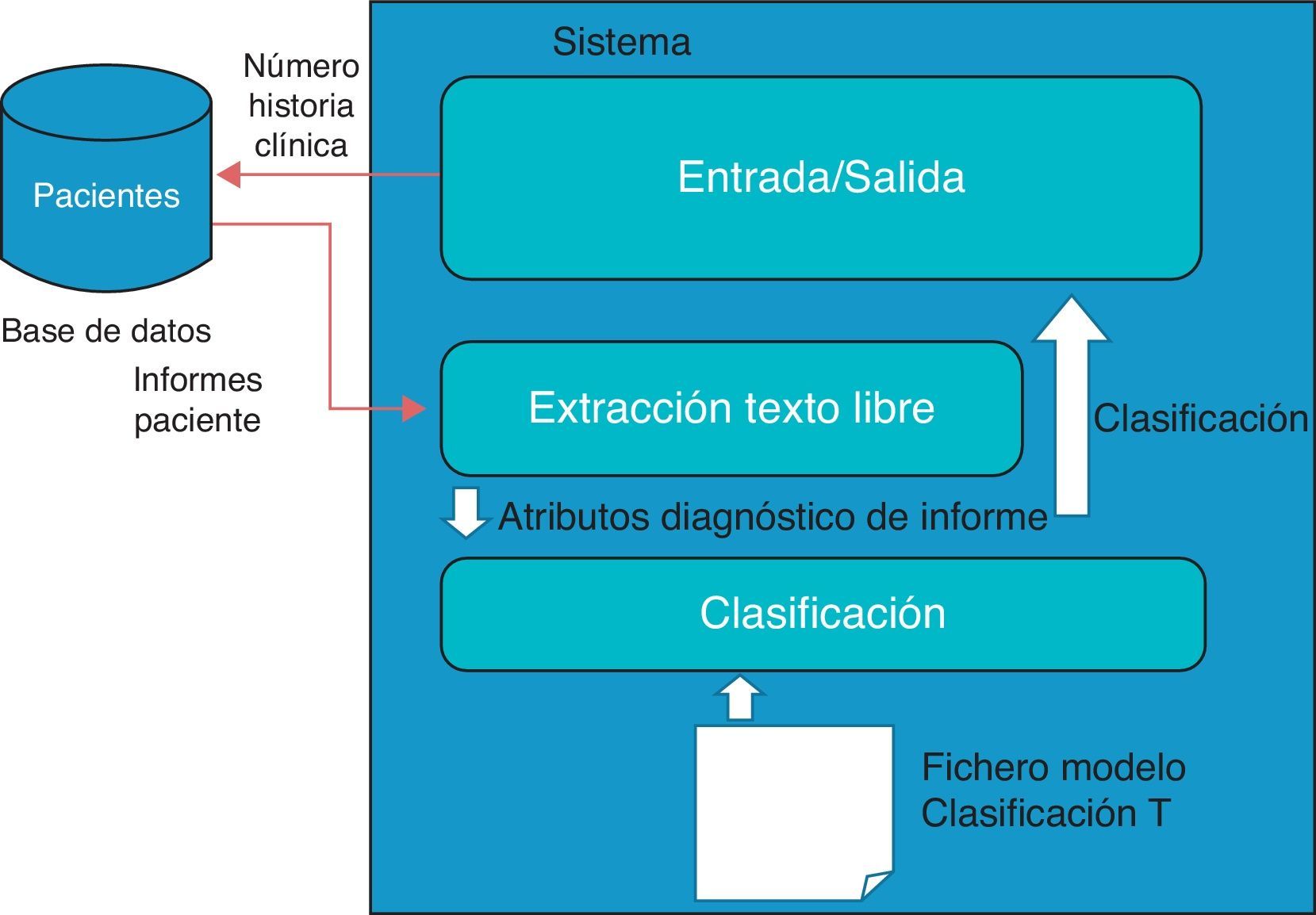
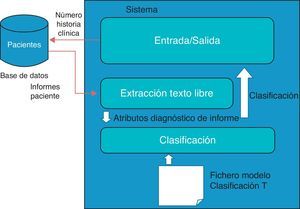
HerramientaLa herramienta consiste en una aplicación Java que obtiene información a partir de los informes de AP y OR, escritos en texto libre, mediante la aplicación de algoritmos de filtrado de texto y extracción de datos y la posterior clasificación de casos. Aquellos casos en los que se observaron divergencias fueron revisados por un médico especialista de OR experto. El diseño de la aplicación se muestra en la figura 1. El primer paso fue evaluar si los informes clínicos contenían la información necesaria para la clasificación del tamaño del tumor. A continuación, se desarrolló una herramienta basada en la detección de términos relacionados con el tamaño del tumor. Por último, se evaluó si la aplicación podría recuperar y clasificar la información sobre el tamaño del tumor de los informes no estructurados con una precisión similar a la de un clínico.
El módulo NLP de la herramienta busca las características del tumor escritas en el área de diagnóstico de los informes de AP mediante la detección de palabras clave. Algunas de esas palabras clave están asociadas a características que definen el tumor (tipo, tamaño, capacidad de invasión, etc.). Estos atributos se establecieron para el reconocimiento de la clasificación T en los informes electrónicos. La variabilidad entre los distintos redactores de informes ha sido tenida en cuenta dotando a la herramienta informática de los diferentes atributos que representan esta variabilidad (por ejemplo, escribir las unidades en acrónimos [cm vs. centímetro], escribir los números con cifras o letras [4 vs. cuatro], etc.). El diagnóstico del paciente queda definido en forma de vector, y posteriormente este diagnóstico es clasificado usando distintos algoritmos de clasificación.
Algoritmos de clasificaciónLos algoritmos empleados fueron: J48 basado en el algoritmo C4.54, LADtree5 y NaiveBayes6. Todos ellos forman parte de Weka7. Weka se ha utilizado en muchos estudios médicos y aplicaciones8, especialmente relacionados con el cáncer9,10. Estos algoritmos fueron previamente entrenados en la obtención de la clasificación T. Para ello se usó un conjunto de entrenamiento compuesto por los vectores de características de 65 pacientes «virtuales» (existiendo al menos 5 ejemplos de cada tipo de clasificación) y su clasificación T, dada por el experto. La decisión de tomar 65 pacientes virtuales vino dada por la necesidad de cubrir las 13 categorías T (T0, Tx, T1mi, Tis, T1a, T1b, T1c, T2, T3, T4a, T4b, T4c y T4d) y de este modo evitar construir un grupo de entrenamiento inadecuado que pudiera no incorporar alguna de las categorías. Para ello, debíamos tener al menos 2 ejemplos por categoría, que representasen el margen superior e inferior de cada una. Decidimos dar 5 ejemplos por categoría para obtener un grupo de entrenamiento con una muestra parecida a la del grupo de prueba (N=100). En ocasiones sí se tomaron pacientes reales, pero no todas las categorías disponían de estos en nuestro grupo de prueba, por esa razón se recurrió a pacientes virtuales. El árbol de decisión obtenido apenas variaba su precisión por mucho que se aumentara el número de casos en el grupo de entrenamiento. Se trata, pues, de un aprendizaje supervisado del modelo de clasificación, que posteriormente fue empleado para clasificar a los 100 pacientes de nuestro estudio.
Los algoritmos anteriormente mencionados fueron seleccionados por varias razones. En primer lugar, J48 y LADtree son algoritmos que devuelven un árbol de decisión11, estos son usados en aplicaciones y estudios médicos actuales12,13. La segunda razón fue el alto grado de acierto que tuvieron ambos algoritmos cuando procedimos a la validación del modelo aprendido. La validación del modelo la hicimos mediante el proceso de validación cruzada por 10 que ofrece Weka14. La selección del algoritmo NaiveBayes para este estudio se debió a otras razones. NaiveBayes no proporciona un árbol de decisión como en los 2 casos anteriores, ni fue uno de los algoritmos que más aciertos consiguió con la validación cruzada, sin embargo, NaiveBayes combina eficiencia y buena precisión y suele ser usado como línea base en multitud de estudios y artículos de investigación15. De esta forma, nos sirve para comparar las tasas de aprendizaje de los distintos algoritmos, independientemente del escenario que se quiera aprender.
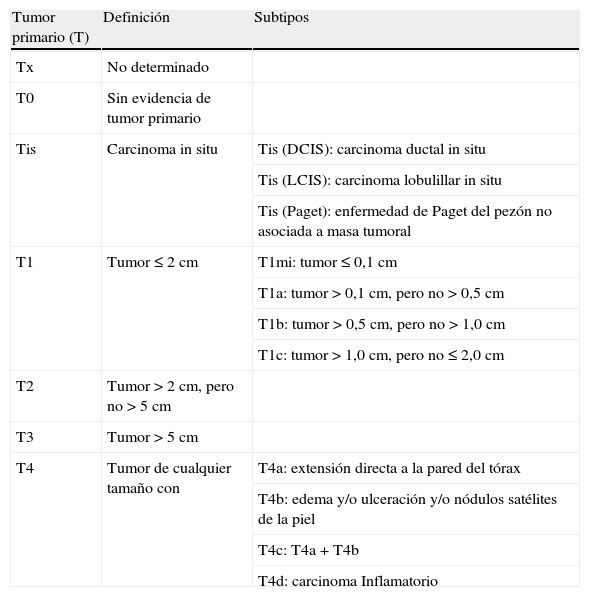
AnálisisLos datos acerca del tamaño tumoral se obtuvieron a partir de los informes clínicos seleccionados (AP y OR), los cuales fueron revisados por un médico en formación especializada de tercer año en OR de acuerdo con el sistema de estadificación que se detalla en la guía TNM (séptima edición; tabla 1). Posteriormente, un médico especialista en radioterapia revisó las divergencias encontradas al realizar la comparación entre los resultados de la clasificación obtenida por el médico en formación especializada y la obtenida por la herramienta (software) desarrollada para conocer el grado de acierto de esta, así como los errores cometidos. Calculamos la sensibilidad de la clasificación automática realizada por la herramienta en comparación con la realizada por el oncólogo experto. Definimos sensibilidad como el número de casos acertados (verdaderos positivos [VP]) dividido por la suma de los casos acertados (VP) y los no acertados (falsos negativos [FN]). La ecuación queda como se muestra a continuación: VP/(VP+FN). Además, se calculó el intervalo de confianza (IC) correspondiente
Clasificación del tamaño tumoral (T) en cáncer de mama según la séptima edición TNM de la American Joint Committee on Cancer
| Tumor primario (T) | Definición | Subtipos |
| Tx | No determinado | |
| T0 | Sin evidencia de tumor primario | |
| Tis | Carcinoma in situ | Tis (DCIS): carcinoma ductal in situ |
| Tis (LCIS): carcinoma lobulillar in situ | ||
| Tis (Paget): enfermedad de Paget del pezón no asociada a masa tumoral | ||
| T1 | Tumor≤2cm | T1mi: tumor≤0,1cm |
| T1a: tumor>0,1cm, pero no>0,5cm | ||
| T1b: tumor>0,5cm, pero no>1,0cm | ||
| T1c: tumor>1,0cm, pero no≤2,0cm | ||
| T2 | Tumor>2cm, pero no>5cm | |
| T3 | Tumor>5cm | |
| T4 | Tumor de cualquier tamaño con | T4a: extensión directa a la pared del tórax |
| T4b: edema y/o ulceración y/o nódulos satélites de la piel | ||
| T4c: T4a+T4b | ||
| T4d: carcinoma Inflamatorio |
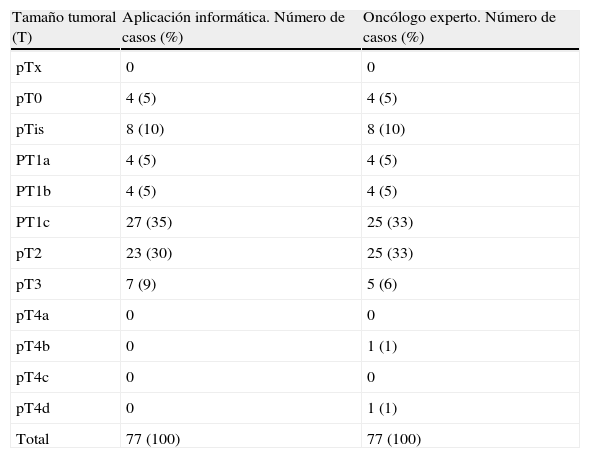
La aplicación fue capaz de detectar el 62% de los casos tras examinar los informes de AP, y el 77% al añadir el examen de los informes de OR (tabla 2). La sensibilidad de la aplicación fue del 93,5% (IC 95%: 85,49-97,85).
Distribución de los casos detectados por la aplicación y clasificados por el oncólogo experto en función de cada categoría T
| Tamaño tumoral (T) | Aplicación informática. Número de casos (%) | Oncólogo experto. Número de casos (%) |
| pTx | 0 | 0 |
| pT0 | 4 (5) | 4 (5) |
| pTis | 8 (10) | 8 (10) |
| PT1a | 4 (5) | 4 (5) |
| PT1b | 4 (5) | 4 (5) |
| PT1c | 27 (35) | 25 (33) |
| pT2 | 23 (30) | 25 (33) |
| pT3 | 7 (9) | 5 (6) |
| pT4a | 0 | 0 |
| pT4b | 0 | 1 (1) |
| pT4c | 0 | 0 |
| pT4d | 0 | 1 (1) |
| Total | 77 (100) | 77 (100) |
El médico en formación especializada en OR realizó la clasificación manual de los 100 pacientes incluidos en el estudio. El tiempo medio dedicado por el médico en formación especializada para la clasificación de un solo paciente fue de 5min, mientras que nuestra aplicación fue capaz de clasificar cerca de 100 pacientes en menos de 1segundo.
Análisis comparativoComparando los resultados entre la aplicación y la recogida manual, hubo una diferencia del 13% (10/77). Al examinar estos casos, se observó que en el 50% (5/10) la aplicación era correcta, mientras que en el otro 50% lo fue la recogida manual. El error más común (N=3) cometido por la aplicación fue la falta de capacidad para estadificar el carcinoma inflamatorio (pT4d) debido a que el estadio posquirúrgico se sigue considerando como carcinoma inflamatorio, aunque se resuelva la inflamación tras quimioterapia neoadyuvante. El error más común en la recogida manual (N=3) fue designar con el estadio inicial clínico aquellos casos con respuesta completa a la quimioterapia tras la cirugía (ypT0).
DiscusiónDisponer de datos estructurados para poderlos analizar es una norma en la medicina basada en la evidencia. El tratamiento oncológico, y en particular el de cáncer de mama, han evolucionado en las últimas décadas gracias a los análisis de datos relacionados con los pacientes, el tumor y el tratamiento. En la mayoría de los centros oncológicos aún no se dispone de una base de datos estructurada para la recogida prospectiva de datos en la práctica clínica diaria. Esto hace que se tenga que dedicar un tiempo adicional y en muchos casos contar con un personal específico para esta tarea. Nuestro trabajo se encamina a automatizar esta tarea de manera que se puedan obtener datos estructurados a partir de texto libre para su posterior análisis. Los resultados obtenidos en este estudio preliminar pueden resumirse en los siguientes puntos: primero, nuestro equipo multidisciplinar de innovación tecnológica y OR ha desarrollado una herramienta informática capaz de detectar el tamaño tumoral postoperatorio a partir de texto libre. Segundo, la herramienta desarrollada detectó la información mencionada en la mayoría de los casos (>75%). Los casos no detectados se debieron a que no existían informes de AP almacenados en la estación clínica porque las pacientes habían sido biopsiadas e intervenidas en otro hospital (pero realizando la radioterapia en nuestro centro), no disponiendo, por tanto, de los informes patológicos originales. Tercero, la herramienta tuvo un gran número de aciertos (>93%) en cuanto a la obtención de datos tras compararla con la extracción manual. Por último, la herramienta obtuvo los datos de manera automática mientras que los profesionales de OR precisaron varias horas y la ayuda de libros de texto (clasificación TNM) para realizar la clasificación de forma precisa.
Este problema ha sido analizado en los estudios de Nguyen et al.16 y Cheng et al.17, pero con enfoques diferentes. Nuestra herramienta no identifica términos propios del SNOMED-CT18 como en los casos mencionados, ni hace uso de ninguna ontología. En nuestro caso la información se encuentra en forma de texto libre, principalmente en el campo diagnóstico de los informes de AP y en el campo juicio clínico, en los informes de OR, y hemos empleado el mismo NLP en los distintos tipos de informes, con lo que la extracción de información sigue siempre el mismo proceso, independientemente del tipo de informe. La diferencia observada en los resultados viene del uso de distintos algoritmos para el aprendizaje de la clasificación T.
Nuestra herramienta funciona como clasificador preciso y automático. También permite adaptación si decidimos cambiar los criterios de clasificación de tamaño T, por ejemplo, el cambio de rango de tamaño para el estado T. Todo esto nos da la oportunidad de modificar la herramienta y actualizarla ante un cambio en los criterios de clasificación de la guía a lo largo del tiempo.
Las limitaciones de la herramienta se podrían solventar mediante la utilización de bases de datos estructuradas en lugar de texto libre. Una aplicación de este tipo puede ahorrar tiempo, esfuerzos y también puede reducir los errores derivados de la actividad diaria. El ahorro en tiempo es uno de los factores más fácilmente cuantificables. La herramienta supuso en este estudio (N=100) un ahorro en tiempo de unos 500min u 8horas de trabajo, que equivale a una jornada laboral. Otro beneficio que nos aporta el uso de esta herramienta es su eficacia, aunque se produzca un cambio en la guía TNM de clasificación, que, como sabemos, es susceptible de llevarse a cabo en futuras ediciones. El uso de algoritmos de aprendizaje nos permite modificar el sistema de clasificación sin cambiar la codificación de nuestra herramienta software. Lo único que nos haría falta es un nuevo conjunto de entrenamiento cuyos ejemplos estén clasificados siguiendo los nuevos criterios descritos en la nueva revisión de la guía.
Este innovador sistema permite recoger automáticamente el tamaño tumoral en el cáncer de mama y llevar a cabo una estadificación más precisa al evitar errores humanos en la clasificación manual (por ejemplo, utilizar una versión de la clasificación TNM anterior a la más actualizada o interpretar el estadio patológico como clínico cuando hay respuesta completa a la quimioterapia neoadyuvante). El siguiente paso consiste en incluir el estadio de los ganglios (N) y, por último, la presencia o no de metástasis a distancia en el diagnóstico (M) para poder calcular el estadio TNM automáticamente. Por tanto, consideramos que puede ser una herramienta aplicable a las unidades multidisciplinares de cáncer de mama que ayudaría a ahorrar tiempo en la recogida de datos, evitar errores en la clasificación tumoral, orientar la decisión terapéutica y mejorar la calidad de los estudios de investigación.
FinanciaciónProyecto financiado por el Ministerio de Ciencia e Innovación. Ayudas al Subprograma INNPACTO, Plan Nacional de Investigación Científica, Desarrollo e Innovación Tecnológica 2008-2011. Subdirección General de Estrategias Público-Privadas. Proyecto: Modelo semántico y algoritmos de DM aplicados al tratamiento del cáncer de mama. Expediente: IPT-2011-1126-900000. Red de Innovación en Tecnología Médica y Sanitaria (Red ITEMAS). Expediente: RD09/0077/0025.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.