Staphylococcus aureus resistente a la meticilina (SARM) es un patógeno nosocomial, con reservorio en portadores o infectados y que tiene como principal mecanismo de transmisión el contacto con las manos del personal.
MétodosSe puso en marcha una investigación epidemiológica y se realizaron determinaciones de laboratorio para abordar el estudio de la situación provocada por la aparición de nuevos casos de SARM resistente a los descolonizadores habituales.
ResultadosDesde septiembre de 2010 a febrero de 2012, en nuestro centro, 16 pacientes y una trabajadora tuvieron un aislamiento de SARM resistente a los descolonizadores habituales (mupirocina y ácido fusídico). Se detectaron casos esporádicos no relacionados, y a su vez brotes epidémicos relacionados con la ubicación de la actividad del personal sanitario portador de SARM. El análisis mediante electroforesis en campo pulsado de las muestras de pacientes y de la trabajadora puso de manifiesto la clonalidad de las cepas, lo que sugiere que el reservorio pudiera ser la trabajadora afectada. La descontaminación con antibióticos sistémicos no tuvo éxito y la trabajadora fue adscrita a otro puesto de trabajo sin contacto directo con pacientes, con lo que se consiguió que no aparecieran más casos hasta el momento actual (septiembre de 2012).
ConclusionesEste trabajo ilustra el riesgo de transmisión nosocomial relacionada con la atención prestada por los trabajadores sanitarios.
Methicillin-resistant Staphylococcus aureus (MRSA) has become an important hospital-acquired pathogen, with transfer of the organism from a carrier or infected patient to uninfected patients by the hands or clothing of staff as the main mode of transmission.
MethodsInvestigation of a cluster of new cases of MRSA resistant to mupirocin and fusidic acid, using epidemiological and microbiological resources.
ResultsFrom September 2010 to February 2012, sixteen patients had at least one culture positive for MRSA resistant to mupirocin and fusidic acid. Some not apparently related cases and outbreaks appeared. By analysing cultures taken from patients and staff using pulsed-field gel electrophoresis, it was demonstrated that most likely this situation was started by an auxiliary nurse who was a carrier of the MRSA. Healthcare worker decontamination using oral antibiotic therapy was unsuccessful. Eventually, the situation was controlled by placing the carrier in a different job, with no further cases to date (September, 2012).
ConclusionThis report illustrates the risk of nosocomial transmission linked to care delivered by healthcare workers.
Staphylococcus aureus resistente a la meticilina (SARM) ocasiona brotes epidémicos intrahospitalarios, y en muchos casos se está comportando como un microorganismo endémico. Los reservorios son principalmente los pacientes colonizados, pero el personal sanitario también puede actuar como tal, aunque el verdadero papel del personal sanitario en la transmisión de este patógeno puede ser distinta para cada centro o situación, a veces difícil de determinar, pero con evidencias de su implicación1,2.
La mupirocina es uno de los antibióticos más usados para erradicar el estado de portador nasal de SARM, pero el incremento en su uso está llevando al aumento de la incidencia de su resistencia desde hace algunos años3,4, hecho que está recibiendo mayor atención.
La resistencia a la mupirocina puede ser de bajo nivel (CMI=8-256μg/ml) o de alto nivel (RAN) (CMI≥512μg/ml). Esta última es la de mayor interés epidemiológico, pues puede favorecer la extensión de cepas resistentes al estar mediada dicha resistencia por plásmidos4,5.
La resistencia al ácido fusídico está menos documentada. Un estudio realizado en hospitales canadienses puso de manifiesto que la probabilidad de resistencia al ácido fusídico es más frecuente entre las cepas resistentes a la mupirocina que entre las sensibles5. En otro estudio se ha visto que la resistencia al ácido fusídico puede ser del 90% en las cepas con alto nivel de resistencia a la mupirocina4. Se piensa que los genes que codifican la resistencia al ácido fusídico y la de alto nivel a la mupirocina se encuentran en el mismo plásmido6.
Entre septiembre de 2010 y febrero de 2012 aparecieron en el Hospital Universitario Insular de Gran Canaria (HUIGC) 16 muestras positivas de SARM resistente a los descolonizadores tópicos habituales (RAN a mupirocina y ácido fusídico), con posible reservorio detectado en un trabajador sanitario. Se describen los hallazgos y el comportamiento de esas cepas de SARM no habituales en nuestro centro y las medidas para su control.
Material y métodosVigilancia epidemiológicaEstudio descriptivo retrospectivo de nuevos casos de SARM resistentes a la mupirocina y al ácido fusídico entre septiembre de 2010-febrero de 2012 en un hospital de agudos de tercer nivel de 445 camas(24 de cuidados intensivos) que tiene 411.618 habitantes asignados y ubicado en el área sanitaria sur de Gran Canaria (España). Es el centro regional de referencia para las prestaciones a pacientes con lesión medular, implante coclear y medicina tropical.
En el centro existe un programa de vigilancia, prevención y control de la infección nosocomial, entre cuyas actividades se encuentra la vigilancia epidemiológica del SARM, que incluye una estrategia de detección activa de portadores al ingreso mediante el estudio de frotis nasales en pacientes admitidos en las áreas en las que las tasas de infección son más elevadas, entre ellas: planta de hospitalización de medicina interna, de cuidados intensivos, planta de cirugía general y digestiva y otra planta de hospitalización de especialidades quirúrgicas (neurocirugía, urología y cirugía vascular). Según este programa, cada aislamiento de SARM es notificado diariamente por el servicio de microbiología al servicio de medicina preventiva, donde personal debidamente entrenado realiza un estudio pormenorizado de cada caso de infección y/o colonización por cualquier cepa de SARM, el cual es estudiado de forma individualizada para clasificarlo y valorar la relación epidemiológica de los casos y su posible adquisición nosocomial.
En nuestro centro, la resistencia de Staphylococcus aureus resistente a la oxacilina está en torno al 20%. La tasa de incidencia de casos de SARM de adquisición nosocomial era de 0,20 casos por 1.000 estancias en 2005, ascendiendo paulatinamente hasta alcanzar un máximo de 0,33 casos por 1.000, para luego disminuir después de comenzar con el programa de vigilancia activa de SARM en 2009, 2010 y 2011, donde la tasa de incidencia está estabilizada en torno a los 0,25 casos por 1.000 estancias. Ocasionalmente aparecen 1-2 brotes epidémicos al año, habitualmente de escasa repercusión (3-4 casos por brote) y con adecuada respuesta a las medidas de control que se instauran.
Los pacientes ingresados en los que se detecta colonización nasal por SARM son sometidos habitualmente a descolonización mediante las pautas recomendadas para el tratamiento descritas en el documento de consenso de Vigilancia y control de SARM en los hospitales españoles7, con mupirocina mediante administración tópica en ambas fosas nasales, 3 veces al día, más higiene con gel de clorhexidina durante 5 días, o ácido fusídico tópico (2%) 2 veces al día durante 5 días en aquellos pacientes con cepas resistentes a la mupirocina. En los casos de resistencia a ambos no se ha realizado tratamiento descolonizador de pacientes.
Se definió el caso como muestra positiva para SARM resistente a la mupirocina (RAN) y al ácido fusídico (SARM MupFusr). Se tuvo en cuenta una sola muestra por paciente, excepto si hubiera indicios de una nueva situación clínica de infección o colonización. Cuando el paciente presentó síntomas o signos de infección se consideró como infectado; en otro caso, se consideró colonizado. La clasificación de los casos como nosocomiales o comunitarios se hizo teniendo en cuenta criterios epidemiológicos bien definidos en nuestro medio7.
IntervencionesCon motivo de la aparición de estos casos no habituales en nuestro centro, se instauraron medidas de aislamiento de contacto, se reforzaron las precauciones estándar (higiene de manos, limpieza medioambiental, desinfección de material clínico) y se hizo un cribado con frotis nasales de todo el personal sanitario adscrito a la sala de hospitalización con brote por esta cepa, siguiendo recomendaciones científicas1,2,7.
Métodos de laboratorioTransporte y conservación de la muestraPara los frotis nasales se empleó una torunda humedecida para ambas fosas nasales, rotándola al menos 5 veces, y posteriormente se introdujo en un medio de transporte. Estas muestras se conservaron a temperatura ambiente durante un tiempo ≤24h, y las que superaban este tiempo se conservaron en nevera entre 2 y 8°C1,8.
Selección de medios de cultivo y condiciones de incubaciónPara el cultivo se emplearon agar sangre de cordero 5% y un medio selectivo y diferencial para SARM (MRSA-ID bioMérieux). Las placas inoculadas se incubaron en estufa a 35-37°C. Se realizó una primera lectura a las 24h del cultivo, y cuando no se detectó crecimiento se prolongó la incubación hasta las 48h9,10.
Identificación bacteriana y sensibilidadPara la identificación presuntiva se realizó tinción de Gram, catalasa y aglutinación para Staphylococcus aureus (BioRad®). En caso de resultar un coco grampositivo, catalasa y aglutinación positiva con crecimiento en agar MRSA, el microorganismo se identificó como SARM y se realizaron pruebas complementarias para la identificación definitiva y la sensibilidad a antimicrobianos11. Para ello, se realizó un panel comercial de pruebas bioquímicas y de sensibilidad mediante microdilución (paneles 3w Wider®, Soria-Melguizo). Para la confirmación de la resistencia a meticilina se usó un disco de cefoxitina (30μg) mediante el método de disco-difusión en agar Mueller Hinton Medium (método recomendado por el CLSI). Para la confirmación de la resistencia a los descolonizadores tópicos (mupirocina y ácido fusídico) se usaron tiras de Etest® (bioMérieux) de ambos antimicrobianos utilizando los criterios publicados (resistencia de alto nivel a mupirocina: CLSI≥512mg/l; resistencia a ácido fusídico: EUCAST>1mg/l)12,13.
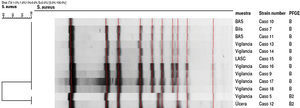
Relación clonalEl análisis genotípico de las cepas se realizó mediante electroforesis en campo pulsado (PFGE). Para ello se utilizó la enzima de restricción SmaI y el protocolo de pulsos Harmony para Staphylococcus aureus (pulsos 5-15s; 10h y 15-60s; 13h)14. Para el análisis de patrones de bandas se utilizaron los criterios de Tenover, complementados con el programa PFQuest (BioRad®), utilizando la comparación de bandas de Dice y el dendrograma tipo UPGMA15.
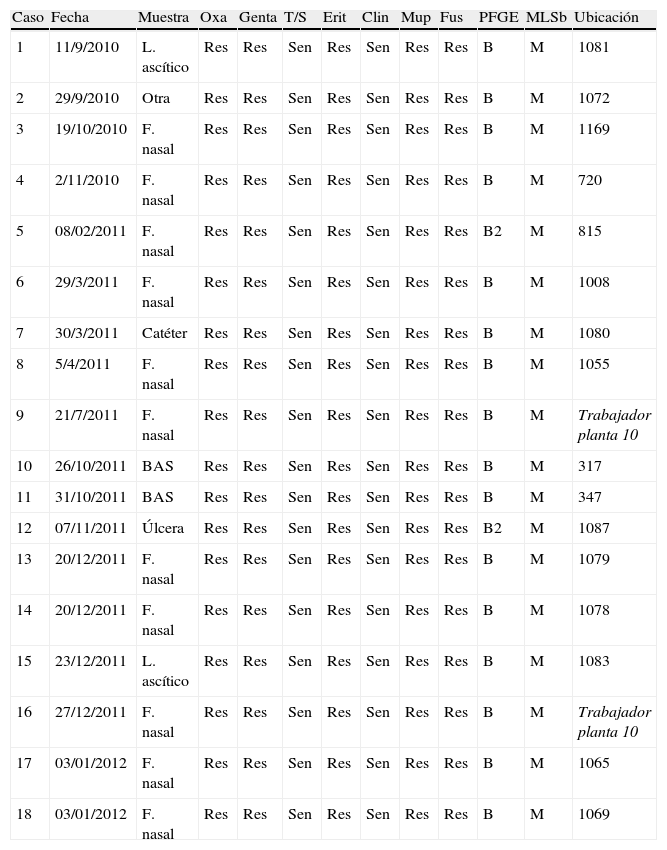
ResultadosLa tabla 1 refleja la relación de casos, ordenados por fecha de aparición. En total se afectaron 16 pacientes distintos y una trabajadora (2 muestras) adscrita a la planta 10 del centro.
Características de pacientes afectados y perfil de sensibilidad a antibióticos
| Caso | Fecha | Muestra | Oxa | Genta | T/S | Erit | Clin | Mup | Fus | PFGE | MLSb | Ubicación |
| 1 | 11/9/2010 | L. ascítico | Res | Res | Sen | Res | Sen | Res | Res | B | M | 1081 |
| 2 | 29/9/2010 | Otra | Res | Res | Sen | Res | Sen | Res | Res | B | M | 1072 |
| 3 | 19/10/2010 | F. nasal | Res | Res | Sen | Res | Sen | Res | Res | B | M | 1169 |
| 4 | 2/11/2010 | F. nasal | Res | Res | Sen | Res | Sen | Res | Res | B | M | 720 |
| 5 | 08/02/2011 | F. nasal | Res | Res | Sen | Res | Sen | Res | Res | B2 | M | 815 |
| 6 | 29/3/2011 | F. nasal | Res | Res | Sen | Res | Sen | Res | Res | B | M | 1008 |
| 7 | 30/3/2011 | Catéter | Res | Res | Sen | Res | Sen | Res | Res | B | M | 1080 |
| 8 | 5/4/2011 | F. nasal | Res | Res | Sen | Res | Sen | Res | Res | B | M | 1055 |
| 9 | 21/7/2011 | F. nasal | Res | Res | Sen | Res | Sen | Res | Res | B | M | Trabajador planta 10 |
| 10 | 26/10/2011 | BAS | Res | Res | Sen | Res | Sen | Res | Res | B | M | 317 |
| 11 | 31/10/2011 | BAS | Res | Res | Sen | Res | Sen | Res | Res | B | M | 347 |
| 12 | 07/11/2011 | Úlcera | Res | Res | Sen | Res | Sen | Res | Res | B2 | M | 1087 |
| 13 | 20/12/2011 | F. nasal | Res | Res | Sen | Res | Sen | Res | Res | B | M | 1079 |
| 14 | 20/12/2011 | F. nasal | Res | Res | Sen | Res | Sen | Res | Res | B | M | 1078 |
| 15 | 23/12/2011 | L. ascítico | Res | Res | Sen | Res | Sen | Res | Res | B | M | 1083 |
| 16 | 27/12/2011 | F. nasal | Res | Res | Sen | Res | Sen | Res | Res | B | M | Trabajador planta 10 |
| 17 | 03/01/2012 | F. nasal | Res | Res | Sen | Res | Sen | Res | Res | B | M | 1065 |
| 18 | 03/01/2012 | F. nasal | Res | Res | Sen | Res | Sen | Res | Res | B | M | 1069 |
En septiembre de 2010 se aislaron 2 cepas de SARM MupFusr de muestras clínicas de 2 pacientes ingresados en una planta de hospitalización del HUIGC (planta 10, cirugía general y digestiva) y que resultaron ser idénticas al compararlas mediante electroforesis en campo pulsado (fig. 1).
Entre octubre y noviembre de 2010, como resultado de las actividades del programa de vigilancia activa de SARM, se encontraron muestras de frotis nasales SARM MupFusr idénticos (fenotipo y genotipo) en 2 pacientes de plantas distintas (plantas 7 y 11).
En febrero de 2011, en la planta 8 apareció otra cepa de SARM con el mismo fenotipo y genotípicamente relacionado con las anteriores.
Entre marzo y abril de 2011 se aisló la misma cepa en 3 pacientes en la planta 10: una en punta de catéter y 2 en frotis nasales, idénticas a las anteriores mediante PFGE. Los 3 pacientes presentaban un ingreso prolongado en esta planta, y la vigilancia activa de SARM de todos ellos, al ingreso y en los meses sucesivos, había sido negativa hasta el momento de la detección de la infección o el estado de portador nasal. Ante esta situación epidémica se consideró necesario realizar un estudio de portadores nasales a todos los trabajadores de la planta 10 (61 trabajadores, incluyendo FEA, MIR, DUE, auxiliares de enfermería y celadores). Se encontraron 4 personas portadoras de S. aureus sensible a la meticilina y una portadora de un SARM MupFusr, que fue retirada de su puesto de trabajo temporalmente y tratada durante 10 días con la siguiente pauta: trimetoprim-sulfametoxazol por vía oral, 160-800mg cada 12h, higiene corporal diaria con gel de clorhexidina jabonosa al 4% y enjuagues bucales 3 veces al día con clorhexidina al 0,2%. Además de ello se le recomendó lavado diario de ropa y lencería a una temperatura superior a los 60°C.
Entre octubre de 2011 y enero de 2012 se detectaron 2 pacientes ingresados en el servicio de medicina intensiva y otros 6 pacientes más en la planta 10 con la cepa SARM MupFusr, detectándose nuevamente el estado de portador en la trabajadora con dicha cepa. Se tomaron frotis nasales y faríngeos semanales al inicio de la jornada laboral. Tras comprobar el estado de portadora crónica, en marzo de 2012 fue adscrita a otro puesto de trabajo sin contacto directo con pacientes.
No se han detectado más casos por esa cepa desde marzo de 2012 hasta la fecha (septiembre de 2012).
DiscusiónSe describe una situación poco frecuente en nuestro medio, y en la literatura en general. Diversos estudios han puesto de manifiesto la mayor probabilidad de resistencia al ácido fusídico cuando se detecta resistencia de alto nivel a la mupirocina4-6. Sobre la base de los resultados descritos, se pueden observar los siguientes perfiles epidemiológicos:
- •
Aislamientos no relacionados en tiempo y espacio, genéticamente idénticos o relacionados pero sin un nexo epidemiológico demostrado (casos 3, 4, 5, 10 y 11 de la tabla 1).
- •
Aislamientos relacionados en tiempo y espacio, genéticamente idénticos (casos 1, 2, 6, 7, 8, 12, 13, 14, 15, 17 y 18 de la tabla 1) y con un nexo epidemiológico demostrado en el estado de portador de un mismo trabajador sanitario (casos 9 y 16 de la tabla 1).
Ambos perfiles se corresponden con la misma cepa de SARM MupFusr, con un pulsotipo de PFGE idéntico con SmaI (protocolo de pulsos Harmony).
El portador sanitario cumple con criterios para ser portador persistente de la cepa y con factores de riesgo que pueden favorecer dicho estado, al presentar una sinusitis crónica1.
En nuestro centro no existía esta cepa antes de la detección del primer caso en septiembre de 2010, y en todos los brotes previamente detectados las cepas implicadas fueron sensibles a los descolonizadores habituales. El patrón de PFGE de la cepa epidémica es distinto del resto de patrones de otras cepas de SARM aisladas en nuestro centro durante la situación epidémica y previamente a ella.
La detección de esta cepa plantea un problema serio, pues deja sin herramientas adecuadas para la descolonización de portadores (colonizados o infectados) y por tanto para disminuir finalmente la carga de morbilidad3-6.
Aunque no lo podemos garantizar, se puede pensar que la trabajadora portadora de esta cepa de SARM MupFusr poco frecuente ha podido ser responsable de la diseminación en nuestro centro. Esta afirmación se fundamenta en varios hechos: primero, la identidad clonal de todos los aislados; segundo, el hecho de que los primeros pacientes en que se detectó esta cepa estuvieran ingresados en la planta en la que la trabajadora portadora prestaba sus servicios y no se hubiera identificado previamente en otros pacientes de nuestro centro; y finalmente, el que no hayan aparecido más casos desde que la trabajadora fue apartada de la asistencia directa a pacientes en marzo de 2012 (hasta septiembre de 2012). Es conocido que la colonización por SARM puede ser muy duradera, por lo que el simple hecho de que los pacientes no hayan coincidido en las plantas no descarta la transmisión desde un foco común anterior (atención en servicios extrahospitalarios previos, o en ingresos previos) o actual (sanitarios de retén que van a distintas plantas, radiología, etc.). Esto, no obstante, es complicado de discernir, habida cuenta de la complejidad de la práctica asistencial, a pesar del estudio epidemiológico rutinario de cada caso de SARM que se realiza en nuestro centro por personal entrenado.
En el manejo de esta situación se nos planteó el tomar la decisión de adscribir a un trabajador a otro puesto de trabajo, cuando los datos de la literatura no avalan de forma fehaciente esta recomendación. En el caso concreto de la transmisión del SARM, la revisión de Albrich et al.1 recoge que, debido a las particularidades epidemiológicas del SARM, presente tanto en el hospital como en la comunidad, el trabajador puede ser un vehículo pero también una víctima. Finalmente, tras comprobar el estado de portadora crónica de la trabajadora afectada, tras un seguimiento microbiológico semanal, se decidió que no debía seguir en contacto directo con pacientes ingresados.
Respecto a la resistencia a la mupirocina, en nuestro centro desde inicios de 2009 hasta agosto de 2011 tuvimos una resistencia de alto nivel a la mupirocina (testado mediante Etest® y confirmado mediante PCR del gen IleS2) del 19% de los SARM (datos no publicados). Este dato, muy similar a la media nacional en estudios multicéntricos del 20% de los SARM16, nos hace pensar que nuestra situación epidemiológica respecto al SARM no es desfavorable.
La principal limitación de este estudio es que adolece de un diseño epidemiológico previamente planificado, que por otro lado resultaría imposible en el contexto descrito. Tampoco se ha hecho una investigación de todas las posibles causas suficientes y sus factores de riesgo componentes. Pero el hecho de eliminar al menos uno de ellos (exposición a la trabajadora afectada) parece haber sido suficiente para interrumpir la cadena de transmisión, lo que tiene respaldo científico17. Finalmente, la continuidad de una estrecha vigilancia del comportamiento del SARM en nuestro centro definirá si se ha realizado un control adecuado de la situación.
Este trabajo puede ser de interés para centros que experimenten problemas parecidos al planteado. Pensamos que la rápida identificación de portadores de SARM (con o sin resistencias a los descolonizadores habituales) entre los trabajadores de un centro sanitario se revela como una estrategia necesaria para la implementación de actuaciones adicionales en el caso de situaciones no usuales, que se definen por una vigilancia epidemiológica adecuadamente instaurada.
Conflicto de interesesLos autores declaran que no existe ningún conflicto de intereses.