A nivel mundial, los problemas de salud mental constituyen una carga de enfermedad importante. En Chile, la depresión ocupa el primer lugar en la carga de enfermedad debida a trastornos mentales; según datos de la Encuesta Nacional de Salud 2016-2017 realizada por el Ministerio de Salud, un 15,8% de la población mayores de 18 años presentan depresión según la definición que emplea el CIDI (Composite International Diagnostic Interview) desarrollado por la OMS para estudios de salud mental. Uno de los principales desafíos que enfrenta la psiquiatría actualmente, es poder mejorar el enfoque de prueba y error para el tratamiento farmacológico de la enfermedad mental, puesto que uno de los problemas más importantes es la falta de respuesta al tratamiento (20-30% de los pacientes), sumado a la falta de adherencia al tratamiento y la presencia de reacciones adversas a las drogas.
Dados los avances en la secuenciación del genoma humano, se ha podido establecer que uno de los factores que influyen en el metabolismo de las drogas usadas en pacientes psiquiátricos es la presencia de polimorfismos que afectan distintos genes que modulan la farmacocinética o la farmacodinamia de los fármacos. De ahí que en los últimos años los centros de salud más importantes del mundo realizan estudios de farmacogenética a sus pacientes con el fin de entregarles la mejor terapia de acuerdo con sus características individuales
En este trabajo se presenta una actualización de este tema y, conjuntamente, se comenta la experiencia en Clínica Las Condes de la caracterización realizada en un estudio previo en pacientes que consultan principalmente por trastornos psiquiátricos como cuadros ansiosos y depresivos. Dicho estudio incluyó las variantes genómicas de un grupo de 6 genes pertenecientes a la familia del Citocromo P450 (CYP1A2, CYP2C9, CYP2C19, CYP2D6, CYP3A4 y CYP3A5), responsables en gran medida del metabolismo de la mayoría de las drogas usadas en psiquiatría. Con esta información se ha podido implementar una terapia farmacológica dirigida a cada paciente con el fin de lograr una mejor respuesta terapéutica
Globally, mental health problems constitute a significant disease burden. In Chile, depression ranks first in the burden of disease due to mental disorders, according to data from the 2016-2017 National Health
Survey conducted by the Ministry of Health, 15,8% of the population over 18 years old present depression according to the definition used by CIDI (Composite International Diagnostic Interview) developed by the WHO for mental health studies. One of the main challenges facing psychiatry today is to improve the trial-and-error approach for the pharmacological treatment of mental illness since one of the most important problems is the lack of response to treatment (20-30% of patients), in addition to the lack of treatment adherence and the presence of adverse drug reactions.
Given the advances in the sequencing of the human genome, it has been established that one of the factors that influence the metabolism of drugs used in psychiatric patients is the presence of polymorphisms that affect different genes that modulate the pharmacokinetics or pharmacodynamics of drugs. Hence, in recent years the most important health centers in the world carry out pharmacogenetic studies on their patients to deliver the best therapy according to their individual characteristics.
In this review, we present an update on this topic and also comment Clínica Las Condes’ experience in the implementation of a pharmacogenetic study for patients consulting for psychiatric disorders, mainly anxiety and depression, which analyzed the genomic variants of a group of 6 genes belonging to the cytochrome P450 family (CYP1A2, CYP2C9, CYP2C19, CYP2D6, CYP3A4, and CYP3A5), largely responsible for the metabolism of most drugs used in psychiatry. With this information, it has been possible to implement a pharmacological therapy directed at each patient to achieve a better therapeutic response.
A nivel mundial, los problemas de salud mental constituyen una carga de enfermedad importante. Dentro de ellas, la depresión mayor (DM) es una enfermedad altamente prevalente que se ha convertido en una complicación de salud pública muy relevan- te. Recientemente, se ha estimado que más de 300 millones de personas en el mundo sufren de depresión mayor, lo que equi- vale al 4,4% de la población mundial1 y un número similar sufre de ansiedad lo que conlleva altos gastos en salud de las perso- nas2–4. En Chile, según el Instituto Milenio para la Investigación en Depresión y Personalidad (MIDAP), las cifras de esta patología se sitúan dentro de los rangos más altos internacionalmente. Según los datos entregados por la Encuesta Nacional de Salud 2016-2017 realizada por el Ministerio de Salud, un 15,8% de la población general mayor de 18 años, reporta haber experi- mentado síntomas depresivos según la definición que emplea el CIDI (Composite International Diagnostic Interview) desarrollado por la OMS para estudios de salud mental, siendo un 6,2% de los chilenos diagnosticados con esta patología, evidenciándose una diferencia significativa de la prevalencia nacional entre mujeres y hombres (10,1% y 2,1%, respectivamente)5.
A pesar del aumento del número de fármacos psicotrópicos dis- ponibles, el campo de la psiquiatría se enfrenta al desafío de mejorar el actual modelo de “prueba y error” en la prescripción del tratamiento. Las tasas de respuesta a antidepresivos indi- viduales son bajas, especialmente en pacientes con depresión leve6,7. La depresión resistente a tratamiento, generalmente de- finida como la falta de respuesta a dos o más ensayos adecuados de monoterapia con distintas clases de antidepresivos, cada uno administrado por separado, en una dosis adecuada, durante un periodo de tiempo correcto8 representa al menos el 20 a 30% de los casos de DM. Además, la efectividad del tratamiento se ve limitada por la baja adherencia al mismo9. Altas tasas de falla al tratamiento y efectos adversos inducidos por fármacos traen consigo una importante carga económica y social10–13. En la de- presión refractaria o resistente, la respuesta a los antidepresivos resulta escasa, aunque la estrategia farmacológica sea la ade- cuada. La mayoría de los pacientes presentan respuesta, pero no remisión, es decir, continúan manifestando síntomas pese a experimentar cierta mejoría. La falta de respuesta a tratamien- to se acompaña de altas tasas de morbilidad médica, baja pro- ductividad, disminución de la esperanza de vida, altas tasas de suicidio y ha sido considerado como la primera causa de disca- pacidad laboral en las Américas14. Chile, dentro de la región de las Américas, tiene un 36% de años perdidos por discapacidad (APD) y un 21% de años de vida ajustados en función de la disca- pacidad (AVAD)15. Por ello, la atención al paciente deprimido im- plica, entre otras cosas, el diagnóstico oportuno y el tratamiento adecuado del padecimiento. Aunque un número creciente de agentes antidepresivos está disponible para tratar la depresión, aproximadamente la mitad de los pacientes no responden y hasta dos tercios no logran la remisión después del tratamiento de primera línea14.
Los avances en los estudios farmacogenómicos, han sido de gran relevancia en varias disciplinas médicas y particularmente impor- tante en el área de psiquiatría. De acuerdo con recomendaciones internacionales, los pacientes con diversas patologías complejas como desorden afectivo bipolar, depresión mayor, depresión psi- cótica, desorden de personalidad limítrofe, entre otras, deberían realizarse estos estudios antes de iniciar su tratamiento debido al uso frecuente de terapias combinadas en ellas16. En la medida que la demanda por estos estudios aumente, su costo será menor y más accesible a los pacientes, lo cual permitirá diseñar trata- mientos farmacológicos personalizados en base a las características genéticas de cada paciente en particular; asunto de gran importancia si se considera que las dosis terapéuticas estableci- das para los diversos fármacos, se hacen en base a individuos con metabolismo considerado como normal.
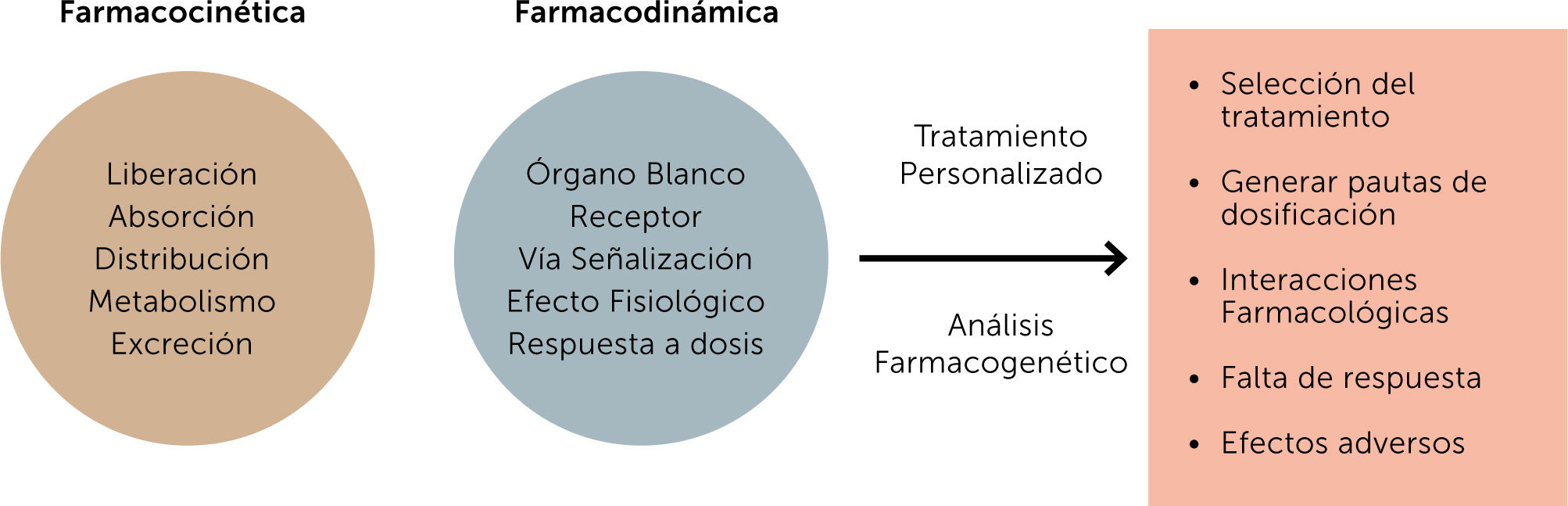
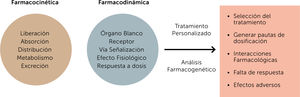
Existen diferentes factores que afectan la respuesta terapéuti- ca de los individuos: estos pueden ser fisiológicos como edad, sexo, peso, grasa corporal; patofisiológicos como función re- nal, hepática, cardiovascular; medioambientales como efecto del tabaco, alcohol, nutrición, contaminantes; y genéticos, que afectan la absorción, distribución, metabolismo y excreción e interacción de los fármacos. Una medicina personalizada con- sistirá en dirigir el tratamiento del paciente basado en su perfil genético-molecular17.
Las variaciones genéticas pueden expresarse a nivel farmacoci- nético así como farmacodinámico, lo que finalmente deriva en una respuesta distinta y particular a un tratamiento específico. Los factores farmacocinéticos influyen en el nivel plasmático y tisular que se alcanza con una determinada dosis y estos niveles están determinados por los procesos de absorción, distribución, metabolismo y excreción de los fármacos. Los factores farma- codinámicos determinan la interacción entre el fármaco y su receptor, dando lugar a respuestas que pueden ser menos o más eficientes que las esperadas, y ello debido a que afectan a receptores, enzimas o proteínas involucradas en la transducción de señales18,19.
Por otra parte, los términos farmacogenética y farmacogenó- mica se han utilizado muchas veces indistintamente. El término farmacogenética, se entiende como el estudio de la variabilidad genética de los individuos de genes involucrados en la respuesta a fármacos, el término farmacogenómica, se utiliza para desig- nar el proceso de usar la variación genética documentada para guiar la selección de medicamentos y sus dosis20. Es así como los estudios farmacogenéticos/farmacogenómicos dirigidos a identificar variantes génicas y establecer biomarcadores gené- ticos que puedan modificar la magnitud del efecto farmacoló- gico y los efectos secundarios e interacciones droga-droga, se han convertido en un desafío para poder definir el tratamiento según el perfil genético de cada paciente, idea base para el con- cepto de medicina personalizada17,18.
En un esfuerzo por estandarizar y facilitar la implementación de la farmacogenética en la práctica clínica, consorcios internacio- nales de expertos, tales como el Consorcio para la Implementa- ción de la Farmacogenética en la Clínica (CPIC), el Pharmacogene Variation Consortium (PharmVar), el American College of Medical Genetics and Genomics (ACMG), el American College of Molecular Pathology (ACMP), y la Human Genome Variation Society (HGVS), entre otros, están comprometidos con la emisión y actualización de guías clínicas revisadas y basadas en evidencia con asociaciones fármaco-gen, grado de evidencia, terminología estandariza- da y recomendaciones clínicas21–28. En la actualidad existen más de 35 guías divulgadas en el sitio web de la Pharmacogenomics Knowledge Base (PharmGKB)29 y se han desarrollado diferen- tes plataformas web que disponen de información almacena- da en sus bases de datos con el fin de orientar a los médicos en la práctica clínica o en su uso en investigación17. Dado que el número de fármacos aprobados por la FDA aumenta año a año, existe también un aumento en el número de herramientas basadas en farmacogenética para el apoyo a la decisión de los clínicos, y pruebas disponibles en el mercado30,31. La utilización de las pruebas farmacogenéticas específicas, ofrecidas por dis- tintas empresas del mercado, deben haber sido validadas según lo indica el modelo ACCE (validación Analítica, validación Clínica, utilidad Clínica, e implicaciones Éticas y legales)32 con estudios clínicos. Por ello se han realizado ensayos controlados aleato- rizados que demuestran que el estudio de la farmacogenética definida por un conjunto de variaciones genéticas y un algorit- mo que las traduzca en un conjunto de recomendaciones médi- cas resulta en una mejora de los valores de salud comparado con la práctica clínica habitual. Esos ensayos se han realizado con una población de pacientes representativa de la práctica real, es decir, ensayos pragmáticos33. Además, esto ha sido apoyado por la observación de diferencias significativas en la eficacia intrín- seca antidepresiva entre los ensayos realizados bajo condiciones amplias de selección y los realizados con poblaciones altamente seleccionadas, tipo fase III34,35.
La respuesta de cada paciente a la medicación psiquiátrica es muy diversa, tanto en el efecto terapéutico como en el riesgo de presentar efectos adversos: entre un 60-70% de los pacientes con depresión no experimentan remisión completa con fárma- cos antidepresivos y entre un 30-40% no obtienen respuesta adecuada36. Alrededor del 10% de los pacientes abandona los fármacos antidepresivos debido a efectos adversos. Los fárma- cos antipsicóticos y estabilizadores pueden también producir efectos adversos con frecuencia37–39. Se ha estimado que hasta un 42% de la variabilidad asociada a la respuesta a fármacos an- tidepresivos es debida a variantes genéticas comunes40. De aquí la importancia que reviste el establecimiento de biomarcadores farmacogenéticos, dada su implicancia en la eficacia de la droga y los efectos secundarios asociados a la droga. Estos efectos se- cundarios determinan cerca de un 7% de hospitalizaciones41, un 20% de todas las readmisiones42 y alrededor de un 4% de retiro de circulación de nuevos fármacos43. De este modo, los ingresos hospitalarios debido a efectos adversos por fármacos suponen un elevado costo para el paciente y el país38.
Dentro de la variabilidad genética de los individuos que afectan la farmacocinética de los medicamentos existen diversos genes que codifican para proteínas involucradas en distintas etapas:
1) la absorción y distribución, en la cual participan transportadores de membrana que facilitan la absorción desde el lumen intestinal y su distribución desde sangre a diversos tejidos ta- les como la familia de transportadores ABC que permiten que las moléculas atraviesen las membranas biológicas y cana- les iónicos; 2) el metabolismo o biotransformación, que en el caso de prácticamente todos los antidepresivos está mediado principalmente por un complejo enzimático, denominado cito- cromo P450 (CYP450), que corresponde a una superfamilia de monooxigenasas, responsables del metabolismo oxidativo de un gran número de compuestos endógenos y xenobióticos, in- cluye las enzimas CYP1A2, CYP2C9, CYP2C19, CYP2D6, CYP3A4 y CYP3A5 llamadas también enzimas de fase I (responsables de la biotransformación inicial de la droga) y luego por enzimas de fase II (encargadas de la conjugación del metabolito) como las N-acetiltransferasas (NAT2), sulfotransferasas y metiltransfe- rasas como las tiopurinas S-metiltransferasas (TPMT), las cate- col-O-metiltransferasas (COMT) y la familia de UDP glucorono- siltransferasas (UGT); y 3) la eliminación, en la cual la presencia nuevamente de proteínas transportadoras, así como canales iónicos pueden contribuir a la excreción facilitada de los fármacos desde ciertos tejidos y órganos, como transportadores ABCB2 o ABCC344.
Variaciones en los genes que codifican para estas enzimas y transportadores, inducen cambios individuales en su actividad, lo que se traduce en una alta variabilidad en la respuesta a los fármacos administrados.
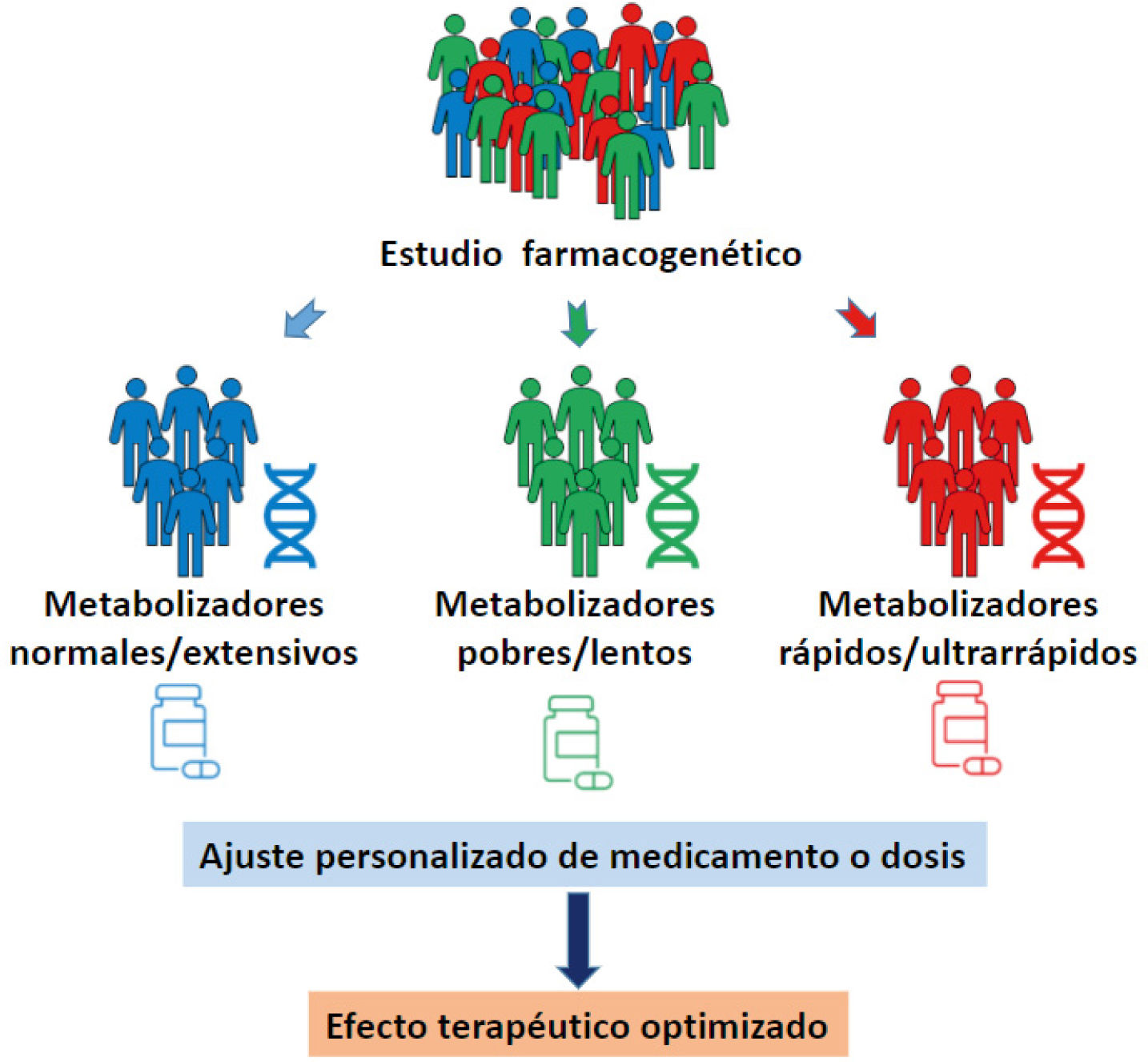
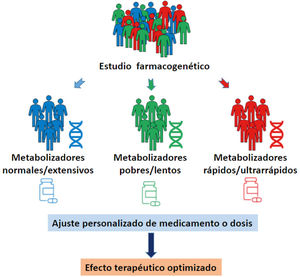
En consecuencia, la identificación de polimorfismos con rele- vancia terapéutica en las enzimas del CYP450, permite clasificar a los pacientes, según su respuesta farmacocinética, como me- tabolizadores pobres (lentos), intermedios (rápidos), normales (extensivos) y ultrarrápidos, permitiendo a su vez el diseño de una estrategia farmacológica personalizada45 (Fig. 1).
Por otra parte, los factores que afectan la interacción droga-re- ceptor en el sitio de acción del fármaco o también llamados factores farmacodinámicos, están principalmente regulados
por genes que codifican para receptores y proteínas funcionales involucradas en las acciones post receptor. Dentro de ellos los genes más estudiados en psiquiatría son aquellos que codifican para los transportadores de serotonina (SLC6A4)46, de dopamina (SLC6A3) y de noradrenalina (SLC6A2), los receptores de seroto- nina (HTR1A, HTR2A, HTR2C), y receptores de dopamina (DRD2, DRD3 y DRD4)18.
El resultado del estudio farmacogenético de los pacientes psi- quiátricos permite por lo tanto ajustar las dosis, cambiar me- dicamentos, evitar combinaciones de algunos fármacos o con- tinuar el uso de altas dosis o terapias combinadas con mayor confianza conociendo las mejores opciones para el paciente en particular47 (Fig. 2).
2Nomenclatura según pharmacogene variation consortium (pharmvar)Los avances en la farmacogenética han llevado además a utili- zar un lenguaje propio con abreviaturas y siglas que se han ido modificando en el tiempo48–50. Para enzimas de fase I pertene- cientes a la familia de CYP450, por ejemplo, se ha diseñado una nomenclatura estándar utilizando la raíz CYP seguida de un nú- mero arábigo que designa la familia de enzimas, una letra para designar la subfamilia y otro número que identifica la enzima individual, por ejemplo, CYP2C1951. Para cada enzima, el alelo silvestre o normal se designa como *1 y las variantes alélicas se enumeran según han sido identificadas de manera secuen- cial (*2, *3, etc.)52. Se define alelo como las múltiples variaciones de un gen o secuencia de DNA en la población, la combinación de ellos conformará un genotipo. Los alelos se encuentran en la misma posición dentro de cromosomas homólogos. Un indi- viduo hereda dos alelos para cada gen, uno del padre y el otrode la madre. Si los dos alelos son idénticos, el individuo es ho- mocigoto para este gen. Se dice que un gen es polimórfico si más de un alelo ocupa el locus de ese gen en más del 1% de la población a diferencia de una mutación que está presente en menos del 1%. A partir del año 2017, PharmVar53 cataloga la nomenclatura de asteriscos para los diversos polimorfismos vi- gente desde el 2000 con el número correspondiente al de iden- tificación “rs” (reference SNP [Single Nucleotide Polymorphism]) o según la posición del cambio nucleotídico con respecto al co- dón de inicio de la traducción. Es así como en el ejemplo de la enzima CYP2C19, el polimorfismo CYP2C19*2 corresponde al rs4244285 o c.681G>A. La nomenclatura de los subalelos de- finidos por letras ha dejado de utilizarse (CYP2C19*2A). La defi- nición de un haplotipo identificado con un asterisco (*) muchas veces involucra más de 1 cambio en el gen, por lo que su iden- tidad está representada por aquellos cambios relevantes que confieren un cambio funcional de la proteína. Cabe destacar que este consorcio es un depósito centralizado para la variación de los polimorfismos que se centra en la estructura del haplo- tipo y la variación alélica. La información de PharmVar53 facilita la investigación básica y clínica, así como la interpretación de los resultados de las pruebas farmacogenéticas para orientar la medicina personalizada54.
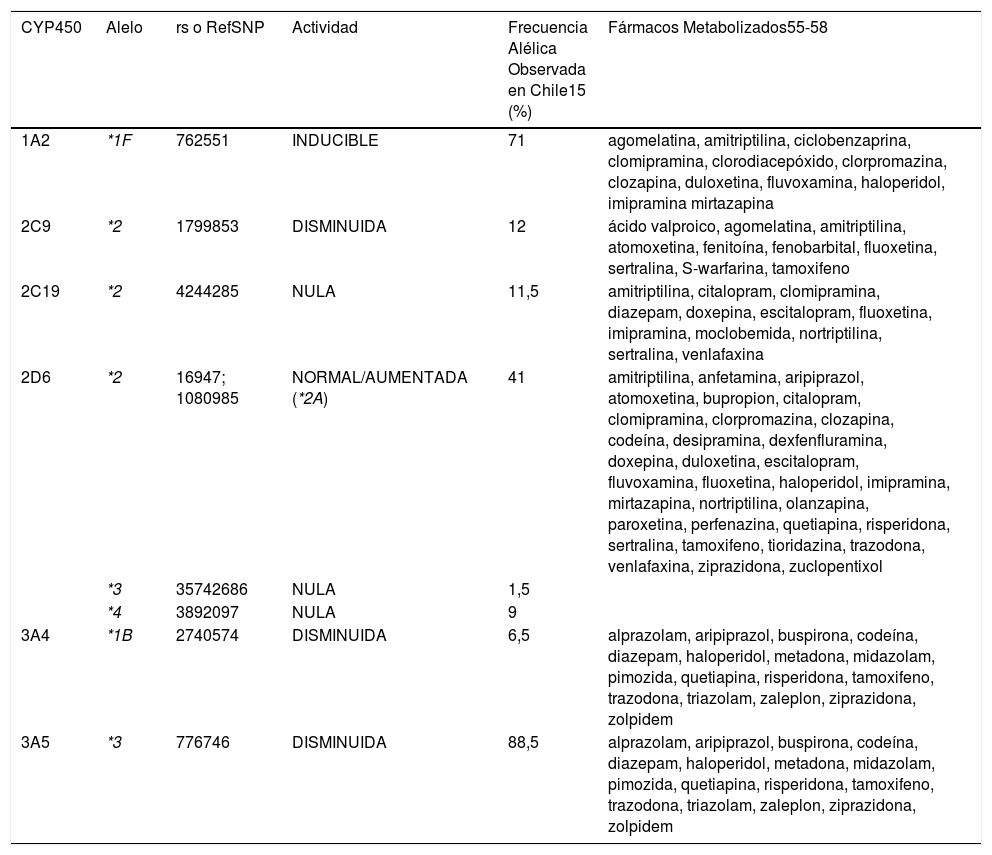
3DESCRIPCIÓN DE LOS GENES CYP1A2, CYP2C9, CYP2C19, CYP2D6, CYP3A4 Y CYP3A5En el contexto de la gran utilidad que tienen los estudios farma- cogenéticos para una medicina personalizada, en Chile, Clínica Las Condes (CLC) implementó a partir del año 2012 el estudio de los polimorfismos más relevantes de los genes menciona- dos anteriormente como CYP1A2, CYP2C9, CYP2C19, CYP2D6, CYP3A4 y CYP3A5 involucrados en el metabolismo de algunos
fármacos y cuya descripción se detalla a continuación. A la fecha se han evaluado 158 pacientes para los cuales se ha recomen- dado el uso de fármacos adecuados a su perfil genético en su tratamiento15 (Tabla 1).
Medicamentos de uso frecuente en psiquiatría y otras patologías. Enzimas asociadas a su metabolismo
| CYP450 | Alelo | rs o RefSNP | Actividad | Frecuencia Alélica Observada en Chile15 (%) | Fármacos Metabolizados55-58 |
|---|---|---|---|---|---|
| 1A2 | *1F | 762551 | INDUCIBLE | 71 | agomelatina, amitriptilina, ciclobenzaprina, clomipramina, clorodiacepóxido, clorpromazina, clozapina, duloxetina, fluvoxamina, haloperidol, imipramina mirtazapina |
| 2C9 | *2 | 1799853 | DISMINUIDA | 12 | ácido valproico, agomelatina, amitriptilina, atomoxetina, fenitoína, fenobarbital, fluoxetina, sertralina, S-warfarina, tamoxifeno |
| 2C19 | *2 | 4244285 | NULA | 11,5 | amitriptilina, citalopram, clomipramina, diazepam, doxepina, escitalopram, fluoxetina, imipramina, moclobemida, nortriptilina, sertralina, venlafaxina |
| 2D6 | *2 | 16947; 1080985 | NORMAL/AUMENTADA (*2A) | 41 | amitriptilina, anfetamina, aripiprazol, atomoxetina, bupropion, citalopram, clomipramina, clorpromazina, clozapina, codeína, desipramina, dexfenfluramina, doxepina, duloxetina, escitalopram, fluvoxamina, fluoxetina, haloperidol, imipramina, mirtazapina, nortriptilina, olanzapina, paroxetina, perfenazina, quetiapina, risperidona, sertralina, tamoxifeno, tioridazina, trazodona, venlafaxina, ziprazidona, zuclopentixol |
| *3 | 35742686 | NULA | 1,5 | ||
| *4 | 3892097 | NULA | 9 | ||
| 3A4 | *1B | 2740574 | DISMINUIDA | 6,5 | alprazolam, aripiprazol, buspirona, codeína, diazepam, haloperidol, metadona, midazolam, pimozida, quetiapina, risperidona, tamoxifeno, trazodona, triazolam, zaleplon, ziprazidona, zolpidem |
| 3A5 | *3 | 776746 | DISMINUIDA | 88,5 | alprazolam, aripiprazol, buspirona, codeína, diazepam, haloperidol, metadona, midazolam, pimozida, quetiapina, risperidona, tamoxifeno, trazodona, triazolam, zaleplon, ziprazidona, zolpidem |
La enzima CYP1A2 es responsable de la hidroxilación o desalqui- lación de muchas drogas prescritas comúnmente. La presencia de polimorfismos observados impacta principalmente en el metabo- lismo de antidepresivos y antipsicóticos. De ellos la presencia del polimorfismo -163C>A (*1F), produce que la enzima sea inducible.
Se considera que el alelo *1A es el alelo silvestre o normal en la po- blación general. Cabe señalar que el alelo *1F es el más presente en nuestra población, representando una frecuencia alélica del 71%15. Esta enzima puede ser inducible por algunas sustancias como café, tabaco, vegetales crucíferos y carnes grilladas entre otros, lo cual puede afectar el comportamiento fenotípico del paciente aun cuando sea heterocigoto para la variante y tenga por ello un aumento en la actividad enzimática. Por lo cual, en pacientes de nuestra población es habitual tener que hacer ajustes a las dosis de medicamentos utilizados en psiquiatría tales como clozapina, fluvoxamina y olanzapina20.
3.2CYP2C9CYP2C9 es una de las 4 isoformas de la familia de CYP2C jun- to a 8, 18 y 19, que se expresa en forma más abundante en el hígado59. La variación genética dentro de este locus impacta el metabolismo o bioactivación de muchas drogas incluyendo antiinflamatorios no esteroidales, agentes antidiabéticos, feni- toína, y bloqueadores del receptor de angiotensina. Su activi- dad es particularmente relevante en la eficacia y seguridad del uso de warfarina y siponimod. Las variantes alélicas c.430C>T y c.1075A>C inicialmente identificadas definen los haplotipos 2C9*2 y 2C9*3, respectivamente. Se define como haplotipo a la presencia simultánea de 1 o más variantes alélicas que se en- cuentran en el mismo cromosoma y tienden a ser heredados juntos. Muchas variantes adicionales se han descrito posterior- mente en la década de los años 1990. Las guías del Consor- cio para la Implementación de la Farmacogenética en la Clínica (CPIC) estableció recomendaciones para el uso de drogas de acuerdo con los diversos haplotipos identificados. Tanto los ha- plotipos *2 como *3 producen una disminución de la actividad enzimática, por lo cual los fármacos sustrato de la isoenzima 2C9, deberían administrarse en dosis menor a la habitual, para evitar efectos adversos del medicamento. Estos dos haplotipos son las variantes más frecuentes en población europea. En la po- blación de estudio chilena15 se ha reportado una frecuencia del 12% del alelo *2 en concordancia con lo descrito para población europea59, mientras que el alelo *3 no se evaluó en nuestra po- blación.
3.3CYP2C19Se describen 35 haplotipos diferentes para la isoforma CYP2C19 (*1 al *35)60. La variante alélica CYP2C19*2 implicada en el me- tabolismo lento o pobre de drogas fue descrito en el año 1994 con una frecuencia alélica de 15% en población europea, 11% en población americana, 17% en población africana y 31-36% en poblaciones asiáticas17. Posteriormente fueron identificadas numerosas variantes con actividad disminuida (*3, *4, *5, *6 y*8) con menor frecuencia de presentación en las distintas po- blaciones a diferencia de *17 que se asocia con un aumento de su actividad y cuya frecuencia alcanza un 22-24% en población europea y africana y un 12-14% en americanos y asiáticos17. La enzima CYP2C19 contribuye al metabolismo de muchas drogas de uso clínico como clopidogrel, voriconazol, inhibidores de bomba de protones, antidepresivos, carosoprodol y diazepam. Además, metaboliza sustratos endógenos como melatonina y progesterona60. El 11% del alelo *2 en la población chilena de estudio es similar a la reportada en población americana, cer- cana al 15% reportada para población europea y menor a las reportadas para población asiática (31-36%)15.
3.4CYP2D6CYP2D6 es una enzima purificada en 1984 y su gen mapeado en el cromosoma 22q13 el año 1987. Con su secuenciación se identificaron diferentes variantes alélicas que presentaban una actividad nula (*3, *4) o disminuida (*5, *6 entre otros) y otros una actividad normal (*2) o aumentada (duplicación*2). CYP2D6 es de gran relevancia en el metabolismo de un 20%-25% de las drogas usadas en la clínica tales como antidepresivos (por ejem- plo, paroxetina, fluoxetina), antipsicóticos (clozapina), agentes antineoplásicos (tamoxifeno), antagonistas adrenérgicos (me- topropol) y analgésicos (codeína, tramadol). Los polimorfismos descritos en esta enzima clasifican a los pacientes en metaboli- zadores lentos, intermedios, normales, rápidos o ultrarrápidos. Sujetos con múltiples copias del gen metabolizarán las drogas rápidamente, por lo cual la concentración de droga en el plasma no llegará a la dosis necesaria para ser efectiva. Los individuos con variantes que metabolizan los sustratos más lento tendrán riesgo de generar efectos adversos o toxicidad. La genotipifica- ción de CYP2D6ha sido particularmente importante en psiquia- tría en al predecir el 50% del metabolismo de antipsicóticos61–64. En algunos centros de genotipificación se considera que CYP2D6*2 tendría una actividad enzimática muy similar al tipo normal, de manera que no influiría en la eficiencia de esta en- zima61,62. En cambio, en otros consideran que *2 tendría una actividad aumentada, determinando que el paciente sea me- tabolizador ultrarrápido, cuando se encuentra la presencia del polimorfismo en forma homocigota. Estas discrepancias se pue- den deber a varias causas, entre ellas a la falta de evaluación de otras variantes consideradas dentro del haplotipo *2 como el polimorfismo -1584C>G ubicado en el extremo 5′-UTR clasi- ficado según la nomenclatura antigua como *2A, rs108098565. En Chile se ha observado que un 19% de los pacientes presenta el polimorfismo c.2850C>T del exón 6 en forma homocigota15 en concordancia con lo descrito por Llenera y cols.63.
La mayor parte de la población metaboliza las drogas en forma normal o extensiva, sin embargo, el 5-10% de los caucásicos y el 1-4% de la mayoría de los grupos étnicos son metabolizado- res lentos con una actividad disminuida del CYP2D6 y riesgo de tener efectos tóxicos si reciben las dosis habituales66–68. En po- blación chilena se ha observado una frecuencia alélica para las variantes que producen una actividad nula de la enzima, 2D6*3 y *4 de 1% y 9%, respectivamente15.
3.5CYP3A4CYP3A4 es una monooxigenasa que participa catalizando mu- chas reacciones involucradas en metabolismo y síntesis de co- lesterol, esteroides y lípidos. Es inducido por glucocorticoides y otros compuestos farmacológicos. Además, es responsable del metabolismo de cerca del 50% de las drogas que sufren me- tabolismo hepático y un primer metabolismo a nivel intestinal incluyendo drogas hipolipidemiantes, acetaminofeno, codeína, ciclosporina, diazepam, eritromicina y cloroquina, entre otros. La enzima también metaboliza algunos carcinógenos. Se han descrito algunos polimorfismos asociados a una disminución de
la actividad enzimática relacionado especialmente con el me- tabolismo de tracolimus. Dentro de los polimorfismos descritos están CYP3A4*1B (rs2740574) y el *1G (rs2242480). Con res- pecto a la variante alélica *1B que corresponde al cambio en el promotor c.-392G>A, es importante señalar que en el pasado esta variante fue definida como A>G usando la secuencia de re- ferencia ya en desuso AF280107.01. Por ejemplo, en el trabajo de Moreno y cols.15, se utilizó la nomenclatura antigua inicial- mente disponible para la caracterización de la frecuencia alélica del polimorfismo descrito como c.-392A>G asignándole *1B a la presencia de G. En la actualidad, de acuerdo con PharmVar este polimorfismo es definido como G>A según la secuencia ac- tual de referencia (NG_008421.1) con la cual se actualizaron todas las definiciones de alelos identificados. Ahora, CYP3A4*1B corresponde a CYP3A4*1.001 (que tiene una G en c.-392) y el alelo CYP3A4*1A corresponde a CYP3A4*1.002 (que tiene una A en c.-392).
La variante alélica*1B ha sido identificada más frecuentemente en africanos que en otras poblaciones (77%) y en Chile se la ha observado con una frecuencia alélica del alelo G de un 7%15. Su presencia tiene al parecer un impacto funcional69,70, en contras- te con algunos reportes que han descrito que estaría influencia- do por estar en desequilibrio genético con CYP3A5*1.
3.6CYP3A5CYP3A5 se expresa pobremente en los adultos y constituye 5 a 20% de la familia 3A, dado que comparte la mayoría de los sustratos con la enzima 3A4. La condición de metabolizador in- termedio (heterocigoto *1A/*3A) para la enzima 3A5 puede ser relevante, determinando mayores concentraciones plasmáticas de ciertos fármacos, con los efectos adversos correspondientes, por lo que se recomienda el ajuste de dosis. La variante alélica 6986A>G (rs776746) presenta una frecuencia de 89% en la po- blación chilena por lo cual podría pensarse que este polimorfis- mo constituye la variante silvestre en ella15.
Aquellos medicamentos que son sustrato de CYP3A5*3A, debe- rían usarse en dosis menor a la habitual en pacientes con am- bos alelos homocigotos para la presencia del alelo 3A, ya que presenta un polimorfismo de baja actividad, que determina su funcionamiento como metabolizador pobre. Es el caso en el uso de tacrolimus71 y en el riesgo de toxicidad renal72.
4Otras proteínas importantes en el metabolismo de fármacos: UGT1A4, UGT2B15, CYP2B6, ABCB1 Y SCL6A4Los polimorfismos presentes en otras enzimas que participan en el metabolismo y transporte de fármacos, influyen también en la farmacocinética y en la farmacodinamia de drogas usadas actualmente en psiquiatría y otras patologías. Debido a esto actualmente sería importante incorporar otros genes como UG- T1A4, UGT2B15, CYP2B6, ABCB1 y SCL6A4 en los estudios, para así seleccionar la mejor terapia requerida por los pacientes psi- quiátricos.
Otras áreas de la medicina cuyos pacientes se ven altamente beneficiados con los análisis farmacogenéticos son en el cán- cer (de mama, colorrectal, pulmonar y leucemia), en la analgesia (artritis entre otras patologías), en el área cardiovascular (en- fermedades cardiovasculares, trombosis, hipercolesterolemia), y cuando hay infecciones bacterianas o por VIH. Solo por citar un ejemplo, las pacientes con cáncer de mama cuyos tumores expresan el receptor de estrógeno (RE), reciben como terapia tamoxifeno que es un potente inhibidor del RE. Esta droga es bioactivada por CYP2D6 generando un metabolito llamado en- doxifeno que tiene una afinidad por el RE 100 veces mayor que el tamoxifeno. Cuando la paciente es portadora de alelos defec- tuosos de CYP2D6, como *4 y *10, tienen un riesgo aumentado de reincidencia de cáncer de mama y menores tasas de sobre- vida libres de eventos cuando se comparan con las pacientes que tienen CYP2D6 metabolizando normalmente. Por lo tanto, el uso de paneles ampliados de varias enzimas metabolizadoras y de transporte son fundamentales para seleccionar la mejor te- rapia para cada paciente.
De acuerdo con el reporte de los años 2016-2017 de la OMS “Depresión y otros Desórdenes Mentales Comunes”, en Chile 844.253 personas mayores de 15 años padecen depresión, es decir, el 5% de la población, y más de un millón sufre de ansie- dad (6,5%). Lo cual nos posiciona en el cuarto lugar dentro de los países con mayor prevalencia de esta enfermedad en la región de las Américas.
En Chile, el tratamiento de la depresión está garantizado en el Plan Auge para las personas mayores de 15 años. Según cifras de la Superintendencia de Salud, desde 2005 a septiembre de 2016, ingresaron más de 1 millón 60 mil casos a través de FO- NASA (Fondo Nacional de Salud en Chile) y cerca de 245 mil a través de ISAPRES (Instituciones de Salud Previsional Privadas en Chile), siendo uno de los diez problemas de salud con más ingresos.
Aunque existen tratamientos eficaces disponibles para la de- presión, más de la mitad de los afectados en todo el mundo no los recibe. La falta de recursos además de la estigmatización de los trastornos mentales y la evaluación clínica inexacta son algunos de los factores que impiden una atención eficaz. Otra barrera es la evaluación errónea. En países de todo tipo de in- gresos, las personas con depresión a menudo no son correcta- mente diagnosticadas, mientras que otras que no la padecen son a menudo diagnosticadas erróneamente y tratadas con antidepresivos.
Actualmente existen empresas, en Chile y el extranjero, que ofrecen la evaluación de un panel de genes para el manejo del tratamiento de pacientes con patologías psiquiátricas, utilizando un estudio patentado para generar recomendaciones basadas en guías farmacogenéticas publicadas disponibles (como la guía CPIC para los ajustes de dosis basados en genotipos combinados), la información farmacogenética incluida en etiquetas de fárma- cos aprobados por la FDA u otros ensayos clínicos seleccionados; y una alerta de seguridad para clasificar las variaciones genéticas que contribuyen a un mayor riesgo de efectos adversos por enci- ma de otras variaciones. Su efectividad se ha demostrado en es- tudios clínicos realizados en otras poblaciones34, sin embargo, su efectividad en población chilena aún no ha sido probada. Por otra parte, debido a su alto costo y la falta de cobertura por el sistema de salud, lo hace de difícil acceso para los pacientes en general. Un estudio más acotado de menor costo ha permitido orientar el tratamiento de estos pacientes en una institución privada en Chile15. Sin embargo, a pesar de lograr modificar empíricamente la terapia para lograr una respuesta adecuada o mejorada, fal- ta una evaluación sistemática de su eficacia que demuestre sus beneficios. Actualmente esta institución es la única que ofrece este estudio y el resultado del examen es entregado de forma confidencial y personalmente por el médico psiquiatra con las recomendaciones clínicas de acuerdo con los medicamentos in- dicados al paciente por su médico tratante. De gran relevancia sería el registro por parte de los psiquiatras de la mejoría y eficacia del tratamiento en los pacientes después de guiar el tratamiento según la genotipificación, con aplicación de encuestas tales como la Clinical Global Impression of Severity Scale (CGI-S)73 o la Escala de Depresión de Hamilton de 17-ítems (HDRS-17)74,75. Una estima- ción más efectiva sería también medir niveles de metabolitos del fármaco en orina y ver como ellos se modifican después de ajustar las dosis de acuerdo con el fenotipo del paciente.
En resumen, el ajuste de dosis con base en el fenotipo meta- bolizador de los distintos genes permite: a) dar dosis estándar de inicio a pacientes que van a tolerar bien la terapia (fenotipo metabolizador normal); b) reducir la dosis de aquellos pacientes que tolerarían mal la dosis de inicio estándar (fenotipo metabo- lizador intermedio); c) evitar el uso de estos fármacos o reducir drásticamente su dosis en aquellos pacientes que vayan a tolerar muy mal la terapia evitando la aparición de efectos secundarios por la droga (fenotipo metabolizador lento); d) cambio de fárma- co por uno de mayor tolerancia de acuerdo a su perfil genético y; e) poder rescatar drogas que habían sido sacadas de circulación previamente debido a efectos secundarios. En la actualidad la farmacogenómica ha permitido conocer cómo las variaciones de un gen están ligadas a la función de éste y ha permitido mejorar la respuesta al tratamiento de los pacientes basándose en estu- dios farmacogenéticos que permiten identificar las diferencias interindividuales en la respuesta a medicamentos siendo la base de la medicina personalizada. El conocimiento de la variabilidad genética de un individuo puede ser clínica y económicamente importante, y podría proporcionar las bases para una farmaco- terapia racional en los trastornos neuropsiquiátricos y en otras patologías.
Declaración de conflicto de interésLos autores declaran no tener conflicto de intereses.