Los cultivos de vigilancia epidemiológica y la tipificación molecular han sido importantes aportaciones de la Microbiología Clínica al control de la infección nosocomial. En este documento se ofrece información sobre recogida, transporte, conservación y procesamiento de muestras para cultivos de vigilancia, criterios para la interpretación de los resultados y la emisión de los mismos en relación con las bacterias de mayor interés en infección nosocomial. Se incluyen Staphylococcus aureus resistente a meticilina (SARM), Enterococcus spp. resistentes a glucopéptidos, enterobacterias productoras de betalactamasas de espectro extendido (BLEE), Acinetobacter baumannii multirresistente y Pseudomonas aeruginosa resistente a carbapenems. Esta información pretende aportar una aproximación general al problema, a partir de la cual el laboratorio desarrolle directrices propias, en función de las necesidades acordadas con el equipo multidisciplinar de control de infección nosocomial.
Implementation of surveillance culture programs and molecular typing are important contributions of Clinical Microbiology to the control of nosocomial infections. This document provides information on collection, transport, preservation, and processing of samples for surveillance culture, as well as the criteria for interpreting and reporting the results of relevant etiologic agents in nosocomial infection. This includes methicillin-resistant Staphylococcus aureus (MRSA), glycopeptide-resistant Enterococcus spp., enterobacteria producing extended-spectrum β-lactamases (ESBLs), multiresistant Acinetobacter baumannii and carbapenem-resistant Pseudomonas aeruginosa. Details on the available methods for rapid diagnosis are also presented. The information in this document attempts to provide a general approach to the problem and may be considered a starting point for laboratories that are developing their own guidelines, according to needs defined by the multidisciplinary nosocomial infection control team.
La infección nosocomial es actualmente uno de los principales problemas sanitarios, y son de particular importancia las infecciones causadas por bacterias multirresistentes. Aunque no hay una definición precisa de bacteria multirresistente, se ha sugerido que el término debiera aplicarse a aquellos microorganismos que son resistentes a dos o más grupos de antimicrobianos habitualmente empleados en el tratamiento de las infecciones por el microorganismo considerado y que esta resistencia tenga relevancia clínica1. Probablemente, también sea razonable aplicar el término a los microorganismos que presentan de forma natural resistencia a múltiples antimicrobianos de uso clínico habitual y que han sido capaces de adquirir resistencia a alguno de los restantes grupos de antimicrobianos con posible utilidad clínica.
La multirresistencia aparece como consecuencia de mecanismos bioquímicos codificados en el cromosoma o por diversos elementos móviles. Esta última posibilidad añade mayor gravedad al problema, pues la diseminación del correspondiente elemento móvil favorece la aparición de brotes nosocomiales.
Múltiples estudios han demostrado que es útil realizar cultivos de vigilancia epidemiológica para conocer la verdadera dimensión del problema de la multirresistencia en un centro o en una unidad, pues la información que puede inferirse de los resultados de los cultivos de muestras clínicas obtenidas con fines diagnósticos sólo representa una parte (con frecuencia la menor) del problema. Estos estudios deben considerarse una herramienta adicional en los programas de control de la transmisión nosocomial de estos microorganismos. Desgraciadamente, su puesta en marcha supone una carga económica importante, tanto en personal como en medios materiales, por lo que debieran aplicarse en el contexto de un programa global, encaminado en última instancia al control de la infección nosocomial.
En este documento se abordarán los agentes etiológicos de mayor interés en infección nosocomial, por su frecuencia y por las dificultades terapéuticas que suponen. Se incluyen, Staphylococcus aureus resistente a meticilina, Enterococcus spp. resistentes a glucopéptidos, enterobacterias productoras de betalactamasas de espectro extendido (BLEE), Acinetobacter baumannii multirresistente y Pseudomonas aeruginosa resistente a carbapenems. No abordaremos otros microorganismos que en determinados circunstancias también adquieren especial importancia, como enterobacterias productoras de betalactamasa cromosómica o plasmídica de clase C, Stenotrophomonas maltophilia, Staphylococcus coagulasa negativa resistentes a meticilina, Clostridium difficile, etc.
El laboratorio de microbiología es fundamental para detectar e identificar correctamente los microorganismos multirresistentes presentes en muestras clínicas y para contribuir al diseño e implementación de programas para su vigilancia y control. La aplicación de métodos de tipificación molecular que ayuden a establecer la relación clonal de los aislamientos y la caracterización de los mecanismos de resistencia implicados y de los elementos genéticos que los codifican son también de gran utilidad en este caso.
La información que se recoge en este documento pretende aportar una aproximación bastante general al problema, y puede considerarse un punto de partida para que cada laboratorio pueda desarrollar sus propias directrices, en función de las necesidades acordadas con el equipo multidisciplinar de control de infección nosocomial del centro.
Muestras para cultivos de vigilancia: consideraciones generalesLa toma de la muestra, su transporte y su conservación para cultivos de vigilancia epidemiológica se realizarán siguiendo las recomendaciones generales de la Sociedad Española de Enfermedades Infecciosas y Microbiología Clínica (SEIMC) en su documento Recogida, transporte y procesamiento general de las muestras en el laboratorio de Microbiología (www.seimc.org/protocolos/microbiologia/).
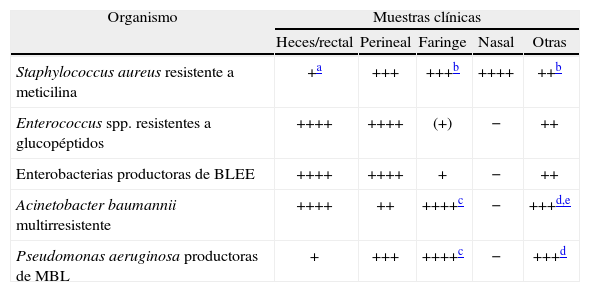
A título orientativo, en la tabla 1 se recogen las localizaciones de mayor interés para la búsqueda con fines epidemiológicos de los patógenos multirresistentes considerados.
Indicaciones orientativas sobre el interés cualitativo de diferentes muestras clínicas para la investigación de patógenos multirresistentes con fines epidemiológicos
| Organismo | Muestras clínicas | ||||
| Heces/rectal | Perineal | Faringe | Nasal | Otras | |
| Staphylococcus aureus resistente a meticilina | +a | +++ | +++b | ++++ | ++b |
| Enterococcus spp. resistentes a glucopéptidos | ++++ | ++++ | (+) | − | ++ |
| Enterobacterias productoras de BLEE | ++++ | ++++ | + | − | ++ |
| Acinetobacter baumannii multirresistente | ++++ | ++ | ++++c | − | +++d,e |
| Pseudomonas aeruginosa productoras de MBL | + | +++ | ++++c | − | +++d |
BLEE: betalactamasas de espectro extendido; MBL: metalo-betalactamasa.
Los S. aureus resistente a meticilina (SARM) son aquéllos para los que la concentración mínima inhibitoria (CMI) de oxacilina es igual o superior a 4 μg/ml o la de meticilina es igual o superior a 16 μg/ml. La resistencia es cromosómica y se debe a la transcripción del gen mecA, que genera una nueva proteína fijadora de penicilina (PBP) denominada PBP2a o 2′, con muy baja afinidad por los antibióticos betalactámicos actualmente disponibles para uso clínico. El gen mecA es transportado por un elemento genético móvil denominado casete cromosómico mec (SCCmec).
En las últimas dos décadas la expansión y prevalencia de este microorganismo ha aumentado de forma importante en todos los países, hasta convertirse en uno de los patógenos nosocomiales de mayor trascendencia. En España, la prevalencia aumentó del 1,5% en 1986 al 31,2% en 20022. La importancia de este microorganismo se debe a su resistencia a múltiples antimicrobianos (no sólo a betalactámicos), lo que hace difícil el tratamiento de las infecciones que produce3.
SARM ocasiona brotes epidémicos en los hospitales y en muchos casos se está comportando ya como un microorganismo endémico, contribuyendo al aumento de morbimortalidad y del coste hospitalario. En los últimos años se están documentando, de manera cada vez más frecuente, infecciones extrahospitalarias causadas por SARM en pacientes ingresados en centros de crónicos, asilos, etc. Estas infecciones, que muchos autores consideran "asociadas al sistema sanitario" se relacionan estrechamente, desde un punto de vista epidemiológico, con las infecciones clásicamente definidas como nosocomiales. Además, en la última década se están describiendo infecciones auténticamente comunitarias por SARM pero con características microbiológicas (sensibilidad antimicrobiana, virulencia, etc.) y clínicas claramente diferentes de las de adquisición nosocomial. Más recientemente, se está comprobando que estas cepas comunitarias están comenzando a describirse como causantes de infecciones nosocomiales2.
Consideraciones clínicasLos reservorios de S. aureus son principalmente los pacientes colonizados, pero el personal sanitario (que puede estar colonizado de forma permanente o temporal) también puede actuar como tal. La transmisión se produce fundamentalmente de forma cruzada a través de las manos del personal sanitario. No hay que olvidar, aunque tenga menor importancia, el papel que desempeña el ambiente hospitalario (superficies, objetos de uso común, etc.)4.
Las infecciones más frecuentes causadas por SARM son bacteriemia primaria, relacionada con catéter, o secundaria, infección quirúrgica, neumonía (especialmente la asociada a ventilación mecánica) y otras infecciones (infección de piel y tejidos blandos, osteomielitis, endocarditis, etc.)2.
Recogida, transporte, conservación y procesamiento de la muestraLas muestras adecuadas para realizar la vigilancia epidemiológica de SARM van encaminadas a detectar pacientes o personal sanitario colonizados.
En general, se recomienda:
- 1.
Exudado nasal: si se elige una única muestra para realizar cultivos de vigilancia, ésta es la más adecuada. En ocasiones, podría sustituirse por un frotis faríngeo, pero la extracción de esta última muestra es más incómoda para el paciente. Con una mayor sensibilidad está la triple muestra de frotis nasal, faríngeo y perirrectal o perineal.
- 2.
Exudado de piel de la zona perineal-perirrectal: en determinada población tiene alta sensibilidad, pero no se aconseja como muestra única.
- 3.
Muestras respiratorias: en pacientes con ventilación mecánica o traqueotomía.
- 4.
Exudados de úlceras o heridas: en pacientes con solución de continuidad en la piel.
- 5.
Urocultivo: en pacientes con sonda vesical.
Para los exudados nasal, faríngeo y de piel se empleará una torunda con medio de transporte. Estas muestras se pueden conservar durante un tiempo inferior o igual a 24 h a temperatura ambiente o en nevera entre 2 y 8 °C.
Las muestras para vigilancia epidemiológica para detectar SARM no precisan la realización de tinción de Gram, dado que los cocos grampositivos en racimos forman parte de la flora normal de la mayoría de las localizaciones de interés para cultivo de vigilancia epidemiológica. No es necesario ningún tipo de preparación ni pretratamiento previo a la inoculación de los medios de cultivo. La muestra se sembrará de forma cualitativa para obtener colonias aisladas. Se deben emplear medios selectivos y, preferiblemente diferenciales, a los que se identifique SARM fácil y rápidamente. De entre las múltiples posibilidades existentes, los medios más empleados son el de agar manitol-sal (medio de Chapman) y los de agar cromogénico5. Los medios cromogénicos tienen la ventaja de una identificación presuntiva más rápida de SARM y de que existen ya preparados comercialmente. Es recomendable usar medios selectivos suplementados con oxacilina (se ha usado desde 0,5 a 6 μg/ml) o de cefoxitina (desde 4 a 8 μg/ml) como agentes selectivos para las cepas resistentes a meticilina. Las placas inoculadas deben incubarse en estufa a 35-37 °C.
Criterios para la interpretación de resultadosSe realizará una primera lectura a las 24 h del cultivo; si en las placas no se detecta aislamiento de microorganismos, se prolongará la incubación hasta las 48 h, y se volverán a leer las placas en ese momento.
Si se ha empleado agar manitol-sal, se realizará la lectura a las 24 y 48 h. Cualquier organismo manitol positivo (color amarillo) será sospechoso de ser S. aureus. Se realizará un subcultivo de la colonia sospechosa en agar sangre y en agar MRSA (meticilina-resistente-screenire.g'agar, medio recomendado por el Clinical Laboratory Standards Institute (CLSI), consistente en agar Mueller-Hinton suplementado con 4% de ClNa y 6 μg/ml de oxacilina) que se leerá 24 h después. El crecimiento en agar MRSA nos indicará que el microorganismo es resistente a meticilina. Éste en agar sangre se someterá a identificación definitiva realizando tinción de Gram, catalasa y coagulasa (en tubo o aglutinación). En caso de resultar un coco grampositivo, catalasa y coagulasa positivo con crecimiento en agar MRSA, el microorganismo se identificará como SARM y se podrá realizar sensibilidad antimicrobiana y los estudios que se crean oportunos (tipificación molecular, etc.).
Si se ha empleado agar cromogénico selectivo, se realizará también la lectura a las 24 y 48 h. Cualquier microorganismo con una coloración compatible con lo especificado por el fabricante del medio, se identificará como SARM. Se realizará un subcultivo de esta cepa en agar sangre para realizar las pruebas de sensibilidad antimicrobiana y los estudios que se crean oportunos (biología molecular, etc.). Algunos autores recomiendan, en cualquier caso, una identificación definitiva, con un esquema similar al señalado previamente, al menos cuando se detecte el microorganismo en un paciente por primera vez.
Con independencia de estos métodos fenotípicos, la confirmación de la resistencia a meticilina en S. aureus se puede realizar detectando el gen mecA por reacción en cadena de la polimerasa (PCR) convencional o en tiempo real (método que en la actualidad se considera de referencia)6,7 o mediante la aglutinación con látex para detectar la presencia de PBP2a8.
Información sobre los resultadosCuando se ha empleado agar manitol-sal, en caso de aislarse un microorganismo manitol positivo, coco en racimo grampositivo, catalasa y coagulasa positivos sin crecimiento en agar MRSA, se informará: "Se aísla S. aureus sensible a meticilina". Si se aísla un microorganismo manitol positivo, coco en racimo, catalasa y coagulasa positivo con crecimiento en agar MRSA, se informará: "Se aísla S. aureus resistente a meticilina". Si a las 48 h no hay colonias compatibles con S. aureus se informará: "No se aísla S. aureus".
Cuando se ha empleado agar cromogénico selectivo, si se aísla un microorganismo con la coloración característica definida por el fabricante para S. aureus, se informará: "Se aísla S. aureus resistente a meticilina". Si a las 48 h no hay colonias con la coloración característica definida por el fabricante para S. aureus resistente a meticilina, se informará: "No se aísla S. aureus".
Técnicas rápidas de diagnósticoEn la última década se han empezado a describir métodos rápidos, con resultados disponibles en horas, empleando PCR a tiempo real para detectar SARM directamente en muestras clínicas6,7. Los estudios realizados hasta el momento dan a esta técnica una sensibilidad y especificidad altas (mayor del 90%). Uno de los problemas de esta aproximación es que al realizar la toma de exudado nasal o de piel, pueden coexistir cepas de S. aureus y de Staphylococcus coagulasa negativo resistente a meticilina. Si se emplea el gen mecA para realizar la detección, la técnica puede ser positiva tanto en ese caso como si existiera verdaderamente SARM. Por este motivo, algunos estudios se basan en la detección del casete cromosómico SCCmecA, con una sensibilidad del 98% y una especificidad de prácticamente del 100%. Probablemente, en cuanto a vigilancia epidemiológica, éste sea el futuro en la detección rápida de SARM en cultivos de vigilancia.
Enterococcus resistente a glucopéptidosEl género Enterococcus está integrado por más de 20 especies, de las que E. faecalis es la que se ha venido aislando con mayor frecuencia en muestras clínicas (80-90%), seguida de E. faecium (5-10%)9.
En 1988 se describieron las primeras cepas de Enterococcus resistentes a glucopéptidos (ERG) en Europa. En la actualidad se conocen varios fenotipos de resistencia a glucopéptidos (VanA, VanB, VanC, VanD, VanE y VanG) con diferente grado de resistencia a vancomicina y teicoplanina10. Los fenotipos más frecuentemente detectados son el VanA (alta resistencia a vancomicina y teicoplanina) y VanB (resistencia entre moderada y alta a vancomicina pero no a teicoplanina). Ambos mecanismos son adquiridos, inducibles y capaces de ser transferidos a otros cocos grampositivos, incluyendo S. aureus. El gen vanA se encuentra en el transposón Tn1546 o en elementos relacionados. Tn1546 suele localizarse en plásmidos, lo que facilita su diseminación. El gen vanB se localiza habitualmente en el transposón compuesto Tn1547 o en el transposón conjugativo Tn5382. Este último se suele insertar cerca del gen pbp5 (que confiere resistencia a penicilina), lo que ayuda a explicar la transferencia simultánea de resistencia a vancomicina y ampicilina en cepas de E. faecium10.
Algunas especies, como E. gallinarum y E. casseliflavus presentan resistencia intrínseca de bajo nivel a vancomicina, mediada por el gen vanC, pero carecen de la importancia epidemiológica de E. faecium y E. faecalis.
La mayor parte de los ERG corresponden a E. faecium con el genotipo vanA, pero también se han descrito epidemias debidas a esta especie con el genotipo vanB y a E. faecalis, en el que son igualmente más frecuentes esos dos genotipos. En nuestro país, donde por el momento los ERG son poco frecuentes, las cepas más habituales han sido E. faecium vanA. Se ha prestado particular atención en los últimos años a los E. faecium vanA incluidos en el denominado complejo clonal 17, que además de resistencia a glucopéptidos presentan resistencia a ampicilina y a fluoroquinolonas, varios determinantes de patogenicidad, y están bien adaptados al entorno hospitalario.
Consideraciones clínicasLos enterococos forman parte de la flora gastrointestinal normal del ser humano. E. faecalis se aísla en altas concentraciones en el colon de más del 90% de individuos sanos, mientras que el resto de especies se encuentran menos frecuentemente y en menor cantidad. También se pueden encontrar colonizando la cavidad oral, vagina, área perineal, tracto hepatobiliar y tracto respiratorio superior. Otros reservorios en pacientes hospitalizados incluyen la piel, las heridas abiertas y las úlceras de decúbito.
Estos microorganismos poseen la capacidad de sobrevivir largos períodos de tiempo en condiciones desfavorables, por lo que también pueden recuperarse del suelo, alimentos y agua. En los hospitales, tanto el equipamiento médico como el ambiente que rodea a pacientes colonizados o infectados constituyen otro reservorio.
La vía más importante de transmisión nosocomial de ERG es el contacto directo o indirecto a través de las manos contaminadas del personal sanitario. Otras vías incluyen el contacto paciente-paciente y el contacto con el equipamiento médico y las superficies que rodean al paciente. En el caso de pacientes colonizados que presentan diarrea, el riesgo de transmisión aumenta4.
Los enterococos son considerados agentes de importancia en la infección nosocomial, fundamentalmente en pacientes críticos, quirúrgicos o de áreas especiales como quemados, hematooncológicos y trasplantados. Son causa importante de infecciones urinarias, bacteriemias, infecciones de herida quirúrgica, quemaduras, úlceras de pie diabético y úlceras de decúbito9.
Recogida, transporte conservación y procesamiento de la muestraEl tracto gastrointestinal de los pacientes ingresados constituye el principal reservorio de ERG en el ambiente hospitalario. Por este motivo, las muestras más habituales para el cultivo de vigilancia epidemiológica de ERG son las de frotis rectal o perianal y las de las heces. En ocasiones se pueden aceptar otras muestras como la orina y los exudados de herida, y si se requiere el estudio de contaminación ambiental se analizarán muestras procedentes de las superficies próximas al paciente y del instrumental médico en contacto con él9.
Las muestras no requieren condiciones especiales de transporte y conservación. Si embargo debido a las características de las muestras habitualmente seleccionadas para la detección de ERG (heces o frotis rectal), si el procesamiento no va a ser inmediato se recomienda su conservación a 4 °C, para evitar el sobrecrecimiento de la flora comensal acompañante.
Las muestras para vigilancia epidemiológica de ERG se inocularán directamente en los medios de cultivo, ya que no requieren la realización de medidas previas de pretratamiento. La inoculación de los medios de cultivo sólidos debe realizarse por agotamiento con el fin de obtener colonias aisladas.
Los medios más frecuentemente utilizados son medios selectivos y diferenciales que permiten la rápida detección de ERG a partir de muestras muy contaminadas11. Estos medios contienen vancomicina (como agente selectivo, habitualmente en concentración de 6 a 8 mg/l) y esculina, que es hidrolizada por enterococos y estreptococos grupo D dando lugar a esculetina, que al reaccionar con el citrato férrico del medio forma un complejo de color negro o marrón. Este fenómeno se traduce en un oscurecimiento del medio que rodea a las colonias crecidas en medio sólido y un oscurecimiento completo del caldo si se trata de medios líquidos. Además, contienen sales biliares para inhibir el crecimiento de otras bacterias grampositivas y azida sódica para inhibir el crecimiento de bacterias gramnegativas. Existen comercializados diferentes tipos de medios, líquidos y sólidos, que se pueden utilizar en el cribado de ERG. Incluso existen medios cromogénicos que permiten la detección de ERG. Algunos de estos medios permiten diferenciar E. faecium y E. faecalis. Las placas inoculadas deben incubarse a 35-37 °C durante 48 h con lecturas a las 24 y 48 h.
Criterios para la interpretación de resultadosLa aparición de pequeñas colonias translúcidas acompañadas de una pigmentación negra o marrón del medio antes descrito nos hará sospechar la presencia de ERG. Si se ha empleado caldo de cultivo se observará un oscurecimiento del medio. Si se utiliza otro medio de cultivo se buscarán colonias con las características que especifique el fabricante.
Las colonias sospechosas se someterán a tinción de Gram y catalasa. Si se trata de cocos grampositivos y catalasa negativo, serán subcultivadas a placa de agar sangre y placa de cribado de resistencia a vancomicina (agar BHI suplementada con 6 μg/ml de vancomicina) según recomendaciones del CLSI.
A partir del crecimiento en agar sangre se realizarán los estudios oportunos para su identificación en cuanto a especie y estudios de sensibilidad. La identificación se llevará a cabo mediante la utilización de pruebas clásicas (PYR, LAP, movilidad, pigmentación, fermentación de azúcares) o utilizando galerías comerciales, sistemas automatizados de identificación o técnicas de microbiología molecular.
Información de resultadosSi el cultivo es positivo para ERG se informará como "se aísla Enterococcus [faecalis/faecium] resistente a glucopéptidos". Si después de 48 h el cultivo es negativo para ERG se informará como "no se aísla Enterococcus resistente a glucopéptidos".
El aislamiento de E. gallinarum y E. casseliflavus, con baja resistencia intrínseca a vancomicina (vanC), no debe informarse como Enterococcus resistentes a glucopéptidos cuando se aíslen en cultivos de vigilancia, por carecer de significado epidemiológico.
Técnicas rápidas de diagnósticoEn los últimos años se han desarrollado técnicas de PCR en tiempo real que permiten detectar la presencia de los genes vanA y vanB directamente de muestras clínicas12,13. Los diferentes estudios realizados al respecto parecen demostrar una mayor sensibilidad de estas técnicas con respecto al método de cultivo en medios selectivos, además de una mayor rapidez en la obtención de los resultados y la posibilidad añadida de detectar ERG tanto de la muestra directa o como del crecimiento en cultivo convencional14.
Enterobacterias productoras de BLEEUno de los grupos de betalactamasas15 con mayor trascendencia clínica es el de las BLEE, codificadas por plásmidos. Estas enzimas se inhiben por inhibidores de betalactamasas de serina (como ácido clavulánico, tazobactan y sulbactam) e hidrolizan in vitro todos los betalactámicos de uso clínico, salvo los carbapenemes y las cefamicinas16.
La primera BLEE se describió en Alemania en la década de 1980, derivaba de la betalactamasa de amplio espectro SHV-1 y fue denominada SHV-2. Posteriormente, se describieron nuevas BLEE derivadas de TEM-1 y TEM-2. Más recientemente, se ha descrito un nuevo gran grupo de BLEE genéricamente conocido como CTX-M. Se conocen también otras BLEE aisladas menos frecuentemente que pertenecen a diferentes familias como OXA, PER, VEB, BES, GES, SFO, IBC, etc.17.
Aunque las BLEE son en su gran mayoría sensibles al ácido clavulánico, se han descrito variantes que siendo capaces de hidrolizar oxyimino-betalactámicos no se inhiben por los inhibidores de serina habituales.
Puede encontrarse una relación detallada de las diferentes enzimas incluidas en las distintas familias de BLEE en la siguiente dirección de Internet: http//www.lahey.org/studies/webt.htm.
Consideraciones clínicasHasta hace pocos años la mayoría de las infecciones por enterobacterias productoras de BLEE estaban causadas por Klebsiella pneumoniae o por Escherichia coli, y se producían en pacientes hospitalizados o en centros de enfermos crónicos. Sin embargo, en los últimos años han cobrado gran relevancia las infecciones de origen estrictamente comunitario producidas por estos microorganismos, en particular E. coli, productor de enzimas de la familia CTX-M. También están adquiriendo importancia creciente las infecciones producidas por otras especies de enterobacterias (Enterobacter spp., Proteus mirabilis y Salmonella enterica, entre otras). En un estudio multicéntrico sobre microorganismos productores de BLEE realizado en España18,19 en el año 2000, el 93% de las K. pneumoniae se aislaron en pacientes hospitalizados, pero el 51% de E. coli con BLEE se aislaron en pacientes que en el momento de toma de la muestra clínica no estaban hospitalizados.
El principal mecanismo de transmisión de estos microorganismos ocurre a través de las manos del personal sanitario, que se coloniza cuando entran en contacto con pacientes que, a su vez, están colonizados. El aparato digestivo es el principal reservorio de estas cepas. La tasa de portadores fecales en situaciones de endemia en unidades de cuidados intensivos puede llegar al 30-70% de los pacientes ingresados. Se han descrito otros mecanismos de transmisión que han involucrado a los objetos que rodean a los pacientes o a productos utilizados en la higiene de los mismos, pero la contribución real de estas fuentes en el desarrollo del brote es difícil de determinar. Los pacientes con mayor riesgo de desarrollar colonización o infección por microorganismos productores de BLEE son aquellos que tienen una enfermedad de base grave, estancias prolongadas en el hospital, diferentes objetos médicos de soporte vital (sonda urinaria, catéteres intravasculares, tubos endotraqueales) y reciben tratamiento antimicrobiano durante períodos prolongados17. Varios estudios han relacionado el uso de cefalosporinas de amplio espectro, fluoroquinolonas, cotrimoxazol y aminoglucósidos con la adquisición de infección por cepas productoras de BLEE20.
Recogida, transporte, conservación y procesamiento de la muestraMuchos pacientes sufren una colonización asintomática por enterobacterias productoras de BLEE. Como el principal reservorio es el aparato digestivo, las muestras de elección son un frotis rectal o las heces21.
En una unidad en la que no se hubieran detectado previamente cepas productoras de BLEE, se aconseja tomar un frotis rectal/heces de los pacientes ingresados para determinar el grado de colonización. También deben tomarse muestras ambientales. Una vez que la situación se hace endémica, el control es mucho más complejo, pero la toma de muestras de vigilancia sigue siendo importante para determinar la utilidad de las medidas de control que puedan implementarse.
No se precisan condiciones especiales de transporte y conservación para el cultivo de enterobacterias productoras de BLEE.
Hay descritos en la literatura médica diferentes medios selectivos (p. ej., MacConkey, agar Driglasky y agar nutritivo con vancomicina y anfotericina B) para un rendimiento óptimo en la recuperación de enterobacterias productoras de BLEE a partir de frotis rectales y heces, siendo los más habituales los que están suplementados con cefotaxima o ceftacidima. También se ha comercializado recientemente un medio cromogénico (ESBL-Bx)22 que permite una identificación preliminar de las enterobacterias productoras de BLEE en 24 h.
Las concentraciones de cefotaxima o ceftacidima empleadas en diferentes estudios han oscilado entre 0,5 y 4 mg/l, aunque una concentración demasiado alta (4 mg/l) podría determinar una menor sensibilidad en la detección. Aunque las BLEE hidrolizan con mayor o menor eficacia tanto cefotaxima como ceftacidima, algunas CTX-M hidrolizan muy poco ceftacidima. Las placas se incubarán a 35 °C en aerobiosis, con lecturas a las 24 y 48 h de la siembra.
Criterios para la interpretación de resultadosAunque los medios selectivos están diseñados para garantizar una alta sensibilidad en la detección de cepas con BLEE, el criterio de selección resulta poco específico. Por tanto, deben seleccionarse las colonias con morfología de enterobacteria y proceder a su identificación como especie. El CLSI ha estandarizado métodos de cribado y de confirmación fenotípica de producción de BLEE en E. coli, Klebsiella spp. y P. mirabilis23. La caracterización de la presencia de BLEE en otras enterobacterias (o incluso en bacterias gramnegativas no fermentadoras) es mucho más compleja y no se desarrollará en este documento. En cualquier caso, la detección de BLEE en otras especies sin AmpC cromosómica, como S. enterica, podría estar basada en los criterios que se exponen a continuación, mientras que en aquellos que sí expresan habitualmente AmpC (Enterobacter spp., Citrobacter freundii, Morganella morganii, Serratia marcescens, etc.) la sospecha de la presencia de BLEE puede inferirse (entre otros marcadores) de la actividad comparada de cefepima y cefepima más ácido clavulánico.
Para E. coli, K. pneumoniae y P. mirabilis, el CLSI ha propuesto sendos métodos de cribado para la detección de BLEE mediante la realización de un antibiograma por el método de disco difusión o mediante el método de dilución23. En este último caso se indica el uso de ceftacidima, aztreonan, cefotaxima o ceftriaxona a una concentración de 1 μg/ml. Puesto que las placas con medio selectivo de alguna forma remedan esta última situación, es razonable asumir que los microorganismos que crecen en las placas selectivas son sospechosos de producir BLEE, por lo que es aconsejable realizar directamente una prueba de confirmación de la producción de BLEE.
Para llevar a cabo esta confirmación, el CLSI también propone dos opciones basadas en los métodos de disco difusión y de microdilución, respectivamente. Para la confirmación de la producción de BLEE mediante disco difusión, se recomienda la utilización de discos de cefotaxima (30 μg) o ceftacidima (30 μg) con y sin ácido clavulánico (10 μg). La prueba se realiza en agar Mueller-Hinton con un inóculo de 0,5 McFarland y un tiempo de incubación de 16-18 h. Una diferencia de a partir de 5 mm en el diámetro del halo de cualquiera de las cefalosporinas y el correspondiente halo del disco con ácido clavulánico confirma la producción de BLEE. La confirmación mediante microdilución en caldo se basa en el uso de ceftacidima (0,25-128 μg/ml) y cefotaxima (0,25-64 μg/ml) con y sin una concentración fija de ácido clavulánico (4 μg/ml). Se considera que la prueba es positiva cuando la CMI de una o de las dos cefalosporinas disminuye 3 o más diluciones en presencia de ácido clavulánico. Igual que en el método de disco difusión, es necesario utilizar las dos cefalosporinas.
La prueba de confirmación fenotípica puede ocasionar falsos positivos en cepas de K. pneumoniae y de E. coli que hiperproducen SHV-124 y que han sufrido una pérdida de porinas y en cepas de Klebsiella oxytoca que hiperproducen la betalactamasa cromosómica K1; el incremento de CMI de aztreonam junto con CMI de ceftriaxona y de cefotaxima mucho mayores que la CMI de ceftacidima ayuda a sospechar esta última posibilidad. Se han descrito falsos negativos en cepas de K. pneumoniae que producen, además de BLEE, una betalactamasa plasmídica de tipo AmpC, por la incapacidad del ácido clavulánico para inhibir esta última enzima.
La detección de BLEE se puede determinar también con otras metodologías (método de difusión del doble disco, método de difusión con discos comparando los resultados en agar Mueller-Hinton y en agar Mueller-Hinton suplementado con ácido clavulánico, y prueba tridimensional entre otros), incluyendo sistemas comerciales, como tiras de Etest (cuyo principal inconveniente es el elevado coste). Varios sistemas automatizados de antibiograma25 como Vitek (BioMerieux), MicroScan (Dade Behring) y BD Phoenix (Becton Dickinson Biosciences) disponen en sus paneles/tarjetas comerciales de pocillos específicamente diseñados para el reconocimiento de enterobacterias productoras de BLEE. En general, se obtienen buenos resultados con estos sistemas, que, además, suelen disponer de un sistema experto que alerta de la presencia de este mecanismo de resistencia.
Información de los resultadosSi el cultivo es positivo para E. coli, Klebsiella spp. o P. mirabilis y se confirma la producción de BLEE, se informará como "se aísla [nombre de la especie] productora de BLEE". Si tras 48 h el cultivo es negativo, o si en algunas de las tres especies indicadas no se demuestra la producción de BLEE se informará como "no se aísla E. coli, Klebsiella spp. o P. mirabilis productores de BLEE". La identificación de alguna otra enterobacteria en la que sin género de duda se haya demostrado la presencia de BLEE se informará de igual forma "se aísla [nombre de especie] productora de BLEE", teniendo en cuenta los objetivos perseguidos en el programa de vigilancia que se esté llevando a cabo.
Acinetobacter baumannii multirresistenteEl género Acinetobacter incluye un amplio número de especies/genospecies de las que la de mayor importancia clínica es A. baumannii. La compleja taxonomía de estos microorganismos dificulta bastante su identificación precisa en cuanto a especie, lo que suele requerir el uso de métodos moleculares26. Con gran frecuencia las cepas de A. baumannii nosocomiales son resistentes a betalactámicos (incluyendo los carbapenems en bastantes ocasiones), fluoroquinolonas, aminoglucósidos, tetraciclinas y cotrimoxazol. Algunas cepas sólo son sensibles a las polimixinas, pero incluso se han descrito ya aislados resistentes también a estos compuestos.
La resistencia a betalactámicos en A. baumannii se relaciona con la producción de diferentes betalactamasas (AmpC, oxacilinasas, metalo-betalactamasas), con alteraciones de la permeabilidad y con la expresión de PBP de baja afinidad27. La producción de enzimas modificadoras de aminoglucósidos y de mecanismos de expulsión activa se relaciona con la resistencia a aminoglucósidos. En el caso de la resistencia a las fluoroquinolonas se han descrito mutaciones cromosómicas que conducen a alteraciones de las topoisomerasas de clase II (ADN-girasa y topoisomerasa IV) y mecanismos de expulsión activa. Muchas cepas albergan integrones de clase 1 que contienen los genes que codifican oxacilinasas, metaloenzimas y enzimas modificadoras de aminoglucósidos.
Los porcentajes de sensibilidad a distintos antimicrobianos en 211 aislados de A. baumannii obtenidos en un estudio multicéntrico español (25 centros) del año 2000 fueron: polimixina B, 100%; minociclina, 66%; imipenem, 52%; rifampicina, 49%; sulbactam, 47%; meropenem, 43%; amicacina, 35%; doxiciclina, 32%; tobramicina, 21%, y para piperacilina, ceftacidima, cefepima, gentamicina, cotrimoxazol, tetraciclina, ciprofloxacino y gemifloxacino la sensibilidad fue inferior al 20%28.
En la mayoría de los centros, tras una primera fase de epidemia que no llega a controlarse se produce una situación de endemicidad. Los estudios de epidemiología molecular indican que incluso en esta última situación sólo persisten uno o dos clones mayoritarios, aunque también se han descrito situaciones en las que en un mismo centro coexisten múltiples clones29.
Consideraciones clínicasComo A. baumannii puede sobrevivir en el medio ambiente durante largos períodos, la contaminación ambiental es importante porque permite la transmisión del patógeno al paciente directamente o a través de las manos del personal sanitario, que suele estar colonizado sólo de forma transitoria. Además, los pacientes colonizados o infectados son un importante reservorio del microorganismo30.
A. baumannii puede causar una amplia variedad de infecciones, incluyendo bacteriemia, neumonía nosocomial (en particular en el paciente con ventilación mecánica), meningitis, infecciones urinarias, infecciones de heridas quirúrgicas e infecciones de tejidos blandos, entre otras.
A. baumannii es típicamente un patógeno nosocomial. Aunque se han descrito brotes en unidades médicas y quirúrgicas, la mayoría han tenido lugar en unidades de cuidados intensivos (UCI). Los factores de riesgo relacionados con su adquisición incluyen una estancia hospitalaria prolongada, empleo de maniobras invasoras, inmunosupresión, enfermedad de base grave y uso previo de antimicrobianos. Se ha observado que las infecciones nosocomiales por A. baumannii (y en particular la bacteriemia) siguen cierta estacionalidad, con un incremento de casos en los meses de verano. A diferencia de lo que ocurre con SARM, no parece que la expansión de A. baumannii multirresistente al medio extrahospitalario haya sido muy importante29,31.
Recogida, transporte conservación y procesamiento de la muestraLas muestras de vigilancia en pacientes más frecuentemente evaluadas incluyen esputo y exudado de traqueotomía, heridas, axila/ingle y frotis rectal. En un estudio español sobre detección de A. baumannii en distintas muestras de vigilancia en pacientes de UCI, se identificó el microorganismo en el 75% de las muestras axilares o faríngeas y en el 77% de los frotis rectales; la combinación de muestras axilar-faríngea y axilar-rectal permitió la identificación en el 90% de los pacientes, cifra que alcanzó el 96% para la combinación de la muestra faríngea-rectal. Las muestras ambientales que hay que considerar pueden ser muy diversas. Se ha descrito contaminación por A. baumannii en componentes de los equipos de respiración asistida, líquidos diversos, medicaciones multidosis, ropa de cama, transductores de presión no desechables, etc.
No es necesario observar condiciones especiales de transporte y conservación de las muestras de vigilancia epidemiológica. Como en otros casos, si el procesamiento de los frotis rectales no va a ser inmediato, se recomienda su conservación a 4 °C.
Las muestras procedentes de pacientes se procesarán siguiendo las pautas habituales. Las muestras ambientales tomadas con torunda en medio líquido se cultivarán en un medio sólido para identificación de colonias, manteniendo la punta de la torunda en el tubo con un medio líquido para asegurar el crecimiento de pequeñas cantidades de inóculo.
A. baumannii crece sin dificultad en los medios de cultivo habituales. Las muestras de vigilancia procedentes de los pacientes se pueden sembrar en cualquiera de los medios selectivos habituales para bacterias gramnegativas, (como MacConkey). Teniendo en cuenta que el objetivo del estudio es la detección de las cepas multirresistentes, es preferible emplear medios de cultivo diferenciales suplementados con un antimicrobiano al que el microorganismo sea resistente. Se ha empleado el medio LAM (Leeds Acinetobacter Medium), que está suplementado con vancomicina (10 mg/l), cefsulodina (15 mg/l) y cefradina (50 mg/l) para el aislamiento selectivo de Acinetobacter en muestras clínicas o ambientales. Como una gran mayoría de cepas son resistentes a gentamicina, muchos protocolos aconsejan también el uso de agar MacConkey suplementado con gentamicina en una concentración de 8 μg/ml. Podría aprovecharse el uso de placas de agar MacConkey suplementado con cefotaxima/ceftazidima empleadas en la vigilancia de otros patógenos (como se ha explicado con anterioridad). Las muestras ambientales pueden procesarse en infusión cerebro corazón (BHI), y el subcultivo puede realizarse en el mismo tipo de medio sólido empleado para las muestras clínicas descritas previamente. Como en otros casos, los medios de cultivo se incubarán en aerobiosis a 35 °C durante 48 h.
Criterios para la interpretación de resultadosSe realizará una lectura de los cultivos a las 24 y a las 48 h de su inoculación. La identificación presuntiva de las colonias de A. baumannii se basará en la morfología de las colonias. El microorganismo es productor de catalasa pero no de oxidasa. La identificación como especie, como se ha dicho, es compleja y mediante el uso de galerías o de paneles comerciales no se garantiza completamente. Por esta razón, en muchos laboratorios la identificación bioquímica se completa sólo con un tubo de tres azúcares hierro (TSI) (A. baumannii es no fermentador y crece visiblemente en la superficie de la lengüeta del medio) y a lo sumo de movilidad (A. baumannii es inmóvil). Si se emplean los referidos métodos bioquímicos comerciales y el método en cuestión señala la identificación de A. baumannii, es recomendable casi siempre hacer referencia en la identificación al "complejo A. baumannii" antes que la especie A. baumannii propiamente dicha. La identificación de especie definitiva se realiza por métodos moleculares, habitualmente en centros de referencia.
Información de resultadosLa identificación de Acinetobacter sp. (o A. baumannii si se ha realizado una identificación precisa) multirresistente se informará como "se aísla Acinetobacter sp. multirresistente". Si tras 48 h de incubación no se aísla el microorganismo, se informará "No se aísla Acinetobacter sp.". Si la cepa aislada no fuera multirresistente se informará "No se aísla Acinetobacter sp. multirresistente".
Pseudomonas aeruginosa productora de metalo-betalactamasaP. aeruginosa es un microorganismo que sobrevive fácilmente en el ambiente hospitalario, y en la actualidad constituye uno de los principales patógenos nosocomiales oportunistas, sobre todo en pacientes inmunodeprimidos y grandes quemados. Presenta resistencia natural a muchos antimicrobianos (la mayoría de las penicilinas, cefalosporinas de primera, segunda y muchas de las de tercera generación, tetraciclinas, cloranfenicol, cotrimoxazol, rifampicina), y con gran facilidad desarrolla mutaciones cromosómicas y adquiere material genético que incrementan su resistencia.
En un estudio multicéntrico español con 1.014 cepas procedentes de 136 hospitales32 se comprobó que ninguno de los antimicrobianos evaluados era activo frente a todos los aislados. Las tasas de resistencia para los principales compuestos fueron: piperacilina-tazobactam, 7%; meropenem, 8%; amicacina, 9%; tobramicina, 10%; imipenem, 14%; ceftacidima, 15%; cefepima, 17%; aztreonam y ciprofloxacino: 23%, y gentamicina: 31%.
La resistencia intrínseca de P. aeruginosa depende de la baja permeabilidad de su membrana externa y de varios sistemas de expulsión activa33. Casi la totalidad de los aislamientos clínicos de P. aeruginosa expresan la betalactamasa AmpC, cuya desrepresión puede ser parcial o total. La práctica totalidad de cepas clínicas poseen también 0XA-50, aunque su contribución a la resistencia parece irrelevante. Algunas cepas producen, además, otras betalactamasas adquiridas, de las que son particularmente importantes las metalo-betalactamassas (MBL), carbapenemasas que se inhiben por EDTA, pero no por ácido clavulánico34. Se han descrito varios grupos de MBL: IMP, VIM, SPM, GIM, SIM y AIM (de ellos, los de mayor importancia son IMP y VIM)34.
La pérdida de la porina OprD causa una disminución drástica en la penetración de carbapenems, lo que junto con la expresión de AmpC y de sistemas de expulsión activa acaba determinando también la aparición de resistencia clínica a estos compuestos. El sistema de expulsión MexAB-OprM se expresa de forma basal y su actividad afecta a meropenem, pero no a imipenem. Como la desrepresión de AmpC es más frecuente que la hiperexpresión de MexAB-OprM, en cepas clínicas la frecuencia de resistencia a imipenem es mayor que la de resistencia a meropenem. La resistencia a aminoglucósidos se debe a la producción de enzimas modificadoras y al sistema de expulsión activa MexXY-OprM. La resistencia a quinolonas en P. aeruginosa depende de la existencia de las ya citadas bombas de expulsión y de alteraciones en las topoisomerasas. Aunque la detección de resistencia a carbapenems en P. aeruginosa no es difícil empleando los métodos estandarizados habituales en el laboratorio clínico, sí es complejo precisar cuál es el mecanismo subyacente. La caracterización de estos mecanismos tiene importantes implicaciones epidemiológicas, y es deseable aclarar si obedecen a mutaciones cromosómicas o a la producción de carbapenemasas, en especial de MBL, cuyos correspondiente genes suelen estar codificadas por integrones (de clase 1 y en ocasiones de clase 3) y por transposones.
Las MBL no hidrolizan los monobactams, por lo que la combinación de resistencia a carbapenem y sensibilidad a aztreonam sugiere la presencia de estas enzimas35. Por desgracia, el microorganismo puede expresar otros mecanismos que, independientemente, causan resistencia a aztreonam; por ello, no siempre que existen MBL se observa el fenotipo indicado. Se han desarrollado diferentes métodos que permiten la detección fenotípica de MBL en P. aeruginosa (y por analogía en otros microorganismos), basados en la inhibición del enzima por EDTA o por otro tipo de inhibidores (fenantrolina, derivados del ácido mercaptoacético, del ácido mercaptopropiónico). No se dispone de un método estandarizado para la detección de MBL, y además los métodos fenotípicos indicados no son completamente sensibles ni específicos, por lo que la confirmación definitiva de la presencia de la enzima depende, por el momento, de métodos moleculares36.
Consideraciones clínicasAunque P. aeruginosa no suele formar parte de la flora normal, en ocasiones existe colonización en el tracto gastrointestinal y en otras zonas, como faringe, axila y periné. Puede producirse contaminación de productos y equipos hospitalarios, en particular de aquellos que poseen componentes en contacto con la humedad. También puede encontrarse en la superficie de frutas y verduras.
A pesar de la amplia distribución que puede tener P. aeruginosa en el medio ambiente, varios estudios sugieren que el reservorio de los pacientes colonizados es de mayor importancia para el ulterior desarrollo de brotes epidémicos, en los que los pacientes adquieren el microorganismo por transmisión cruzada.
P. aeruginosa posee múltiples factores de virulencia, pero más allá de su producción, la patogenia de las infecciones por este agente se relaciona estrechamente con la situación del huésped, y resultan importantes la rotura de la barrea cutáneo-mucosa, los trastornos de la inmunidad humoral y la neutropenia.
Los principales factores relacionados con la multirresistencia en P. aeruginosa han sido la gravedad de la infección, el uso de dispositivos invasores, la hospitalización prolongada y la exposición previa a antimicrobianos, en particular (y como para tantas otras bacterias gramnegativas) betalactámicos y quinolonas37,38.
P. aeruginosa puede producir una amplísima variedad de infecciones, como bacteriemia, neumonía asociada a ventilación mecánica, infecciones respiratorias en pacientes con fibrosis quística y neumonía de la comunidad, infecciones urinarias, endocarditis, meningitis, diversas formas de otitis, queratitis y endoftalmitis, osteomielitis, enterocolitis e infecciones perianales, entre otras.
Recogida, transporte y conservación de la muestraLos cultivos de vigilancia epidemiológica para P. aeruginosa multirresistente deben tener en cuenta la toma de muestras de pacientes y muestras del medio ambiente y equipos de atención sanitaria. No son necesarias consideraciones especiales para el transporte y conservación de muestras en que se investigue la presencia de P. aeruginosa.
P. aeruginosa sobrevive en la mayoría de ambientes y temperaturas propias del entorno clínico y crece con facilidad en los medios de cultivo habituales, incluyendo los medios de cultivo diferenciales para bacterias gramnegativas, como agar MacConkey. Se han diseñado medios con agentes selectivos (como cetrimida) para favorecer el crecimiento de P. aeruginosa a partir de muestras clínicas con flora mixta y muestras ambientales.
La gran mayoría de las cepas clínicas de P. aeruginosa son resistentes a las concentraciones de cefotaxima que habitualmente se emplean en los medios selectivos de vigilancia epidemiológica diseñados para el aislamiento de otros microorganismos multirresistentes, por lo que su uso garantizaría el crecimiento de P. aeruginosa con independencia de si el aislado es multirresistente. La adición de ceftacidima, aminoglucósidos, o carbapenems a agar MacConkey u otro medio equivalente favorecerá específicamente la selección de cepas con más resistencia.
Para el cultivo de muestras ambientales y de equipos médicos pueden seguirse las recomendaciones señaladas para A. baumannii, con las particularidades que acaban de indicarse. Los medios de cultivo deben incubarse en aerobiosis a 35 °C durante 48 h.
Criterios para la interpretación de resultadosLa lectura de los medios de cultivo en busca de P. aeruginosa (de cepas resistentes al antimicrobiano deseado si se han empelado medios selectivos específicos) se realizará a las 24 y a las 48 h. P. aeruginosa se identifica fácilmente en función del aspecto de la colonia, la pigmentación y algunas pruebas bioquímicas sencillas: producción de oxidasa, oxidación de la glucosa pero no fermentación de la misma, presencia de arginina-dehidrolasa y crecimiento a 42 °C. Los sistemas comerciales de identificación habitualmente empleados en el laboratorio son también bastante fiables para la identificación del microorganismo, excepto en el caso de cepas mucoides.
La confirmación de la resistencia a los diferentes antimicrobianos (y de la multirresistencia, por tanto) se hará en función de los resultados del antibiograma. La identificación de cepas que presuntamente producen MBL se basará en los resultados de pruebas fenotípicas35. En ausencia de un método estandarizado para llevar a cabo esta detección se han empleado múltiples ensayos, basados en la capacidad de inhibir la actividad de la MBL con un agente quelante. Como agente indicador se han usado los carbapenems, pero también ceftacidima (que es hidrolizada por la MBL), lo que aumenta la sensibilidad del estudio. Con relación a los métodos de difusión, se han empleado el método de doble disco con EDTA e imipenem o ceftacidima, o con ácido mercaptopropiónico e imipenem o ceftacidima, comprobando que el quelante es capaz de aumentar el diámetro del halo del betalactámico. En otros casos, empleando una aproximación similar a la que recomienda el CLSI para la detección de BLEE, se han usado discos que incorporan simultáneamente EDTA y un carbapenem o ceftacidima, comparando el diámetro del halo de inhibición con el del correspondiente disco sin EDTA. También se ha usado un extracto del microorganismo obtenido por sonicación con el que se impregnan discos de carbapenem o ceftacidima con y sin EDTA, comparando luego los correspondientes halos de inhibición.
Otro método alternativo basado en la microdilución se basa en la comparación de la CMI de carbapenem solo y en combinación con dos inhibidores, EDTA (0,4 mM) y 1,10 fenantrolina (0,04 mM). Por analogía con el ensayo de detección de BLEE, la disminución de la CMI de carbapenem en combinación con los quelantes de al menos 8 veces sugiere la presencia de MBL. Con el mismo fundamento metodológico se dispone de tiras de Etest que contienen imipenem e imipenem + EDTA. Un incremento de la CMI de imipenem en presencia de EDTA de al menos 8 veces es sugestivo de la presencia de MBL. Como en otros casos el principal inconveniente de este último método es el precio de las tiras.
Estos métodos no son completamente sensibles ni específicos y en ciertos casos tienen difícil interpretación, con frecuencia subjetiva. Por esta razón se han puesto a punto métodos de detección molecular de MBL tipo IMP y VIM (en principio sería extensible a otros grupos de MBL) mediante PCR36. Además, como las MBL suelen estar incluidas en integrones, también se ha demostrado la presencia de un gen que codifica MBL empleando cebadores específicos para los integrones de clase 1 en combinación con cebadores específicos para MBL.
Información de resultadosEn función de la metodología empleada, el informe de resultados cuando se haya identificado P. aeruginosa será: "Se aísla Pseudomonas aeruginosa [multirresistente/productoras de metalo-betalactamasa]". Si tras 48 h de incubación no se aísla el microorganismo se informará "No se aísla Pseudomonas aeruginosa".