La infección del tracto urinario (ITU) es una patología frecuente, de gran preocupación por la resistencia creciente de los microorganismos causantes frente a los antibióticos de primera línea y la aparición de cepas resistentes productoras de betalactamasas de espectro extendido en la comunidad.
MétodosEstudio analítico tipo casos y controles de 12 meses, en 9 hospitales de Colombia. Se analizaron aislamientos de Escherichia coli, Klebsiella spp. y Proteus spp. de pacientes con ITU de inicio comunitario. Se determinó la presencia de betalactamasas de espectro extendido, AmpC y KPC por métodos microbiológicos y moleculares. Se establecieron factores relacionados con la presencia de estos mecanismos de resistencia a cefalosporinas de tercera generación.
ResultadosSe recolectaron 325 aislamientos (287 E. coli, 29 Klebsiella spp. y 9 Proteus spp.). Las comorbilidades más frecuentes fueron hipertensión arterial (n=82; 25,2%) y diabetes mellitus (n=68; 20,9%). Se encontró consumo previo de antibióticos en el 23% y antecedente de ITU previa en el 29%. La resistencia a cefalosporinas de tercera y cuarta generación varió entre el 3,4 y el 6,3% para E. coli y entre el 3,4 y el 17,2% para K. pneumoniae.
Se detectó CTX-M-15 en 7 aislamientos de E. coli (2,4%), 4 pertenecientes al clon ST131. En K. pneumoniae se detectaron 3 aislamientos positivos para KPC-3(10,3%).
ConclusiónSe confirma la emergencia de enterobacterias resistentes a cefalosporinas de tercera generación como causa de ITU de inicio comunitario. Se resalta la circulación en Colombia del clon ST 131 y carbapenemasas tipo KPC en pacientes fuera del ambiente hospitalario.
Urinary tract infection (UTI) is a common disease in the community, and a matter of concern due to the increasing resistance of microorganisms to first line antibiotics and the emergence of multiresistant strains producing extended spectrum beta lactamases (ESBL) in the community.
MethodsAn analytical case-control study was conducted over twelve months in 9 hospitals in Colombia. We collected isolates of E. coli, Klebsiella spp. and Proteus spp. from patients with community-onset UTI. The presence of ESBL, AmpC and KPC betalactamases were characterized by microbiological and molecular methods. The aim of this study was to determine factors related to the presence of these mechanisms of the resistance to third generation cephalosporins.
ResultsA total of 325 isolates (287 E. coli, 29 Klebsiella spp. and 9 Proteus spp.) were included. The most frequent comorbidities among the patients were hypertension (n=82; 25.2%) and diabetes mellitus (n=68; 20.9%). Previous use of antimicrobials was found in 23% of patients, and 29% had a previous UTI. Resistance to third and fourth generation cephalosporins varied between 3.4% and 6.3% in E. coli and between 6.9% and 17.8% in K. pneumoniae. Seven (2.4%) CTX-M-15 ESBL-producing E. coli isolates were detected; four of them belonged to ST 131 clone. In K. pneumoniae we detected three KPC-3 carbapenemases (10.3%).
ConclusionsThis study confirms the emergence of resistance to third generation cephalosporins enterobacteriaceae as a cause of community-onset UTI. We emphasize the presence of ST 131 clone and KPC carbapenemases circulating in Colombia outside the hospital environment.
La infección del tracto urinario (ITU) es una de las principales enfermedades de origen infeccioso. Los patógenos más importantes son las enterobacterias, especialmente Escherichia coli, que produce más de 80% de estas infecciones a nivel comunitario. La presencia de betalactamasas de espectro extendido (BLEE) fue inicialmente descrita en aislamientos de Klebsiella pneumoniae y E. coli de origen nosocomial, pero en los últimos años se ha comprobado la diseminación de BLEE, particularmente del tipo CTX-M, en cepas de E. coli causantes de infecciones de inicio comunitario1-4.
CTX-M-15 es una de las variantes de CTX-M más frecuentes a nivel mundial, en diferentes enterobacterias5,6. Actualmente su diseminación global se asocia con cepas de E. coli virulentas pertenecientes al clon pandémico ST131 y que causa con frecuencia infecciones adquiridas en la comunidad. Las cepas de este clon pueden ser portadoras de otras variantes de CTX-M y de otras familias de BLEE, como TEM y SHV, y también pueden presentar enzimas AmpC tipo CMY5,7. En Colombia se ha descrito la circulación de cepas de E. coli productoras de CTX-M-15 pertenecientes a los clones ST131 y ST405 en diferentes ciudades8.
La magnitud y los factores de riesgo asociados a ITU de inicio comunitario causada por enterobacterias productoras de BLEE han sido descritos en diversas partes de mundo3,4,9-11. Sin embargo, en América Latina existe poca información sobre esta problemática.
El objetivo del presente estudio fue determinar los perfiles de susceptibilidad a los principales agentes antimicrobianos utilizados en el manejo de infecciones urinarias de inicio en la comunidad que consultan a servicios de urgencias de hospitales de tercer nivel de atención, caracterizar molecularmente y describir los factores de riesgo asociados a la presencia de resistencia a cefalosporinas de tercera generación.
MétodosSe realizó un estudio analítico tipo casos y controles durante 12 meses de aislamientos provenientes de 9 hospitales de tercer nivel de complejidad en 3 ciudades de Colombia. Los 9 hospitales eran hospitales generales, cuya admisión se producía primordialmente por el servicio de urgencias, de carácter público (2 instituciones) o privado (7 instituciones). Se incluyeron todos los pacientes de ambos sexos mayores de 18 años con diagnóstico al ingreso de ITU (pielonefritis) de inicio comunitario que requirieran tratamiento hospitalario por orden médica en razón de la gravedad de su presentación en urgencias o por la comorbilidad subyacente y cuyo aislamiento microbiológico incluyera E. coli, K. pneumoniae o Proteus mirabilis. La definición de ITU de inicio comunitario se consideró en todos los pacientes cuyos síntomas se hubieran iniciado al menos 48h previas al ingreso a la institución y no llevaran sonda vesical permanente, ni se hubieran realizado procedimientos quirúrgicos de la vía urinaria en la semana previa o hubieran estado hospitalizados en los últimos 7 días. Si un episodio de ITU fue separado por más de un mes desde el alta hospitalaria previa hasta el nuevo episodio, este se consideró como un nuevo caso.
La captación de los pacientes se realizó mediante una búsqueda activa a partir de los diagnósticos de ingreso y de los hallazgos microbiológicos en cada una de las instituciones. La información de cada uno de los pacientes se obtuvo a partir de la revisión de sus respectivas historias clínicas o por información directa de estos, utilizando un formato de recogida de datos que incluía las siguientes variables: edad, género, comorbilidades, ITU previa (uno o más episodios en los últimos 3 meses), presencia de síntomas, antecedentes quirúrgicos, uso previo de antibióticos (en los últimos 3 meses), tipo y duración del antibiótico.
Todos los aislamientos de E. coli, Klebsiella spp. y Proteus spp. obtenidos a partir de urocultivos en cada una de las instituciones se enviaron al laboratorio de microbiología de la Facultad de Medicina de la Universidad Nacional de Colombia, para confirmar su identificación y susceptibilidad por método automatizado (Phoenix, BD, Estados Unidos) a los siguientes antibióticos: amikacina, cefepima, cefazolina, cefotaxima, ceftazidima, ceftriaxona, ciprofloxacino, ertapenem, gentamicina, imipenem, meropenem, piperacilina/tazobactam, trimetoprim/sulfametoxazol, amoxicilina-ácido clavulánico y cefoxitina. La susceptibilidad a ampicilina-sulbactam fue determinada por difusión en agar por el método de E-test (AB Biodisk, Solna, Suecia). Para confirmar la presencia de BLEE se utilizó el método de doble disco, y para confirmar la producción de carbapenemasas se utilizó la prueba de Hodge. Las pruebas de susceptibilidad se realizaron de acuerdo a las recomendaciones de manufactura de los paneles (Phoenix, BD, Estados Unidos), y la interpretación de la CIM se realizó siguiendo las recomendaciones de CLSI 201012.
Como control de calidad de las pruebas de susceptibilidad antimicrobiana se utilizaron las cepas de E. coli ATCC 25922, Pseudomonas aeruginosa ATCC 27853 y K. pneumoniae ATCC 700603.
En los aislamientos con fenotipo BLEE se evaluó la presencia de los genes blaTEM, blaSHV y blaCTX-M por medio de PCR multiplex13. Los aislamientos positivos para CTX-M se sometieron a una amplificación adicional para identificación de grupos de CTX-M 1, 2, 8, 9 y 2514. Para los aislamientos con fenotipo AmpC se detectó la presencia de betalactamasas AmpC plasmídicas utilizando los iniciadores MOX, CIT, DHA, ACC, EBC y FOX en un ensayo de PCR multiplex15, y en los aislamientos con resistencia a carbapenémicos se evaluó por PCR la presencia de genes codificantes de carbapenemasas blaKPC16, blaIMP y blaVIM17 y blaNDM18. Los productos de amplificación obtenidos fueron secuenciados y analizados con el programa BLAST.
La relación genética de aislamientos de E. coli productores de enzimas CTX-M, SHV y AmpC plasmídicas se realizó por REP- PCR, y la de K. pneumoniae productores de KPC, CTX-M y SHV, por ERIC-PCR utilizando los iniciadores previamente descritos19. Los patrones electroforéticos obtenidos se analizaron usando el software Fingerprinting II (Bio-Rad); para el análisis de agrupamiento se usó el algoritmo UPGMA (unweight pair-group meted with arthmetic averages), con el 1% de optimización y de tolerancia, y los aislamientos con más del 85% de similitud fueron considerados del mismo genotipo.
En los aislamientos de E. coli productores de CTX-M-15 se determinó el grupo filogenético (A, B1, B2 o D) por medio de la amplificación de los genes genes ChuA, YjaA y TSPE4 utilizando los iniciadores descritos por Clermont et al.20. Se seleccionaron para ser identificados los siguientes factores de virulencia como representantes de las 5 clases descritas: FimG/H, papC, usp, SfaD/sfaE, hly y cnf. Para evaluar la resistencia a quinolonas, sulfonamidas y tetraciclinas se detectó por PCR la presencia de los genes qnrA, qnrB y qnrS; sul1, sul2 y sul3; y tetA, tetB y tetC, respectivamente21.
La relación genética de los aislamientos de E. coli con el clon ST131 se determinó de acuerdo con la metodología publicada por Dhanji et al. en 201022 y confirmada por secuenciación directa de los productos de amplificación.
El análisis estadístico incluyó inicialmente un análisis univariado, para conocer la distribución de todas las variables del estudio. Posteriormente se realizó un análisis bivariado para determinar la relación de cada una de las variables de interés clínico con la presencia o no de cualquier tipo de resistencia a cefalosporinas de tercera generación. Para las variables continuas se realizó una prueba de suma de rangos sin signos, y para las variables categóricas se realizó chi cuadrado. Para el análisis de los factores de riesgo de resistencia a cefalosporinas de tercera generación se realizó un análisis multivariado tipo regresión logística, en el que se asociaban las variables que tuvieran alguna relación clínica o que en el análisis bivariado mostraran asociación estadística, como predictoras de resistencia a cefalosporinas de tercera generación. El análisis se realizó con stepwise. Con el mejor modelo predictivo, se calculó el área bajo la curva ROC para conocer la predicción del modelo. Todos los análisis fueron realizados con Stata (versión 11.0, Texas, Estados Unidos).
ResultadosSe obtuvieron 325 aislamientos provenientes de 323 pacientes. De los pacientes analizados, 18 (5,6%) fueron menores de 50 años y 271 (83,4%) del género femenino, de las cuales 74 (22,8%) estaban en embarazo. La muestra que predominó fue la micción espontánea en 174 pacientes (53,5%), y la sintomatología más frecuente fue disuria en 182 pacientes (56%). Las patologías de base más frecuentes fueron hipertensión arterial en 82 pacientes (25,2%), diabetes mellitus en 68 (21%), insuficiencia cardiaca congestiva en 27 (8,3%), enfermedad pulmonar obstructiva crónica (EPOC) en 26 (8%) y enfermedad renal crónica en 21 casos (6,5%). La ITU previa (uno o más episodios en los últimos 3 meses) se presentó en 94 pacientes (28,9%), de los cuales 62 (66%) habían tenido un episodio y 20 (21,3%) 2 episodios. Se documentó consumo previo de antibióticos en 75 pacientes (23%); los más utilizados fueron cefalosporinas de primera generación (46,7%), ampicilina sulbactam (17,3%) y ciprofloxacino (14,7%).
Susceptibilidad antimicrobianaLos resultados de susceptibilidad a los antimicrobianos se presentan en la tabla 1. Se obtuvieron 287 aislamientos de E. coli (88,3%). En estos, los mayores porcentajes de resistencia se presentaron frente a ampicilina (55,1%), trimetoprim-sulfametoxazol (42,9%) y ciprofloxacino (25,1%). En menor proporción se presentó resistencia a amoxicilina/ácido clavulánico y cefoxitina (4,2 y 5,6%, respectivamente). Todos los aislamientos fueron sensibles a carbapenémicos y amikacina. La resistencia a cefalosporinas de tercera y cuarta generación varió entre el 3,4 y el 6,3%. La confirmación fenotípica para BLEE fue positiva en 9 aislamientos (3,1%); estos presentaron además resistencia a ciprofloxacino (79%), a gentamicina (42%), a trimetoprim-sulfametoxazol y a piperacilina tazobactam (28% para ambos casos).
Perfiles de susceptibilidad en aislamientos de Escherichia coli y Klebsiella spp
| Antiobiótico | E. coli (n=287) | Klebsiella spp. (n=29) | ||||
| %R | %I | %S | %R | %I | %S | |
| Amikacina | 0 | 0 | 100 | 0 | 10,3 | 89,7 |
| Amoxicilina clavulánico | 5,6 | 16 | 78,4 | 10,3 | 13,8 | 75,9 |
| Ampicilina | 55,1 | 0,3 | 44,6 | 75,9 | 10,3 | 13,8 |
| Ampicilina sulbactam | 11,5 | 8 | 80,5 | 27,6 | 6,9 | 65,5 |
| Cefazolina | 10,1 | 3,5 | 86,4 | 20,7 | 6,9 | 72,4 |
| Cefepima | 3,8 | 0 | 96,2 | 6,9 | 0 | 93,1 |
| Cefotaxima | 3,4 | 2,4 | 94 | 3,4 | 10,3 | 86,2 |
| Ceftazidima | 6,3 | 0 | 93,7 | 17,2 | 0 | 82,8 |
| Cefoxitina | 4,2 | 0,3 | 95,5 | 10,3 | 3,4 | 86,2 |
| Ciprofloxacino | 25,1 | 1,4 | 73,5 | 17,2 | 6,9 | 75,9 |
| Gentamicina | 10,8 | 0 | 89,2 | 20,7 | 0 | 79,3 |
| Ertapenem | 0 | 0 | 100 | 6,9 | 0 | 93,1 |
| Imipenem | 0 | 0 | 100 | 3,4 | 0 | 96,6 |
| Meropenem | 0 | 0 | 100 | 6,9 | 0 | 93,1 |
| Piperacilina tazobactam | 3,8 | 3,5 | 92,7 | 24,1 | 10,3 | 65,5 |
| Trimetoprim sulfametoxazol | 42,9 | 0 | 57,1 | 20,7 | 0 | 79,3 |
R: resistencia; I: intermedia; S: sensibilidad.
Se obtuvieron 29 aislamientos de Klebsiella spp. (8,9%). Los mayores porcentajes de resistencia se presentaron frente a ampicilina (75,9%), ampicilina sulbactam (27,6%), piperacilina tazobactam (24,1%), cefazolina y gentamicina (20,7% para cada uno). Para amoxicilina/clavulánico y cefoxitina, la resistencia fue del 10,3%. La resistencia a cefalosporinas de tercera y cuarta generación osciló entre el 3,4 y el 17,2%. La confirmación fenotípica para BLEE fue positiva en 5 aislamientos (17,2%). Todos los aislamientos fueron resistentes, además, a piperacilina tazobactam, a trimetoprim-sulfametoxazol y a ciprofloxacino. En 3 aislamientos se observó resistencia a carbapenémicos, uno de los cuales solo presentó resistencia a ertapenem. En los 9 aislamientos de P. mirabilis (2,7%), solo se presentó resistencia frente a ampicilina sulbactam (11,1%) y a piperacilina tazobactam (10%). Todos los aislamientos presentaron resistencia a cefazolina (tabla 1).
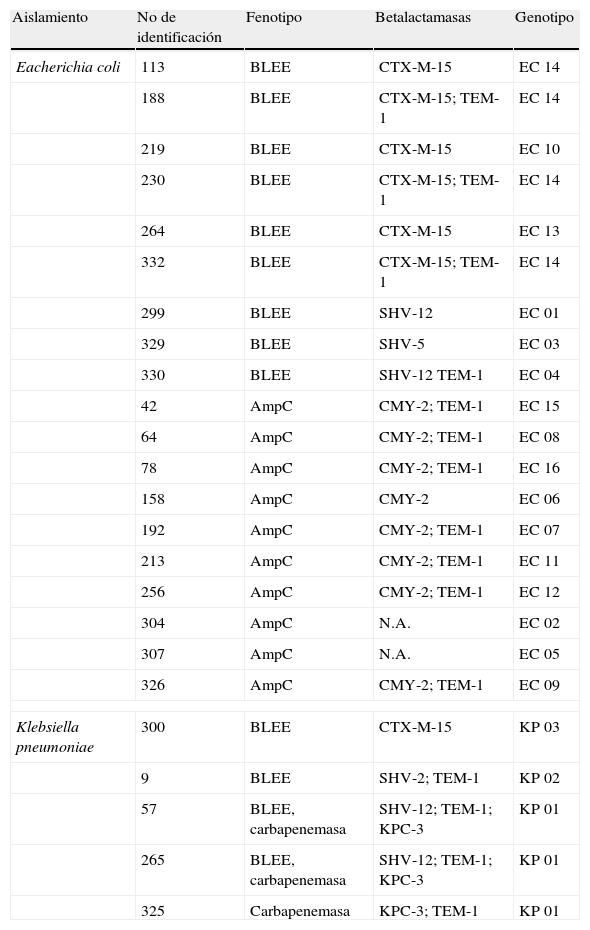
Características genotípicasEl patrón electroforético mostró que en los aislamientos de E. coli se presentaron 16 huellas genómicas, lo que incluyó un grupo clonal conformado por 4 aislamientos y 15 perfiles únicos; para el caso de K. pneumoniae se encontraron 3 huellas genómicas correspondientes a un grupo clonal conformado por 3 aislamientos y 2 perfiles únicos (tabla 2). No se identificó relación epidemiológica entre pacientes con similares mecanismos de resistencia o patrón molecular similar.
Características de los aislamientos resistentes a cefalosporinas de tercera generación
| Aislamiento | No de identificación | Fenotipo | Betalactamasas | Genotipo |
| Eacherichia coli | 113 | BLEE | CTX-M-15 | EC 14 |
| 188 | BLEE | CTX-M-15; TEM-1 | EC 14 | |
| 219 | BLEE | CTX-M-15 | EC 10 | |
| 230 | BLEE | CTX-M-15; TEM-1 | EC 14 | |
| 264 | BLEE | CTX-M-15 | EC 13 | |
| 332 | BLEE | CTX-M-15; TEM-1 | EC 14 | |
| 299 | BLEE | SHV-12 | EC 01 | |
| 329 | BLEE | SHV-5 | EC 03 | |
| 330 | BLEE | SHV-12 TEM-1 | EC 04 | |
| 42 | AmpC | CMY-2; TEM-1 | EC 15 | |
| 64 | AmpC | CMY-2; TEM-1 | EC 08 | |
| 78 | AmpC | CMY-2; TEM-1 | EC 16 | |
| 158 | AmpC | CMY-2 | EC 06 | |
| 192 | AmpC | CMY-2; TEM-1 | EC 07 | |
| 213 | AmpC | CMY-2; TEM-1 | EC 11 | |
| 256 | AmpC | CMY-2; TEM-1 | EC 12 | |
| 304 | AmpC | N.A. | EC 02 | |
| 307 | AmpC | N.A. | EC 05 | |
| 326 | AmpC | CMY-2; TEM-1 | EC 09 | |
| Klebsiella pneumoniae | 300 | BLEE | CTX-M-15 | KP 03 |
| 9 | BLEE | SHV-2; TEM-1 | KP 02 | |
| 57 | BLEE, carbapenemasa | SHV-12; TEM-1; KPC-3 | KP 01 | |
| 265 | BLEE, carbapenemasa | SHV-12; TEM-1; KPC-3 | KP 01 | |
| 325 | Carbapenemasa | KPC-3; TEM-1 | KP 01 | |
BLEE: betalactamasas de espectro extendido; genotipos EC 01-EC 16: genotipos REP-PCR para E. coli; genotipos KP 01-KP 03: genotipos ERIC-PCR para K. pneumoniae; N.A.: no amplificación; en estos aislamientos no se obtuvo amplificación con los iniciadores utilizados para la detección de enzimas AmpC plasmídicas.
De los aislamientos de E. coli resistentes a cefalosporinas de tercera generación, en 9 se detectó BLEE, 6 aislamientos producían CTX-M-15, 2 aislamientos SHV-12 y un aislamiento SHV-5. En 8 aislamientos se confirmó la presencia de la enzima AmpC plasmídica tipo CMY-2, mientras que 2 aislamientos con fenotipo AmpC no amplificaron con los iniciadores utilizados. De los 6 aislamientos productores de CTX-M-15, uno se clasificó como filogrupo A (aislamiento 219) y 5 como filogrupo B2 (aislamientos 113, 188, 230, 264 y 332); 4 de estos pertenecieron al clon ST131 y se clasificaron como genotipo 14. Todos los aislamientos del clon ST131 presentaron el factor de virulencia fimbria (fimG/H) y el gen de resistencia tetA. En 3 aislamientos (aislamientos 113, 188 y 230) se detectó el factor pili (papC) y el gen tetC. El aislamiento 113 fue el único que presentó la proteína específica de uropatógeno (usp) y el gen qnrB, mientras que el aislamiento 332 fue el único en el que se detectó el gen qnrA. En los aislamientos CTX-M-15 no ST131 (aislamientos 219 y 264) se detectaron los genes tetB y sul1, y en el aislamiento 219, los factores de virulencia sfaD/E, usp y fimG/H. Ninguno de los aislamientos de E. coli CTX-M-15 presentó los genes qnrS, sul2 o sul3, ni los factores cnf (factor nefrotóxico necrotizante tipo 1) y hly (hemolisina α).
Para el caso de K. pneumoniae, en 3 aislamientos se detectó carbapenemasa KPC-3 y en 2 aislamientos una BLEE (SHV-1 y CTX-M-15, respectivamente). Dos de las 3 cepas productoras de KPC producían además la BLEE SHV-12 (tabla 2).
Factores asociados con resistencia a cefalosporinas de tercera generaciónA partir de los 24 aislamientos resistentes a cefalosporinas de tercera generación, el análisis bivariado mostró que únicamente la variable 2 comorbilidades o más presentó una asociación estadísticamente significativa (Pr chi2=0,0009) (tabla 3). No se observaron diferencias estadísticamente significativas entre la presencia de factores de comorbilidad específicos entre los grupos de pacientes sensibles o resistentes a cefalosporinas de tercera generación.
Análisis bivariado con relación a la presencia de resistencia a cefalosporinas de tercera generación
| Variable | Resistentes (n=24) (%) | Sensibles (n=301) (%) | Valor p (chi2) |
| Edad | 57,5a | 48,7a | 0,07 |
| Género femenino | 18 (75) | 253 (84) | 0,25 |
| Disuria | 13 (54) | 171 (57) | 0,67 |
| Polaquiuria | 7 (29) | 122 (40) | 0,28 |
| Orina fétida | 9 (37) | 78 (26) | 0,20 |
| Turbidez | 9 (37) | 73 (24) | 0,14 |
| Hematuria | 2 (8) | 37 (12) | 0,59 |
| Embarazo | 5 (21) | 71 (23) | 0,51 |
| ITU previa | 9 (37) | 91 (30) | 0,80 |
| AB previo | 8 (33) | 90 (30) | 0,72 |
| Dos comorbilidades o más | 16 (67) | 99 (33) | 0,0009b |
ITU: infección del tracto urinario; AB: antibiótico.
El modelo multivariado de regresión logística con stepwise sacó a todas las variables, con excepción de 2 comorbilidades o más, con un OR de 4,7 y un valor de p de 0,002. El área bajo la curva ROC de este modelo fue de 0,68.
DiscusiónTradicionalmente, la presencia de BLEE se ha relacionado con infecciones hospitalarias. Sin embargo, han empezado a emerger aislamientos de E. coli productores de CTX-M-15 como un importante agente causal de infecciones de inicio comunitario, principalmente ITU23. En Colombia, en el año 2010 se describió por primera vez aislamientos de E. coli productores de CTX-M-15 pertenecientes al clon ST1318. En este trabajo se destaca la emergencia de aislamientos de enterobacterias resistentes a cefalosporinas de tercera generación e incluso a carbapenémicos causantes de infecciones de inicio en la comunidad. Adicionalmente, se destaca la presencia de aislamientos de E. coli productores de CTX-M-15 pertenecientes al grupo filogenético B2 (clon pandémico ST131), los cuales provenían de 2 ciudades de Colombia, lo que confirma la circulación y la diseminación de este clon. Si bien en este trabajo solamente se analizaron los factores de virulencia más representativos de E. coli, la frecuencia encontrada de los mismos concuerda con lo informado en la literatura, donde se han descrito como factores importantes en aislamientos extraintestinales y en el clon ST13124.
Otro aspecto relevante en este estudio es la detección de aislamientos de K. pneumoniae productores de KPC-3 en infecciones de pacientes que se encontraban fuera del ambiente hospitalario. La rápida diseminación de K. pneumoniae productora de carbapenemasa tipo KPC se ha convertido en un problema global, y las variantes KPC-2 y KPC-3 son las más reportadas a nivel mundial25. En Colombia, en el año 2006 se describió por primera vez la presencia de KPC-2 en pacientes hospitalizados, y en 2008 el primer brote por KPC-326. A partir de este momento se han informado brotes en hospitales de distintas ciudades del país por K. pneumoniae KPC, pero no existen datos sobre este fenómeno en la comunidad. En este estudio, los 3 aislamientos KPC correspondieron a mujeres de edades avanzadas (72, 77, 91 años), con patologías de base (diabetes mellitus, EPOC, enfermedad renal crónica y cirrosis, entre otras) y sin antecedentes de ITU previa ni de hospitalización un mes antes de su ingreso. Aunque la evidencia en la literatura de este fenómeno en la comunidad es limitada, las características de los pacientes son similares con lo informado en un estudio previo27. Dentro de las explicaciones a este fenómeno, algunos autores lo correlacionan con estancias hospitalarias previas prolongadas27 y factores como la presencia de familiares hospitalizados28.
Es importante resaltar que en Colombia este es el primer reporte de enzimas CMY-2 detectadas en E. coli y asociadas a infecciones de origen comunitario. Según la tipificación, los aislamientos portadores de CMY-2 no presentaron relación clonal, lo que sugiere una ocurrencia esporádica. Sin embargo, estos aislamientos pueden estar asociados con brotes, y no solo han sido aislados en muestras de origen humano, sino también en animales de granja y domésticos29. Los aislamientos de E. coli con fenotipo AmpC y en los cuales no se obtuvo amplificación con los iniciadores para AmpC plasmídicas son probablemente aislamientos con hiperexpresión de AmpC cromosómica.
Diversos autores han publicado como factores de riesgo relacionados con infecciones comunitarias por BLEE el sexo masculino, ser mayor de 65 años, el uso previo de fluoroquinolonas o cefalosporinas de tercera generación, el fallo cardiaco, la diabetes mellitus y la cirrosis hepática, entre otros, además de factores ambientales como el uso de antimicrobianos en alimentos9. En este estudio se observó que la presencia de 2 o más comorbilidades fue el factor asociado a la ITU adquirida en la comunidad.
Para el tratamiento de las infecciones urinarias de la comunidad se han planteado diversos antimicrobianos: fluoroquinolonas (ciprofloxacino), cefalosporinas de tercera generación (ceftriaxona) y, en infecciones no complicadas, el uso de trimetoprim-sulfametoxazol, basándose en las recomendaciones publicadas por diversas sociedades científicas como la Sociedad Americana de Enfermedades Infecciosas (IDSA) o la guía NICE de Gran Bretaña30,31. Sin embargo, este estudio muestra que el uso de fluoroquinolonas y sulfas en el medio local no es eficaz, pues se presentan tasas elevadas de resistencia frente a estas moléculas.
Para pacientes con 2 o más comorbilidades que requieran manejo en cuidado intensivo sería prudente el uso de un antimicrobiano que cubra enterobacterias productoras de BLEE. Sin embargo, es importante señalar que la infrecuente identificación del uso previo de antibióticos puede ser un sesgo de información de este estudio y limita la posibilidad de identificar este antecedente como un factor de riesgo de resistencia a cefalosporinas de tercera generación. Otra limitación que se debe señalar es que solo identificaron pacientes que requirieran hospitalización para el tratamiento de la ITU, lo cual pudo sesgar el factor de riesgo de la resistencia a las cefalosporinas.
Los datos del presente estudio muestran la preocupante emergencia de perfiles de resistencia a cefalosporinas de tercera generación en enterobacterias causantes de infecciones de inicio comunitario, y se evidencia la necesidad de controlar la diseminación de estos uropatógenos, así como determinar el impacto clínico y terapéutico de este problema de salud pública emergente a nivel no solo regional, sino global.
FinanciaciónEste estudio fue financiado por un Grant de investigación de la Asociación Colombiana de Infectología y Laboratorios Merck Sharp&dome y convocatoria para apoyo de la investigación Facultad de Medicina Universidad Nacional de Colombia.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.
El Grupo para el control de la Resistencia Bacteriana (GREBO) está formado por: Nubia Escobar, Hospital Kennedy; Carlos Álvarez, Hospital San Ignacio; Claudia Clavijo, Hospital San Rafael; Claudia Pilar Botero, Clínica La Presentación; William Buitrago, Clínica Corpas; Jorge Cortes, Clínica Colombia; José Yesid Rodríguez, Hospital Rosario Pumarejo; German Esparza, Hospital Santa Clara.
El listado de integrantes del GREBO se detalla en el Anexo 1.