El objetivo principal de esta revisión sistemática es evaluar la evidencia científica disponible respecto al PGT-A no invasivo en la detección de ADN en el medio de cultivo embrionario (SCM), la amplificación de cf-DNA y las tasas de concordancia respecto al PGT-A convencional.
MetodologíaSe realizó una revisión exhaustiva a través de bases de datos, en búsqueda de artículos originales experimentales de un periodo desde su inicio hasta marzo 2020, mediante el uso de una combinación de las siguientes palabras clave: líquido blastocele, blastoscentesis, cf-DNA embrionaria, aneuplodía embrionaria, preimplantación, biopsia embrionaria preimplantatoria, PGT no invasivo, diagnóstico preimplantacional, medio cultivo embrionario y biopsia trofoectodermo.
ResultadosEncontramos 26 estudios para el análisis cualitativo y 15 para el cuantitativo. El cf-DNA fue detectado en un 100% de los estudios con tratamientos de FIV/ICSI con una tasa de 44,4-100% de detección en las muestras analizadas. La concentración promedio fue de 37,42 ng/μL. La concordancia entre cf-DNA y la biopsia de TE/WE osciló entre el 54,9-100%, (promedio de 78,32% ± 14,55). Encontramos discrepancias entre los métodos de niPGT-A en cuanto a: el volumen de incubación, la temperatura de almacenamiento, las edades de las mujeres que donaron los embriones, la calidad embrionaria, el uso de control negativo para la detección de cf-DNA, el tiempo de recolección de SCM, y los sistemas de análisis genético.
ConclusionesExisten medidas para optimizar el proceso de niPGT-A según la evidencia científica actual; sin embargo, aún persisten incógnitas pendientes de estudio para poder utilizar el niPGT-A en la práctica rutinaria de TRA.
The primary aim of this systematic review is to evaluate the available current scientific evidence on non-invasive PGT-A (niPGT-A) in regards to cell free DNA (cf-DNA) detection in spent culture media (SCM), cf-DNA amplification, and concordance rates against conventional PGT-A.
Search MethodsWe performed an exhaustive systematic search through major databases for original experimental research, from a period of conception to March 2020, using a combination of the following search words: blastocele/blastoceol fluid, blastoscentesis, cell-free DNA embryo, embryo aneuploidy preimplantation, embryo biopsy preimplantation, noninvasive pgt, preimplantation diagnosis, spent embryo culture media, and trophoectoderm (TE) biopsy.
ResultsOur search yielded 26 studies for a qualitative and 15 for quantitative analysis. Our main results report that cf-DNA is detectable in SCM in 100% of ART (IVF/ICSI) treatments at a rate of 44.4-100% per sample. The average concentration of cf-DNA is 37.42 ng/μL. General concordance rates between cf-DNA and TE biopsy range from 54.9 to 100% (average 78.32% ± 14.55). We found important discrepancies between niPGT-A methodologies carried out by different ART centers, such as culture volume, SCM storage temperature, women embryo donors' age, embryo quality, use of negative/sham controls, time of SCM collection, and genetic analysis platforms/systems.
ConclusionsThere are many ways in which niPGT-A may be optimized, according to current scientific evidence. However, there are many important questions that remain unanswered in order to standardize niPGT-A as routine practice in ART.
Uso del diagnóstico preimplantacional en la reproducción asistida
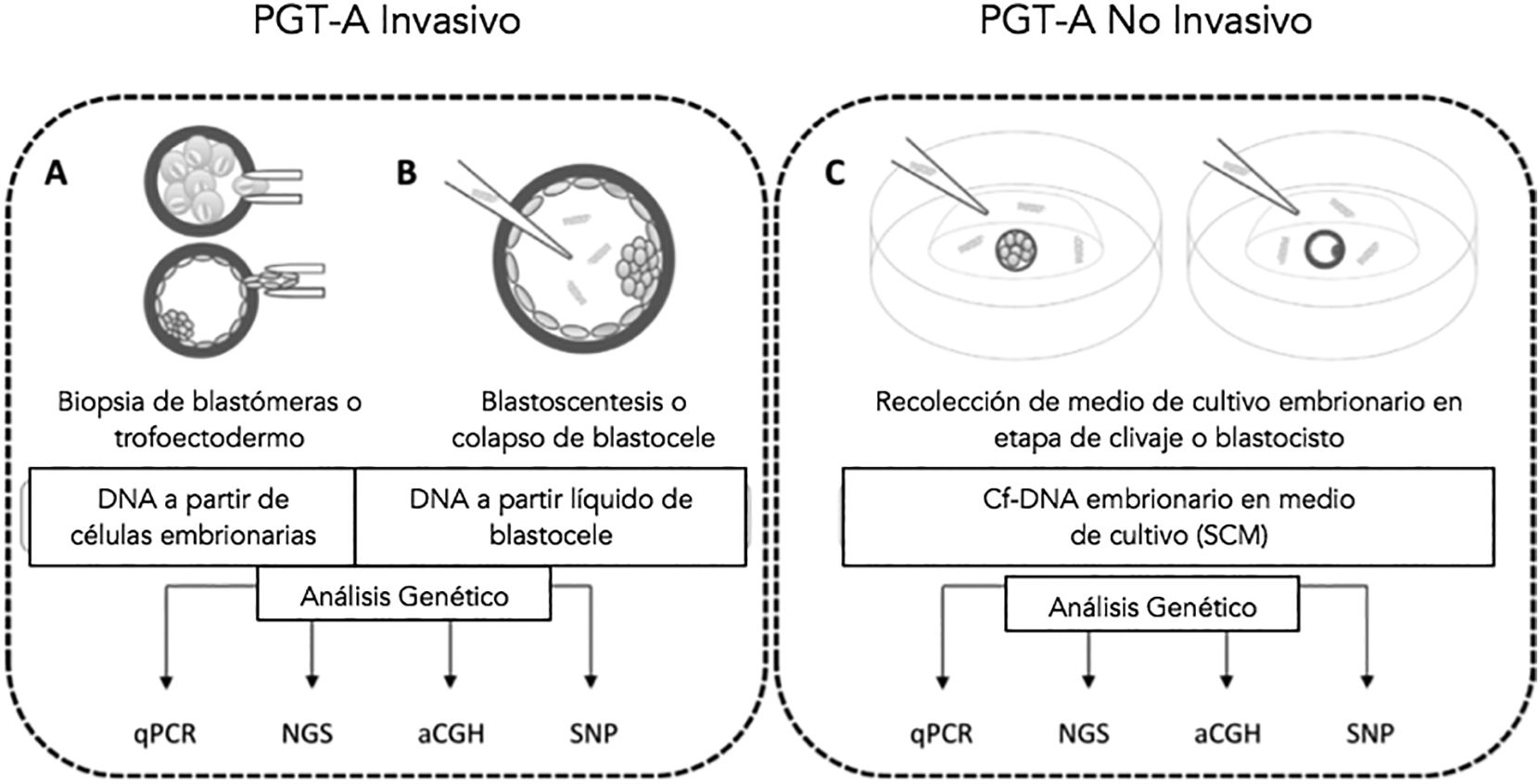
El objetivo principal del diagnóstico genético preimplantacional (PGT) es seleccionar un embrión para ser transferido con mayores posibilidades de progresar a un recién nacido (RN) sano1–3. Actualmente, es un método de rutina en muchas clínicas de reproducción asistida (De Rycke et al., 2015). Entre los principales factores genéticos responsables del fallo de progresión de un embrión, están las aneuploidías, tanto en embarazos espontáneos, como en aquellos obtenidos por las técnicas de reproducción asistida (TRA) (Fragouli et al., 2019). El genotipo tiene un impacto modesto en el fenotipo embrionario, por lo que embriones de «buena calidad» (seleccionados por morfología) pueden no implantarse tras la transferencia embrionaria o presentar abortos tempranos (Fragouli et al., 2019). Además, los embriones con aneuploidías cromosómicas se consideran una causa importante del fallo de la implantación y de los resultados adversos en el proceso de fertilización in vitro (FIV), especialmente en las mujeres de edad avanzada (> 38 años) (Tsuiko et al., 2020). El PGT se utiliza para diagnosticar genéticamente embriones obtenidos por FIV, evitar la transmisión y/o detectar defectos genéticos monogénicos (PGT-M), alteraciones cromosómicas estructurales (PGT-SR) y aneuploidías (PGT-A) (Brouillet et al., 2020; Leaver and Wells, 2020). Para realizar los diversos tipos de PGT es necesario obtener el material genético, esto es, ADN del embrión mediante 2 procedimientos: invasivos (iPGT) y recientemente, no invasivos (niPGT) (Brouillet et al., 2020). Un resumen de las técnicas de PGT-A se ofrece en la figura 1.
Métodos invasivos y no invasivos utilizados en diagnóstico de PGT-A.A) En los métodos de PGT invasivos, encontramos la biopsia de blastómera, ya sea en la etapa de clivaje o al momento de blastocisto, utilizando células del TE. B) Se observa la blastoscentesis para el análisis de líquido del blastocele, el cual se forma durante la etapa de expansión del blastoscisto (D5 de desarrollo embrionario). C) Encontramos la aspiración de SCM de un embrión cultivado, en búsqueda de cf-DNA, independiente de la etapa de desarrollo en la que se pueda encontrar ese embrión. Tanto las blastómeras, como el líquido de blastocisto y el SCM, se pueden analizar por medio de qPCR, aCGH, polimorfismos de nucleótidos puntuales, o el más frecuentemente utilizado, NGS. Adaptada de Brouillet et al.
El iPGT se caracteriza por la biopsia embrionaria, ya sea de blastómera (una o 2 blastómeras en etapa de clivaje, día 3) o de células del trofoectodermo (TE) (5-10 células en etapa de blastocisto, día 5/6) (Brouillet et al., 2020; Leaver and Wells, 2020).
1. Biopsia de blastómeras: para el análisis de la biopsia de blastómeras en la etapa de clivaje (día 3), se realiza una micro-manipulación de embriones, los cuales quizá no alcancen la etapa de blastocisto (sin embargo, esto no implica la imposibilidad de ser transferido). Esta técnica tiene la ventaja de que al hacer la biopsia en día 3 del desarrollo embrionario, existe la posibilidad de realizar la transferencia embrionaria en fresco o por desvitrificación.
2. Biopsia de TE: se realiza en embriones que alcanzan la etapa de blastocisto, lo cual implica un mayor potencial de implantación.
Aunque ambas ténicas se siguen utilizando en la actualidad, es evidente que la biopsia de TE se ha posicionado por encima de la biopsia en etapa de clivaje, ya que se obtiene una mayor cantidad de ADN al conseguir un mayor número de células pero con un menor impacto en el potencial de desarrollo del embrión, reduce el riesgo de fallo de amplificación y ADO (allele dropout) y permite una mayor posibilidad de detección de un porcentaje de mosaicismo (Chang et al., 2016).
Sin embargo, ambas técnicas de biopsia por iPGT conllevan limitaciones importantes: primero, se realiza en un momento específico y requiere de al menos 6 blastómeras o en el caso del blastocisto, una clara diferenciación entre el embrioblasto (masa celular interna) y el trofoblasto. Además, ya que la biopsia se limita a un número definido de células, existe un potencial falso positivo/negativo, en especial en el caso de mosaicismos. Segundo, la pérdida de células previo a la transferencia embrionaria se puede asociar a un efecto deletéreo en cuanto a la implantación/viabilidad, ya sea por fragmentación celular, vitrificación y desvitrificación o biopsia por iPGT (Brouillet et al., 2020; Cohen et al., 2007; Fragouli et al., 2019; Huang et al., 2019). Tercero, esta técnica conlleva un coste elevado y consume una gran cantidad de tiempo. Además, requiere de una formación profesional constante y de material adecuado (Brouillet et al., 2020).
Es importante mencionar que la biopsia de cuerpo polar, originalmente propuesta en 1996 por Verlinsky y Kuliev (Gleicher and Orvieto, 2017), tiene limitaciones importantes, por lo cual actualmente se encuentra en desuso.
Por ejemplo, la ausencia de material genético paterno, el hecho de que requiere una labor intensa, pues conlleva analizar todos los ovocitos maduros obtenidos de los cuales no todos progresarán a embriones maduros viables; por último, tiene una dudosa utilidad diagnóstica, pues los cambios post-meióticos no son valorados. Además, este procedimiento puede aumentar la tasa de fragmentación embrionaria y el arresto del desarrollo (Brouillet et al., 2020).
PGT no invasivo y el análisis genético del SCMEstos 3 motivos, crean la necesidad fundamental de métodos de niPGT, que parten de la detección de ácidos nucleicos celulares libres (cf-DNA) u otros componentes del secretoma (proteínas, interleucinas, micro-RNA) al medio de cultivo utilizado (SCM) por el embrión. Este cf-DNA se ha detectado en fluido del blastocele (BF) y en el SCM. (Brouillet et al., 2020; Leaver and Wells, 2020) El método de blastocentesis fue descrito originalmente en 2013, pero se debe considerar como un método de «mínima invasión», pues conlleva una disrupción de la capa TE por una aguja para acceder al blastocele, en donde es posible aspirar el contenido (blastoscentesis), o bien con láser fenestrar el TE y recuperar fluido del blastocele del medio de cultivo (Brouillet et al., 2020; Palini et al., 2013). Además, se debe realizar durante la expansión del blastocisto, para obtener así una muestra por BF, descartando la posibilidad de obtener BF de blastocistos tempranos o la etapa de clivaje (Brouillet et al., 2020); sin embargo, el BF se puede retirar con otro motivo, como el de proteger al embrión previo al proceso de vitrificación, para evitar la formación de cristales al momento de la congelación (vitrificación) y mejorar el supervivencia embrionaria (Tsuiko et al., 2020). Aunque el embrión previo a la implantación se encuentra rodeado de una membrana glicoproteíca (zona pelúcida), esta tiene un alto grado de permeabilidad con respecto a macromoléculas, lo cual permite el paso libre de ácidos nucleicos del embrión hacia el SCM (Leaver and Wells, 2020).
Por lo tanto, el único método verdaderamente de niPGT es la obtención de cf-DNA a partir del medio de cultivo embrionario (Brouillet et al., 2020). Existe evidencia que demuestra que la combinación de niPGT con BF para el análisis de ADN conlleva una mayor concordancia que el PGT-A de solamente biopsia de TE (Kuznyetsov et al., 2018).
La técnica de niPGT inició en 2016, cuando se analizó ADN para la detección de aneuploidías en el SCM de los blastocistos humanos (Xu et al., 2016). Este contiene ADN nuclear (nDNA) y mitocondrial (mtDNA) (Brouillet et al., 2020). Las tasas de detección de cf-DNA y amplificación son dependientes de la cantidad de medio recogido, la cantidad de cf-DNA detectada, la etapa embrionaria de su desarrollo y el periodo de incubación al momento de la recogida de SCM (Brouillet et al., 2020). Por ejemplo, no se ha detectado cf-DNA en el periodo de cigoto. Asimismo, aunque el cf-DNA se libera durante el periodo embrionario temprano, las cantidades y la calidad de cf-DNA parecen ser mayores en la etapa de blastocisto (≥ D5) que en la de clivaje (Brouillet et al., 2020; Yang et al., 2017). Al parecer, el tiempo óptimo de incubación para la obtención de una cantidad adecuada de cf-DNA y sin degradación de ADN, es de 24-48 h (Hammond et al., 2017; Kuznyetsov et al., 2018).
El tiempo del proceso se considera relativamente corto, < 12 h de la recolección al análisis (Li et al., 2018). El hecho de que la cantidad de ADN aumenta durante el cultivo embrionario, nos sugiere que el material genético detectado en el SCM es de origen embrionario, a pesar de que ciertos medios contengan contaminantes de ADN (Leaver and Wells, 2020). Al parecer, el origen del ADN en el SCM proviene de los fragmentos citoplastmáticos anucleares y no de la apoptosis (Leaver and Wells, 2020).
Además de la técnica de obtención del material genético, el uso de un medio único o secuencial y el día de desarrollo embrionario, existen también otras variables presentes en el análisis de PGT-A por niPGT: el uso de una eclosión asistida, métodos de amplificación, métodos de tamizaje comprensivo cromosómico (CCS), plataformas de secuenciación, análisis bioinformático y estrategias de identificación de contaminación por ADN materno (Belandres et al., 2019).
En cuanto al tipo de medio, tanto en el medio único, como en el secuencial se ha detectado cf-DNA. El medio único tiene la desventaja de una mayor degradación de ADN y por consiguiente, más cf-DNA de mala calidad, mientras que, con el uso de medio secuencial, aunque disminuye el problema de la degradación, se podría asociar a una menor concentración de cf-DNA (Belandres et al., 2019). En referencia al momento de vitrificación y desvitrificación, no hay resultados concluyentes por evaluación sistemática que sea estadísticamente significativa (Belandres et al., 2019; Ho et al., 2018; Kuznyetsov et al., 2018). Se podría intuir que con el uso de la eclosión asistida (assisted hatching), se obtendría una mayor cantidad de cf-DNA en el medio, esta opción se ha descartado recientemente (Yeung et al., 2019).
Métodos de análisis genético de cf-DNA en SCMLos métodos reportados en la detección de cf-DNA en SCM incluyen: 1) cuantificación de ADN por espectrofotometría (NanoDrop, Thermo Fisher Scientific™, Massachusetts, USA) (Li et al., 2018), la cual mide el ADN de doble cadena y otros ácidos nucleicos (ARN, ADN de una cadena y nucleótidos libres, lo cual podría sobreestimar la concentración de cf-DNA) (Brouillet et al., 2020; Garcia-Elias et al., 2017); 2) fluorescencia (Qubit, Fisher Scientific™, Massachusetts, USA) (Vera-Rodriguez et al., 2018), el cual solo mide ADN de doble cadena, pero no discrimina entre las distintas longitudes de moléculas de ADN, lo cual podría cuantificar dímeros cebadores y de adaptación (Brouillet et al., 2020); 3) electroforesis (Bioanalyzer, Agilent Technologies, California, USA) que permite la valoración de la calidad del amplicón y excluye a las pequeñas moléculas. Además, la PCR cuantitativa (qPCR) se considera el abordaje más sensible (Brouillet et al., 2020; Klein, 2002). Sin embargo, la presencia de cf-DNA extraembrionaria puede aumentar de forma engañosa la detección y amplificación de cf-DNA (Brouillet et al., 2020).
Previo al análisis genético, se aumenta la cantidad de ADN por medio de la amplificación genómica (WGA) basado en 3 estrategias: amplificación por desplazamiento múltiple (multiple displacement amplification, MDA), PCR y una combinación de ambos (ciclos de amplificación basados en el bucle y múltiple hibridación, MALBAC, Yikon Genomics). Al parecer, los métodos de WGA basados en PCR reportan la mejor reproducibilidad y precisión para la detección de la variación del número de copias. Por otra parte, MDA y MALBAC pueden detectar de manera precisa las variaciones puntuales de nucleótidos con mayor eficiencia y sensibilidad que el método de WGA basado en PCR. (Belandres et al., 2019; Brouillet et al., 2020) A pesar de que existe un riesgo teórico de error al amplificar el ADN, este riesgo es minimizado al aplicar la técnicas de secuenciación de siguiente generación (NGS, next generation sequencing), por su alta tasa de cobertura de secuencias que reducen la contaminación y errores aleatorizados (Brouillet et al., 2020).
Se ha reportado el análisis de PGT-M, PGT-SR y PGT-A a partir de SCM. (Jiao et al., 2019; Leaver and Wells, 2020) A medida que evoluciona el uso de PGT-A en TRA, también el CCS, en el que se ha progresado de técnicas de hibridación fluorescente in situ (FISH) a otras más sensibles, como qPCR, hibridación genómica comparada (aCGH), microarreglos de polimorfismos de nucleótidos puntuales y NGS. (Belandres et al., 2019) Sin embargo, actualmente el NGS se asocia a una mayor sensibilidad, precisión y es una plataforma más sensible para detectar mosaicos (Fiorentino et al., 2014; Tortoriello et al., 2016).
El análisis mediante WGA y NGS del SCM ha reportado el estado de ploidia exitosamente, aunque el nivel de concordancia fue variado (Leaver and Wells, 2020; Shamonki et al., 2016).
El nivel de concordancia tiene distintas implicaciones: estado de la ploidía (euploide vs aneuploide), concordancia del sexo, números enteros de cromosomas, concordancia de las aberraciones (mosaicos y aneuploidías segmentarias), y concordancia de un cromosoma entero (Belandres et al., 2019).
Al parecer, los resultados genéticos obtenidos de iPGT (biopsia TE) concordaban más con SCM que con BF. (Capalbo et al., 2018) Sin embargo, el éxito de obtener ADN de SCM, la concentración de ADN y la concordancia con biopsia de TE varían según el año, las técnicas y los materiales empleados (Belandres et al., 2019).
Es importante validar el niPGT a partir de SCM por muchas razones: es un procedimiento seguro, no invasivo, con mayor ventana diagnóstica que algunas técnicas de iPGT, apto para la mayoría de embriones, con buen coste-efectividad, utiliza poca manipulación y además no requiere habilidades adicionales por parte del embriólogo (Belandres et al., 2019; Brouillet et al., 2020).
Debido a esto, la presente revisión sistematizada de la evidencia de niPGT tiene como objetivo evaluar: 1) el éxito o tasa de detección de cf-DNA en SCM, 2) la amplificación de cf-DNA en SCM y sus técnicas y 3) el nivel de concordancia con PGT-A por biopsia de blastómeras.
MetodologíaEste trabajo es retrospectivo, descriptivo, llevado a cabo mediante una revisión sistemática y realizado según las recomendaciones publicadas en Preffered Reporting of Systematic Reviews and Meta-Analyses (PRISMA, 2009). (Moher et al., 2009) Por este motivo, no fue necesario conseguir ninguna aprobación por el comité de bioética en investigación.
Estrategia de búsquedaSe realizó una búsqueda electrónica de la bibliografía publicada desde el inicio (concepción) hasta marzo de 2020 en inglés y en español, en las bases de datos de Medline a través de PubMed, EMBASE, ClinicalTrials.gov y Cochrane. Utilizamos distintas combinaciones de los siguientes términos y palabras (Medical Subject Headings, MeSH): preimplantation genetic testing, preimplantation genetic diagnosis, preimplantation genetic screening, noninvasive pgt, embryo biopsy, blastocentesis, blastocoel fluid, trophectoderm biopsy, cell-free DNA, spent embryo culture media, aneuploidy.
Selección de estudiosEn este estudio incluimos ensayos clínicos, revisiones sistemáticas, ensayos controlados aleatorizados, estudios de casos y controles, reporte de casos, estudios de tipo cohorte, comparativos retrospectivos y transversales que cumplieran los siguientes criterios: 1) ensayos que incluyen detección de ADN obtenido de embriones de manera no invasiva (SCM), 2) detección y amplificación del ADN obtenida de embriones de manera no invasiva con fines de estudio para valoración de aneuplodías y 3) medición de concordancia entre los resultados del diagnóstico pre-implantacional de ADN obtenida de embriones manera invasiva (TE/embrión completo, WE) y no invasiva (SCM). Se hizo uso de aquellos estudios en inglés o en español, en humanos, artículos indexados en las 4 bases de datos principales utilizadas que contaran con texto publicado en revistas (ya sea texto completo o presentación en congresos). Todos los artículos fueron evaluados por un revisor y supervisados por una persona más, de manera independiente. La elegibilidad de los estudios inicialmente fue valorada por el título del artículo, posteriormente mediante la lectura del abstract; finalmente, el texto completo de los artículos potencialmente relevantes fue evaluado para su inclusión. Cualquier duda de inclusión fue resuelta a través del supervisor de la búsqueda.
Extracción de informaciónSe retiró toda la información duplicada, se realizó una revisión por parte de 2 revisores (autores del estudio) en 2 pasos: la valoración del título del estudio, abstract, y la revisión completa del texto del estudio en búsqueda de información elegible para esta revisión sistemática. Las discrepancias y diferencias de opinión en cuanto a los estudios incluidos se resolvieron por medio de consenso. La información extraída incluye los siguientes dominios: autores del estudio, año de la publicación, edad promedio de la población, método de fecundación de FIV, muestras recolectadas, volumen de incubación, temperatura de almacenamiento de muestra, número de embriones, uso de embriones en fresco/criopreservación, calidad embrionaria utilizada, día del estado de desarrollo embrionario, uso de los controles negativos en la detección de cf-DNA, detección de cf-DNA, protocolo diagnóstico preimplantacional utilizado, tiempo de secreción de cf-DNA en SCM, duración de incubación embrionaria en SCM, método de amplificación (kit comercial), análisis genético de ADN, sistema/plataforma de secuenciación utilizada, concentración promedio de cf-DNA, referencia utilizada para evaluar la concordancia y la tasa de concordancia general reportada en porcentaje. Toda esta información fue extraída de manera independiente y luego evaluada por el supervisor del estudio (JM Moreno-Garcia). Cualquier desacuerdo fue resuelto por medio de discusión.
Valoración del riesgo de sesgo (Risk-of-Bias)La calidad de los estudios obtenidos se valoró según Cochrane Handbook of Systematic Reviews of Interventions, 6.0 (Julio, 2019). Los riesgos valorados fueron: sesgo de selección (generación en secuencia, secuencia de alocación y ocultar), sesgo de desempeño (cegar participantes y personal), sesgo de detección (cegar la valoración de los resultados), sesgo de atrición (información de los resultados incompleta) y sesgo de reporte (reporte selectivo de los resultados). El juicio de los autores se categorizó como «bajo riesgo», «alto riesgo» o «riesgo incierto» de sesgo. Para la evaluación de la calidad de los estudios se utilizó la misma fuente mencionada.
Síntesis de información y análisis estadísticoLos resultados agrupados presentados en este trabajo de revisión sistemática en los métodos de niPGT fueron la cantidad de ADN obtenida, la amplificación obtenida y el nivel de concordancia con los métodos de iPGT. Toda la información obtenida fue evaluada por un software estadístico R: A Language and Environment for Statistical Computing (R Foundation for Statistical Computing, Vienna, Austria, 2019) y su package «netmeta» para el análisis de las medidas de tendencia central. No se utilizó el análisis inferencial sobre los datos obtenidos.
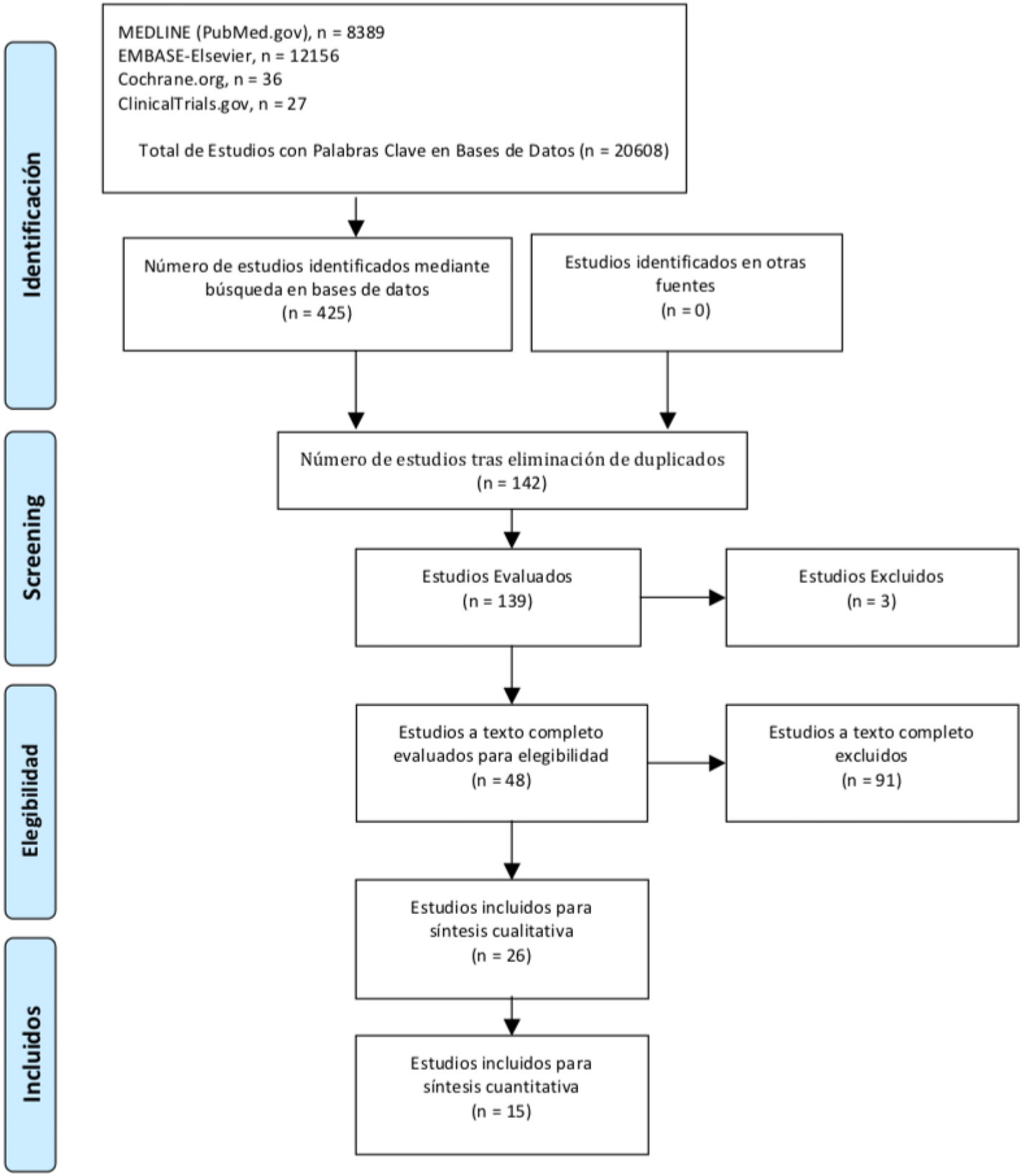
ResultadosResultados de búsqueda sistematizadaLa combinación de términos utilizados en las distintas bases de datos arrojó un total de 20.608 resultados provenientes de 4 de las bases de datos más importantes. En total, se analizaron 425 artículos de investigación experimental que cumplían con los criterios de búsqueda. Se eliminaron los duplicados entre bases de datos y estos fueron valorados para cumplir con los requisitos de contestar las preguntas de investigación formuladas. Para la evaluación de las características cualitativas, se obtuvieron 26 estudios en cuanto a la detección o no de cf-DNA en SCM y se obtuvieron 15 artículos para las características cuantitativas de la amplificación de cf-DNA y la concordancia de detección de euploidía/aneuploidía por medio de niPGT-A vs iPGT-A. El periodo de los estudios recolectados fue de 2013-2019. El diagrama de flujo PRISMA de la estrategia de búsqueda del estudio se presenta en la figura 2.
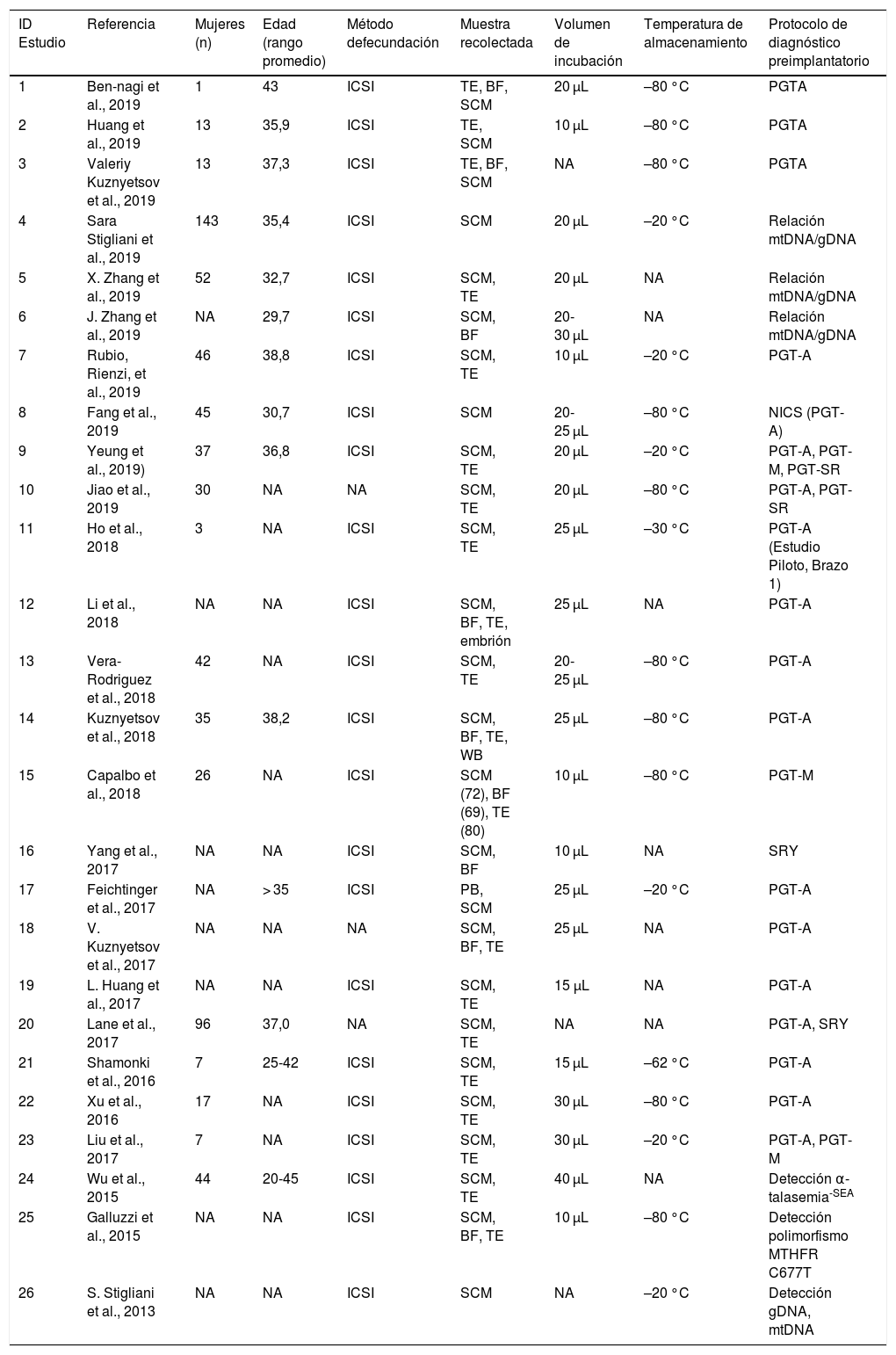
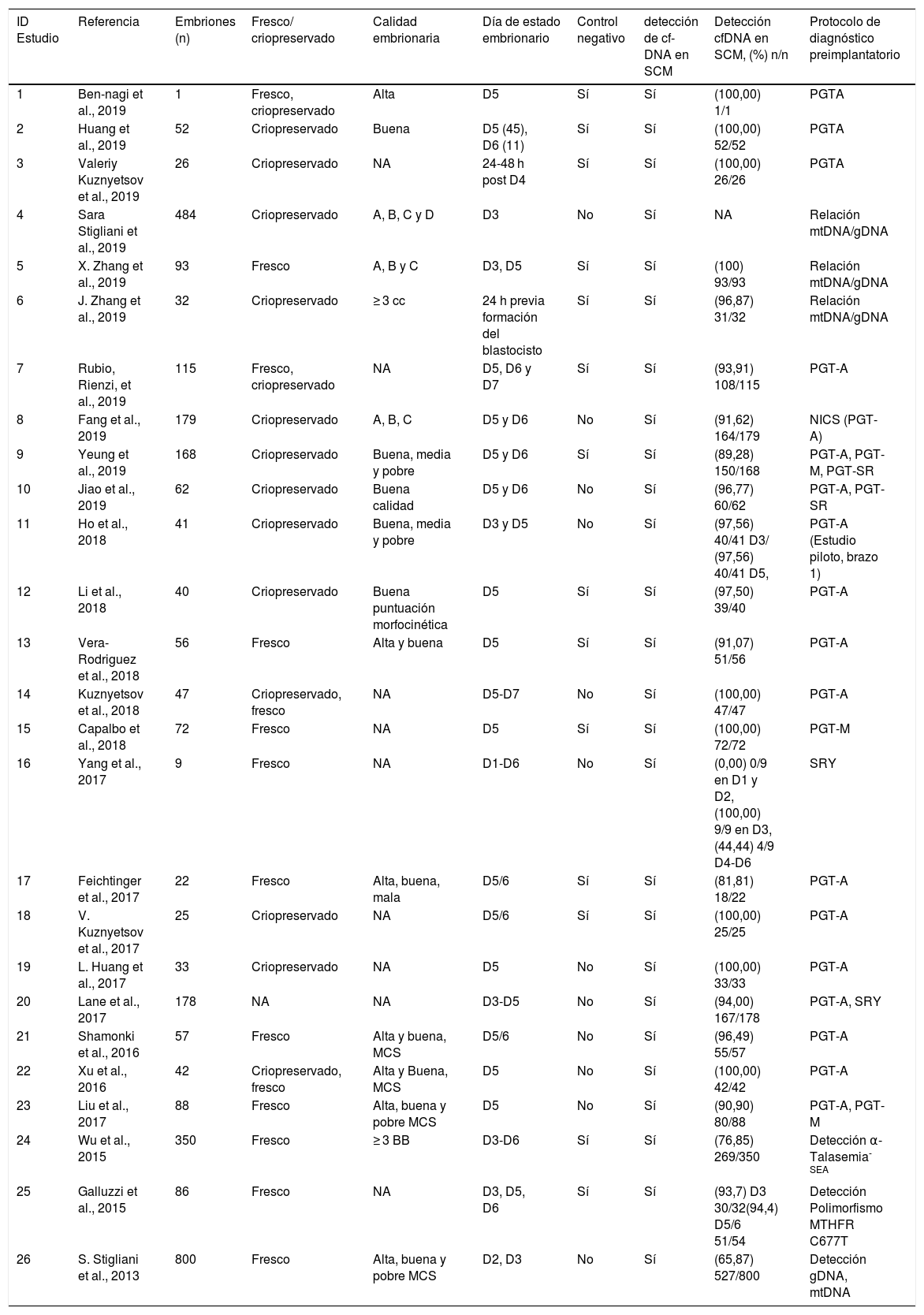
Detección de cf-DNA en SCMUno de los principales aspectos negativos del carácter invasivo del PGT convencional es la obtención de material genético representativo del genotipo embrionario por medio de la biopsia de trofoectodermo (TE, con fines de diagnóstico clínico) y el embrión completo (WE, con fines de investigación). Aunque existen métodos no invasivos como el perfil metabólico embrionario, morfología embrionaria y el morfocinética embrionaria que podrían aportar información sobre la ploidía embrionaria, no se valora el material genético directamente. Dentro de los resultados de la revisión sistematizada, encontramos 26 estudios que analizaron el material genético en el SCM. El SCM puede ser evaluado con fines de valorar la relación mtDNA/gDNA (4/26, 15,38%), selección de sexo (SRY) y polimorfismos (3/26, 11,53%), y por supuesto, PGT (PGT-A, PGT-M y PGT-SR) (19/26, 73,03%). En estos estudios, la población de mujeres que recibieron tratamientos de TRA, oscilaba entre 1 a 143 (18/26 estudios, 8/26 no reportan población). Dentro de las edades reportadas de las mujeres de estudio, 11/26 (42,30%) reportan edades promedio de 29,7-43 años, 3/26 reportan rangos de edad de 20 a 45 años y el resto de estudios 12/26 no revelan edades utilizadas. En cuanto a la metodología de fertilización de gametos, 23/26 (88,46%) estudios reportan ICSI, mientras que 3/26 no revelan metodología. La metodología de fecundación es importante, pues evita la contaminación paterna y limita la contaminación por parte de las células del complejo de cumulus oophorus (CCO). (Hammond et al., 2016) En cuanto a la muestra analizada, en 100% (26/26) de los estudios recopilados se evaluó la presencia de cf-DNA en SCM. El volumen del medio en el que se cultivó al embrión oscilaba entre 10-40 μL en 23/26 (88,46%) estudios y el resto (3/26) no reveló el volumen de cultivo. Una vez que el SCM fue recolectado, se almacenó a un temperatura que oscilaba entre –20 °C a –80 °C en 18/26 (69,23%) de los estudios y el resto (8/26) no revelaron la temperatura de almacenamiento. Cabe mencionar que los estudios no mencionaron el tiempo que los embriones estuvieron en almacenamiento a la temperatura reportada, ni el sustento teórico de por qué congelar a –20 °C, –60 °C o –80 °C (ver tabla 1).
Resultados de las condiciones generales para detección de cf-DNA en SCM (n = 26).
| ID Estudio | Referencia | Mujeres (n) | Edad (rango promedio) | Método defecundación | Muestra recolectada | Volumen de incubación | Temperatura de almacenamiento | Protocolo de diagnóstico preimplantatorio |
|---|---|---|---|---|---|---|---|---|
| 1 | Ben-nagi et al., 2019 | 1 | 43 | ICSI | TE, BF, SCM | 20 μL | –80 °C | PGTA |
| 2 | Huang et al., 2019 | 13 | 35,9 | ICSI | TE, SCM | 10 μL | –80 °C | PGTA |
| 3 | Valeriy Kuznyetsov et al., 2019 | 13 | 37,3 | ICSI | TE, BF, SCM | NA | –80 °C | PGTA |
| 4 | Sara Stigliani et al., 2019 | 143 | 35,4 | ICSI | SCM | 20 μL | –20 °C | Relación mtDNA/gDNA |
| 5 | X. Zhang et al., 2019 | 52 | 32,7 | ICSI | SCM, TE | 20 μL | NA | Relación mtDNA/gDNA |
| 6 | J. Zhang et al., 2019 | NA | 29,7 | ICSI | SCM, BF | 20-30 μL | NA | Relación mtDNA/gDNA |
| 7 | Rubio, Rienzi, et al., 2019 | 46 | 38,8 | ICSI | SCM, TE | 10 μL | –20 °C | PGT-A |
| 8 | Fang et al., 2019 | 45 | 30,7 | ICSI | SCM | 20-25 μL | –80 °C | NICS (PGT-A) |
| 9 | Yeung et al., 2019) | 37 | 36,8 | ICSI | SCM, TE | 20 μL | –20 °C | PGT-A, PGT-M, PGT-SR |
| 10 | Jiao et al., 2019 | 30 | NA | NA | SCM, TE | 20 μL | –80 °C | PGT-A, PGT-SR |
| 11 | Ho et al., 2018 | 3 | NA | ICSI | SCM, TE | 25 μL | –30 °C | PGT-A (Estudio Piloto, Brazo 1) |
| 12 | Li et al., 2018 | NA | NA | ICSI | SCM, BF, TE, embrión | 25 μL | NA | PGT-A |
| 13 | Vera-Rodriguez et al., 2018 | 42 | NA | ICSI | SCM, TE | 20-25 μL | –80 °C | PGT-A |
| 14 | Kuznyetsov et al., 2018 | 35 | 38,2 | ICSI | SCM, BF, TE, WB | 25 μL | –80 °C | PGT-A |
| 15 | Capalbo et al., 2018 | 26 | NA | ICSI | SCM (72), BF (69), TE (80) | 10 μL | –80 °C | PGT-M |
| 16 | Yang et al., 2017 | NA | NA | ICSI | SCM, BF | 10 μL | NA | SRY |
| 17 | Feichtinger et al., 2017 | NA | > 35 | ICSI | PB, SCM | 25 μL | –20 °C | PGT-A |
| 18 | V. Kuznyetsov et al., 2017 | NA | NA | NA | SCM, BF, TE | 25 μL | NA | PGT-A |
| 19 | L. Huang et al., 2017 | NA | NA | ICSI | SCM, TE | 15 μL | NA | PGT-A |
| 20 | Lane et al., 2017 | 96 | 37,0 | NA | SCM, TE | NA | NA | PGT-A, SRY |
| 21 | Shamonki et al., 2016 | 7 | 25-42 | ICSI | SCM, TE | 15 μL | –62 °C | PGT-A |
| 22 | Xu et al., 2016 | 17 | NA | ICSI | SCM, TE | 30 μL | –80 °C | PGT-A |
| 23 | Liu et al., 2017 | 7 | NA | ICSI | SCM, TE | 30 μL | –20 °C | PGT-A, PGT-M |
| 24 | Wu et al., 2015 | 44 | 20-45 | ICSI | SCM, TE | 40 μL | NA | Detección α-talasemia-SEA |
| 25 | Galluzzi et al., 2015 | NA | NA | ICSI | SCM, BF, TE | 10 μL | –80 °C | Detección polimorfismo MTHFR C677T |
| 26 | S. Stigliani et al., 2013 | NA | NA | ICSI | SCM | NA | –20 °C | Detección gDNA, mtDNA |
En cuanto a la detección de cf-DNA en el SCM, el 100% (26/26) de los estudios reportaron el número de embriones utilizados para evaluar la presencia de cf-DNA en SCM, el cual oscilaba entre 1 y 800 (promedio de 121,46) embriones. De estos estudios, 11/26 (42,30%) eran criopreservados, 10/26 (28,46%) eran en fresco, (15,38%) utilizaron en fresco y criopreservados, y 1/26 no lo reportó. En cuanto al día de recolección, muestras de SCM se analizaron de manera heterogénea en distintos días desde D1 hasta D7 en 26/26 (100%) de los estudios. Los SCM se compararon con un control negativo en 14/26 (53,84%). En cuanto a la detección de cf-DNA en SCM, es reportada de manera heterogénea en los estudios, pero 26/26 (100%) reportaron su detección (tabla 2).
Detección de cf-DNA en SCM.
| ID Estudio | Referencia | Embriones (n) | Fresco/ criopreservado | Calidad embrionaria | Día de estado embrionario | Control negativo | detección de cf-DNA en SCM | Detección cfDNA en SCM, (%) n/n | Protocolo de diagnóstico preimplantatorio |
|---|---|---|---|---|---|---|---|---|---|
| 1 | Ben-nagi et al., 2019 | 1 | Fresco, criopreservado | Alta | D5 | Sí | Sí | (100,00) 1/1 | PGTA |
| 2 | Huang et al., 2019 | 52 | Criopreservado | Buena | D5 (45), D6 (11) | Sí | Sí | (100,00) 52/52 | PGTA |
| 3 | Valeriy Kuznyetsov et al., 2019 | 26 | Criopreservado | NA | 24-48 h post D4 | Sí | Sí | (100,00) 26/26 | PGTA |
| 4 | Sara Stigliani et al., 2019 | 484 | Criopreservado | A, B, C y D | D3 | No | Sí | NA | Relación mtDNA/gDNA |
| 5 | X. Zhang et al., 2019 | 93 | Fresco | A, B y C | D3, D5 | Sí | Sí | (100) 93/93 | Relación mtDNA/gDNA |
| 6 | J. Zhang et al., 2019 | 32 | Criopreservado | ≥ 3 cc | 24 h previa formación del blastocisto | Sí | Sí | (96,87) 31/32 | Relación mtDNA/gDNA |
| 7 | Rubio, Rienzi, et al., 2019 | 115 | Fresco, criopreservado | NA | D5, D6 y D7 | Sí | Sí | (93,91) 108/115 | PGT-A |
| 8 | Fang et al., 2019 | 179 | Criopreservado | A, B, C | D5 y D6 | No | Sí | (91,62) 164/179 | NICS (PGT-A) |
| 9 | Yeung et al., 2019 | 168 | Criopreservado | Buena, media y pobre | D5 y D6 | Sí | Sí | (89,28) 150/168 | PGT-A, PGT-M, PGT-SR |
| 10 | Jiao et al., 2019 | 62 | Criopreservado | Buena calidad | D5 y D6 | No | Sí | (96,77) 60/62 | PGT-A, PGT-SR |
| 11 | Ho et al., 2018 | 41 | Criopreservado | Buena, media y pobre | D3 y D5 | No | Sí | (97,56) 40/41 D3/ (97,56) 40/41 D5, | PGT-A (Estudio piloto, brazo 1) |
| 12 | Li et al., 2018 | 40 | Criopreservado | Buena puntuación morfocinética | D5 | Sí | Sí | (97,50) 39/40 | PGT-A |
| 13 | Vera-Rodriguez et al., 2018 | 56 | Fresco | Alta y buena | D5 | Sí | Sí | (91,07) 51/56 | PGT-A |
| 14 | Kuznyetsov et al., 2018 | 47 | Criopreservado, fresco | NA | D5-D7 | No | Sí | (100,00) 47/47 | PGT-A |
| 15 | Capalbo et al., 2018 | 72 | Fresco | NA | D5 | Sí | Sí | (100,00) 72/72 | PGT-M |
| 16 | Yang et al., 2017 | 9 | Fresco | NA | D1-D6 | No | Sí | (0,00) 0/9 en D1 y D2,(100,00) 9/9 en D3, (44,44) 4/9 D4-D6 | SRY |
| 17 | Feichtinger et al., 2017 | 22 | Fresco | Alta, buena, mala | D5/6 | Sí | Sí | (81,81) 18/22 | PGT-A |
| 18 | V. Kuznyetsov et al., 2017 | 25 | Criopreservado | NA | D5/6 | Sí | Sí | (100,00) 25/25 | PGT-A |
| 19 | L. Huang et al., 2017 | 33 | Criopreservado | NA | D5 | No | Sí | (100,00) 33/33 | PGT-A |
| 20 | Lane et al., 2017 | 178 | NA | NA | D3-D5 | No | Sí | (94,00) 167/178 | PGT-A, SRY |
| 21 | Shamonki et al., 2016 | 57 | Fresco | Alta y buena, MCS | D5/6 | No | Sí | (96,49) 55/57 | PGT-A |
| 22 | Xu et al., 2016 | 42 | Criopreservado, fresco | Alta y Buena, MCS | D5 | No | Sí | (100,00) 42/42 | PGT-A |
| 23 | Liu et al., 2017 | 88 | Fresco | Alta, buena y pobre MCS | D5 | No | Sí | (90,90) 80/88 | PGT-A, PGT-M |
| 24 | Wu et al., 2015 | 350 | Fresco | ≥ 3 BB | D3-D6 | Sí | Sí | (76,85) 269/350 | Detección α-Talasemia-SEA |
| 25 | Galluzzi et al., 2015 | 86 | Fresco | NA | D3, D5, D6 | Sí | Sí | (93,7) D3 30/32(94,4) D5/6 51/54 | Detección Polimorfismo MTHFR C677T |
| 26 | S. Stigliani et al., 2013 | 800 | Fresco | Alta, buena y pobre MCS | D2, D3 | No | Sí | (65,87) 527/800 | Detección gDNA, mtDNA |
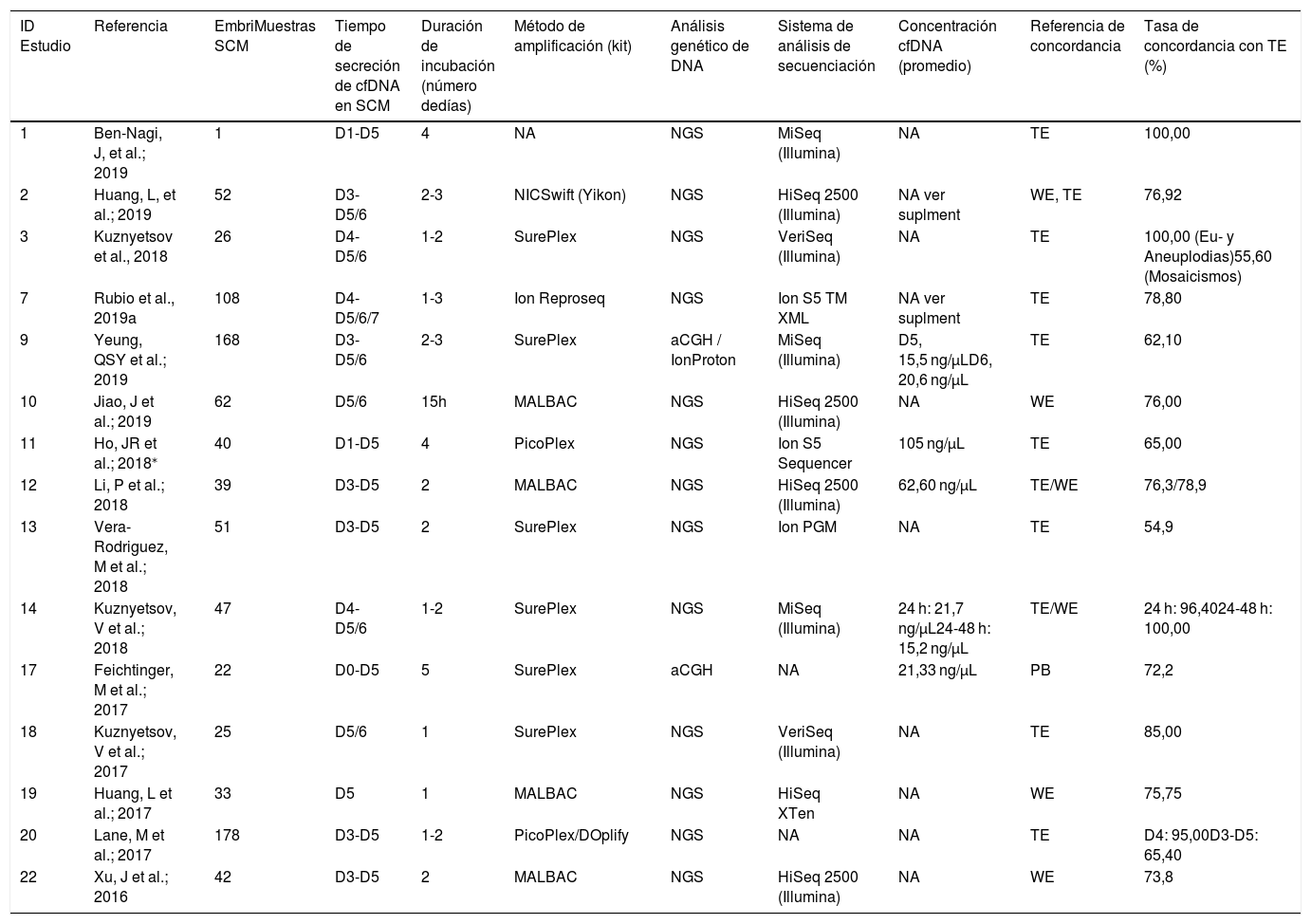
De los 26 estudios que reportaron detección exitosa de cf-DNA en el SCM, utilizamos únicamente aquellos que habían evaluado PGT-A en su análisis genético (n = 15). De los 26 estudios que reportaron detección de cf-DNA en el SCM, un total de 25/26 (96,15%) reportaron el número de muestras de SCM en las que detectaron cf-DNA. Este número oscila entre 0 y 100%. Sin embargo, si se consideran únicamente los estudios en los cuales el SCM se analizó en embriones mayores de D3, el porcentaje de detección de cf-DNA osciló entre 44,44-100% (26/26, 100%). Cabe mencionar que un estudio analizó el SCM de 9 embriones en D1 y D2, de los cuales no se reportó detección de cf-DNA. (Yang et al., 2017) De los estudios en los que se detectó cf-DNA, se analizaron 15 estudios que evaluaron amplificación y concordancia de cf-DNA con el TE o embrión para el análisis de PGT-A. Estos estudios analizaron el SCM de embriones entre D1 a D7, con periodos de incubación entre 15 h y 5 días (15/15, 100%). El kit de amplificación varía entre los estudios que lo reportaron (14/15, 93,33%) entre SurePlex, PicoPlex/DOplify, MALBAC y Ion ReproSeq. El análisis genético de ADN se llevó a cabo por medio de NGS (13/15, 86,66%) y aCGH/Ion Proton (2/15, 13,33%). La plataforma utilizada para la secuenciación se reportó en 13/15 estudios (86,66%); por ejemplo: HiSeq 2500, HiSeq XTen, MiSeq y VeriSeq de Illumina (10/15, 66,66%), Ion PGM, Ion S5 Seq., y Ion S5 XML.
En cuanto a la concentración de cf-DNA, esta fue reportada en 6/15 (40%) de los estudios con una concentración promedio de 37,42 ng/μL ± 34,06 (rango de 15,2-105 ng/μL) (tabla 3).
Amplificación de cf-DNA obtenido de SCM y nivel de concordancia con biopsia TE (n = 15).
| ID Estudio | Referencia | EmbriMuestras SCM | Tiempo de secreción de cfDNA en SCM | Duración de incubación (número dedías) | Método de amplificación (kit) | Análisis genético de DNA | Sistema de análisis de secuenciación | Concentración cfDNA (promedio) | Referencia de concordancia | Tasa de concordancia con TE (%) |
|---|---|---|---|---|---|---|---|---|---|---|
| 1 | Ben-Nagi, J, et al.; 2019 | 1 | D1-D5 | 4 | NA | NGS | MiSeq (Illumina) | NA | TE | 100,00 |
| 2 | Huang, L, et al.; 2019 | 52 | D3-D5/6 | 2-3 | NICSwift (Yikon) | NGS | HiSeq 2500 (Illumina) | NA ver suplment | WE, TE | 76,92 |
| 3 | Kuznyetsov et al., 2018 | 26 | D4-D5/6 | 1-2 | SurePlex | NGS | VeriSeq (Illumina) | NA | TE | 100,00 (Eu- y Aneuplodias)55,60 (Mosaicismos) |
| 7 | Rubio et al., 2019a | 108 | D4-D5/6/7 | 1-3 | Ion Reproseq | NGS | Ion S5 TM XML | NA ver suplment | TE | 78,80 |
| 9 | Yeung, QSY et al.; 2019 | 168 | D3-D5/6 | 2-3 | SurePlex | aCGH / IonProton | MiSeq (Illumina) | D5, 15,5 ng/μLD6, 20,6 ng/μL | TE | 62,10 |
| 10 | Jiao, J et al.; 2019 | 62 | D5/6 | 15h | MALBAC | NGS | HiSeq 2500 (Illumina) | NA | WE | 76,00 |
| 11 | Ho, JR et al.; 2018⁎ | 40 | D1-D5 | 4 | PicoPlex | NGS | Ion S5 Sequencer | 105 ng/μL | TE | 65,00 |
| 12 | Li, P et al.; 2018 | 39 | D3-D5 | 2 | MALBAC | NGS | HiSeq 2500 (Illumina) | 62,60 ng/μL | TE/WE | 76,3/78,9 |
| 13 | Vera-Rodriguez, M et al.; 2018 | 51 | D3-D5 | 2 | SurePlex | NGS | Ion PGM | NA | TE | 54,9 |
| 14 | Kuznyetsov, V et al.; 2018 | 47 | D4-D5/6 | 1-2 | SurePlex | NGS | MiSeq (Illumina) | 24 h: 21,7 ng/μL24-48 h: 15,2 ng/μL | TE/WE | 24 h: 96,4024-48 h: 100,00 |
| 17 | Feichtinger, M et al.; 2017 | 22 | D0-D5 | 5 | SurePlex | aCGH | NA | 21,33 ng/μL | PB | 72,2 |
| 18 | Kuznyetsov, V et al.; 2017 | 25 | D5/6 | 1 | SurePlex | NGS | VeriSeq (Illumina) | NA | TE | 85,00 |
| 19 | Huang, L et al.; 2017 | 33 | D5 | 1 | MALBAC | NGS | HiSeq XTen | NA | WE | 75,75 |
| 20 | Lane, M et al.; 2017 | 178 | D3-D5 | 1-2 | PicoPlex/DOplify | NGS | NA | NA | TE | D4: 95,00D3-D5: 65,40 |
| 22 | Xu, J et al.; 2016 | 42 | D3-D5 | 2 | MALBAC | NGS | HiSeq 2500 (Illumina) | NA | WE | 73,8 |
Concordancia cf-DNA detectado en SCM y TE/WE para diagnóstico de aneuploidías
De los 15 estudios utilizados para el análisis cuantitativo de esta revisión sistemática, 15/15 (100%) reportaron la concordancia. La concordancia que se evaluó fue aquella «concordancia general», esto es, sano vs no sano. Sin embargo, algunos estudios también incluyeron «concordancia completa», esto es, el número específico y el tipo de alteraciones genéticas. Debido a la disparidad entre los reportes de la concordancia general y completa, se decidió reportar la concordancia general, ya que esta se incluía en los estudios seleccionados. Esta se evaluó de acuerdo a la muestra comparativa de biopsia de TE (8/15, 53,33%), análisis de WE (3/15, 20,00%) o ambos (3/15, 20,00%). Inclusive en un estudio (1/15, 6,66%) se comparó con la biopsia de cuerpo polar. De los estudios que reportaron concordancia, el rango osciló entre 54,9-100%. El promedio de concordancia de los 15 estudios fue de 78,32% ± 14,55. Sin embargo, los promedios según la muestra de comparación de concordancia fueron de 76,18% con biopsia por TE, 75,18% con WE y 85,70 en comparación con TE/WE (ver tabla 3). No obstante, estos resultados pueden ser producto de las heterogeneidades entre las diversas metodologías, como el tiempo de cultivo, el día de recolección, la manipulación embrionaria (eclosión asistida, colapso de blastocele, etc.), los sistemas de amplificación y secuenciación distintos utilizados, y la concentración de cf-DNA amplificada.
DiscusiónLas aneuploidías cromosómicas tienen una prevalencia de entre el 20-80% de los blastocistos humanos (Vera-Rodriguez et al., 2018).
Las aneuploidías ocurren en 2 momentos importantes: 1) gametogénesis (errores meióticos) y 2) post-fertilización (errores mitóticos). (Fragouli et al., 2019) Los errores mitóticos ocurren durante las divisiones de clivaje embrionarias, con un resultado de viabilidad inversamente proporcional al momento de presentación. Además, se ha detectado una elevada cantidad de mosaicismo (presencia de 2 linajes celulares cariotípicamente distintos en un mismo embrión) en embriones en etapa de clivaje. Existen mosaicos sin células euploides, que han ocasionado errores mitóticos sobre aneuploidías meióticas, o alguna combinación de anomalías mitóticas. Los mosaicos afectan al 30-40% de los blastocistos humanos. (Huang et al., 2019; Vera-Rodriguez et al., 2018) Este tipo de aneuploidías en mosaicos conllevan una tasa muy baja de viabilidad. (Fragouli et al., 2019) Por otra parte, aquellos mosaicos con presencia de células euploides (diploide-aneuploide), con incidencia de 2,0 a 17,3%, también tienen un potencial reducido de viabilidad, pues el linaje aneuploide interrumpe interacciones críticas entre célula y célula. (Fragouli et al., 2019; Huang et al., 2019) Es importante mencionar que algunas guías recomiendan un linaje adicional menor al 20% para considerarse euploide y no mosaico. (Gleicher and Orvieto, 2017) Cabe mencionar, que el sistema regulador de arresto del ciclo celular o las vías de apoptosis que «regularían» a las células aneuploides, pueden tener efecto deletéreo sobre la viabilidad embrionaria al reducir el número celular total. Aunque embriones mosaicos de embriones diploides-aneuploides pueden tener un potencial implantatorio, este es muy limitado (Fragouli et al., 2019).
Por lo tanto, no es sorprendente que dentro los protocolos de PGT, el diagnóstico de aneuploidías (PGT-A) es el más utilizado (90%) para el análisis del número de copias de los 24 cromosomas, con el fin de transferir solo aquellos embriones con diagnóstico euploide. (Munné, 2018; Penzias et al., 2018) Igualmente, hasta un 40% de los ciclos de FIV en Estados Unidos se acompañan de PGT-A. (Munné, 2018) Las indicaciones de PGT-A incluyen: edad materna avanzada (≥ 35 años), pérdida gestacional recurrente, fallo de implantación recurrente, factor masculino de infertilidad severo y para aumentar el potencial de implantación de un embrión. (Munné, 2018; Rubio, Rodrigo, et al., 2019) Para valorar la utilidad de PGT-A, es importante mencionar que el beneficio de este ha mejorado considerablemente desde sus inicios, cuando solo valoraba por FISH de 5 a 10 cromosomas. (Penzias et al., 2018) Actualmente, contamos con ensayos clínicos aleatorizados (ECA) que han corroborado la utilidad de este estudio, pero con una mayor utilidad en poblaciones específicas. Un ECA de 155 pacientes de 32 años (21-42 años de edad) aleatorizadas reportaron la transferencia de un número mayor de embriones en el grupo control que en el grupo de embriones con CCS (1,8 vs. 2,0; P = 0,0001). (Scott et al., 2013) Incluso, la tasa de implantación fue mayor en el grupo de CCS vs. control (79,8% vs. 62,2%, P = 0,002) (Scott et al., 2013). De la misma forma, en el grupo con CCS se reportó una tasa mayor de RN (RR 1,39, IC 95% 1,07-1,60, P = 0,001) y de embarazo evolutivo (RR 1,15, IC 95% 1,03-1,43; P = 0,03). (Scott et al., 2013) En otro estudio prospectivo de intervención a un ciego, se comparó el uso de la valoración embrionaria convencional (n = 48) vs. el análisis genético por aCGH en la biopsia de TE (n = 55), en pacientes menores de 35 años. (Yang et al., 2012) Los resultados reportaron la detección de aneuploidías en 44,9% de los blastocistos biopsiados (35,5% monosomías, 20,9% trisomías). A todas las pacientes del grupo de estudio (aCGH) se les transfirió al menos un blastocisto diagnosticado como euploide y se reportó que este grupo tuvo una tasa mayor de embarazo evolutivo (69,1 vs. 41,7%, P = 0,009) y embarazo clínico (70,9 vs. 45,8%, P = 0,017). La tasa de aborto fue menor en el grupo de aCGH, pero de manera estadísticamente no significativa (2,6% vs. 9,1%, P = 0,597). (Yang et al., 2012) A pesar de estos resultados de ECA, una revisión del sistema nacional de supervisión de TRA reportó que el PGT no se asocia a mejores tasas de RN vivo por transferencia de blastocisto en fresco autógeno y embarazo clínico en las pacientes ≤ 37 años de edad, sin importar la indicación. (Chang et al., 2016; Kushnir et al., 2016; Penzias et al., 2018) Sin embargo, aquellos embriones tratados con PGT-A tuvieron mejores tasas de RN vivo en las pacientes mayores de 37 años. (Kushnir et al., 2016) Sin embargo, es importante destacar que este reporte incluye ciclos de PGT-A con metodología FISH, actualmente en desuso, (Penzias et al., 2018) pero podemos concluir que el PGT-A y la subsecuente transferencia de embriones euploides tienen una utilidad obvia en TRA y específicamente en las mujeres de edad materna avanzada, por lo que las ventajas de poder realizar este diagnóstico a través de métodos no invasivos, sería un beneficio obvio.
Como se mencionó previamente, el 100% de los ensayos estudiados en esta revisión sistemática detectaron cf-DNA dentro del SCM. Es importante partir de la noción que la cantidad de cf-DNA detectada es mayor en SCM de cultivos expuestos a embrión, en comparación a cultivos que no han sido expuestos a embrión. Esto se traduce en que cierta cantidad de ADN embrionario es liberado en el SCM (Cree and Farquhar, 2020; Hammond et al., 2017; Vera-Rodriguez et al., 2018); por lo tanto, aunque se ha propuesto que este ADN proviene de blastómeras sometidas a apoptosis en corrección de aneuploidías (Hammond et al., 2017), se reportó que la cantidad de cf-DNA no es mayor en embriones aneuploides en comparación con embriones euploides. (Vera-Rodriguez et al., 2018) Si esta hipótesis fuera correcta, los embriones aneuploides presentarían mayor secreción de cf-DNA al medio debido a una mayor tasa de apoptosis. Ahora, es importante establecer que biológicamente, es el TE y no la masa celular interna (MCI) el que está directamente en contacto con el SCM, los cuales pueden diferir. Aunque previamente se ha reportado una concordancia de 100% en biopsias de TE y MCI, a pesar de obtener una tasa de aneuploidías de 38,8-51%. (Fragouli et al., 2008; Fragouli et al., 2019) Esto es importante, ya que en el estado de blastocisto, las células de TE son mayores en cantidad en comparación de la MCI. Más aún, las blastómeras del TE evolucionan a la formación de la placenta, la cual puede contener segmentos compuestos de islotes aneuploides (Gleicher and Barad, 2019; Orvieto et al., 2016).
Además, existen 4 grandes problemas con la detección de cf-DNA en el SCM: 1) la contaminación materna (la contaminación paterna puede ser excluida en gran medida mediante ICSI), 2) la diferencia en los cromosomas implicados en muestras concordantes (TE vs. SCM) de embriones aneuploides, 3) la cantidad de ADN embrionario presente en SCM dependiendo del día de cultivo y 4) los niveles bajos de cf-DNA en el SCM (Cree and Farquhar, 2020; Hammond et al., 2016; Lane et al., 2017; Vera-Rodriguez et al., 2018).
En cuanto a la contaminación materna, su presencia se ha reportado hasta en un 60% (Ho et al., 2018; Vera-Rodriguez et al., 2018), la cual puede provenir de líquido folicular, células del CCO, pero es importante recordar que la presencia de cromosoma Y (SRY) detectada en SCM comprueba que sí existen fragmentos de ADN que originan de la mórula/blastocisto(Cree and Farquhar, 2020; Lane et al., 2017; Vera-Rodriguez et al., 2018; Yang et al., 2017); por lo tanto, para evitar en mayor medida la contaminación del SCM, sería importante realizar la fecundación por ICSI y la denudación extensa del cúmulo de los ovocitos obtenidos por medio de la captación de ovocitos, así como el el lavado para eliminar restos de líquido folicular en los ovocitos captados (Cree and Farquhar, 2020).
En cuanto a las diferencias de concordancia entre las biopsia de TE y SCM, pueden ser muchos los factores implicados. No está claramente establecido en qué porcentaje el cf-DNA presente del SCM es proveniente de la MCI o de las células del TE, si es a través de un mecanismo de necrosis o apoptosis, o el grado de mosaicismo. Hasta ahora, distintos autores utilizan diversos umbrales para establecer mosaicismo, pero existe un consenso general de establecerlo en cuanto este se encuentra presente en un grado mayor al 30%. (Cree and Farquhar, 2020; Rubio, Rodrigo, et al., 2019) Sin embargo, este mosaicismo, aunque sea inferior al 30% tiene mayores implicaciones al establecer la euploidía en un embrión por medio del análisis de SCM: el tipo de mosaicismo y la línea celular afectada. Este puede ser a) mosaico total: este puede presentar un mosaico en MCI y TE; b) mosaico de MCI: puede presentar un mosaico dentro de la MCI y contar con un TE euploide; c) mosaico de TE: puede presentar un mosaico dentro del TE y contar con una MCI euploide; d) mosaicismo MCI/TE Tipo I: MCI aneuploide con un TE euploide; e) mosaicismo MCI/TE Tipo II: MCI euploide con un TE aneuploide. Este puede ser un motivo de las discordancias entre cf-DNA y la biopsia de TE (Vera-Rodriguez and Rubio, 2017).
Entre todas las técnicas de análisis genético, actualmente la NGS es la técnica más comúnmente aplicada para el análisis de PGT-A. (Rubio, Rodrigo, et al., 2019) Esto se refleja en nuestro estudio, en el cual el NGS se utilizó en 86% de los estudios. Esta tecnología permite que posterior al WGA, cada secuencia pueda ser alineada con un genoma humano de referencia, en búsqueda de variaciones en el número de copias de cromosomas o grandes deleciones/duplicaciones mediante la utilización de software específico. Sin embargo, el protocolo de NGS debe ser modificado en niPGT-A con el fin de obtener un resultado mayor amplificación de ADN y mayores resultados informativos (Rubio, Rodrigo, et al., 2019).
En un estudio se analizó el SCM de 9 embriones posterior a las 24 h del cultivo de D1 a D2, y se analizó cada 24 h hasta el D6. Sin embargo, no se detectó material genético en SCM durante los primeros 2 días de desarrollo embrionario, solo se detectó cf-DNA a partir de D3-D6; por lo que el día de desarrollo embrionario juega un papel crucial en la evaluación de niPGT-A. Esto es una importante ventaja sobre la técnica de blastoscentesis, ya que el líquido del blastocele solo es recuperable una vez que se ha formado el blastocisto (≥ D5), mientras que el cf-DNA es detectable en el SCM a partir del día 3. Sin embargo, la cantidad/calidad no parece seguir un patrón lineal; si bien hay estudios que reportan la ausencia de cf-DNA en D1-D2, se ha reportado una tasa mayor en concordancia en cultivos de SCM desde D4 en adelante en comparación a cultivos de D3-D5. (Lane et al., 2017; Yang et al., 2017) Esto se podría explicar por los resultados de numerosos estudios que indican una mayor proporción de blastocistos cromosómicamente normales en comparación con la etapa de clivaje. (Fragouli et al., 2019) Inclusive, se ha reportado mayor cantidad de cf-DNA en día 3, pero mejor calidad de ese ADN en día 5. (Ho et al., 2018) A pesar de esto, los resultados reportados han sido mejor en sistemas de SCM secuenciales que en la recolección en la etapa de blastocisto (Cree and Farquhar, 2020; Leaver and Wells, 2020).
El cf-DNA encontrado en SCM proviene de 3 sitios: 1) células (blastómeras) apoptóticas, 2) células necróticas y 3) productos liberados por células en división activa, independientemente de un origen euploide o aneuploide. (Gahan et al., 2008; Ho et al., 2018) Normalmente, consiste en fragmentos de una longitud < 200 pb, lo cual es mucho menor si se compara con gDNA. En biopsias de TE, la cobertura por NGS es de 50-60 millones de lecturas, lo que equivale a una cobertura de aproximadamente 2% del genoma; esto se ha podido emular en cf-DNA. Aunque se han reportado tasas de lectura similares de NGS en cf-DNA de SCM en comparación con biopsia de TE, la habilidad de secuenciar cf-DNA en D5 no cuenta con una especificidad suficientemente alta para asegurar la selección de un embrión euploide. (Ho et al., 2018) Sin embargo, para poder analizar por medio de NGS, se puede presentar un obstáculo en la amplificación del cf-DNA en SCM. En un estudio en el que muestras de SCM fueron amplificadas en D3 vs. D5 por medio de PicoPlex-WGA, solo el 39% de las muestras de D3 y 80,4% en D5 generaron suficiente ADN para analizarse por medio de NGS. (Ho et al., 2018; Leaver and Wells, 2020) Una de las maneras en que la amplificación de DNA se podría optimizar, según los resultados de estudios previos presentados en esta revisión, sería el cultivo continuo hasta los días D5-D7. De la misma manera, el volumen ideal de cultivo es algo que no ha sido estandarizado y como pudimos encontrar en este estudio, varía de manera importante a través de cada centro; (Galluzzi et al., 2015; Hammond et al., 2017; Ho et al., 2018; Leaver and Wells, 2020; Yeung et al., 2019) inclusive, el medio de cultivo puede contener componentes inhibitorios para la adecuada amplificación de cf-DNA. (Leaver and Wells, 2020) Por esto y por la característica de los kits comerciales de WGA disponibles de utilizar muestras de ADN en volúmenes < 10 μL, una recomendación inicial podría ser mantener los embriones en volúmenes de cultivo menores, para evitar la dilución del contenido de cf-DNA en el SCM; (Huang et al., 2019; Jiao et al., 2019; Leaver and Wells, 2020; Rubio, Rienzi, et al., 2019) más aún, el cultivo en volumen reducido se ha reportado que se asocia a una tasa mayor de formación de blastocisto (35 μL vs. 7 μL), lo cual sugiere un efecto adicional de factores autocrinos producidos por el embrión, que ejercen un efecto positivo en ausencia de dilución. (Leaver and Wells, 2020; Minasi et al., 2015)
ConclusionesLas ventajas de realizar PGT-A son claras en los casos indicados por el fallo de la implantación repetida, la pérdida gestacional recurrente y la edad materna avanzada, por lo que la posibilidad de realizar niPGT-A es muy atractiva, ya que son indudables las ventajas prácticas en cuanto a las habilidades requeridas por parte del equipo de laboratorio, el coste-efectividad del procedimiento y por supuesto, la disminución de la manipulación embrionaria que harían prácticamente nulos los riesgos que enfrentaría el embrión en cuanto a la posibilidad de presentar un daño.
Hemos encontrado que (a excepción de embriones de D0-D2) el cf-DNA es detectable en el medio de cultivo embrionario en la etapa de mórula y blastocisto. La amplificación de cf-DNA en SCM es completamente factible, aunque la cantidad de cf-DNA amplificada varía en términos de cantidad e integridad, además de que aun persiste una gran cantidad de heterogeneidad entre las técnicas de protocolos que utilizan los distintos centros que realizan este procedimiento. Por último, aunque se ha encontrado un nivel de concordancia con TE de hasta un 100%, el promedio oscila alrededor de un 78%. Además, la concordancia específica en cuanto a cromosomas afectados en embriones aneuploides podría tener un mayor grado de disparidad.
Sin embargo, factores como la contaminación materna, el establecer un día ideal de recolección de SCM durante el desarrollo embrionario, cantidades bajas de cf-DNA presentes en el SCM y las discrepancias en la concordancia entre niPGT-A y biopsia de TE, son obstáculos que aún no permiten el uso de niPGT-A como un método fiable de análisis de detección de aneuploidías. Por lo tanto, se deben tomar medidas de optimización de los protocolos de niPGT-A, como lo son la fecundación por ICSI, el lavado de ovocitos y la denudación del CCO, el cultivo continuo hasta la etapa de blastocisto (D5-D7), mantener un volumen reducido del cultivo embrionario (≤ 10 μL), y el análisis genético de por medio de NGS. Por otra parte, quedan incógnitas pendientes de resolver para la estandarización de niPGTA: el origen de cf-DNA embrionaria y el mecanismo de secreción de este material, la concordancia de mosaicismo en muestras de SCM y TE/WE, o el establecimiento preciso del mejor día de recolección de SCM.
Concluimos que aunque niPGTA es una técnica prometedora que conllevaría un conjunto de beneficios superiores al iPGT-A, por el momento es necesario seguir validando esta técnica, obtener un mayor entendimiento de la fisiología embrionaria, tener el origen concreto del ADN extra-embrionario, hacer un protocolo más homogéneo entre los distintos centros de reproducción que están utilizando esta técnica y una mayor cantidad de ensayos clínicos aleatorizados que puedan brindar un mayor sustento teórico y práctico a este estudio diagnóstico. Por esto y todos los motivos presentados en esta extensa revisión sistemática, concluimos que el niPGT-A no es apto para ser utilizado en el contexto clínico y debe reservarse para uso en estudios de investigación y pre-clínicos hasta que emerja un consenso de todos los interrogantes teóricos y de protocolos de procedimiento que aún ofuscan al niPGT-A como un método alternativo a la biopsia celular.