El papel de la microbiota en los modelos animales de esclerosis múltiple está bien establecido; por el contrario, los estudios en humanos son escasos.
MétodosEstudio transversal descriptivo que compara la composición relativa de la microbiota intestinal en 30 pacientes con esclerosis múltiple (15 tratados con interferónβ-1b, 15 sin tratamiento) y 14 controles sanos utilizando la secuenciación de última generación.
ResultadosLos sujetos control y los pacientes con esclerosis múltiple presentaron diferente abundancia de los filos Euryarchaeota, Firmicutes, Proteobacteria, Actinobacteria, y Lentisphaerae y 17 especies bacterianas. Concretamente, la abundancia en Firmicutes, Actinobacteria y Lentisphaerae y 6 especies mostró diferencias al comparar los grupos control y sin tratamiento, desapareciendo esta diferencia cuando se compararon con los pacientes tratados. Se observó reducción estadísticamente significativa en la abundancia de Prevotella copri en pacientes sin tratamiento en comparación con controles, mientras que los tratados con interferónβ-1b presentaron un aumento significativo frente a pacientes sin tratamiento, asemejándose al grupo de pacientes sanos control.
ConclusiónLa composición de la microbiota intestinal fue diferente entre los pacientes con esclerosis múltiple y los controles, y entre los pacientes sin tratamiento y los tratados con interferónβ-1b. En la mayoría de los casos, no se encontraron diferencias entre los pacientes tratados y los controles sanos, siendo especialmente evidente con P. copri. Esto podría indicar que la influencia del interferónβ-1b sobre la microbiota intestinal podría subyacer en los beneficios clínicos observados en pacientes con esclerosis múltiple que siguen este tratamiento. Serán necesarios estudios longitudinales y funcionales para poder mostrar causalidad.
The association between gut microbiota and animal models of multiple sclerosis has been well established; however, studies in humans are scarce.
MethodsWe performed a descriptive, cross-sectional study comparing the relative composition of gut microbiota in 30 patients with multiple sclerosis (15 treated with interferon β–1b, 15 not receiving this treatment) and 14 healthy controls using next generation sequencing.
ResultsPatients with multiple sclerosis and controls showed differences in the proportion of Euryarchaeota, Firmicutes, Proteobacteria, Actinobacteria, and Lentisphaerae phyla and in 17 bacterial species. More specifically, we found significant differences in the proportion of Firmicutes, Actinobacteria, and Lentisphaerae and 6 bacteria species between controls and untreated patients; however, these differences disappeared when compared with treated patients. Untreated patients showed a significant reduction in the proportion of Prevotella copri compared to controls, while the bacteria was significantly more abundant in patients treated with interferon β–1b than in untreated patients, with levels resembling those observed in the healthy control group.
ConclusionWe observed differences in gut microbiota composition between patients with multiple sclerosis and controls, and between patients treated and not treated with interferon β–1b. In most cases, no differences were observed between treated patients and healthy controls, particularly for P. copri levels. This suggests that the clinical improvements observed in patients with multiple sclerosis receiving interferon β–1b may result from the effect of the drug on gut microbiota. Longitudinal and functional studies are necessary to establish a causal relationship.
La esclerosis múltiple (EM) es una enfermedad desmielinizante que afecta a más de 2,5 millones de personas en el mundo y cuya incidencia y prevalencia, como en otras enfermedades autoinmunes, están aumentando en los últimos años1. Su patogénesis no está totalmente dilucidada, pero se acepta que se requiere predisposición genética y exposición a determinados factores ambientales, algunos bien establecidos, como la infección por el virus de Epstein-Barr, el tabaquismo, la latitud o el déficit de vitamina D, y otros actualmente en investigación, como la ingesta excesiva de sodio, vitamina A o alcohol, el sobrepeso en la adolescencia, los elevados niveles de leptina, la ausencia de contacto con helmintos o con Helicobacter pylori1. Entre estos nuevos factores ambientales podemos situar a la microbiota intestinal (MI)2-9. De hecho, su papel en la fisiología humana está bien descrito, regulando la homeostasis inmunológica tanto a nivel de respuesta innata como adaptativa, en la maduración de órganos linfáticos, producción de células plasmáticas productoras de IgA, células Tγδ intestinales, linfocitos TCD4, expresión génica de receptores toll-like (TLR) y del complejo mayor de histocompatibilidadII, producción de citocinas, etc. Todo esto subyace en su influencia sobre los linfocitos T reguladores y los T helper, y más concretamente en el equilibrio Th1-Th17/Th2, condicionando con ello la susceptibilidad a experimentar infecciones, enfermedades inflamatorias, neoplásicas, y lo que aquí nos ocupa, autoinmunes1.
La disbiosis se ha relacionado con un estado proinflamatorio asociado a numerosas afecciones autoinmunes, incluyendo la enfermedad inflamatoria intestinal, la diabetes tipo 1, la artritis reumatoide, así como a patologías gastrointestinales, neurológicas, respiratorias, metabólicas, hepáticas, cardiovasculares10, a la infección por VIH11 o a la EM. Más específicamente, y en relación con la EM, hay una sólida evidencia científica del papel de la microbiota en la génesis, prevención e incluso tratamiento de la encefalomielitis autoinmune experimental (EAE), el modelo animal de la EM1. Sin embargo, y a diferencia de lo expuesto para modelos animales, los estudios de la microbiota en la EM son escasos y están en las primeras fases de investigación2-9. Ninguno de estos estudios se ha llevado a cabo en población mediterránea ni, por tanto, española, y ningún estudio se ha diseñado específicamente para valorar la influencia en la microbiota del tratamiento de la enfermedad.
Por todo ello se diseñó este estudio, para describir la composición relativa en taxones bacterianos y arqueas de la MI de una muestra de pacientes residentes en España y detectar diferencias entre aquellos pacientes sin tratamiento y los tratados con interferón (IFN) β-1b.
MétodosReclutamiento de sujetosEste estudio transversal se llevó a cabo en el Hospital San Pedro de Logroño, España (septiembre de 2014-abril de 2015). Se invitó a participar en el estudio a pacientes mayores de 18 años atendidos tanto en consultas externas como hospitalizados, que presentaban diagnóstico de EM según los criterios de McDonald (2010)12. Se incluyeron dos grupos de participantes: un grupo sin tratamiento, formado por pacientes que nunca habían sido tratados con ningún fármaco modificador de la enfermedad, y un grupo tratamiento, compuesto por pacientes que se encontraban en tratamiento con IFNβ-1b cada 48h desde hacía al menos 9 meses. Se seleccionaron los 15 primeros pacientes que cumplían las características mencionadas para cada uno de los grupos y que no presentaban criterios de exclusión. No existiendo un consenso para la selección del grupo control4, se seleccionaron 14 voluntarios sanos que no presentaban antecedentes patológicos de enfermedades autoinmunes, ni antecedentes familiares de EM.
Criterios de exclusión: tratamiento en el último mes con antibióticos, corticoides, inmunosupresores o con suplementos nutricionales o medicamentos a base de probióticos, aquellos intervenidos de cirugía gastrointestinal —salvo apendicetomía o colecistectomía— y los que tuviesen antecedentes personales de enfermedad inflamatoria intestinal, celiaquía, pancreatitis crónica o cualquier síndrome de malabsorción.
Este estudio se llevó a cabo siguiendo los principios éticos de investigación recogidos en el informe Blemont y la Declaración de Helsinki, así como los contenidos en la Declaración Universal de la UNESCO referentes al genoma humano, y fue aprobado por el Comité Ético de Investigación Clínica en La Rioja (CEICLAR, Ref. CEICLAR PI 176). Se obtuvo el consentimiento informado de todos los participantes en el estudio.
Recogida de muestras, extracción de ADN, secuenciación de ADNr16s y análisis bioinformáticoDe cada individuo se recogieron las primeras heces de la mañana y se procedió a extraer el ADN en fresco (25mg) utilizando el kit DNeasy Blood & Tissue Kit (Qiagen, Alemania) siguiendo las instrucciones del fabricante. La pureza y concentración se determinaron mediante un espectrofotómetro Nanodrop 1000 (ND-1000; Thermo Scientific, EE.UU.). Para la secuenciación de ADNr se amplificó la región V4 hipervariable del gen 16s empleando los primers 515-806-R mediante la tecnología Illumina MiSeq (2×250pb), con alrededor de 100.000 lecturas por muestra13. El análisis bioinformático fue llevado a cabo por la empresa Era7 Bioinformatics (Granada, España). Para ello, mediante el programa FLASH se procedió al ensamblaje de las dos lecturas obtenidas mediante la tecnología Illumina. Las lecturas se asignaron a un taxón en función de la similitud con las secuencias de ARNr16s incluidas en el Ribosomal Database Project (RDP)14, con base en la especificidad de su asignación taxonómica en la base de datos del NCBI (National Center for Biotechnology Information) disponible en https://github.com/ohnosequences/db.rna16s. Para esto se llevó a cabo un BLAST (basic local alignment 116 search tool) masivo utilizando un análisis con MG715. Para realizar la asignación taxonómica posterior se utilizaron dos métodos, BBH (best blast hit) para estudios a nivel de filo y LCA (lowest common ancestor) para estudios a niveles taxonómicos inferiores. El LCA se basó en herramientas avanzadas de análisis de metagenómica similares a la última versión del MEGAN (MEtaGenome ANalyzer)16. En primer lugar se estudió la riqueza de taxones (α-diversidad) y, en segundo lugar, se llevó a cabo un estudio de las diferencias en taxones que se puedan establecer entre los grupos de estudio (β-diversidad)17.
Análisis estadísticoLos resultados se presentan como media±error estándar de la media (standard error of the mean [SEM]). La significación estadística se fijó en p<0,05. Las variables categóricas se analizaron mediante Chi-cuadrado y la prueba exacta de Fisher. Los análisis de normalidad se llevaron a cabo mediante el test de Shapiro-Wilk. Para comparar ambos grupos se utilizó la t de Student cuando ambos grupos presentaban una distribución normal y el test de Mann-Whitney cuando la distribución no lo era en alguno de ellos. El análisis comparativo de los 3 grupos (control, grupo sin tratamiento y grupo con tratamiento) se realizó mediante análisis de la varianza (ANOVA) y con un análisis post-hoc de comparaciones múltiples mediante la prueba de Tukey cuando la distribución era normal. En aquellos casos en que la distribución no resultaba normal, se empleó el test de Kruskal-Wallis de los 3 grupos, con un estudio post-hoc de comparaciones múltiples mediante el test de comparaciones múltiples de Dunn. El análisis estadístico se llevó a cabo mediante SPSS 19.0 (SPSS® Inc., Chicago, IL, EE.UU.) y GraphPad Prism 6 (GraphPad Prism®, La Jolla, CA, EE.UU.).
La α-diversidad se calculó por medio de 4 índices (Shannon index, Margalef's diversity index, Chao-1 y Alpha index) utilizando R (Vegan Community Ecology Package. R package version 2.3-2: http://CRAN.R-project.org/package=vegan). Para los análisis de β-diversidad, se realizó un análisis comparativo para todos los filos, los 40 taxones más abundantes y aquellos que mostraban diferencias tras aplicar un análisis metagenómico a través del servidor web METAGENassist18. Los análisis solo se realizaron cuando se obtuvieron lecturas en al menos el 25% de los participantes de cada grupo.
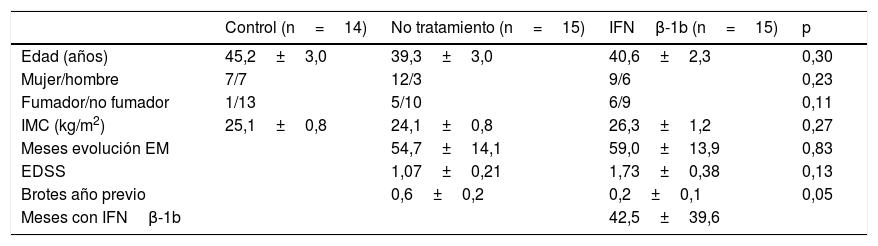
ResultadosTodos los sujetos incluidos en el estudio eran caucásicos y todos los enfermos presentaban la forma remitente recurrente de la enfermedad. Las características epidemiológicas de los grupos se presentan en la tabla 1. Los grupos resultaron homogéneos y solo se encontraron diferencias significativas en los brotes presentados por los pacientes en el último año, que como cabe esperar, son mayores entre los pacientes no tratados.
Características epidemiológicas de la muestra
| Control (n=14) | No tratamiento (n=15) | IFNβ-1b (n=15) | p | |
|---|---|---|---|---|
| Edad (años) | 45,2±3,0 | 39,3±3,0 | 40,6±2,3 | 0,30 |
| Mujer/hombre | 7/7 | 12/3 | 9/6 | 0,23 |
| Fumador/no fumador | 1/13 | 5/10 | 6/9 | 0,11 |
| IMC (kg/m2) | 25,1±0,8 | 24,1±0,8 | 26,3±1,2 | 0,27 |
| Meses evolución EM | 54,7±14,1 | 59,0±13,9 | 0,83 | |
| EDSS | 1,07±0,21 | 1,73±0,38 | 0,13 | |
| Brotes año previo | 0,6±0,2 | 0,2±0,1 | 0,05 | |
| Meses con IFNβ-1b | 42,5±39,6 |
EDSS: Escala Ampliada del Estado de Discapacidad; EM: esclerosis múltiple; IFN: interferón; IMC: índice de masa corporal; SEM: error estándar de la media.
Los datos se expresan como media±SEM.
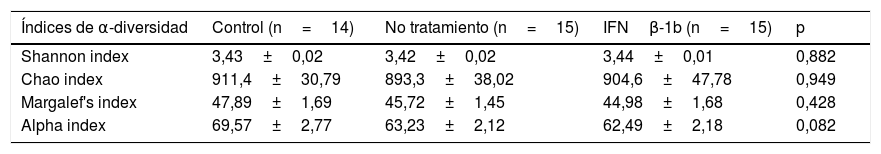
No se encontraron diferencias significativas entre los grupos estudiados con ninguno de los índices analizados (tabla 2), si bien sí que puede observarse una tendencia a la disminución de la riqueza en especies en pacientes con EM frente a controles cuando se calculó el índice alfa (p=0,08).
Medidas de α-diversidad del grupo control comparadas con los pacientes con esclerosis múltiple sin tratamiento y tratados con IFNβ-1b
| Índices de α-diversidad | Control (n=14) | No tratamiento (n=15) | IFNβ-1b (n=15) | p |
|---|---|---|---|---|
| Shannon index | 3,43±0,02 | 3,42±0,02 | 3,44±0,01 | 0,882 |
| Chao index | 911,4±30,79 | 893,3±38,02 | 904,6±47,78 | 0,949 |
| Margalef's index | 47,89±1,69 | 45,72±1,45 | 44,98±1,68 | 0,428 |
| Alpha index | 69,57±2,77 | 63,23±2,12 | 62,49±2,18 | 0,082 |
Los datos se expresan como media±SEM.
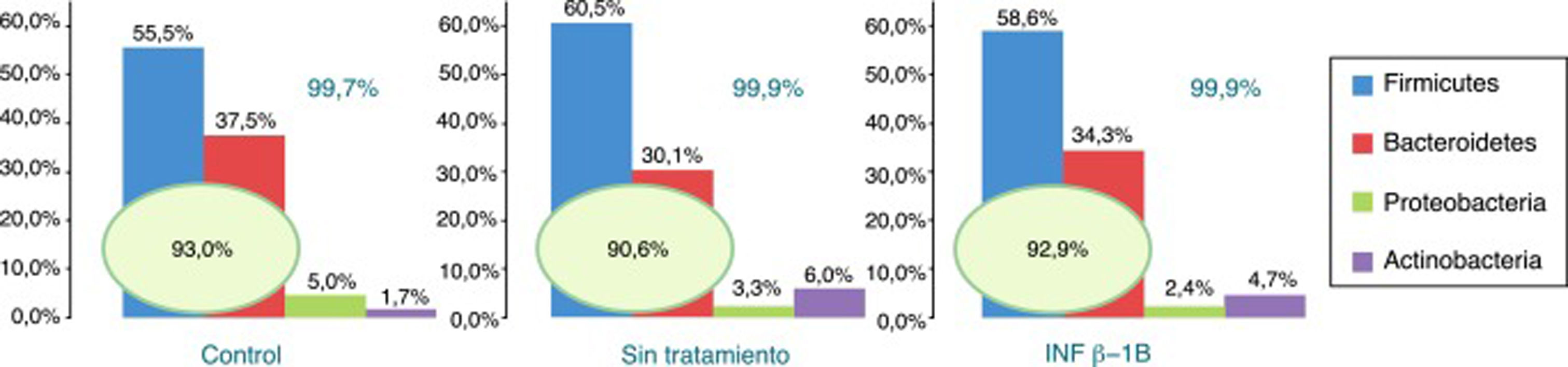
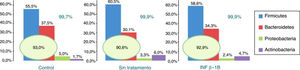
De los más de 30 filos bacterianos presentes en la MI2, se obtuvieron lecturas de 12 filos bacterianos más uno de arqueas. Los filos más abundantes presentes en los 3 grupos fueron Firmicutes, Bacteroidetes, Proteobacteria y Actinobacteria, constituyendo al menos el 99,70% de las lecturas asignadas. Dentro de estos 4 grupos destacaron Firmicutes y Bacteroidetes, representando más del 90% de las lecturas asignadas en todos los grupos, seguidos de Proteobacteria y Actinobacteria en sujetos control, mientras que en los pacientes con EM se encontró más abundancia de Actinobacteria que de Proteobacteria (fig. 1).
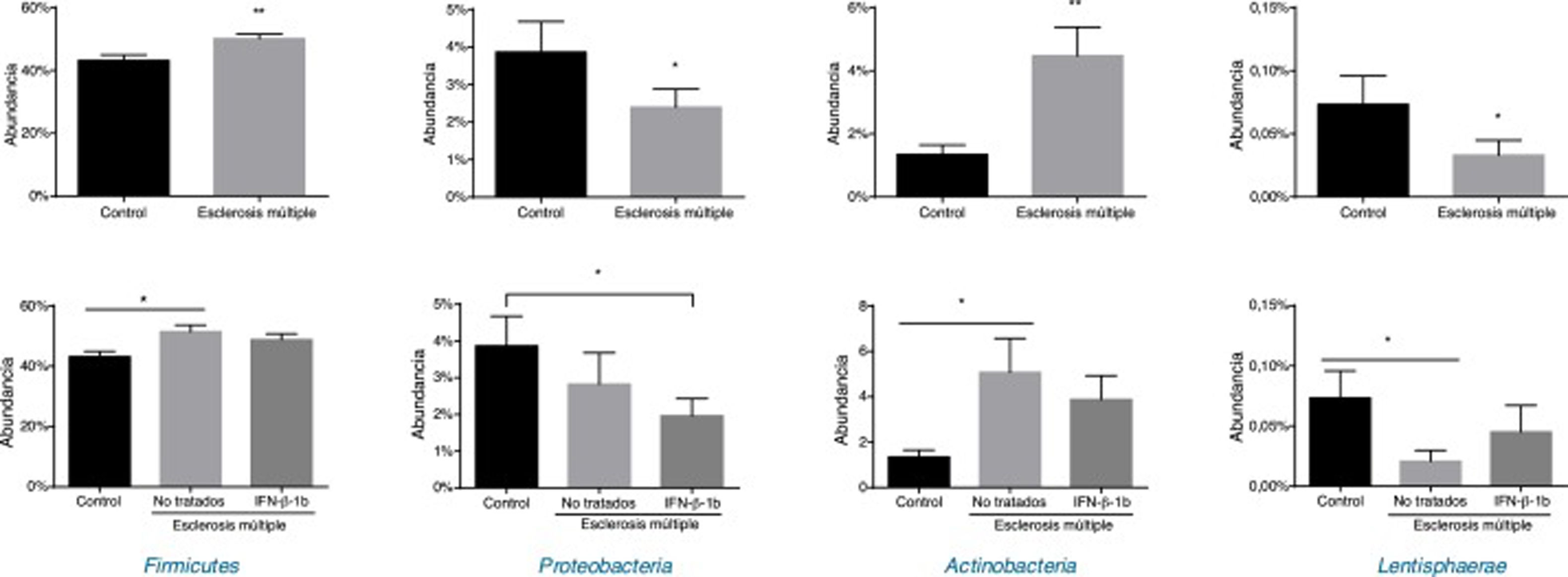
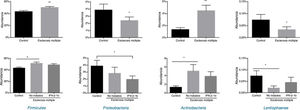
Al comparar los controles frente a los pacientes con EM se observaron diferencias estadísticamente significativas en los filos Firmicutes, Proteobacteria, Actinobacteria y Lentisphaerae (fig. 2). Se observó un aumento en pacientes con EM en Firmicutes y Actinobacteria, mientras que descendieron Proteobacteria y Lentisphaerae al compararlos con los controles. Cabe destacar que cuando se introdujo la variable del tratamiento en el estudio de los filos, Firmicutes, Actinobacteria y Lentisphaerae presentaron diferencias entre el grupo control y los pacientes no tratados; sin embargo, estas diferencias no se observaron al comparar los controles con los pacientes tratados. Por el contrario, la reducción observada en la abundancia de Proteobacteria en pacientes con EM se asoció a la reducción que se presentó entre los pacientes tratados (fig. 2).
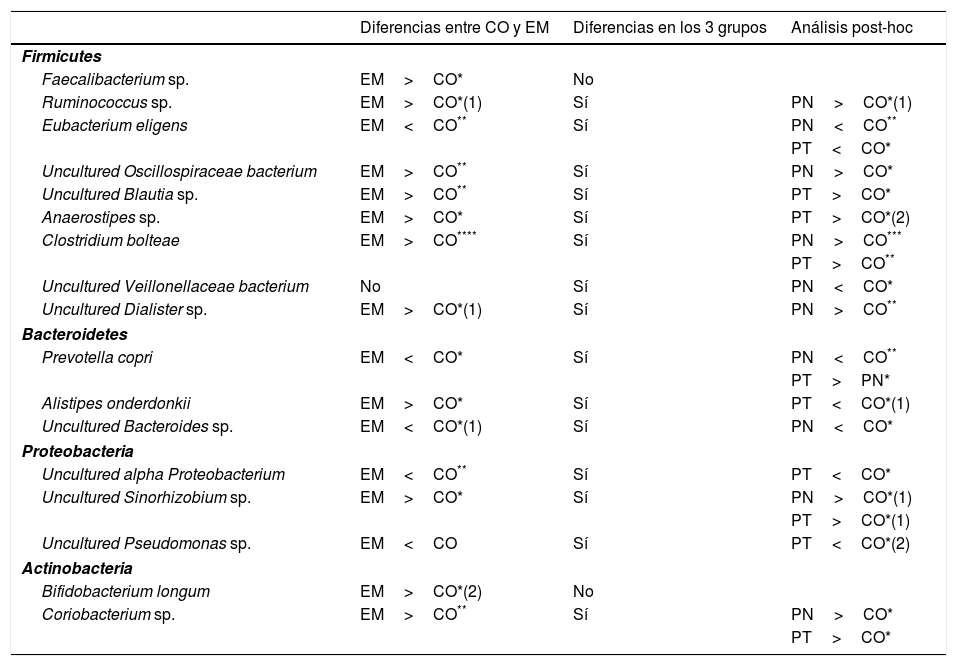
Cuando se comparó el grupo control con los pacientes con EM en unidades taxonómicas operacionales (UTO) correspondientes a género y especie, 11 UTO (Faecalibacterium sp.; Ruminococcus sp.; uncultured Oscillospiraceae bacterium; uncultured Blautia sp.; uncultured Sinorhizobium sp.; Anaerostipes sp.; Clostridium bolteae; uncultured Dialister sp.; Alistipes onderdonkii; Bifidobacterium longum y Coriobacterium sp.) fueron más abundantes en pacientes con EM que en el grupo control (tabla 3). De estos, 7 pertenecían al filo Firmicutes, una a Bacteroidetes, una a Proteobacteria y 2 a Actinobacteria. Por el contrario, 5 UTO fueron menos abundantes entre los enfermos de EM (Eubacterium eligens; Prevotella copri; uncultured Bacteroides sp.; uncultured alpha Proteobacterium; y uncultured Pseudomonas sp.). De estas, una pertenece a Firmicutes, 2 a Bacteroidetes y 2 a Proteobacteria (tabla 3).
Resumen de las diferencias encontradas a nivel de especie bacteriana
| Diferencias entre CO y EM | Diferencias en los 3 grupos | Análisis post-hoc | |
|---|---|---|---|
| Firmicutes | |||
| Faecalibacterium sp. | EM>CO* | No | |
| Ruminococcus sp. | EM>CO*(1) | Sí | PN>CO*(1) |
| Eubacterium eligens | EM<CO** | Sí | PN<CO** |
| PT<CO* | |||
| Uncultured Oscillospiraceae bacterium | EM>CO** | Sí | PN>CO* |
| Uncultured Blautia sp. | EM>CO** | Sí | PT>CO* |
| Anaerostipes sp. | EM>CO* | Sí | PT>CO*(2) |
| Clostridium bolteae | EM>CO**** | Sí | PN>CO*** |
| PT>CO** | |||
| Uncultured Veillonellaceae bacterium | No | Sí | PN<CO* |
| Uncultured Dialister sp. | EM>CO*(1) | Sí | PN>CO** |
| Bacteroidetes | |||
| Prevotella copri | EM<CO* | Sí | PN<CO** |
| PT>PN* | |||
| Alistipes onderdonkii | EM>CO* | Sí | PT<CO*(1) |
| Uncultured Bacteroides sp. | EM<CO*(1) | Sí | PN<CO* |
| Proteobacteria | |||
| Uncultured alpha Proteobacterium | EM<CO** | Sí | PT<CO* |
| Uncultured Sinorhizobium sp. | EM>CO* | Sí | PN>CO*(1) |
| PT>CO*(1) | |||
| Uncultured Pseudomonas sp. | EM<CO | Sí | PT<CO*(2) |
| Actinobacteria | |||
| Bifidobacterium longum | EM>CO*(2) | No | |
| Coriobacterium sp. | EM>CO** | Sí | PN>CO* |
| PT>CO* | |||
Observaciones: (1) la significación se obtiene solo al eliminar los valores atípicos; (2) la significación se pierde al eliminar los valores atípicos. En el resto la significación se obtiene tanto con valores atípicos como al eliminarlos.
CO: grupo control; EM: grupo con esclerosis múltiple; PN: pacientes no tratados; PT: pacientes tratados.
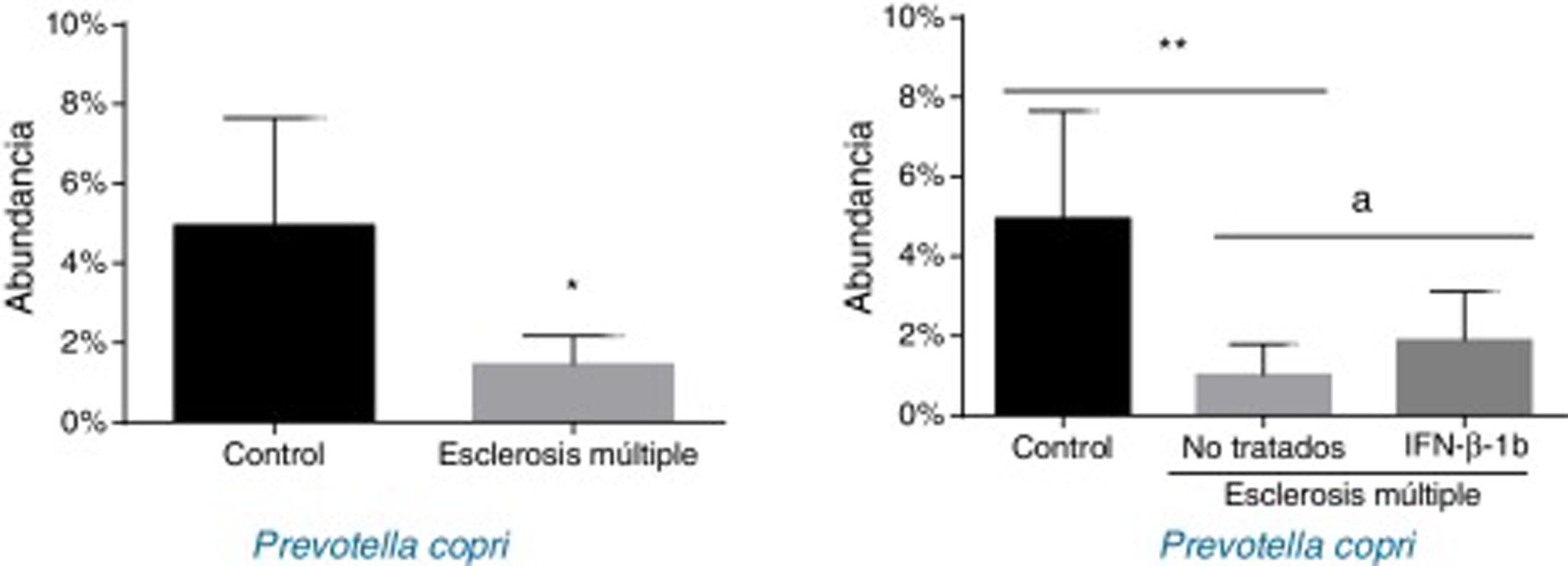
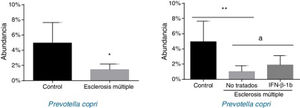
Cuando los pacientes se dividieron en función de su tratamiento, solo se observaron diferencias estadísticamente significativas entre los tratados y no tratados con P. copri (fig. 3). En primer lugar, se observó una reducción significativa en su presencia en pacientes sin tratamiento frente a controles (p<0,01) y, en segundo lugar, los tratados con IFNβ-1b mostraron una mayor abundancia frente a los no tratados (p<0,05) y una abundancia similar a los pacientes control (fig. 3). Esta diferencia observada entre controles y enfermos ya era evidente a nivel de género, donde Prevotella mostró mucha menor abundancia entre los enfermos de EM que en sujetos control (0,041±0,038% vs. 0,003±0,001%, p<0,05).
De las 9 UTO pertenecientes al filo Firmicutes, en que se encontraron mayor abundancia en pacientes con EM que en controles, 7 pertenecían al orden Clostridiales (fig. S1 del material suplementario). En todos los casos, salvo Faecalibacterium sp., encontramos diferencias en el estudio de los 3 grupos. Otras 2 especies del filo Firmicutes en que detectamos diferencias significativas pertenecían al orden Selenomonadales (tabla 3), uncultured Veillonellaceae bacterium y uncultured Dialister sp.; ambas presentan un comportamiento muy interesante cuando se estudian teniendo en cuenta el tratamiento, puesto que en ambos casos se observan diferencias significativas entre los 3 grupos, que recaen en la diferente abundancia de los pacientes sin tratamiento respecto a los controles. Cuando se analiza el grupo de pacientes en tratamiento con IFN, se observa que presentan una abundancia igual a la de los sujetos control, mostrando diferencias con los pacientes no tratados, que si bien no alcanza significación estadística, sí alcanza una interesante tendencia a la misma (fig. S2del material suplementario).
Además de la previamente mencionada P. copri, dentro del filo Bacteroidetes se han encontrado diferencias estadísticas en 2 UTO más, A. onderdonkii y uncultured Bacteroides sp. (fig. S3del material suplementario), en 3 del filo Proteobacteria, uncultured alpha Proteobacterium, uncultured Pseudomonas sp. y uncultured Sinorhizobium sp. (fig. S4del material suplementario) y en 2 pertenecientes al filo Actinobacteria, B. longum y Coriobacterium sp. (fig. S5del material suplementario).
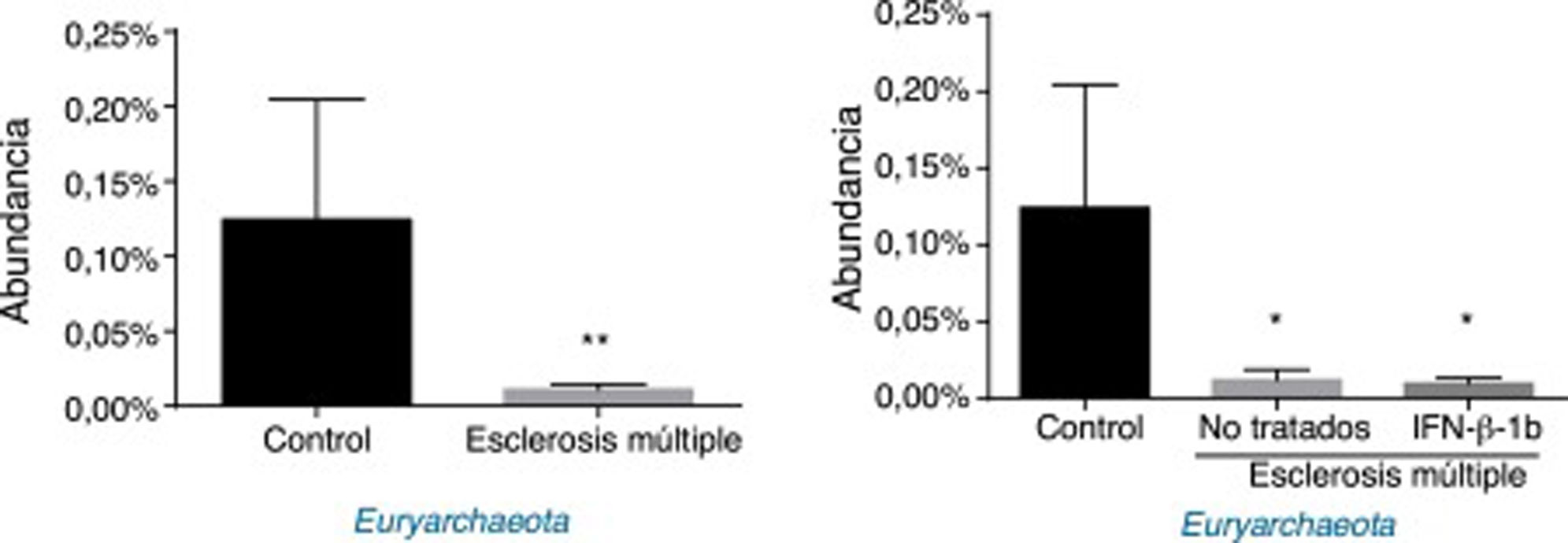
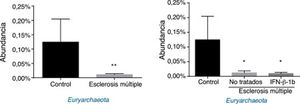
Respecto a las arqueas, se observaron diferencias significativas en la abundancia del filo Euryarchaeota (todas las lecturas pertenecieron a este filo y se obtuvieron de un solo género, Methanobrevibacter). Los pacientes con EM presentaron una abundancia menor que los controles, independientemente del tratamiento o no con IFNβ-1b (fig. 4).
DiscusiónNuestro estudio se encuadra dentro de los pocos trabajos publicados que demuestran diferencias en la MI de pacientes con EM frente a sujetos control, tanto a nivel de filos como en niveles taxonómicos inferiores2,18. Los hallazgos aquí presentados son de gran interés, puesto que la modulación/restauración de la MI se ha propuesto como una estrategia prometedora para el tratamiento de la EM. En este contexto, demostramos que los pacientes tratados con el fármaco modificador de la enfermedad IFNβ-1b presentan una abundancia similar que los sujetos sanos en diferentes taxones que se encuentran alterados en pacientes con EM no tratados. Estos datos abren la puerta a investigar si la mejoría clínica observada en pacientes tratados con este fármaco podría estar, en parte, mediada por el efecto del mismo en la MI.
La riqueza bacteriana (α-diversidad) en la MI se ve alterada en diversas situaciones clínicas, como obesidad, VIH11,19, etc. Sin embargo, nuestros hallazgos muestran una riqueza similar en la microbiota de los pacientes afectados por EM que en los sujetos sanos, como ya se había mostrado previamente, e independientemente del tratamiento con IFNβ-1b. Del mismo modo que lo previamente publicado, encontramos cambios relacionados con determinados taxones entre los pacientes afectados de EM2-6,9.
A nivel de filo, hemos encontrado diferencias estadísticamente significativas en la abundancia entre los pacientes con EM y los controles sanos en Firmicutes, Proteobacteria, Actinobacteria y Lentisphaerae —los 3 primeros pertenecientes a los 4 filos predominantes en el tracto gastrointestinal. En este sentido, Mowry et al. (2012) ya describieron cambios en Firmicutes y Actinobacteria en pacientes con EM frente a controles7, si bien también encontraron diferencias en Bacteroidetes. Una posible explicación a estas diferencias encontradas en cuanto a la abundancia de este último filo puede deberse al diseño diferente del estudio, el cual se llevó a cabo en pacientes deficitarios en vitamina D y mediante PhyloChip Assay (Second Genome, Inc.). En este mismo sentido, Miyake et al. (2015) también encuentran una disminución de la abundancia en Firmicutes y Bacteroidetes, aunque no alcanzó significación estadística2. El aumento que encontramos en la abundancia de Actinobacteria en pacientes con EM ya había sido descrito en niños4 y en adultos en población japonesa2, aunque en este segundo caso sin significación estadística. Esta concordancia entre los 3 estudios apunta a un papel importante del filo Actinobacteria en la EM, que también ha sido descrito en la enfermedad inflamatoria intestinal20,21.
Los cambios observados en los pacientes con EM en los filos Actinobacteria, Firmicutes y Lentisphaerae recaen en el grupo de pacientes sin tratamiento, no existiendo diferencias entre los pacientes tratados con IFNβ-1b y los sujetos sanos. Harán falta estudios diseñados para estudiar causalidad para confirmar si existe una recuperación en estos filos asociada al tratamiento, lo que sería compatible con estos resultados. Los resultados aquí presentados en el filo Actinobacteria son diferentes a los presentados por Tremlett et al. (2016)4, en que el tratamiento inmunomodulador aumenta las diferencias con sujetos sanos, si bien su estudio está realizado en población pediátrica en que la microbiota no está tan bien establecida, siendo la edad un factor clave en la composición de la MI22. Además, el citado estudio no se diseñó para buscar diferencias en el tratamiento, de modo que había niños recibiendo diferentes fármacos (acetato de glatirámero, IFN, natalizumab y corticoides).
Dentro del filo Firmicutes se encuentra el género Clostridium, relacionado con la producción de ácidos grasos de cadena corta, especialmente butirato, e implicado en la diferenciación de células T hacia células Treg, lo que le confiere un papel protector frente a enfermedades autoinmunes e inflamatorias1,23. Dentro de este género, y en contra de lo que cabría esperar, C. bolteae mostró mayor abundancia en pacientes con EM, independientemente de que se encuentren tratados o no con IFN. También dentro de los Clostridiales, hemos puesto de manifiesto un aumento en la abundancia de Faecalibacterium sp. en pacientes con EM. Por el contrario, no hemos encontrado diferencias en Faecalibacterium prausnitzii, especie que se ha descrito como protectora en diversas enfermedades inflamatorias24-27, por lo que no parece que esta última bacteria esté implicada en la génesis o desarrollo de la EM. El resto de Clostridiales que han presentado diferencias en nuestro estudio entre los pacientes con EM pertenecen al Clostridium Cluster XIVa, productor de butirato y que, por tanto, se ha relacionado con un ambiente antiinflamatorio y con la promoción de la salud23,28. Este hecho puede explicar su reducción entre los pacientes enfermos respecto al grupo control en nuestras muestras, independientemente de que los pacientes reciban o no tratamiento. Para terminar con las especies pertenecientes a Firmicutes, cabe destacar las 2 pertenecientes al orden Selenomonadales, y a la familia Veillonellaceae en que se observan un cambio significativo en la abundancia entre los sujetos sanos y los pacientes sin tratamiento, pero cuya abundancia es similar a la de los sujetos tratados. Se observa además una interesante tendencia a la significación estadística cuando se comparan los sujetos tratados y los no tratados, que se hubiese alcanzado con otro diseño de estudio de comparación directa. El papel fisiológico y la relevancia clínica de este hecho podrían ser investigados en el futuro.
En cuanto a las especies pertenecientes al filo Bacteroidetes, orden Bacteroidales, Bacteroides fragilis ha sido ampliamente estudiado como microorganismo protector en la EAE1 y ha mostrado diferente abundancia en población pediátrica entre pacientes con EM y sujetos control4. Por el contrario, y al igual que lo publicado por el grupo de Miyake2, nuestro trabajo no ha evidenciado ningún cambio en la abundancia de B. fragilis, ni en relación con la enfermedad ni con el tratamiento. Habrá que estudiar con más profundidad el papel de esta especie en la EM y el porqué de estos resultados discordantes obtenidos entre la población pediátrica y la población adulta.
En cuanto a Prevotella, a nivel de género, hemos observado una disminución notable en su abundancia en los enfermos de EM, consistente con lo descrito previamente por otros grupos de trabajo2,3,5. Estos datos apoyan la existencia de una posible relación entre la disminución de la abundancia de Prevotella y la EM, relación ya apuntada en otras enfermedades como la diabetes mellitus tipo 229 o el hígado graso no alcohólico30. A nivel de especie, los resultados obtenidos de P. copri resultan muy interesantes, puesto que en este caso, además de encontrar una disminución significativa de su abundancia entre los pacientes afectados por EM, lo cual ya había sido comunicado2,4, demostramos un aumento estadísticamente significativo en la abundancia en los pacientes tratados con IFNβ-1b en comparación con aquellos que no reciben tratamiento. El mecanismo de acción del IFNβ-1b en EM es multifactorial y en parte desconocido. El IFNβ-1b parece aumentar directamente la expresión y concentración de agentes antiinflamatorios y regula a la baja la expresión de citocinas proinflamatorias31, y como parte de los interferones tipo 1, puede interactuar con el epitelio intestinal regulando el ecosistema microbiano32. Nuestro estudio podría abrir la puerta a investigar si el IFNβ1b se puede asociar a cambios en la composición en la MI que resulten en efectos antiinflamatorios, los cuales podrían explicar, en parte, su mecanismo de acción disminuyendo los brotes y la progresión de la EM.
Dentro del filo Actinobacteria se encuentra encuadrado el género Bifidobacterium, algunas de cuyas especies forman parte del VSL#3, combinación de probióticos comercializada con diversos usos33, cuya disminución se ha relacionado con alergias, enfermedad inflamatoria intestinal, síndrome del colon irritable, cáncer colorrectal y diarreas34. La administración de algunas bacterias de este género ha demostrado mejoría en EAE mediada por IL-10 y Treg1. Sin embargo, en este estudio se muestra que la abundancia de B. longum aumenta en pacientes con EM frente a controles sanos. Los resultados son por tanto opuestos a los mostrados en modelos animales, pero consistentes con lo anteriormente publicado en EM a nivel del género Bifidobacterium por los grupos de Tremlett en población pediátrica4 y Miyake, si bien este último sin alcanzar significación estadística2. El papel exacto de este género y de B. longum en particular en el contexto de la EM necesita por tanto de mayor evaluación.
Finalmente, en lo referente a Archaea, dominio relacionado con la inflamación (a través de la producción de TNF), con enfermedad inflamatoria intestinal35, la obesidad y con sus alteraciones metabólicas36, hemos encontrado una disminución de su abundancia en los pacientes con EM respecto al grupo control. En contraste con nuestros resultados, Jangi et al. (2016) observaron un aumento de Methanobrevibacteriaceae (género incluido en el filo Euryarchaeota), así como de la exhalación de metano en pacientes con EM frente a controles sanos5. Estas discrepancias podrían explicarse por las diferencias en las características de los pacientes incluidos en ambos estudios (especialmente en términos de duración de la enfermedad), abriendo la puerta a futuras investigaciones.
A pesar de los interesantes hallazgos que aquí comunicamos, es obvio que nuestro estudio presenta algunas limitaciones, como el número de individuos incluidos en el estudio, o la ausencia de un consenso acerca de los controles más adecuados para el estudio de la microbiota4. Sin embargo, el número de sujetos en estudio no difiere en demasía respecto a los trabajos realizados con metagenómica en individuos con EM2-6,9. Otras limitaciones son la ausencia de control sobre algunos factores que impactan sobre la MI, como por ejemplo el estrés o la dieta. Además, dado el carácter descriptivo de este estudio, no nos permite establecer causalidad, y por tanto, qué fue antes, si el huevo o la gallina, es decir, los cambios en la MI o el desarrollo de la EM, ni afirmar que los cambios observados en los pacientes tratados se deben al IFNβ-1b.
No obstante, nuestro estudio contribuye a ampliar la fotografía de lo que ocurre con la MI en los pacientes con EM, siendo el primer estudio llevado a cabo en España y en países mediterráneos (con hábitos de vida y dietéticos muy diferentes a los grupos de pacientes estudiados hasta ahora). Estas diferencias son de gran valor, ya que permitirán el diseño de futuros estudios que aborden la implicación de la microbiota en la fisiopatología de la enfermedad, su posible papel como marcador pronóstico o diferentes estrategias para valorar el resultado del restablecimiento de la disbiosis. Además, el hecho de que los pacientes tratados con IFNβ-1b presenten un perfil de MI similar al de pacientes control, y diferenciado de los pacientes sin tratamiento, es de gran valor y podría abrir la puerta a futuros trabajos que estudien si las mejorías observadas en los pacientes tras el tratamiento podrían ser debidas, al menos en parte, a la mencionada influencia sobre la composición de la MI. Todo ello abre la puerta a futuros estudios que traten de establecer relaciones de causalidad entre las alteraciones en la MI y la EM, así como la influencia de los tratamientos, permitiendo con ello explorar si la corrección de la disbiosis asociada a la EM podría establecerse como una posible estrategia en la prevención y tratamiento de esta enfermedad.
FinanciaciónEste trabajo ha sido financiado por el servicio de neurología del Hospital San Pedro, La Rioja, España, y la Fundación Rioja Salud (http://www.fundacionriojasalud.org/), los cuales no han tenido ningún papel en el diseño del estudio, la recolección y análisis de los datos, la decisión de publicar ni la preparación del manuscrito.
Conflicto de interesesNinguno de los autores presenta conflicto de intereses relacionados con esta publicación.
Nos gustaría dar las gracias a todos los sujetos participantes en este estudio. Así mismo agradecemos mucho la excelente asistencia técnica prestada por María Jesús Villanueva-Millán y los sabios consejos en materia estadística prestados por Enrique Ramalle Gomara.