Las comunidades microbianas de las colonias de las abejas melíferas contribuyen en la defensa frente a patógenos. El objetivo del presente trabajo fue aislar, identificar y examinar el efecto de la liofilización de bacterias lácticas y bifidobacterias provenientes del intestino de abejas nodrizas y del pan de abejas de colonias de Apis mellifera. A partir del contenido intestinal se realizaron cultivos bacterianos, que posteriormente fueron identificados, secuenciados y liofilizados. Se evaluó su antagonismo cruzado. La secuenciación del gen 16S del ARNr según Sanger mostró los siguientes porcentajes de identidad: cepa MC3, 100% con Bifidobacterium choladohabitans; cepa PP2B, 99,16% con Enterococcus faecium; cepa PP1, 99,49% con Lacticaseibacillus spp.; cepa PP1B, 99,32% con Lacticaseibacillus spp. No se evidenció antagonismo cruzado y hubo buena estabilidad y conservación de las cepas con la liofilización. Este es el primer reporte de aislamiento de B. choladohabitans de intestino de abejas melíferas en Argentina; además, asocia la presencia de E. faecium al pan de abejas.
The microbial communities within honey bee colonies contribute to the defense against pathogens. The goal of this study was to isolate, identify, and lyophilize lactic acid bacteria and bifidobacteria from the gut of nurse bees and bee bread in Apis mellifera colonies. Bacterial cultures from the intestinal content were conducted, and subsequently identified, sequenced, and lyophilized. Cross-antagonism among them was also assessed. Studies based on 16 S rRNA gene Sanger sequencing revealed that the MC3 strain had 100% identity with Bifidobacterium choladohabitans, the PP2B strain showed 99.16% similarity with Enterococcus faecium, while the PP1 strain exhibited 99.49% similarity with Lacticaseibacillus sp. and the PP1B strain showed 99.32% similarity with Lacticaseibacillus sp. There was no evidence of cross-antagonism among the strains, and the lyophilization process showed good stability and conservation. This is the first report of the isolation of B. choladohabitans from honey bee gut in Argentina, and also associates the presence of E. faecium with bee bread.
La abeja melífera es el principal polinizador gestionado y varios cultivos destinados a la producción de alimentos en Argentina requieren de polinizadores de manera obligada3. El paulatino despoblamiento registrado en colonias de abejas melíferas en diferentes regiones del mundo, asociado al efecto de varios estresores ambientales, ha preocupado a la comunidad científica y en los últimos años, las investigaciones se han focalizado en aspectos relacionados con la inmunidad de las abejas.
La microbiota es esencial para la salud de las abejas por sus efectos metabólicos y su función moduladora del sistema inmunológico proporcionando una barrera frente a microorganismos patógenos15. Según Daisley et al.7 el sistema digestivo de las abejas melíferas contiene los siguientes géneros bacterianos predominantes, Bombilactobacillus (previamente conocido como Lactobacillus Firm-4 phylotype), Lactobacillus (Firm-5 phylotype), Bifidobacterium, Gilliamella, Snodgrassella, Frischella,Bartonella y Commensalibacter. Algunas de las bacterias del tracto digestivo son adquiridas por transmisión horizontal desde las flores y se encuentran presentes en el pan de abejas, siendo las responsables de la fermentación del polen, los géneros reportados en la bibliografía son Oenoccus spp., Bifidobacterium spp. y Lactobacillus spp15. El efecto beneficioso de algunas bacterias en la salud y la productividad de las colonias de las abejas ha sido demostrado, por ejemplo, las bacteriocinas producidas por Enterococcus faecium mostraron actividad inhibitoria frente a Paenibacillus larvae, agente causal de la enfermedad de la cría conocida como Loque Americana8. Además, la administración en colonias de abejas de Lactobacillus johnsonii CRL1647, aislado del intestino de Apis mellifera, redujo la prevalencia de nosemosis, una enfermedad de las obreras adultas2. Las bacterias asociadas a las abejas melíferas pueden transmitirse ambientalmente14, por lo que la composición del paisaje y los recursos florales disponibles afectaría directamente a los microorganismos beneficiosos de las colonias5. El avance en el conocimiento de la microbiota de las abejas melíferas en diferentes regiones del país es de relevancia no solo por su importancia en sus funciones fundamentales en el hospedador, sino porque estos microorganismos tienen un potencial biotecnológico para el desarrollo de bioproductos. El objetivo de este trabajo fue aislar, identificar, liofilizar bacterias lácticas y bifidobacterias provenientes del intestino de A. mellifera y de pan de abejas de las colonias. Se realizaron pruebas de antagonismo cruzado entre las cepas en estudio para evaluar la factibilidad de futuras aplicaciones de la comunidad bacteriana en apicultura.
Las muestras fueron obtenidas en primavera del apiario perteneciente al Área de Producción Apícola, Departamento de Producción Animal, ubicado en el Campus Universitario (37° 19́ S, 59° 07́ O) de la Facultad de Ciencias Veterinarias, UNCPBA, Tandil, Bs. As., Argentina. Se muestrearon 20 abejas nodrizas y 1g de pan de abejas de los cuadros de la cámara de cría. Se extrajeron los intestinos en condiciones de asepsia y se realizó un homogenato11. El contenido intestinal se sembró en agar MRS en microaerofilia a 37°C durante 48h, y en MRS con cisteína y medio (Britania, Argentina) modificado en anaerobiosis, 37°C durante 48h para bifidobacterias. El pan de abejas se diluyó al 10% en agua peptonada (p/v). Se sembró en agar MRS se incubó en microaerofilia a 37°, durante 48h. De cada placa de cultivo se replicaron colonias características, que se sometieron a un primer screening mediante tinción de Gram, tinción de esporas y pruebas de catalasa y oxidasa, para seleccionar aquellas que presentaran las características fenotípicas que definen a Lactobacillus spp. y Bifidobacterium spp.
Para diferenciar los géneros Lactobacillus y Bifidobacterium se utilizó la técnica de PCR, usando primers generales. Una vez identificados estos géneros, se enviaron muestras para su secuenciación en un laboratorio de referencia. Para la técnica de PCR, se extrajo el ADN de un cultivo overnight, utilizando kits específicos. Los primers utilizados fueron: g-Bifid-F (CTCCTGGAAACGGGTGG) y g-Bifid-R(GGTGTTCTTCCCGATATCTACA) y LbLMA1-rev (5́-CTC AAA ACT AAA CAA AGT TTC-3́) y R16-1 (5́-CTT GTA CAC ACC GCC CGT CA-3́). La amplificación se realizó en un termociclador marca Ivema T-17, con un programa específico. Los productos de amplificación se visualizaron usando electroforesis en gel de agarosa al 2% y visualizados en un transiluminador con luz UV.
Posteriormente, las cepas que fueron identificadas como Lactobacillus spp.; Bifidobacterium spp. y una que fue caracterizada fenotípicamente como coco gram positivo fueron secuenciadas en el servicio de identificación taxonómica del Laboratorio de Alimentos Stamboulian (CABA, Argentina), utilizando un secuenciador AB 3500 Genetic Analyzer Appl. Los cebadores de oligonucleótidos (P13P REVERSE, 5’-AGGCCCGGGAACGTATTCAC-3’, y PSL FORWARD, 5’-AGGATTAGATACCCTGGTAGTCCA-3’) se utilizaron para amplificar la región variable (VI) del gen de ARN ribosómico 16S. Las secuencias obtenidas se analizaron utilizando el algoritmo de búsqueda BLAST (The Local Alignment Search Tool Basic) utilizando la base de datos NCBI (http://www.ncbi.nlm.nih.gov/BLAST), la fecha de búsqueda se realizó el 14/08/2023. Las cepas secuenciadas, fueron liofilizadas mediante la técnica de Jalali et al.10, con un medio de protección compuesto de 0,8g de lactosa; 0,6g de leche en polvo; en 10ml de agua destilada. Se cuantificó el rendimiento del proceso y la viabilidad mediante el recuento de las UFC/g utilizando la técnica de recuento en placa.
Para la prueba de antagonismo cruzado se realizó un cultivo overnight. Se estandarizó el cultivo hasta llegar a uno en la escala McFarland. Se utilizó la técnica de difusión en agar por pocillos4, utilizando MRS con cisteína para asegurar el correcto crecimiento de todas las cepas seleccionadas. La actividad inhibitoria entre las cepas se determinó observando la presencia o ausencia de halos de inhibición. Para esta prueba se utilizaron las cepas secuenciadas y Weisellia paramesenteroides (K7) previamente aislada de pan de abejas por nuestro grupo de investigación13.
Se aislaron en total 11 microorganismos. Los resultados de la secuenciación mostraron que la cepa MC3 tuvo un 100% de identidad con Bifidobacterium choladohabitans y la cepa PP2B un 99,16% de identidad con E. faecium. Dentro de los aislamientos considerados Lactobacillus, la cepa PP1 mostró un 99,49% de identidad con Lacticaseibacillus rhamnosus cepa JCM 113616 y 99,49% de identidad con Lacticaseibacillus paracasei cepa NBRC 15889, por lo cual solo se pudo identificar como Lacticaseibacillus spp. De manera similar, la cepa PP1B mostró 99,32% de identidad con L. paracasei cepa ATCC 25302 y 99,32% de identidad con L. rhamnosus cepa NBRC 3425, por lo que se la identificó como Lacticaseibacillus spp.
El grupo Lacticaseibacillus está conformado por cepas de especies que presentan características filogenéticas muy cercanas, por lo tanto, son difíciles de diferenciar. El análisis del gen 16S ARNr incluso no permitió diferenciarlas porque comparten más del 98-99% de identidad entre ellas, por lo tanto, para diferenciarlas genéticamente en próximos estudios se deberán utilizar primers específicos de la especie o técnicas específicas como las mencionadas por Hill et al.9. De acuerdo con los resultados, hasta el momento solo se puede concluir que estas cepas se identificaron como cepas del género Lacticaseibacillus spp. Todas las secuencias se introdujeron en la base de datos GenBank (tabla 1).
Caracterización fenotípica y genotípica de las bacterias aisladas del intestino y del pan de abejas de colonias de A. mellifera
| Origen intestino | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| PCR | Secuenciación | ||||||||
| CEPA | Tinción de Gram | Catalasay Oxidasa | Producción de esporas | Lactobacillus | Bifidobacterium | Especie tipo más relacionada.Cepa tipo | Identidad (%) | 16S ARNr(pb) | AccesoGenbank |
| 1(I-3 2) | Coco (+) | (–) | (–) | (-) | |||||
| 2(I-2 1) | Coco (+) | (–) | (–) | (-) | |||||
| 3(MC1) | Bacilos (+) | (–) | (–) | (–) | (-) | ||||
| 4(MC2) | Bacilos (+) | (–) | (–) | (–) | (-) | ||||
| 5(MC3) | Bacilos (+) | (–) | (–) | (–) | (+) | Bifidobacterium choladohabitans | 100% | 595 | OR119753 |
| 6(V1) | Cocos (+) | (–) | (–) | (-) | |||||
| Origen pan de abeja | |||||||||
| PCR | Secuenciación | ||||||||
| CEPA | Tinción de Gram | Catalasa y oxidasa | Producción de esporas | Lactobacillus | Bifidobacterium | Especie tipo másrelacionada.Cepa tipo | Identidad(%) | 16S ARNr(pb) | Acceso |
| 7(PP1) | Bacilos (+) | (–) | (–) | (+) | (–) | Lacticaseibacillus spp. | 99,49% | 597 | OR115616 |
| 8(PP2) | Bacilos (+) | (–) | (–) | (–) | (–) | ||||
| 9(PP3) | Bacilos (+) | (–) | (–) | (–) | (–) | ||||
| 10(PP1B) | Bacilos (+) | (–) | (–) | (+) | (–) | Lacticaseibacillus spp. | 99,32% | 597 | OR115699 |
| 11(PP2B) | Cocos (+) | (–) | (–) | (–) | Enterococcus faecium | 99,16% | 594 | OR085843 | |
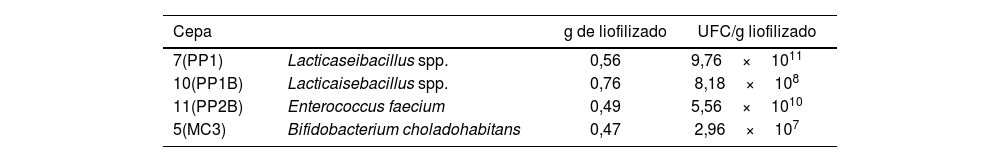
El recuento bacteriano obtenido de la liofilización fue similar al reportado por Strasser et al.16 para E. faecium. Para B. choladohabitans y Lacticaseibacillus spp. los recuentos obtenidos fueron menores en 2órdenes de magnitud de la escala logarítmica (tabla 2) que los reportados por Vinderola et al.17 para Bifidobacterium animalis y Chen et al.6 para Lactobacillus bulgaricus. Estas diferencias se podrían atribuir a que estos autores trabajaron con otras especies y las distintas cepas difieren en su sensibilidad al método de secado utilizado. Todas las cepas se mantuvieron viables 3años posliofilización, mostrando una buena estabilidad y conservación, característica importante para resguardar su potencial biotecnológico. No se evidenció antagonismo cruzado entre las cepas (fig. 1), de modo que es factible su futura aplicación como comunidad bacteriana. Sin embargo, futuros estudios son necesarios para determinar la sinergia y su efecto benéfico para el uso en apicultura.
Recuento bacteriano expresado en UFC/g luego del liofilizado de las cepas
| Cepa | g de liofilizado | UFC/g liofilizado | |
|---|---|---|---|
| 7(PP1) | Lacticaseibacillus spp. | 0,56 | 9,76×1011 |
| 10(PP1B) | Lacticaisebacillus spp. | 0,76 | 8,18×108 |
| 11(PP2B) | Enterococcus faecium | 0,49 | 5,56×1010 |
| 5(MC3) | Bifidobacterium choladohabitans | 0,47 | 2,96×107 |
Cultivo de la cepa MC3 en el césped de la placa y en los pocillos las cepas en estudio (PP1, PP1B y PP2B) y la cepa K7 aislada previamente.
Fuente: Libonatti et al.13.
Este es el primer reporte de aislamiento de B. choladohabitans a partir de intestino de abejas melíferas en Argentina y que asocia la presencia de E. faecium al pan de abejas. Aislamientos previos realizados en el noroeste del país reportaron la presencia del enterococo en el intestino y en las mieles1. La presencia de esta bacteria en los diferentes ambientes de las colonias evidenciaría que integra el núcleo bacteriano de las colonias de abejas melíferas. No obstante, se requieren estudios de prevalencia y estacionalidad para establecer si esta bacteria puede ser considerada parte del microbioma central de la colmena.
El aislamiento del género Lacticaseibacillus, ratifica que el mismo puede ser considerado como parte del microbioma central del intestino y lo que algunos autores sostienen que es un género predominante no solo en las abejas melíferas, sino en otras especies de abejas12.
Conflicto de interesesLos autores declaran que no tienen conflicto de interés.
Este trabajo fue financiado por SECAT (Secretaría de Ciencia, Arte y Tecnología de la Universidad Nacional del Centro de la Provincia de Buenos Aires), Proyecto 03 PIO 27H.