Escherichia coli productor de toxina Shiga (STEC) es un patógeno transmitido por alimentos que puede causar diarrea acuosa, diarrea sanguinolenta (DS) y síndrome urémico hemolítico (SUH). El objetivo de este estudio fue determinar las características fenotípicas y genotípicas de cepas STEC aisladas de niños con DS y SUH atendidos en un hospital pediátrico de la ciudad de La Plata en el período 2006-2012 y establecer la relación clonal de los aislamientos O157:H7 mediante electroforesis de campo pulsado. El porcentaje de muestras positivas fue de 4,9 y 39,2% en los pacientes que presentaron DS y SUH, respectivamente. Se aislaron 77 cepas STEC de 10 serotipos distintos, con el 100% de recuperación de colonias. El serotipo más frecuente fue O157:H7 (71,4%), seguido por O145:NM (15,6%). El 98,2% de los aislamientos O157:H7 correspondió al biotipo C y fue sensible a los antibióticos ensayados. Todos esos aislamientos presentaron el genotipo stx2, eae, fliCH7, ehxA, iha, efa, toxB, lpfA1-3 y lpfA2-2. Al estudiar la relación clonal de las cepas O157:H7, se identificaron un total de 42 patrones con al menos un 88% de similitud y se establecieron 6 clústeres que agruparon cepas con perfiles idénticos. Los aislamientos eae negativos pertenecieron a los serotipos O59:H19, O102:H6, O174:NM y O174:H21. Las cepas O59:H19 y O174:H21 fueron positivas para el gen aggR. Este estudio muestra que en la ciudad de La Plata y alrededores circulan STEC de diferentes serotipos y genotipos. A pesar de la diversidad genética observada entre los aislamientos O157:H7, algunos fueron indistinguibles por las técnicas de subtipificación utilizadas.
Shiga toxin-producing Escherichia coli (STEC) is a foodborne pathogen that can cause watery diarrhea, bloody diarrhea (BD), and hemolytic uremic syndrome (HUS). The objective of this study was to determine the phenotypic and genotypic profiles of STEC strains isolated from children with BD and HUS treated at a pediatric hospital in the city of La Plata in the period 2006-2012, and to establish the clonal relationship of O157:H7 isolates by pulsed field electrophoresis. The percentage of positive samples was 4.9% and 39.2% in patients with BD and HUS, respectively. Seventy-seven STEC strains from 10 different serotypes were isolated, with 100% colony recovery, O157:H7 being the most frequent (71.4%) serotype, followed by O145:NM (15.6%). An average of 98.2% of O157:H7 isolates belonged to biotype C and were sensitive to all the antibiotics tested. All of them (100%) carried genotype stx2, eae, fliCH7, ehxA, iha, efa, toxB, lpfA1-3 and lpfA2-2. When the clonal relationship of the O157:H7 strains was studied, a total of 42 patterns with at least 88% similarity were identified, and 6 clusters with identical profiles were established. The eae-negative isolates belonged to serotypes O59:H19, O102:H6, O174:NM and O174:H21. The strains O59:H19 and O174:H21 were positive for the aggR gene. This study shows that STEC of different serotypes and genotypes circulate in the city of La Plata and surroundings. Despite the genetic diversity observed between the O157:H7 isolates, some were indistinguishable by the subtyping techniques used.
Las enfermedades diarreicas agudas constituyen uno de los problemas de salud pública más serios en los países en vías de desarrollo, con altas tasas de morbimortalidad, y son la segunda causa de mortalidad en menores de 5 años37.
Los agentes enteropatógenos responsables de las diarreas agudas son múltiples y pueden ser de origen viral, bacteriano o parasitario; son frecuentes las asociaciones de 2o más de ellos. Los virus, principalmente los del género Rotavirus, causan del 70 al 80% de las diarreas infecciosas y las bacterias ocasionan el 10-20% de los casos. Entre los agentes bacterianos habituales se encuentran Campylobacter spp., Shigella spp., Salmonella no typhi, Clostridium difficile y Escherichia coli diarreigénicos, y también otros40.
E. coli productor de toxina Shiga (STEC) es un patógeno zoonótico principalmente transmitido por alimentos. Puede causar una variedad de manifestaciones clínicas, desde infecciones asintomáticas o diarrea leve a moderada hasta enfermedad grave, como colitis hemorrágica y síndrome urémico hemolítico (SUH)35. Las cepas STEC pertenecen a un amplio rango de serotipos2, aunque E. coli O157:H7 es el serotipo más frecuentemente asociado a grandes brotes y casos esporádicos de colitis hemorrágica y SUH en todo el mundo. Sin embargo, otros serogrupos de STEC también han sido reconocidos por su potencial patogénico, ya que son aislados frecuentemente de casos de enfermedad humana24.
El principal factor de virulencia de las cepas STEC son las denominadas toxinas Shiga, codificadas por los genes stx38. La adhesina intimina, codificada por el gen eae, está involucrada en la adherencia íntima de las bacterias a los enterocitos33. Sin embargo, las cepas STEC que carecen de este gen también pueden ocasionar enfermedad en el ser humano. Si bien no se ha establecido aún qué combinación de marcadores definen que una cepa STEC resulte patógena, la presencia de los genes stx/eae se asocia con un riesgo de enfermedad más grave14.
En Argentina, el SUH posentérico es endémico y representa la principal causa de insuficiencia renal aguda e hipertensión arterial en lactantes y niños y la segunda causa de insuficiencia renal crónica y de trasplante renal25. Las cepas STEC fueron identificadas como el principal agente etiológico; sin embargo, no son rutinariamente diagnosticadas en todos los laboratorios de microbiología34.
El objetivo del presente trabajo fue determinar mediante un análisis retrospectivo la frecuencia de aparición de STEC en muestras fecales de niños con diagnóstico clínico de diarrea aguda sanguinolenta y SUH atendidos en el Hospital Especializado en Pediatría Sor María Ludovica de la ciudad de La Plata. Asimismo, se buscó determinar el perfil fenotípico y genotípico de las cepas STEC recuperadas y establecer la relación clonal de las cepas de E. coli O157:H7 mediante electroforesis en gel de campo pulsado (PFGE).
Materiales y métodosMuestras estudiadasDesde el 1 de marzo de 2006 hasta el 30 de diciembre de 2012 se estudiaron 1.051 muestras de materia fecal (MF) obtenidas por evacuación espontánea (una por paciente). Estas comprendieron 107 muestras de pacientes con diagnóstico de SUH y 944 muestras de pacientes con diarrea aguda, de los cuales 731 (77,4%) presentaron diarrea sanguinolenta (DS)y 213 (22,6%) diarrea no sanguinolenta, por observación microscópica al momento de la recolección.
Para la selección de los casos se estipularon las siguientes condiciones: persona previamente sana menor de 14 años con diagnóstico presuntivo de DS o caso clínicamente confirmado de SUH posentérico, con no más de 14 días de evolución al momento de la toma de muestra. Los criterios de exclusión fueron los siguientes: a) niños con diarrea de más de 14 días de evolución; b) antecedentes de enfermedad intestinal inflamatoria crónica; c) inmunocomprometidos; d) pacientes con infección nosocomial; e) SUH familiar, recurrente, secundario, no infeccioso o asociado con infección por Streptococcus pneumoniae. La demora promedio en la toma de muestras para el diagnóstico microbiológico en aquellos pacientes que desarrollaron SUH fue de 7 días (intervalo: 1-15 días) desde el comienzo de los síntomas.
Cultivo microbiológico primario y aislamiento de STECLas muestras de MF fueron procesadas por técnicas microbiológicas estandarizadas antes de las 2h de recogidas o conservadas en medio de transporte Cary-Blair y refrigeradas a 4°C hasta su procesamiento22. A partir de las zonas de crecimiento confluente, se realizó la detección de los genes stx1, stx2 y rfbO157 por PCR múltiple30. De cada muestra positiva se analizaron 10 colonias típicas de E. coli, hasta lograr identificar la colonia stx positiva. Las cepas fueron conservadas a −70°C para su posterior caracterización.
Caracterización fenotípica de los aislamientos de STECLos aislamientos fueron identificados por pruebas bioquímicas convencionales y utilizando el sistema automatizado Vitek-2 Compact (bioMérieux, Mercy ĺEtoile, Francia). La identificación de los antígenos somáticos y flagelares fue realizada por aglutinación con antisueros específicos provistos por el Instituto Nacional de Producción de Biológicos-ANLIS Dr. Carlos G. Malbrán. Los aislamientos de E. coli O157:H7 fueron clasificados en 4 biotipos, según la capacidad de fermentar los azúcares rafinosa, dulcitol y ramnosa29. La producción de Stx se determinó mediante ELISA (PremierTM EHEC, Meridian Diagnostics, Cincinnati, Ohio, EE. UU.) y la de enterohemolisina (EHEC-HlyA) fue determinada en placas de agar sangre ovina3.
La sensibilidad antimicrobiana se determinó según el Clinical and Laboratory Standards Institute (CLSI)10. Se ensayaron los siguientes antimicrobianos: ampicilina (10μg), amoxicilina/ácido clavulánico (20/10μg), cefalotina (30μg), cefoxitina (30μg), ceftazidima (30μg), cefotaxima (10μg), piperacilina/tazobactama, imipenem (10μg), meropenem (10μg), gentamicina (10μg), amicacina (30μg), ácido nalidíxico (30μg), ciprofloxacina (5μg), trimetoprima-sulfametoxazol (1,25/23,75μg), nitrofurantoína (300μg), fosfomicina (200μg), tetraciclina (30μg) y colistina (10μg).
Caracterización genotípica de los aislamientos de STECTodos los aislamientos de STEC fueron analizados por PCR para la detección de los genes eae, fliCH7, ehxA, saa, iha, efa/lifA, toxB, lpfA1, lpfA2, cdt-V, subAB y astA19,20. Los genes aggR y aaiC fueron detectados por PCR en tiempo real16.
Subtipificación molecular de los aislamientos de STECEl análisis de los subtipos de stx1 se realizó por PCR, según metodología previamente publicada44. La identificación de los subtipos de stx2 fue realizada por PCR-RFLP49 y por PCR múltiple, según metodología previamente publicada44. La técnica de PFGE se desarrolló utilizando el protocolo PulseNet de 24-48h para E. coli7 O157:H7. El ADN de cada cepa contenido en bloques de agarosa fue digerido con 30 U de la enzima de restricción XbaI (ThermoScientific) a 37°C durante 3h. Los perfiles moleculares obtenidos por XbaI-PFGE fueron documentados utilizando el sistema de adquisición de imágenes MaestroGen slider® imager (Maestrogen Inc., Nevada, EE. UU.). Las imágenes en formato TIFF fueron analizadas con el software BioNumerics versión 6.6 (Applied Maths, Sint-Martens-Latem, Bélgica). La relación entre los perfiles fue estimada mediante la proporción de bandas compartidas aplicando el coeficiente de similitud de Dice y generando dendrogramas basados en el método unweighted pair group method with arithmetic mean (UPGMA). Las cepas fueron agrupadas en clústeres cuando mostraron idéntico patrón XbaI-PFGE (100% de similitud).
ResultadosSe encontraron 77 (7,33%) muestras positivas para al menos uno de los siguientes genes: stx1, stx2 y rfbO157. A partir de esas muestras fueron aisladas 77 cepas STEC. Se recuperaron 35 cepas de los pacientes con diagnóstico de DS (4,8%) y 42 de los pacientes con SUH (39,2%).
Entre los pacientes infectados, el grupo etario con mayor proporción de casos fue el de niños menores de 5 años (75,3%); la edad promedio dentro de ese grupo de afectados fue de 23,5 meses. El grupo de 5 a 9 años representó el 16,9%, mientras que el 2,6% fueron del grupo de 10 a 14 años. El 5,2% restante fueron casos notificados sin especificar la edad. Para todos los grupos, el serotipo O157:H7 fue el de mayor prevalencia.
La media de edad del grupo de pacientes con DS fue 45,3 meses (intervalo: 7-152 meses) y la del grupo con SUH fue 29,1 meses (intervalo: 6-142 meses). El 62% de los pacientes con SUH tenían menos de 2 años y la mayor incidencia se observó en los niños de un año. En cambio, el 63% de los pacientes con DS eran mayores de 2 años; la mayor incidencia se observó en el grupo de 5 a 9 años.
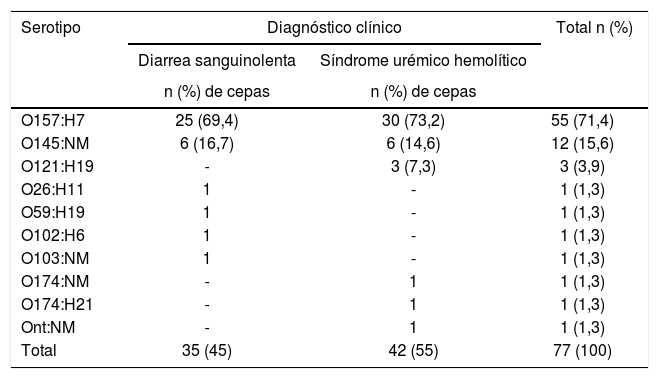
Caracterización fenotípica de los aislamientosSe serotipificaron 76 cepas STEC (98,7%). De ellas, 55 (71,4%) fueron O157:H7 biotipo C, solo una cepa perteneció al biotipo D y 22 cepas (28,6%) fueron no O157. Dentro del grupo no O157, el serotipo O145:NM fue el de mayor prevalencia, seguido por O121:H19. La frecuencia de serotipos de las cepas según el diagnóstico se detalla en la tabla 1.
Frecuencia de serotipos según diagnóstico
| Serotipo | Diagnóstico clínico | Total n (%) | |
|---|---|---|---|
| Diarrea sanguinolenta | Síndrome urémico hemolítico | ||
| n (%) de cepas | n (%) de cepas | ||
| O157:H7 | 25 (69,4) | 30 (73,2) | 55 (71,4) |
| O145:NM | 6 (16,7) | 6 (14,6) | 12 (15,6) |
| O121:H19 | - | 3 (7,3) | 3 (3,9) |
| O26:H11 | 1 | - | 1 (1,3) |
| O59:H19 | 1 | - | 1 (1,3) |
| O102:H6 | 1 | - | 1 (1,3) |
| O103:NM | 1 | - | 1 (1,3) |
| O174:NM | - | 1 | 1 (1,3) |
| O174:H21 | - | 1 | 1 (1,3) |
| Ont:NM | - | 1 | 1 (1,3) |
| Total | 35 (45) | 42 (55) | 77 (100) |
Ont: antígeno O no tipable; NM: no móvil.
La producción de toxina Shiga determinada mediante ELISA se demostró en 76 de las 77 cepas estudiadas. La única excepción fue la cepa O102:H6, asociada a un caso de diarrea leve. La actividad hemolítica, compatible con la presencia de EHEC-Hly, fue detectada en 73 cepas (94,8%). El fenotipo α-hemolítico no fue detectado en ninguna de las cepas estudiadas. Fueron sensibles a todos los antibióticos ensayados 74 cepas (96%). Tres aislamientos fueron resistentes a un solo antibiótico: O157:H7 (AMPr), O145:NM (TETr) y Ont:NM (TMSr).
Caracterización genotípica de los aislamientosLos perfiles genotípicos de las cepas aisladas de pacientes con DS fueron los siguientes: stx2/rfbO157 (68,6%), stx2 (25,8%), stx1/stx2 (2,8%) y stx1/stx2/rfbO157 (2,8%). Los de las cepas aisladas de pacientes con SUH fueron el stx2/rfbO157 (71,5%) y el stx2 (28,5%).
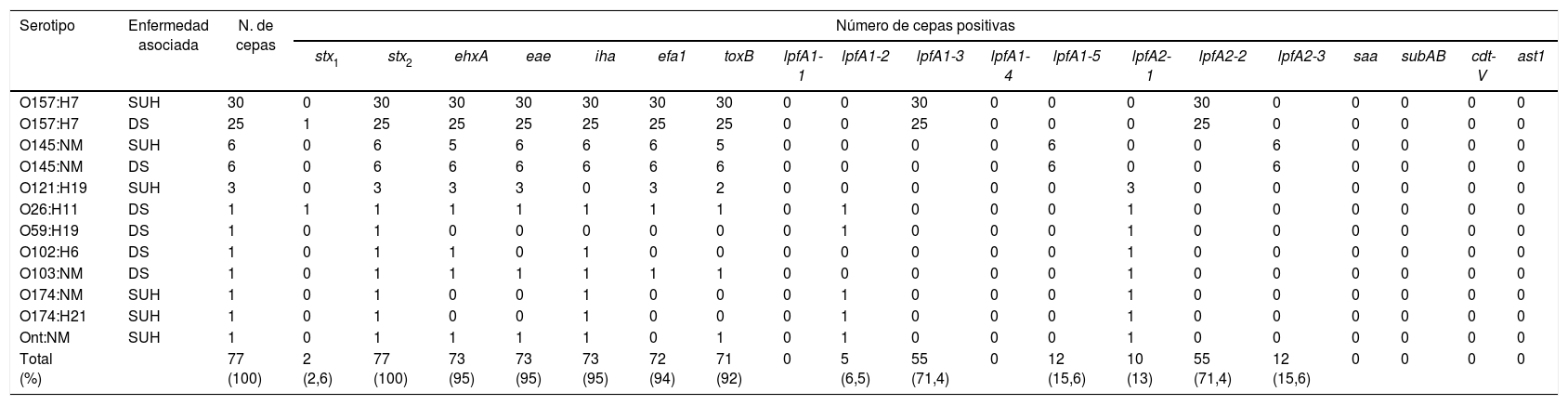
Fueron eae positivas 73 cepas (95%). Las cepas eae negativas pertenecieron a los serotipos O59:H19, O102:H6, O174:NM y O174:H21. En estas 4cepas se ensayó la presencia de los genes aggR y aaiC, las cepas O59:H19 y O174:H21 fueron aggR positivas.
Todos los aislamientos O157:H7 fueron positivos para los genes rfbO157, fliCH7iha, efa1 y toxB. Entre los aislamientos no O157, se detectó el gen de la adhesina iha en 18 de ellos (81,8%), el efa1 en 17 (77,2%) y el toxB en 16 (72,7%). Solo la cepa O59:H19 fue negativa para los 3genes simultáneamente.
Utilizando la clasificación experimental de las distintas variantes de los genes lpfA1 y lpfA248, todas las cepas O157:H7 portaron las variantes lpfA1-3 y lpfA2-2. Las cepas O145:NM portaron las variantes lpfA1-5 y lpfA2-3. Entre los aislamientos distintos a O157:H7 y O145:NM, lpfA2-1 fue la variante más prevalente (100%), 5de esos aislamientos fueron, además, positivos a lpfA1-2. No se pudo establecer ninguna asociación entre la presencia de alguna variante en particular de lpfA y la gravedad de la enfermedad (SUH o DS). Sin embargo, se observó que los aislamientos O157:H7 y O145:NM mantuvieron la misma combinación de variantes lpfA, independientemente de la enfermedad asociada.
No se detectó la presencia de los genes saa, subAB, cdt-V ni astA en ninguna de las cepas estudiadas. La caracterización genotípica de los aislamientos se muestra en la tabla 2.
Caracterización molecular de los aislamientos STEC recuperados de pacientes con DS y SUH atendidos en el Hospital de Niños de La Plata (n=77)
| Serotipo | Enfermedad asociada | N. de cepas | Número de cepas positivas | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| stx1 | stx2 | ehxA | eae | iha | efa1 | toxB | lpfA1-1 | lpfA1-2 | lpfA1-3 | lpfA1-4 | lpfA1-5 | lpfA2-1 | lpfA2-2 | lpfA2-3 | saa | subAB | cdt-V | ast1 | |||
| O157:H7 | SUH | 30 | 0 | 30 | 30 | 30 | 30 | 30 | 30 | 0 | 0 | 30 | 0 | 0 | 0 | 30 | 0 | 0 | 0 | 0 | 0 |
| O157:H7 | DS | 25 | 1 | 25 | 25 | 25 | 25 | 25 | 25 | 0 | 0 | 25 | 0 | 0 | 0 | 25 | 0 | 0 | 0 | 0 | 0 |
| O145:NM | SUH | 6 | 0 | 6 | 5 | 6 | 6 | 6 | 5 | 0 | 0 | 0 | 0 | 6 | 0 | 0 | 6 | 0 | 0 | 0 | 0 |
| O145:NM | DS | 6 | 0 | 6 | 6 | 6 | 6 | 6 | 6 | 0 | 0 | 0 | 0 | 6 | 0 | 0 | 6 | 0 | 0 | 0 | 0 |
| O121:H19 | SUH | 3 | 0 | 3 | 3 | 3 | 0 | 3 | 2 | 0 | 0 | 0 | 0 | 0 | 3 | 0 | 0 | 0 | 0 | 0 | 0 |
| O26:H11 | DS | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 0 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| O59:H19 | DS | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| O102:H6 | DS | 1 | 0 | 1 | 1 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| O103:NM | DS | 1 | 0 | 1 | 1 | 1 | 1 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| O174:NM | SUH | 1 | 0 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| O174:H21 | SUH | 1 | 0 | 1 | 0 | 0 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| Ont:NM | SUH | 1 | 0 | 1 | 1 | 1 | 1 | 0 | 1 | 0 | 1 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 |
| Total (%) | 77 (100) | 2 (2,6) | 77 (100) | 73 (95) | 73 (95) | 73 (95) | 72 (94) | 71 (92) | 0 | 5 (6,5) | 55 (71,4) | 0 | 12 (15,6) | 10 (13) | 55 (71,4) | 12 (15,6) | 0 | 0 | 0 | 0 | |
NM: no móvil; Ont: antígeno somático no tipable; ehxA: hemolisina de EHEC; eae: intimina; iha: adhesina homóloga a IrgA; efa: factor para la adherencia de E. coli enterohemorrágico; toxB: proteína plasmídica que participa en la adherencia de STEC; lpfA: subunidad mayor de la fimbria polar larga; saa: adhesina autoaglutinante de STEC; subAB: subtilasa; cdt-V: toxina citoletal; ast1: enterotoxina termoestable de E. coli enteroagregativa.
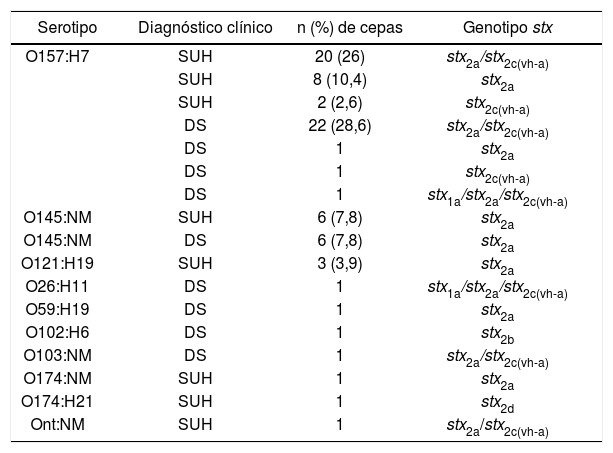
Al estudiar el genotipo de stx de las 55 cepas O157:H7, se halló que 42 de ellas (76,4%) fueron stx2a/stx2c(vh-a), este fue el subtipo predominante, independientemente de que se tratara de cepas aisladas de pacientes con DS o con SUH. En las cepas no O157, el subtipo más frecuente fue stx2a (77%) (tabla 3).
Genotipo stx de las cepas aisladas en el período de estudio (n=77)
| Serotipo | Diagnóstico clínico | n (%) de cepas | Genotipo stx |
|---|---|---|---|
| O157:H7 | SUH | 20 (26) | stx2a/stx2c(vh-a) |
| SUH | 8 (10,4) | stx2a | |
| SUH | 2 (2,6) | stx2c(vh-a) | |
| DS | 22 (28,6) | stx2a/stx2c(vh-a) | |
| DS | 1 | stx2a | |
| DS | 1 | stx2c(vh-a) | |
| DS | 1 | stx1a/stx2a/stx2c(vh-a) | |
| O145:NM | SUH | 6 (7,8) | stx2a |
| O145:NM | DS | 6 (7,8) | stx2a |
| O121:H19 | SUH | 3 (3,9) | stx2a |
| O26:H11 | DS | 1 | stx1a/stx2a/stx2c(vh-a) |
| O59:H19 | DS | 1 | stx2a |
| O102:H6 | DS | 1 | stx2b |
| O103:NM | DS | 1 | stx2a/stx2c(vh-a) |
| O174:NM | SUH | 1 | stx2a |
| O174:H21 | SUH | 1 | stx2d |
| Ont:NM | SUH | 1 | stx2a/stx2c(vh-a) |
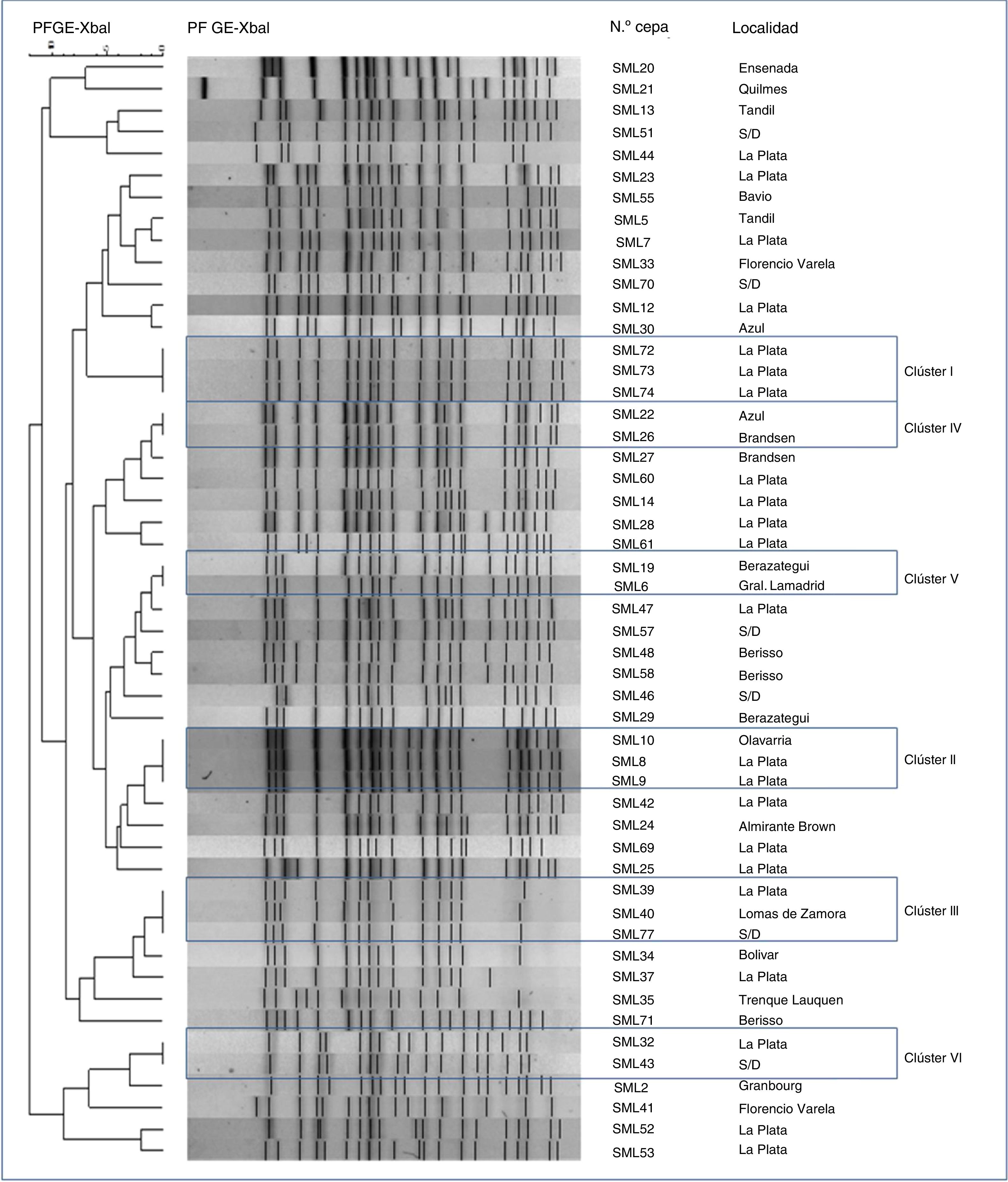
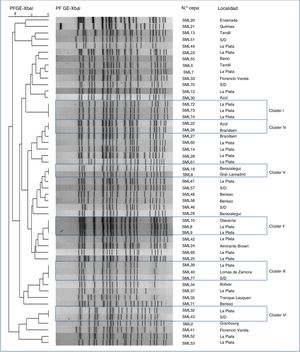
Al analizar los productos de restricción obtenidos tras digerir el genoma de 51 cepas de E. coli O157:H7 con la enzima de restricción XbaI, se encontró que se generaron 42 patrones de bandas diferentes, con al menos un 88% de similitud entre ellos. Cuatro cepas quedaron excluidas del análisis porque su ADN se degradó repetidas veces. Los patrones presentaron entre 16 y 22 bandas, con tamaños aproximados de 40 a 600 kpb. El porcentaje de similitud entre las cepas se representó en un dendrograma de homología utilizando el algoritmo UPGMA (fig. 1). De los 42 patrones de bandas obtenidos, 36 resultaron ser únicos y 15 cepas fueron agrupadas en 6 clústeres: clúster I (SML72, 73 y 74), clúster II (SML8, 9, y 10), clúster III (SML39, 40 y 77), clúster IV (SML22 y 26), clúster V (SML6 y 19) y clúster VI (SML32 y 43).
Las 3cepas agrupadas en el clúster I se aislaron de 3pacientes con DS que no requirieron internación. Estos 3pacientes vivían en la ciudad de La Plata, en un radio de 500 m y el comienzo de los síntomas difirió en 32 días. Aunque no fue posible establecer la fuente de infección en ninguno de los casos, mediante la información epidemiológica disponible se identificó como la única variable en común el consumo de cremas heladas compradas en el mismo local comercial.
Las cepas agrupadas en el clúster II fueron aisladas de 3casos de SUH ocurridos en enero de 2007, en un período de 15 días y en 2ciudades distintas, La Plata y Olavarría, distantes 330km entre sí. Sin embargo, los datos epidemiológicos fueron insuficientes para establecer un nexo entre los casos. Las cepas del clúster IV poseían idéntico genotipo stx y fueron recuperadas de pacientes que desarrollaron SUH, uno con domicilio en Brandsen y el otro en Azul, pero los datos epidemiológicos disponibles no permitieron establecer un nexo entre ellos.
Las cepas agrupadas en el clúster VI poseían idéntico genotipo stx y fueron aisladas en La Plata de un caso de diarrea con sangre y de otro caso con SUH, con 5 años de diferencia entre ellos. Las cepas agrupadas en los clústeres III y V fueron diferenciadas por el genotipo de stx.
DiscusiónMediante técnicas microbiológicas estándar y tamizaje por PCR se investigó la presencia de STEC en MF de niños con diagnóstico presuntivo de DS y SUH atendidos en el Hospital de Niños Sor María Ludovica» de la ciudad de La Plata desde el año 2006. Un análisis retrospectivo de pacientes con diarrea aguda basado en los resultados obtenidos durante 6 años mostró la presencia de STEC en el 4,8% de aquellos que presentaban DS y fue negativo el tamizaje en todos los pacientes con diarrea. Estos resultados no difieren de los obtenidos en otros estudios realizados en nuestro país, a pesar de las limitaciones en la vigilancia, y de los observados en el resto del mundo.
En países poco desarrollados, la recuperación de STEC es menor del 2% en niños pequeños con gastroenteritis9,12 y en los EE. UU. la tasa de detección de STEC va del 0 al 4,1% de las muestras enviadas a los laboratorios23.
En Argentina, Rivero et al.43 detectaron la presencia de STEC en el 10,1% (44/437) de las muestras analizadas de niños con diarrea aguda; en el 27,3% de ellos la diarrea era no sanguinolenta, a diferencia de lo que sucedió en nuestro estudio. En 2012, López et al.32 efectuaron un estudio prospectivo de 2.435 niños (edades: 0,5-15 años) con diarrea acuosa o sanguinolenta, y detectaron la presencia de STEC en el 4,1% de los pacientes que presentaron diarrea acuosa de menos de 5 días de evolución, DS de menos de 36 h de duración, o ambas. Sin embargo, la incidencia de infección por STEC fue significativamente mayor en pacientes con DS.
Al estudiar a los pacientes que desarrollaron SUH posentérico, pudimos evidenciar la presencia de STEC en el 39,2% de la cohorte analizada. Estos resultados son similares a los reportados por otros autores en nuestro país41,43 y en Chile39, pero inferiores a los obtenidos en Francia15 y en un estudio que abarcó Austria y Alemania21. Estas diferencias en las tasas de detección o recuperación de STEC pueden depender de diversos factores, entre los que se encuentran el tiempo transcurrido entre el inicio de los síntomas y la recolección de la muestra, la metodología utilizada para la detección o el aislamiento de los microorganismos, el uso de terapia antimicrobiana antes del diagnóstico certero y otros.
A partir de un estudio prospectivo, Tarr et al.46 informaron la recuperación de E. coli O157:H7 en el 91,4% de las muestras de pacientes que desarrollaron SUH posentérico cuando aquellas eran obtenidas entre los 3 y 6 días desde el inicio de la diarrea, pero la recuperación bajaba al 33% si se demoraban más de 7 días en recolectarlas. Es decir, al momento que se manifestaba el SUH, el 67% de los pacientes no tenían O157:H7 en la MF.
Otro factor importante para asociar el SUH con la infección por STEC es la metodología utilizada para la detección del microorganismo. La búsqueda de citotoxinas libres en MF, el uso de anticuerpos antitoxina Shiga o anti-LPS O157 en suero, la utilización de separación inmunomagnética como técnica de inmunoconcentración de STEC y la amplificación por PCR como técnica de tamizaje permiten aumentar sustancialmente la evidencia de infección por STEC21,41. En el presente trabajo, la combinación de procedimientos bacteriológicos y la amplificación por PCR múltiple aplicada permitieron mejorar el diagnóstico de STEC en nuestro hospital.
En un análisis retrospectivo realizado en nuestro laboratorio22 se estudiaron 7.075 pacientes con diarrea. Utilizando agar MacConkey sorbitol para la recuperación de STEC y sin efectuar tamizaje por PCR, solo se recuperaron 5 aislamientos de E. coli O157:H7 a lo largo de 3 años de estudio.
Cuando se analizó la edad de los pacientes con SUH, la media del grupo fue de 29,1 meses (intervalo: 6-142 meses), el 62% era menor de 2 años y la mayor incidencia se observó en los niños de un año. Estos hallazgos son un reflejo de la situación observada en nuestro país1. En cambio, la edad promedio del grupo de pacientes con DS fue 45,3 meses (intervalo: 7-152 meses).
Diversos autores han reportado que los pacientes menores expuestos a STEC presentan más riesgo de evolucionar a enfermedad grave4. Este fenómeno podría responder a un problema de competencia bacteriana y de inmunidad cruzada, ya que los niños mayores o los que proceden de ambientes con condiciones sanitarias deficientes a menudo presentan —o han presentado— otras infecciones intestinales causadas por microorganismos que podrían impedir o limitar el desarrollo de STEC.
Las cepas STEC caracterizadas pertenecieron a varios serotipos (tabla 1), la mayoría asociados con enfermedad grave en los humanos. E. coli O157:H7 fue el principal agente etiológico de SUH posentérico (73,2%) y el serotipo STEC fue el prevalente entre los pacientes con DS estudiados (69,4%). Este serotipo es responsable de más del 70% de los casos de SUH posentérico en Argentina34 y es el principal serotipo aislado en Chile, Japón, Canadá, EE. UU. y países de la UE11,36,39. Con respecto a STEC no O157, los serotipos implicados varían según el país, aunque ciertos serogrupos, como O26, O103, O111 y O145, son los descriptos con mayor frecuencia12,21,27. En el presente estudio, se recuperaron cepas STEC no O157 en el 28,5% de los casos y el serotipo más frecuente fue O145:NM, al igual que en el resto de nuestro país34.
Al estudiar la expresión fenotípica de las toxinas Shiga, se pudo comprobar la producción de estas en 76 de las 77 cepas estudiadas. La única excepción fue la cepa O102:H6 portadora del gen stx2b, el cual codifica una toxina que no puede ser detectada por el ensayo inmunológico utilizado17.
El presente estudio refleja una elevada sensibilidad a los antimicrobianos en las cepas STEC de origen humano circulantes en nuestra región. A pesar del uso inadecuado de antibióticos en casos de diarrea que se observa en Argentina43 —a lo que se suma el uso de diversos antibióticos como promotores de crecimiento en ganado o en medicina veterinaria45—, la presión de selección que estos ejercen no se ve reflejada en la pérdida de sensibilidad a los antimicrobianos de las cepas STEC de origen humano50. En contraste, los aislamientos europeos de STEC muestran grados variables de resistencia, y la proporción de aislamientos multirresistentes (aquellos que resisten a 3 o más antimicrobianos) es cercana al 20%36.
La vigilancia molecular de estos patógenos humanos permitió caracterizar las cepas circulantes en la región de La Plata y alrededores. Todas las cepas O157:H7 y el 82% de las no O157 portaron el gen eae. Este gen se detecta más frecuentemente en cepas aisladas de pacientes con enfermedad grave como SUH o DS que en aquellos pacientes con enfermedad menos grave o en controles sanos4,26. Sin embargo, la presencia de eae no es esencial para la patogénesis.
El brote ocurrido en Europa en mayo/julio de 2011, asociado al nuevo patotipo de EAEC productor de toxina Stx2a y al serotipo O104:H4, provocó 3.141 casos de diarrea/colitis hemorrágica, 881 casos de SUH y 50 muertes5. Luego de ocurrido ese brote, el comité científico para peligros biológicos de la UE14 propuso ensayar la presencia de los genes aggR (regulador transcripcional) y aaiC (proteína secretada), ambos marcadores genéticos característicos de EAEC, en todas las cepas STEC eae negativas. Siguiendo esa recomendación, todas las cepas eae negativas analizadas en este trabajo fueron evaluadas para estos genes. El hallazgo en las cepas SML49 serotipo O59:H19 y SML67 serotipo O174:H21 de secuencias relacionadas con EAEC (presencia de aggR) en cepas productoras de toxina Shiga es un hecho poco frecuente en nuestro país6. Probablemente estas cepas STEC adquirieron por transferencia horizontal el plásmido de virulencia de cepas EAEC, ya que el gen aaiC (codificado en el cromosoma) está ausente en ambas. Si bien es interesante este hallazgo, se desconoce el impacto de este probable híbrido en la salud pública y se requieren estudios adicionales sobre estas cepas.
En estudios previos se ha mostrado que la virulencia de las cepas STEC para los humanos está relacionada con el tipo de toxina Shiga producida por la bacteria18. En este estudio se detectó la presencia de stx2 en el 100% de las cepas estudiadas y solo 2 cepas portaban conjuntamente stx1 y stx2. Al estudiar los subtipos de stx, la presencia simultánea de stx2a y stx2c(vh-a) se detectó en el 76% de los aislamientos O157:H7. Estos hallazgos son similares a los reportados previamente en Argentina, donde más del 90% de las cepas O157:H7 presentan esta combinación13,31. Este genotipo fue asociado con enfermedad grave en los humanos y se demostró que está presente más frecuentemente en pacientes con DS y SUH que otros subtipos18.
La virulencia de las cepas STEC puede no estar relacionada exclusivamente con la producción de toxinas Shiga e intimina, sino también con la presencia de otras adhesinas y toxinas. La frecuencia de aparición de las adhesinas putativas estudiadas no difiere de la reportada por otros autores8,19,20,47. A pesar de no estar relacionadas con el potencial patogénico de las cepas28, las adhesinas podrían ser un importante factor de colonización especie-específico. En este punto cabe resaltar que la combinación lpfA1-5/lpfA2-3 hallada en las cepas O145:NM se describe por primera vez en el presente trabajo, y es distinta de otras cepas O145 descriptas con anterioridad48. Asimismo, no debemos excluir la posibilidad de complicaciones de la enfermedad atribuibles a estos factores. Es de destacar que ninguna de las cepas STEC caracterizadas en el presente estudio presentó secuencias asociadas a las toxinas subtilasa, Cdt-V y East-1. Sin embargo, su relevancia clínica no debe ignorarse, debido al demostrado potencial patogénico de las cepas con estos factores de virulencia.
Las cepas de E. coli O157:H7 presentaron un alto grado de diversidad genética, tal como se describe en trabajos previos realizados en Argentina42. Además, en el presente trabajo se identificaron cepas con idéntico patrón XbaI-PFGE recuperadas en distintas localidades y en diferentes años, lo que implicaría que están ampliamente diseminadas y persisten a través de los años.
La aplicación de técnicas de epidemiología molecular contribuye a mejorar la vigilancia de STEC y permite integrar los resultados de laboratorio con los datos epidemiológicos, pero su implementación en tiempo real y el acceso a bases de datos nacionales y regionales es fundamental para identificar en forma temprana la probable ocurrencia de un brote y notificarla a las autoridades, que deben ocuparse de las acciones de control. Esto quedó demostrado al identificar un probable brote por cepas del clúster I varios años después de haber ocurrido.
La gravedad de las complicaciones que suelen acompañar a las infecciones asociadas con STEC pone en evidencia la necesidad de reforzar la vigilancia activa en los laboratorios de microbiología clínica en nuestro país, implementar protocolos para la detección de STEC y mejorar la capacidad diagnóstica y analítica de aquellos. Además, nuestros resultados ponen de manifiesto la importancia de completar la caracterización fenotípica y genotípica de los aislamientos en tiempo real, ya que estos datos permiten determinar el tipo de cepas que están circulando en nuestro país.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.
Los autores agradecen a la Bqca. Silvina Giugno por su colaboración en la recolección, recepción y conservación de las muestras. A la Dra. Nora L. Padola por su colaboración en la serotipificación de las cepas STEC no-O157 y a la Dra. Alejandra Londero por su colaboración en el análisis de los patrones XbaI-PFGE.