La espectrometría de masas, conocida como matrix-assisted laser desorption/ionization time-of-flight mass spectrometry (MALDI-TOF MS), es una técnica utilizada en la identificación de microorganismos mediante la creación de un espectro basado en el perfil de proteínas, que es único para una especie dada. El objetivo del presente trabajo fue evaluar la identificación de aislamientos clínicos de levaduras por MALDI-TOF MS en un hospital universitario de Argentina y analizar 2 procedimientos para la extracción de proteínas: el recomendado por el fabricante del equipo y una técnica abreviada rápida. Utilizando el primero de estos procedimientos se analizaron 201 aislamientos identificados previamente por métodos convencionales y se obtuvo coincidencia en la identificación a nivel de especie en el 95,38% de los aislamientos analizados. Con 100 de estos aislamientos se utilizó, además, el procedimiento abreviado para la extracción de proteínas; se obtuvo una identificación correcta a nivel de género y especie en el 98,0% de ellos. La espectrometría de masas MALDI-TOF MS demostró ser una técnica rápida, sencilla y precisa para la identificación de levaduras.
The matrix-assisted laser desorption/ionization time-of-flight mass spectrometry technique known as MALDI-TOF MS is a tool used for the identification of clinical pathogens by generating a protein spectrum that is unique for a given species. In this study we assessed the identification of clinical yeast isolates by MALDI-TOF MS in a university hospital from Argentina and compared two procedures for protein extraction: a rapid method and a procedure based on the manufacturer's recommendations. A short protein extraction procedure was applied in 100 isolates and the rate of correct identification at genus and species level was 98.0%. In addition, we analyzed 201 isolates, previously identified by conventional methods, using the methodology recommended by the manufacturer and there was 95.38% coincidence in the identification at species level. MALDI TOF MS showed to be a fast, simple and reliable tool for yeast identification.
Los hongos levaduriformes como Candida, Cryptococcus y otros géneros producen infecciones con una alta morbimortalidad, fundamentalmente en pacientes críticos.Candida albicans continúa siendo la especie más frecuente; sin embargo, en los últimos años se ha observado un incremento en la incidencia de Candidaspp. no C.albicans y de otros hongos levaduriformes6,8. Además, debemos considerar que algunas especies de levaduras presentan resistencia innata o adquirida a los antifúngicos, y por ello la correcta y rápida identificación es crítica para la toma de decisiones terapéuticas oportunas. El inicio temprano del tratamiento es fundamental para controlar la infección y disminuir la mortalidad7,8.
La identificación de levaduras se realiza habitualmente a través de estudios morfológicos, cultivo en medios cromogénicos, métodos comerciales basados en la asimilación de hidratos de carbono o mediante métodos automatizados. Estas metodologías presentan como inconvenientes su lentitud, la existencia de bases de datos limitadas y algunas identificaciones incorrectas4,9.
La identificación de microorganismos basada en la espectrometría de masas conocida como matrix-assisted laser desorption/ionization time-of-flight mass spectrometry (MALDI-TOF MS) es una técnica que existe hace más de 30años. Sin embargo, en los últimos años han aparecido diversas plataformas comerciales que han permitido a los laboratorios de microbiología clínica acceder más fácilmente a esta tecnología y aplicarla como un método confiable, específico y rápido de identificación de bacterias, micobacterias y hongos basado en el perfil de proteínas1,3,12,15. Esta técnica permite la identificación de microorganismos mediante el análisis de proteínas, principalmente ribosómicas, a través de la creación de un espectro de masas que es específico para cada especie. Un microorganismo dado presentará siempre una serie de picos característicos en el espectro, y esto permite la creación de bases de datos con los espectros de masas que presentan los distintos microorganismos. El espectro obtenido para un determinado microorganismo se compara automáticamente con la base de datos, y el resultado se emite junto a un puntaje o score1,3.
Este sistema constituye una herramienta útil para el laboratorio microbiológico, pues permite una identificación rápida de bacterias y levaduras a partir de colonias aisladas, e incluso es capaz de diferenciar a nivel de subespecie5,11,12.
Los objetivos de este trabajo fueron evaluar la identificación de levaduras por la técnica de MALDI-TOF MS utilizando el equipo Bruker® Daltonics (Bremen, Alemania) y evaluar 2 procedimientos de extracción de proteínas: el recomendado por el fabricante del equipo (en adelante, EXT) y una técnica abreviada rápida (en adelante, MR)14.
Se evaluaron 193 aislamientos de levaduras provenientes de materiales clínicos obtenidos en un hospital universitario de Argentina, previamente identificados por métodos convencionales como Candida parapsilosis (57), Candida albicans (46), Candida glabrata (19), Candida tropicalis (18), Candida guilliermondii (10), Candida krusei (7), Candida dubliniensis (4), Candida kefyr (3), Candida colliculosa/sphaerica (1), Candida famata (1), Candida sake/famata/lipolytica (1), Candida haemulonii (1), Candida lusitaniae (1), Candida inconspicua/norvegensis (1), Candida intermedia (1), Cryptococcus neoformans (10), Cryptococcus gattii (1), Cryptococcus albidus (1), Saccharomyces cerevisiae (2), Trichosporon asahii (7) y Rhodotorula mucilaginosa (1), más 8 cepas de referencia: C.parapsilosis ATCC 22019, C.albicans ATCC 8548, C. glabrata ATCC 90030, C.krusei ATCC 6258, Candida orthopsilosis NRRL Y-48468, C.orthopsilosis NRRL Y-27733, Candida metapsilosis NRRL Y-48470 y C.metapsilosis NRRL Y-48469. Los aislamientos fueron seleccionados teniendo en cuenta su frecuencia de aislamiento en el ámbito de nuestro hospital (hospital polivalente de alta complejidad). Todos los aislamientos fueron identificados por métodos convencionales (en adelante, MC) mediante el estudio micromorfológico, el cultivo en agar cromogénico para levaduras (CHROMagar™ Candida, París, Francia), el sistema API ID 32C (bioMèrieux, Marcy l’Etoile, Francia) o el de tarjetas YST del sistema Vitek 2 (bioMèrieux, Marcy l’Etoile, Francia).
La identificación mediante MALDI-TOF MS se efectuó a partir de aislamientos en agar Sabouraud glucosado incubados a 35°C durante 24-48h. Cuando la extracción de proteínas se realizó según las instrucciones del fabricante (EXT), una a tres colonias se transfirieron a un tubo Eppendorf de 1,5ml y se mezclaron con 300μl de agua destilada, se agregaron 900μl de etanol (70%) (Sigma-Aldrich) y se agitó en vórtex. A continuación se centrifugó a 13000 rpm durante 3min, el precipitado se dejó secar al aire y se resuspendió en 50μl de ácido fórmico (70%) (Sigma-Aldrich), seguido del agregado de 50μl de acetonitrilo (Sigma-Aldrich). Esta suspensión se centrifugó a 13000 rpm por 3min y 1μl del sobrenadante se depositó por duplicado en una placa metálica de 96 pocillos (Bruker Daltonics) y se dejó secar. Se cubrió con 1μl de matriz (solución saturada de ácido α-ciano-4-hidroxicinámico [HCCA; Bruker Daltonics] en 50% de acetonitrilo y 2,5% de ácido trifluoroacético).
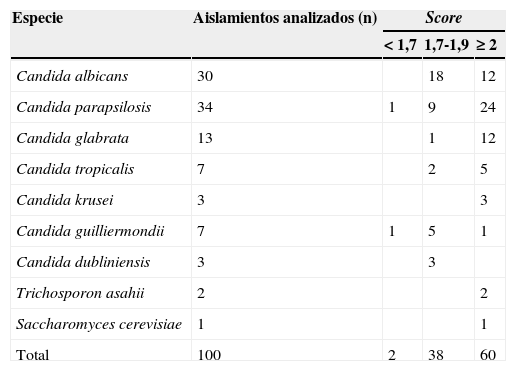
Del total de aislamientos analizados se seleccionaron 100, correspondientes a las especies de levaduras que se aíslan con mayor frecuencia en el laboratorio de micología clínica: C.albicans (30), C.parapsilosis (34), C.glabrata (13), C.tropicalis (7), C.krusei (3), C.guilliermondii (7), C.dubliniensis (3), T.asahii (2) y S.cerevisiae (1); a estos se les realizó, por separado, el procedimiento abreviado de preparación de la muestra (MR), que consistió en colocar una colonia directamente sobre la placa metálica, que luego se cubrió con 1μl de ácido fórmico (70%) y se dejó secar, para luego agregarle 1μl de matriz14.
Las lecturas se realizaron con un espectrómetro de masas Microflex LT y el software Flex Control (versión 3.0, Bruker Daltonics).
El espectrómetro se calibró utilizando un extracto de proteínas de Escherichia coli (Bruker Bacterial Test Standard).
Se utilizaron los siguientes criterios de identificación: puntuación o score ≥2, identificación a nivel de especie; score entre 1,7 y 1,9, identificación a nivel de género; score <1,7, falta de identificación. Consideramos como válidos para identificación a nivel de género y especie los scores comprendidos entre 1,7 y 2 realizados por duplicado con ambas metodologías, que brindaban el mismo resultado y coincidían con la MC. Los resultados obtenidos por MALDI-TOF MS se compararon con los obtenidos por la MC. Las discordancias se resolvieron mediante secuenciación de los espacios intergénicos (ITS1 e ITS2) del ADNr16. Para ello se realizó la extracción del ADN genómico utilizando el QIAmp DNA Mini Kit (Qiagen AG, Suiza); posteriormente se amplificó la región ITS del ADNr utilizando los primers ITS1 (TCCGTAGGTGAACCTGCGG) e ITS4 (TCCTCCGCTTATTGATATGC), el producto fue secuenciado empleando los mismos primers utilizados en la amplificación en un secuenciador ABI Prism 3100/3100-Avant Genetic Analyser (Applied Biosystems, EE.UU.). Las secuencias obtenidas se compararon con las depositadas en el GenBank utilizando la BLASTn Database (National Center for Biotechnology Information [NCBI], http://blast.ncbi.nlm.nih.gov/).
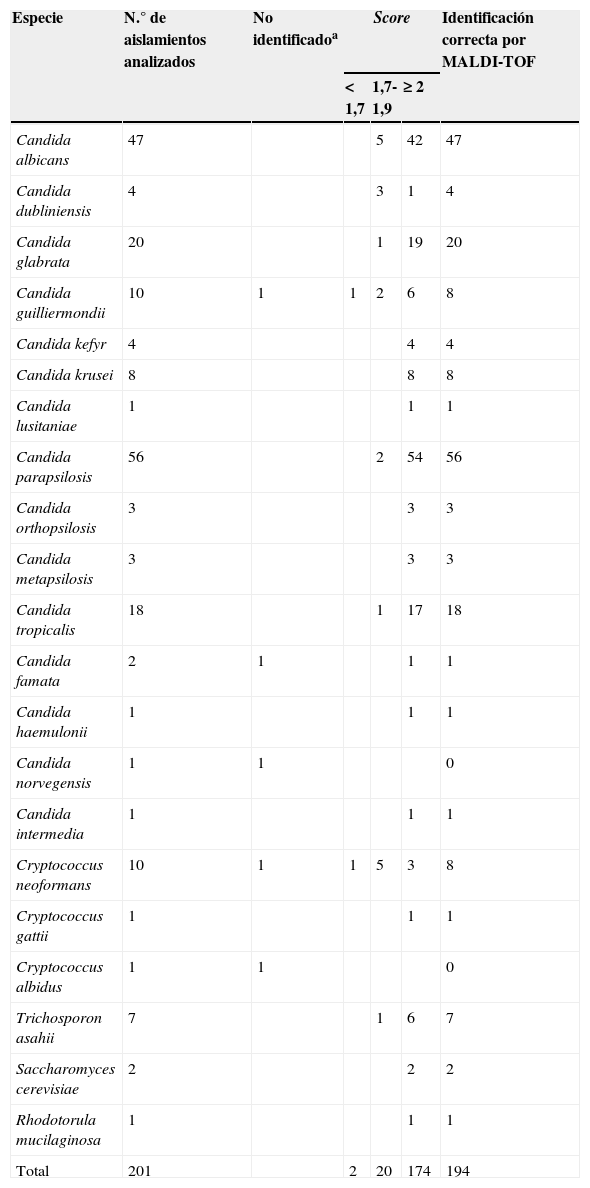
Seis aislamientos no pudieron ser identificados por MALDI-TOF MS, ya sea por presentar un score <1,7 o porque no se obtuvieron espectros de calidad para ser analizados, aun después de repetir el procedimiento de extracción y análisis. La identificación por MALDI-TOF MS coincidió a nivel de especie con la realizada por MC en 95,38% (186/195) de aislamientos.
Dos aislamientos clínicos previamente identificados como C.parapsilosis por MC fueron identificados como C.metapsilosis (3) y C.orthopsilosis (3) por MALDI-TOF MS, y estos resultados fueron confirmados por secuenciación.
En otros 3 casos de discrepancia, 2 fueron resueltos a favor de MALDI-TOF MS usando secuenciación (un aislamiento de C.sake/famata/lipolytica por MC se confirmó como C.famata; otro de C.colliculosa/sphaerica por MC se confirmó como C.kefyr), mientras que un aislamiento de C.inconspicua/norvegensis por MC, que MALDI-TOF identificó como C.inconspicua, fue confirmado como C.norvegensis por secuenciación.
La identificación por MALDI-TOF MS utilizando EXT coincidió en un 97% a nivel de género y en un 96,5% a nivel de especie con la identificación realizada por MC más secuenciación en los casos de discrepancia (tabla 1).
Identificación de levaduras por MALDI-TOF MS empleando para la extracción de proteínas el procedimiento recomendado por el fabricante
| Especie | N.° de aislamientos analizados | No identificadoa | Score | Identificación correcta por MALDI-TOF | ||
|---|---|---|---|---|---|---|
| < 1,7 | 1,7-1,9 | ≥ 2 | ||||
| Candida albicans | 47 | 5 | 42 | 47 | ||
| Candida dubliniensis | 4 | 3 | 1 | 4 | ||
| Candida glabrata | 20 | 1 | 19 | 20 | ||
| Candida guilliermondii | 10 | 1 | 1 | 2 | 6 | 8 |
| Candida kefyr | 4 | 4 | 4 | |||
| Candida krusei | 8 | 8 | 8 | |||
| Candida lusitaniae | 1 | 1 | 1 | |||
| Candida parapsilosis | 56 | 2 | 54 | 56 | ||
| Candida orthopsilosis | 3 | 3 | 3 | |||
| Candida metapsilosis | 3 | 3 | 3 | |||
| Candida tropicalis | 18 | 1 | 17 | 18 | ||
| Candida famata | 2 | 1 | 1 | 1 | ||
| Candida haemulonii | 1 | 1 | 1 | |||
| Candida norvegensis | 1 | 1 | 0 | |||
| Candida intermedia | 1 | 1 | 1 | |||
| Cryptococcus neoformans | 10 | 1 | 1 | 5 | 3 | 8 |
| Cryptococcus gattii | 1 | 1 | 1 | |||
| Cryptococcus albidus | 1 | 1 | 0 | |||
| Trichosporon asahii | 7 | 1 | 6 | 7 | ||
| Saccharomyces cerevisiae | 2 | 2 | 2 | |||
| Rhodotorula mucilaginosa | 1 | 1 | 1 | |||
| Total | 201 | 2 | 20 | 174 | 194 | |
a No identificados por MALDI-TOF MS por tener un score <1,7 o espectros no analizables o identificación incorrecta.
En la tabla 2 se muestra el número de aislamientos correspondientes a cada categoría de score predefinida utilizando la MR de extracción de proteínas. Este procedimiento permitió identificar en forma rápida a nivel de especie el 98% de los aislamientos evaluados, aunque los scores fueron generalmente inferiores a los obtenidos por EXT.
Identificación de levaduras por MALDI-TOF MS usando para la extracción de proteínas una técnica abreviada
| Especie | Aislamientos analizados (n) | Score | ||
|---|---|---|---|---|
| < 1,7 | 1,7-1,9 | ≥ 2 | ||
| Candida albicans | 30 | 18 | 12 | |
| Candida parapsilosis | 34 | 1 | 9 | 24 |
| Candida glabrata | 13 | 1 | 12 | |
| Candida tropicalis | 7 | 2 | 5 | |
| Candida krusei | 3 | 3 | ||
| Candida guilliermondii | 7 | 1 | 5 | 1 |
| Candida dubliniensis | 3 | 3 | ||
| Trichosporon asahii | 2 | 2 | ||
| Saccharomyces cerevisiae | 1 | 1 | ||
| Total | 100 | 2 | 38 | 60 |
Si bien C.albicans continúa siendo la especie de levadura más frecuente en infecciones fúngicas invasoras, las producidas por otras especies de Candida, así como por otros géneros de levaduras, se han incrementado en los últimos tiempos, principalmente debido al aumento de la población de huéspedes inmunosuprimidos6,8. En estos pacientes el diagnóstico temprano y el comienzo del tratamiento antifúngico adecuado es fundamental para disminuir la mortalidad. Dada la variabilidad en la sensibilidad a antifúngicos inherente a las diferentes especies de levaduras, la correcta identificación es a menudo crítica para tomar decisiones terapéuticas adecuadas7,8.
Los procedimientos habituales de identificación fúngica pueden demorar varios días, y en muchos casos los resultados no son confiables, principalmente con especies poco frecuentes9. En los últimos años, la aparición de la metodología MALDI-TOF ha surgido como una herramienta para acelerar y mejorar la identificación de bacterias y hongos en el laboratorio de microbiología clínica12,15. En nuestro trabajo, el 96,5% de las cepas fueron correctamente identificadas a nivel de especie. Marklein et al.5 evaluaron 18 cepas de referencia de 8 especies de levaduras y 267 aislamientos clínicos (28 especies de 7 géneros) y hallaron que todas las cepas de referencia y el 92,5% de los aislamientos clínicos fueron identificados correctamente a nivel de especie. Por otra parte, merece mencionarse que los aislamientos no identificados no se encontraban en la base de datos del sistema. Sendid et al.11 evaluaron 1.207 aislamientos (25 especies) de levaduras y encontraron que el 97,5% de ellos se identificaron correctamente. Podemos resaltar que actualmente esta metodología se usa en forma más frecuente, y prueba de ello son los numerosos trabajos que dan cuenta del uso del sistema MALDI-TOF MS para la identificación de levaduras aisladas de materiales clínicos. Estos muestran que esta metodología permite la identificación correcta de levaduras en el 92,5 al 98% de los casos1,2,5,13.
La técnica de MALDI-TOF MS nos permitió diferenciar entre especies fenotípicamente indistinguibles, como las pertenecientes al complejo formado por C.parapsilosis sensu stricto, C.orthopsilosis y C.metapsilosis, las cuales solo podrían ser diferenciadas por técnicas moleculares. El interés en la identificación correcta de los miembros de este complejo de especies no es solo epidemiológico, también es importante debido a las posibles diferencias en los estudios de sensibilidad a los antifúngicos, en sus virulencias y en la patogenia de las infecciones10.
En 6 aislamientos (2 C.guilliermondii, 1 C.famata, 2 C.neoformans y 1 C.albidus) no se pudo realizar la identificación mediante MALDI-TOF MS por presentar estos un bajo score o espectros de mala calidad. Stevenson et al.13 también mostraron dificultades en la identificación de C.guilliermondii y C.neoformans, y esto lo atribuyeron a propiedades inherentes a los aislados que proporcionan espectros de baja calidad o a que los espectros de proteínas presentes en la base de datos no son suficientes para ser representativos de la variabilidad en la especie. Esto también puede explicar la falta de identificación de C.albidus, ya que se encuentra un solo espectro en la base de datos del sistema. En los casos en los que existió discrepancia entre MALDI-TOF y la MC, la secuenciación mostró una excelente concordancia con la espectrometría de masas.
Las levaduras, al igual que otras especies fúngicas, poseen una pared celular gruesa, por lo que es importante la preparación de la muestra para favorecer la liberación de las proteínas intracelulares. El procedimiento de extracción con etanol, ácido fórmico y acetonitrilo recomendado por el fabricante permite obtener espectros de buena calidad para su análisis y brinda altos niveles de identificación a nivel de género o especie1,2. Sin embargo, la aplicación de una metodología abreviada de extracción directa en placa (MR) nos permitió identificar el 98% de levaduras representativas de las especies más frecuentes en el laboratorio de micología clínica con menores tiempos de procesamiento y costos de reactivos, lo que facilita la incorporación de esta metodología al trabajo del laboratorio.
Cabe señalar que el sistema MALDI-TOF MS ha sido utilizado para la identificación de levaduras directamente de los frascos de hemocultivos positivos, con la ventaja de arrojar resultados en menos tiempo. Esto permite seleccionar el tratamiento antifúngico apropiado más tempranamente2,3.
En conclusión, podemos decir que la metodología MALDI-TOF MS constituye una herramienta útil y confiable para identificar correctamente a nivel de especie y en forma rápida aislamientos de levaduras, permitiendo un diagnóstico temprano de las infecciones por hongos levaduriformes de importancia clínica.
Responsabilidades éticasProtección de personas y animalesLos autores declaran que para esta investigación no se han realizado experimentos en seres humanos ni en animales.
Confidencialidad de los datosLos autores declaran que en este artículo no aparecen datos de pacientes.
Derecho a la privacidad y consentimiento informadoLos autores declaran que en este artículo no aparecen datos de pacientes.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.