Las amebas de vida libre del género Acanthamoeba incluyen cepas patógenas y no patógenas que actualmente se encuentran clasificadas en 18 genotipos diferentes (T1-T18). Se llevó a cabo un relevamiento para determinar la presencia de cepas de Acanthamoeba en agua de bebida para consumo ganadero en la provincia de La Pampa, Argentina. Las muestras fueron sembradas en agar no nutritivo. La identificación de cepas de Acanthamoeba se realizó mediante polymerase chain reaction con los primers JDP1/JDP2 específicos para género. De las 65 muestras tomadas, 13 fueron positivas a Acanthamoeba spp. Estas fueron caracterizadas a nivel de genotipo mediante la secuenciación del fragmento DF3. Los resultados de la secuenciación revelaron la presencia de los genotipos T4, T5 y T15 dentro de las muestras estudiadas, siendo la más frecuente la T4. Nuestro estudio revela importancia de la presencia de Acanthamoeba en el ambiente ganadero y la necesidad de realizar más estudios, para asociar la presencia de estos organismos y el papel que cumplen en patologías veterinarias. Este es el primer estudio en la provincia de La Pampa que demuestra la presencia de Acanthamoeba y el primero estudiado a nivel de genotipo de Argentina.
Free-living Amoebae of Acanthamoeba genus include non-pathogenic and pathogenic strains that are currently classified in 18 different genotypes, T1-T18. In this study, a survey was carried out to evaluate the presence of Acanthamoeba strains inwatering trough sample in La Pampa province, Argentina. Sample were inoculated onto non-nutrient agar plates and were checked for the presence of Acanthamoeba. Polymerase chain reaction was performed with genus-specific primers JDP1/JDP2, followed by direct sequencing of the polymerase chain reaction product for molecular identification. Sequencing results revealed the presence of T4, T5 and T15 genotypes within the studied samples. Sequencing revealed presence of T4, T5 and T15 in the samples studied genotypes, the most frequent T4. Our study reveals importance of the presence of Acanthamoeba in the livestock environment and the need for further studies to associate the presence of these organisms and the role in veterinary pathology. To the best of our knowledge, this is the first report demonstrating the presence Acanthamoeba in La Pampa province and the first study at the genotype level in Argentina.
Las amebas de vida libre (AVL) son un grupo heterogéneo de protozoos de distribución cosmopolita. Pueden desarrollarse en condiciones de vida libre y algunas especies pueden comportarse como parásitos del hombre y otros animales30. Actualmente existen más de 100 especies de AVL y algunas de ellas pertenecen al género Acanthamoeba. Las especies del género Acanthamoeba son las más frecuentemente aisladas, tanto en ambientes naturales como artificiales17. Su ciclo biológico tiene dos etapas, una etapa activa o trofozoíto que exhibe crecimiento vegetativo y una etapa latente de quiste con mínima actividad metabólica26. En los últimos diez años ha aumentado el interés en este organismo por su capacidad de producir lesiones cutáneas e infecciones de senos nasales, queratitis que afecta la visión, encefalitis granulomatosa amebiana11,17,30 como también neumonía e infecciones cutáneas. Además de sus características patogénicas propias, el interés en Acanthamoeba radica en su capacidad de actuar como reservorio/vector de otros agentes patógenos2,11. Por otra parte, Acanthamoeba puede tener importancia veterinaria como lo demuestran algunos trabajos13,27,29. Actualmente son 18 los genotipos (T1-T18) conocidos de Acanthamoeba basados en el análisis del gen del ADNr 18S21. El objetivo de este trabajo fue el aislamiento y la caracterización molecular de los genotipos de Acanthamoeba en agua para consumo ganadero en La Pampa, Argentina.
Materiales y métodosÁrea de estudioSe colectaron muestras de agua que los rumiantes usan para bebida en establecimientos ganaderos de la provincia de La Pampa, Argentina. La provincia cuenta con una superficie de 14,34 millones de hectáreas, de las cuales 12,74 millones se dedican a producción ganadera.
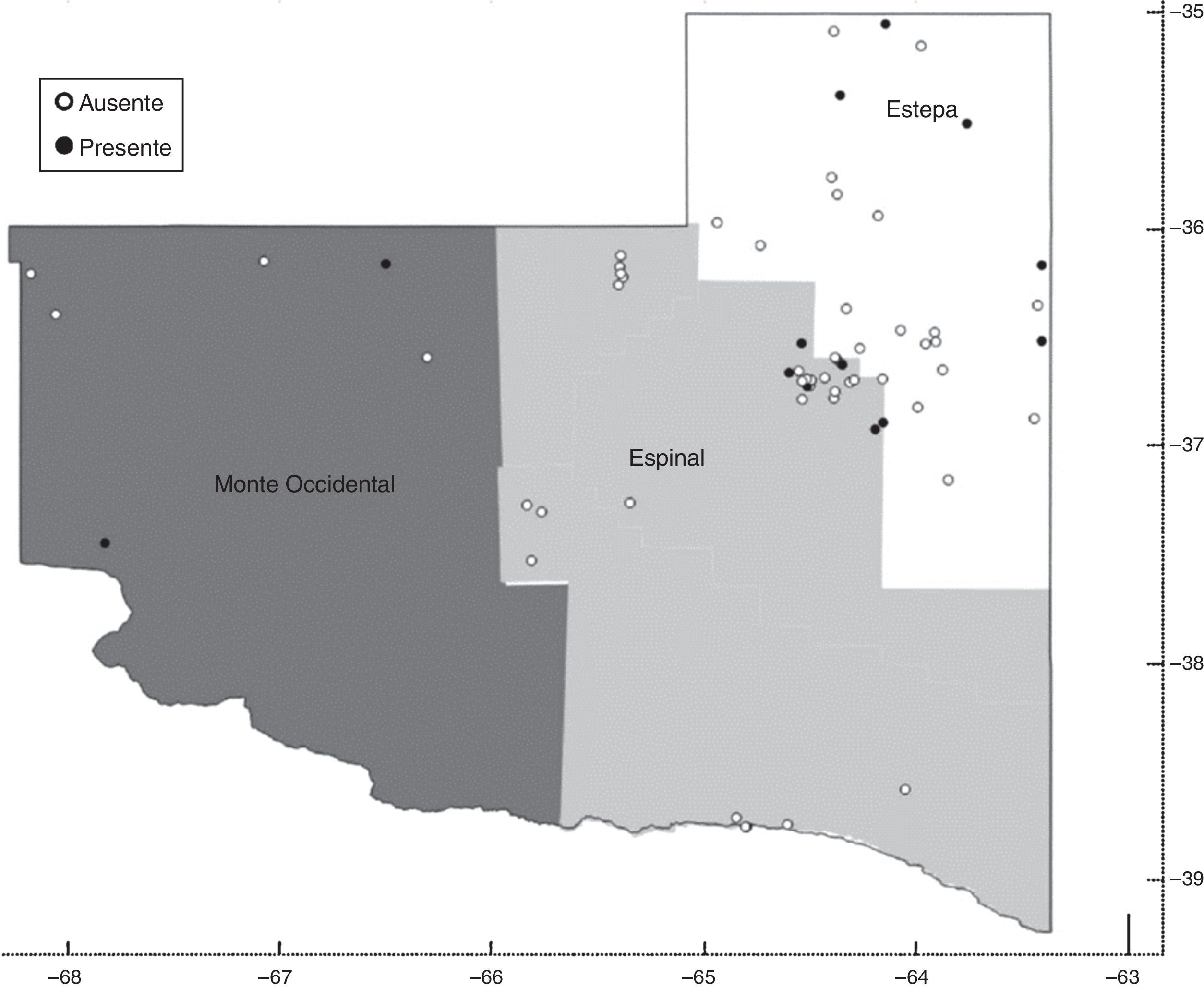
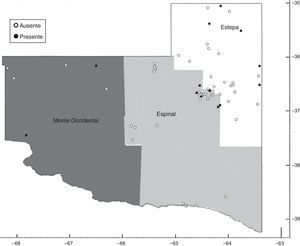
La provincia se puede dividir en tres regiones productivas16: Estepa (noroeste), Espinal (Caldenal) y Monte Occidental (Oeste). La región del noroeste es la más pequeña en cuanto a superficie y es la que presenta mejores aptitudes agroecológicas para el desarrollo de diferentes actividades agropecuarias, con un régimen hídrico de 800mm. La región del Caldenal abarca la región central de la provincia, con una precipitación media anual de 600mm. Representa el 40% de la superficie total, y se caracteriza por la presencia dominante de bosques de caldén. La principal actividad productiva es la ganadería extensiva (cría y recría bovina).
El Monte Occidental representa el 35% de la superficie total de la provincia. Sus características agroecológicas determinan que sea una zona principalmente ganadera (caprina y bovina); el 98% de la región es de monte y campo natural con un régimen hídrico de 400mm anuales (fig. 1).
Cada establecimiento fue ubicado espacialmente y se registraron, para cada muestra, los datos de temperatura del agua y pH.
Muestreo de agua y cultivo de AVLLas muestras de agua se obtuvieron de aguadas (fig. 2) pertenecientes a 65 establecimientos ganaderos, desde junio 2013 a enero 2015. La temperatura de las aguas estudiadas osciló entre 10°C (en invierno) y 28°C (en verano) y el pH entre 6 y 9. En ocho oportunidades las muestras se tomaron de aguadas naturales. De cada lugar se colectaron 500ml de agua en frascos estériles y se dejaron decantar en el laboratorio durante 24 h. Se desechó el sobrenadante y de cada muestra se retuvo un volumen de 40ml. El volumen retenido se centrifugó a 1.500rpm durante 10min. Del sedimento se obtuvieron los inóculos para los cultivos. Se sembraron 100μl del sedimento en placas de Petri con agar no nutritivo cubierto con una suspensión de Escherichia coli en medio de Page, como nutriente para el protozoo20. Las placas fueron incubadas a 37°C y monitoreadas diariamente durante 15 días. Se consideraron negativas cuando pasado este tiempo no se detectó desarrollo.
Extracción de ADNLos cultivos fueron concentrados raspando con asas los anillos de crecimiento de las amebas en las placas de agar utilizando 500μl de ClK0.15M en frío, centrifugando a 1.000rpm por 10 min a 4°C. Se descartó el sobrenadante y se repitió el proceso. El ADN se extrajo utilizando el método UNSET10. El método consistió en lisar las células con 500μl de buffer de lisis (a temperatura ambiente) e igual volumen de fenol-cloroformo-alcohol isoamilíco (25:24:1). La suspensión se centrifugó a 8.000rpm a 10 min. La fase acuosa se transfirió a un microtubo nuevo, se trató con 500μl fenol-cloroformo-alcohol isoamílico. Para precipitar el ADN, se añadieron 10μl de NaCl 3M a la solución acuosa y 1ml etanol absoluto (frío) y se mezcló suavemente. Las suspensiones se mantuvieron a -20°C durante toda la noche. A continuación, se centrifugaron a 15.000rpm durante 30 min. Los sedimentos se lavaron con 500μl de etanol al 70% (frío) y nuevamente se centrifugaron a 15.000rpm por 30 min. Los sedimentos se secaron en un bloque de calor a 55°C, se añadieron 100μl de agua MiliQ y se conservó a -20°C hasta su análisis.
Identificación y genotipificación de Acanthamoeba mediante PCRLos cebadores JDP1 y JDP2 se utilizaron para amplificar la región del ASA.S1 (Rns) que codifica la subunidad pequeña del ARN ribosomal nuclear de la ameba25. Así se obtiene un amplímero designado como DF3 (fragmento de diagnóstico 3). La secuencia de este fragmento dentro del ASA.S1 permite la identificación de los genotipos de Acanthamoeba25. La mezcla de reacción, para un volumen final de 25μl se preparó con 1,25 U Taq (Promega), 0,5μl mix de dNTPs (10mM de cada dNTP, Promega), 2,5μl buffer (Promega), cebador JDP1 1,5μl (1μM), JDP2 1,5μl (1μM) y 5,0μl de templado de ADN. Para el desarrollo de la PCR se utilizó un termociclador IVEMA T18 y las condiciones de reacción fueron: 95°C por 7min, seguido por 45 ciclos de 95°C por 1 minuto, 60°C por 1 minuto, 72°C por 2min, y una extensión a 72°C por 10min.
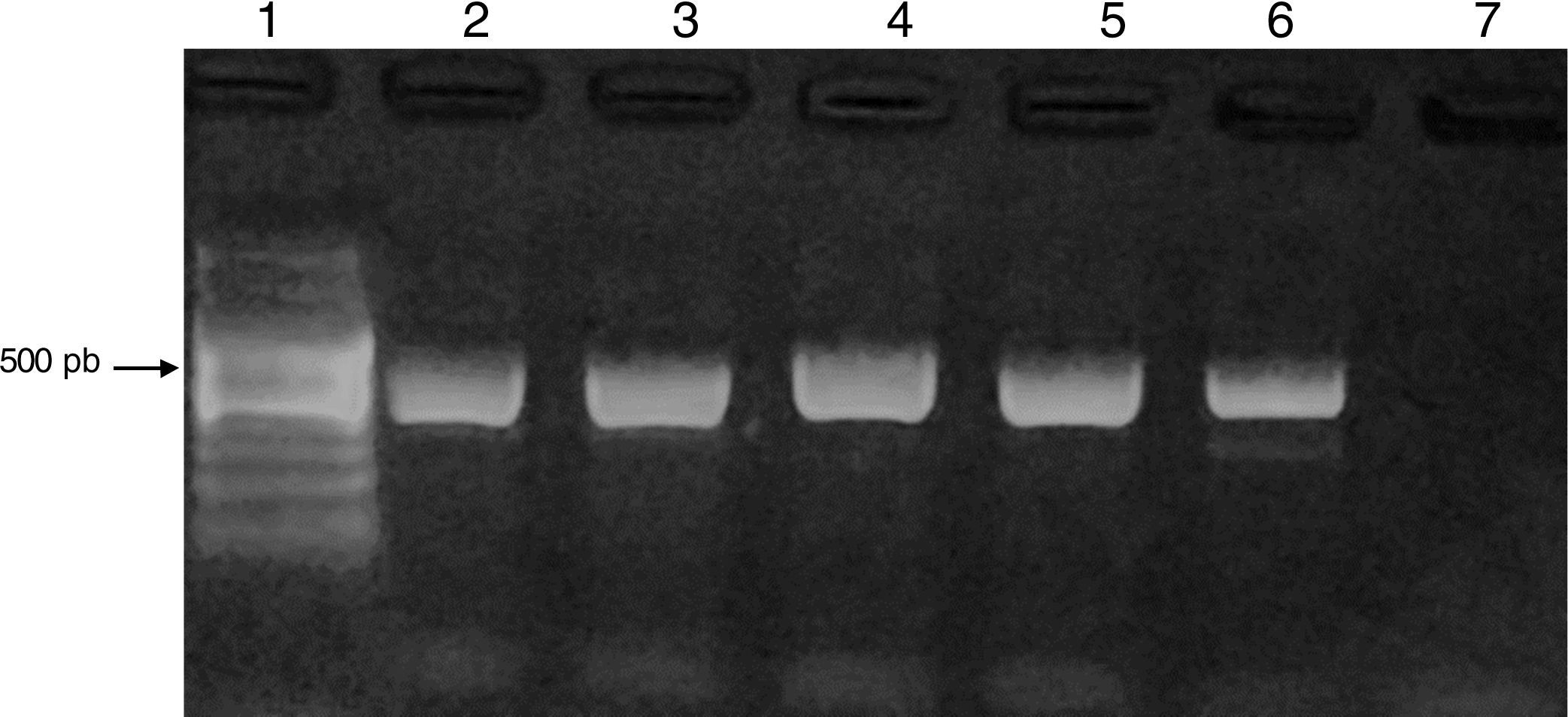
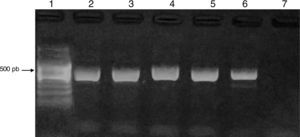
Los productos de amplificación fueron visualizados mediante electroforesis en geles de agarosa (Biodynamics) al 1,5%, se tiñeron con 5μl de GEL RED (BIOTIUM) y se visualizaron bajo luz UV con un transiluminador DyNA Light Dual LABNET. Se observó un producto entre 423-521 pb. Como control positivo se utilizó una cepa de Acanthamoeba polyphaga ATCC 30461.
Secuenciación de los aislados de AcanthamoebaLos productos obtenidos de la PCR fueron purificados y concentrados utilizando el kit DONGSHENG BIOTECH (Montebio). Posteriormente fueron secuenciados en la Unidad Genómica –Instituto de Biotecnología– CICVyA, Instituto Nacional de Tecnología Agropecuaria (INTA Castelar, Buenos Aires). La identificación genotípica se basó en la técnica publicada por Booton3. Con el fin de clasificar los aislamientos, se realizó la búsqueda de secuencias homólogas depositadas en Genbank mediante la herramienta Basic Local Alignment Search Tool (https://blast.ncbi.nlm.nih.gov/Blast.cgi) para buscar la mayoría de las secuencias de referencias similares. Los alineamientos y los árboles filogenéticos se construyeron con el programa Molecular Evolution Genetic Analysis software (MEGA7) (http://megasofware.net), utilizando el método de vecino más cercano (Neighbor-joining) y el modelo de Maximum Composite Likelihood, con un bootstrap de 1.000 muestreos al azar.
ResultadosEl 83% de los casos estudiados (54/65) presentó crecimiento de AVL en placas de agar no nutritivo. En el 24% de las muestras con presencia de AVL (13/54), por la morfología, se las identificó como pertenecientes al género Acanthamoeba, confirmándose por PCR utilizando cebadores específicos de género (fig. 3).
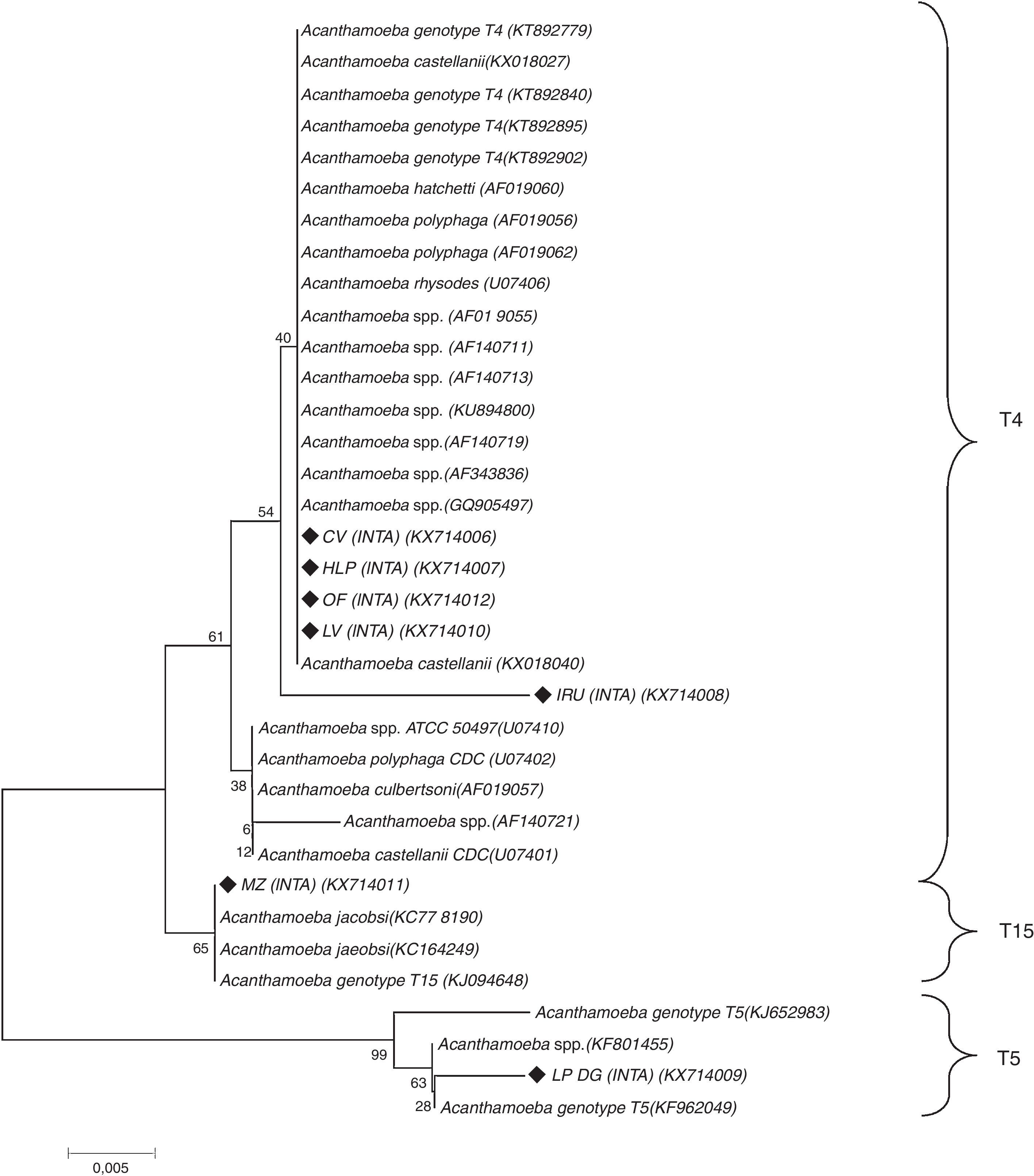
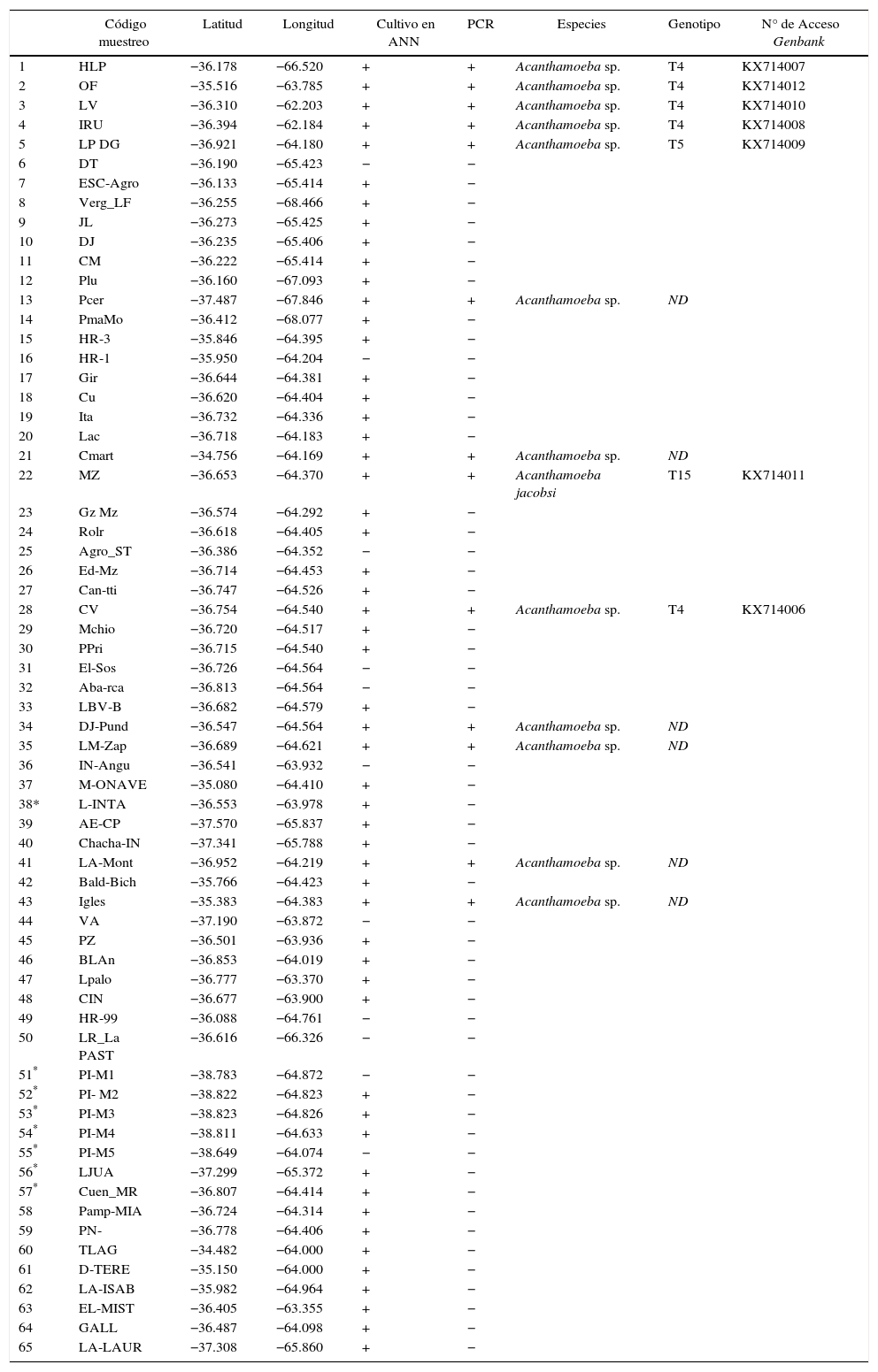
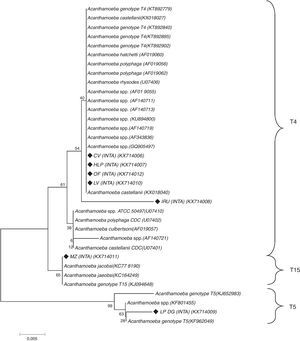
Como parte de un estudio más exhaustivo, se identificaron los genotipos de Acanthamoeba spp. mediante secuenciación del gen del ADNr 18S (tabla 1). La secuenciación se efectuó sobre siete cepas y los aislamientos fueron alineados con secuencias de referencia del GenBank y con las identificadas por Booton et al.3. El análisis filogenético de las secuencias identificó cinco aislamientos (KX714006, KX714007, KX714008, KX714010 y KX714012) dentro del genotipo T4, uno (KX714009) dentro del T5 y el restante (KX714011) como T15. Todos presentaron identidades nucleotídicas superiores al 85% (fig. 4).
Genotipos de Acanthamoeba identificados en la provincia de La Pampa, Argentina
| Código muestreo | Latitud | Longitud | Cultivo en ANN | PCR | Especies | Genotipo | N° de Acceso Genbank | |
|---|---|---|---|---|---|---|---|---|
| 1 | HLP | −36.178 | −66.520 | + | + | Acanthamoeba sp. | T4 | KX714007 |
| 2 | OF | −35.516 | −63.785 | + | + | Acanthamoeba sp. | T4 | KX714012 |
| 3 | LV | −36.310 | −62.203 | + | + | Acanthamoeba sp. | T4 | KX714010 |
| 4 | IRU | −36.394 | −62.184 | + | + | Acanthamoeba sp. | T4 | KX714008 |
| 5 | LP DG | −36.921 | −64.180 | + | + | Acanthamoeba sp. | T5 | KX714009 |
| 6 | DT | −36.190 | −65.423 | − | − | |||
| 7 | ESC-Agro | −36.133 | −65.414 | + | − | |||
| 8 | Verg_LF | −36.255 | −68.466 | + | − | |||
| 9 | JL | −36.273 | −65.425 | + | − | |||
| 10 | DJ | −36.235 | −65.406 | + | − | |||
| 11 | CM | −36.222 | −65.414 | + | − | |||
| 12 | Plu | −36.160 | −67.093 | + | − | |||
| 13 | Pcer | −37.487 | −67.846 | + | + | Acanthamoeba sp. | ND | |
| 14 | PmaMo | −36.412 | −68.077 | + | − | |||
| 15 | HR-3 | −35.846 | −64.395 | + | − | |||
| 16 | HR-1 | −35.950 | −64.204 | − | − | |||
| 17 | Gir | −36.644 | −64.381 | + | − | |||
| 18 | Cu | −36.620 | −64.404 | + | − | |||
| 19 | Ita | −36.732 | −64.336 | + | − | |||
| 20 | Lac | −36.718 | −64.183 | + | − | |||
| 21 | Cmart | −34.756 | −64.169 | + | + | Acanthamoeba sp. | ND | |
| 22 | MZ | −36.653 | −64.370 | + | + | Acanthamoeba jacobsi | T15 | KX714011 |
| 23 | Gz Mz | −36.574 | −64.292 | + | − | |||
| 24 | Rolr | −36.618 | −64.405 | + | − | |||
| 25 | Agro_ST | −36.386 | −64.352 | − | − | |||
| 26 | Ed-Mz | −36.714 | −64.453 | + | − | |||
| 27 | Can-tti | −36.747 | −64.526 | + | − | |||
| 28 | CV | −36.754 | −64.540 | + | + | Acanthamoeba sp. | T4 | KX714006 |
| 29 | Mchio | −36.720 | −64.517 | + | − | |||
| 30 | PPri | −36.715 | −64.540 | + | − | |||
| 31 | El-Sos | −36.726 | −64.564 | − | − | |||
| 32 | Aba-rca | −36.813 | −64.564 | − | − | |||
| 33 | LBV-B | −36.682 | −64.579 | + | − | |||
| 34 | DJ-Pund | −36.547 | −64.564 | + | + | Acanthamoeba sp. | ND | |
| 35 | LM-Zap | −36.689 | −64.621 | + | + | Acanthamoeba sp. | ND | |
| 36 | IN-Angu | −36.541 | −63.932 | − | − | |||
| 37 | M-ONAVE | −35.080 | −64.410 | + | − | |||
| 38* | L-INTA | −36.553 | −63.978 | + | − | |||
| 39 | AE-CP | −37.570 | −65.837 | + | − | |||
| 40 | Chacha-IN | −37.341 | −65.788 | + | − | |||
| 41 | LA-Mont | −36.952 | −64.219 | + | + | Acanthamoeba sp. | ND | |
| 42 | Bald-Bich | −35.766 | −64.423 | + | − | |||
| 43 | Igles | −35.383 | −64.383 | + | + | Acanthamoeba sp. | ND | |
| 44 | VA | −37.190 | −63.872 | − | − | |||
| 45 | PZ | −36.501 | −63.936 | + | − | |||
| 46 | BLAn | −36.853 | −64.019 | + | − | |||
| 47 | Lpalo | −36.777 | −63.370 | + | − | |||
| 48 | CIN | −36.677 | −63.900 | + | − | |||
| 49 | HR-99 | −36.088 | −64.761 | − | − | |||
| 50 | LR_La PAST | −36.616 | −66.326 | − | − | |||
| 51* | PI-M1 | −38.783 | −64.872 | − | − | |||
| 52* | PI- M2 | −38.822 | −64.823 | + | − | |||
| 53* | PI-M3 | −38.823 | −64.826 | + | − | |||
| 54* | PI-M4 | −38.811 | −64.633 | + | − | |||
| 55* | PI-M5 | −38.649 | −64.074 | − | − | |||
| 56* | LJUA | −37.299 | −65.372 | + | − | |||
| 57* | Cuen_MR | −36.807 | −64.414 | + | − | |||
| 58 | Pamp-MIA | −36.724 | −64.314 | + | − | |||
| 59 | PN- | −36.778 | −64.406 | + | − | |||
| 60 | TLAG | −34.482 | −64.000 | + | − | |||
| 61 | D-TERE | −35.150 | −64.000 | + | − | |||
| 62 | LA-ISAB | −35.982 | −64.964 | + | − | |||
| 63 | EL-MIST | −36.405 | −63.355 | + | − | |||
| 64 | GALL | −36.487 | −64.098 | + | − | |||
| 65 | LA-LAUR | −37.308 | −65.860 | + | − |
Árbol construido con el programa MEGA 7.0 utilizando el parámetro Neighbor-joinig (NJ) y el modelo de Maximum Composite Likelihood. Los valores de bootstrap (1.000 repeticiones) se muestran arriba y debajo de los nodos. Los genotipos y especies obtenidos de la base de datos Genbank incluyen los números de acceso (ACCN). Los aislamientos de este estudio están marcados con rombos negros rellenos (♦) (barra horizontal=0,05 sustituciones por posición de nucleótido).
Existen estudios ambientales en todo el mundo donde se informa la presencia de cepas de Acanthamoeba spp.1,4,6–8,14,19,22,28.
Hasta el momento, se han descrito 18 genotipos diferentes (T1- T18) de Acanthamoeba spp.21. El genotipo T4 es el más común tanto en el ambiente como en aislados de individuos con patología15,26 y es el que presenta la mayor variabilidad genética. Asimismo, se han descrito otros genotipos como causantes de infecciones en humanos y en animales13,15,21.
En Argentina existen reportes de Acanthamoeba spp. aisladas de agua de piscina, en casos de queratitis humanas5,9,17,18,30. Sin embargo, no se han realizado genotipificaciones. A pesar de la naturaleza ubicua de esta especie, no existen estudios a nivel del ambiente ganadero en el mundo. El agua destinada al ganado en la provincia varía entre apta para consumo humano hasta aguas muy salinas, muy duras, o con exceso de arsénico, flúor y sulfatos; aun así, este medio permite el desarrollo de Acanthamoeba spp. Un problema en este tipo de muestras es la frecuente contaminación con otros microorganismos, ya que al cuerpo de agua sirve de bebedero para los animales domésticos y silvestres, incluyendo aves migratorias, lo que dificulta la amplificación y genotipificación de una muestra directa de campo. Cabe mencionar que los diferentes repiques de las muestras favorecen a la pérdida de cepas por la falta de adaptación al ambiente de laboratorio. Los genotipos encontrados en este trabajo están asociados a patologías humanas como queratitis (T4, T5 y T15) y encefalitis (T4). En tres de los establecimientos ganaderos en los que se identificó la presencia de Acanthamoeba spp., se tiene registro de problemas de queratoconjuntivitis infecciosa bovina recurrente, pero en ningún momento se ha estudiado la presencia de Acanthamoeba spp. asociada a esta patología. Otro problema registrado en los establecimientos muestreados es la mortandad de terneros en verano por problemas nerviosos. La casuística de enfermedades con signos nerviosos en nuestro país aumentó con la necesidad del país de ser declarado libre de encefalopatía espongiforme bovina. El estudio de la encefalitis amebiana debería ser incluido dentro del diagnóstico diferencial de las patologías del sistema nervioso central en el ganado, en especial en verano.
En todos los casos mencionados no se asoció a la presencia de Acanthamoeba spp., debido a la falta de conocimiento de este organismo como causante de estas patologías. Asimismo, cabe mencionar que son portadores de otros organismos (por ejemplo, Chlamydia) que también podrían favorecer a la presencia de estas y otras patologías. Este estudio permite confirmar la presencia de Acanthamoeba spp. genotipos T4, T5 y T15 en aguas utilizadas para consumo ganadero, todas ellas de importancia zoonótica. Esto abre la posibilidad de estudios futuros ya que estos organismos actúan como vehículos o vectores de transmisión (como «caballos de Troya») para otros patógenos12,23,24, potencial adicional para la ganadería y de precaución para el personal rural.
La información proporcionada en este estudio sirve como línea de base para futuros estudios sobre el papel del género Acanthamoeba como reservorio y vector de enfermedades bacterianas o virales fundamentalmente, en el ambiente ganadero de la provincia.
ConclusiónAcanthamoeba ha ganado una creciente atención por parte de la comunidad científica debido a su capacidad para capturar microorganismos por fagocitosis, actuar como vectores y/o reservorios para los patógenos microbianos, y para producir infecciones graves en humanos y en los animales. Este trabajo permitió confirmar la presencia de Acanthamoeba en aguadas de sistemas ganaderos en la provincia de La Pampa y que las cepas secuenciadas pertenecen a los genotipos T4, T5 y T15. Se requieren más estudios para evaluar cómo actúa Acanthamoeba como fuente potencial de infección para el ganado y el personal rural. Este es el primer estudio epidemiológico referido a presencia de AVL en aguadas de la Provincia de La Pampa, Argentina.
Responsabilidades éticasProtección de personas y animalesLos autores declaran que para esta investigación no se han realizado experimentos en seres humanos ni en animales.
Confidencialidad de los datosLos autores declaran que en este artículo no aparecen datos de pacientes.
Derecho a la privacidad y consentimiento informadoLos autores declaran que en este artículo no aparecen datos de pacientes.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.
Agradecemos al Área de Sanidad Animal de INTA Anguil, a la Lic. María Sol Poey (INTA, Agencia de Extensión Rural Victorica), a los profesionales y a los productores que colaboraron en ofrecer sus establecimientos. Se agradece, además, al Instituto Nacional de Tecnología Agropecuaria (INTA) por financiar parcialmente este trabajo (Resol. N.° 1029/12 y 743/12).