Las mujeres jóvenes (≤40 años) con cáncer de mama suelen tener un peor pronóstico que las mujeres posmenopáusicas, con repercusiones reproductivas y familiares importantes. El tratamiento quimioterapéutico, así como la hormonoterapia, pueden mejorar el pronóstico y la supervivencia libre de enfermedad (SLE); sin embargo, la capacidad individual de metabolizar los fármacos puede modificar la respuesta al tratamiento. Las diferencias interpersonales de esta capacidad tienen una explicación en las variaciones de los genes que codifican las enzimas que metabolizan los quimioterapéuticos y las hormonas endógenas y exógenas. Los genes del citocromo P450 (CYP450), CYP3A4, CYP2B6, CYP2D6 y CYP2C19, están involucrados en el metabolismo de los estrógenos y la ciclofosfamida, los taxanos y el tamoxifeno. La presente revisión expone la evidencia científica del efecto de los polimorfismos funcionales de estos genes sobre la SLE, y sus implicaciones, en las mujeres jóvenes con cáncer de mama.
Breast cancer prognosis tends to be worse in young women (≤40 years) than in postmenopausal women, with significant reproductive and family repercussions. Both chemotherapy and hormonal therapy can improve prognosis and disease-free survival but treatment response may be influenced by the individual's ability to metabolize drugs. Individual differences in metabolic ability can be explained by variations in the genes encoding the enzymes that metabolize chemotherapeutic agents and hormones. The cytochrome P450 genes CYP3A4, CYP2B6, CYP2D6 and CYP2C19 are involved in the metabolism of estrogen, cyclophosphamide, taxanes, and tamoxifen. This review discusses the scientific evidence of the effect of functional polymorphisms in these genes on disease-free survival and overall survival and its implications for young women with breast cancer.
El cáncer de mama se define como un crecimiento desordenado de células provenientes de cualquiera de los tejidos de la glándula mamaria (conductos o lobulillos) con la capacidad de diseminarse1. Es considerado el cáncer más común entre las mujeres tanto en los países desarrollados como en los países en vías de desarrollo2. Las mujeres de 40 años o menos diagnosticadas con cáncer de mama se consideran casos jóvenes3. Este es el cáncer más prevalente en el mundo entre las mujeres de este grupo de edad, con una tasa de 22.2 por cada 100 000 mujeres. Es también el cáncer más incidente, solo en 2018 hubo 243 910 casos, y una tasa de 8.3 por cada 100 000 habitantes. Ese año ocupó el primer lugar en mortalidad por cáncer de mujeres menores de 40 años, con 44 840 muertes4.
Las mujeres jóvenes con cáncer de mama son diagnosticadas en la etapa productiva y fértil de su vida, y pierden alrededor de 40 años de vida saludable5. De aquí que entender los factores que modifican la respuesta al tratamiento sea esencial para mejorar la supervivencia libre de la enfermedad (SLE). La SLE es un indicador de la respuesta tumoral al tratamiento, que permite identificar tempranamente la recurrencia y ajustar el tratamiento. En este sentido, la farmacogenética juega un papel fundamental para entender los factores individuales que modifican la respuesta al tratamiento del cáncer de mama, particularmente el estudio de los genes involucrados en el metabolismo de los fármacos que se utilizan en la quimioterapia y en la hormonoterapia. En este caso, los de la vía del citocromo P450 (CYP450). De aquí que el presente trabajo revise y sintetice la información científica relevante sobre el tema.
MetodologíaPara cumplir ese objetivo, se hizo una búsqueda bibliográfica de la evidencia del efecto de los polimorfismos funcionales tanto en el tratamiento de las mujeres jóvenes con cáncer de mama, como en su pronóstico. La información del sistema del CYP450 y sus polimorfismos funcionales se consultó en la base de datos del consorcio PharmGKB, que incluye las vías metabólicas curadas del CYP4506. Para conocer el panorama epidemiológico del cáncer de mama en las mujeres jóvenes, se consultaron las plataformas de GLOBOCAN4 y Global Burden of Disease5. Para describir su manejo clínico, se consultaron las guías internacionales de diagnóstico y tratamiento del cáncer de mama.
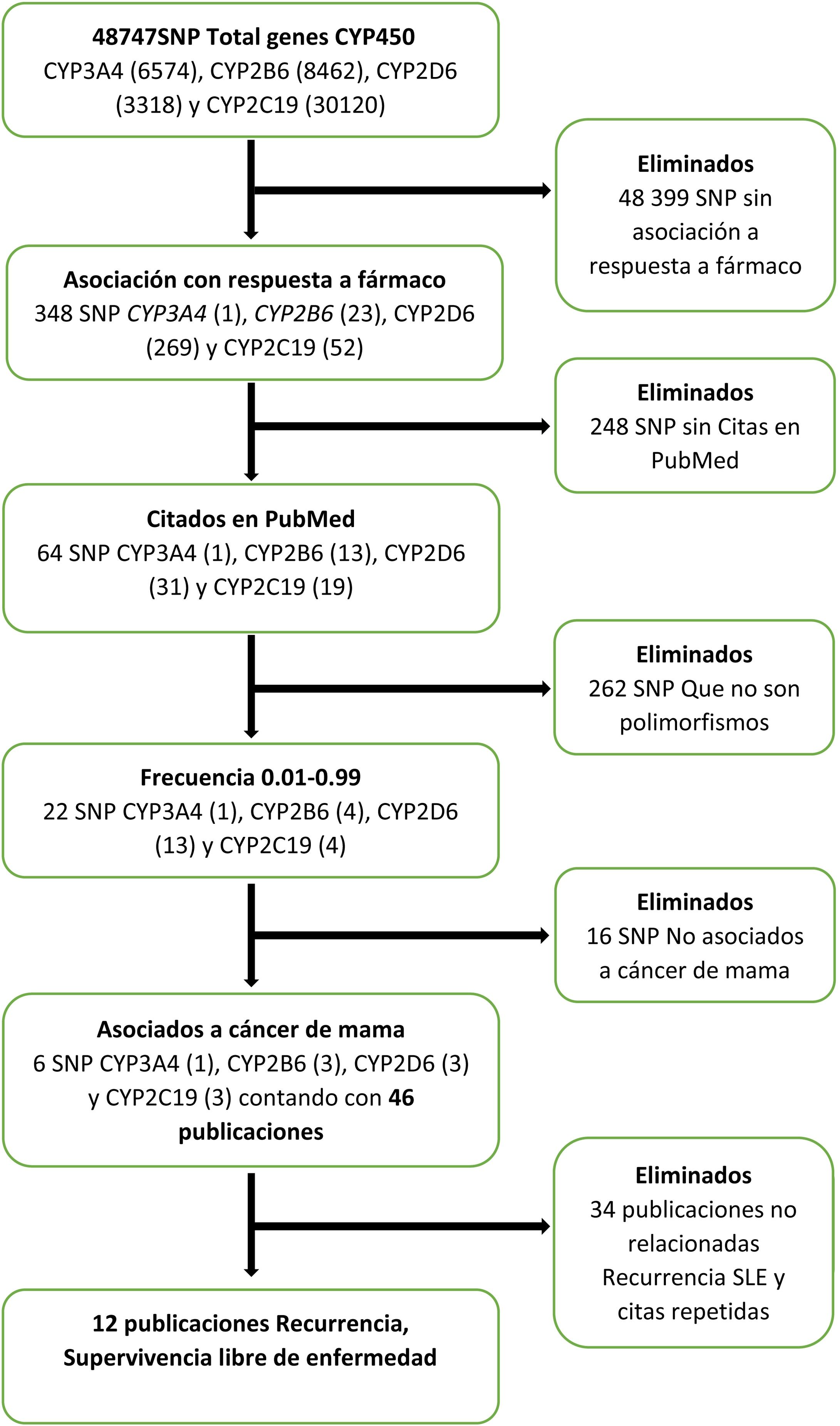
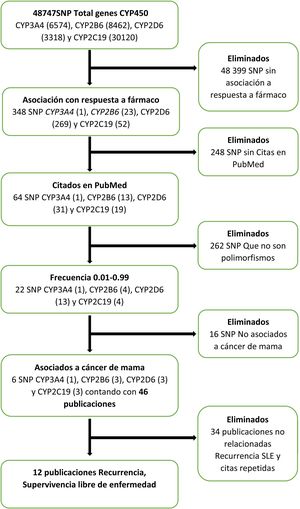
La búsqueda bibliográfica de los efectos de los polimorfismos funcionales de los genes del citocromo P450 sobre la SLE se hizo del 1° al 15 de noviembre de 2020, en las bases de datos del Centro Nacional para la Información Biotecnológica (NCBI, por sus siglas en inglés)7 mediante el algoritmo mostrado en la figura 1. En la base de datos del NCBI7, se introdujeron individualmente los genes del citocromo P450 que intervienen en el metabolismo de la ciclofosfamida, los taxanos y el tamoxifeno, que son: CYP3A4, CYP2B6, CYP2D6 y CYP2C19. Después se aplicaron los filtros “Respuesta a fármaco”, “Citados en Pubmed”8, frecuencia de 0.01-0.99 y “cáncer de mama”, y se obtuvieron únicamente los seis SNP con estas características, que estaban citados en 46 publicaciones. Se eliminaron los que no estaban relacionados con la recurrencia o con la SLE, y las citas repetidas.
Se hizo una segunda búsqueda en la plataforma Pubmed8, utilizando diversas combinaciones de los términos MeSH9 “cáncer de mama” (Breast Neoplasms), “supervivencia libre de enfermedad” (Disease-Free Survival), “CYP3A4”, “CYP2B6”, “CYP2D6”, “CYP2C19”, “ciclofosfamida“(Cyclophosphamide), “paclitaxel” “docetaxel” y “tamoxifeno” (Tamoxifen). Al final se incluyeron 19 estudios.
Características tumorales de las mujeres jóvenes con cáncer de mamaEn las mujeres jóvenes, el cáncer de mama tiene características consideradas de mal pronóstico, tales como una alta proliferación celular del tumor10, una mayor probabilidad de ser triple negativo, menor expresión de receptores hormonales (estrógenos y progesterona) y mayor expresión de HER2, con un grado histológico más alto11,12. La enfermedad suele diagnosticarse en una etapa clínica más avanzada y estas mujeres suelen tener una mayor probabilidad de recurrencia local y de cáncer contralateral12–15. Hay, además, una mayor proporción de mutaciones germinales en los genes del cáncer hereditario, como el BRCA 1, 2 y el TP5316. Si bien los tumores de las mujeres jóvenes presentan una sobreexpresión del marcador de proliferación celular Ki67, el mal pronóstico es independiente de este marcador10,16,17.
Factores pronósticos de la SLE en las mujeres con cáncer de mamaPara estudiar el pronóstico del cáncer de mama, en especial en las mujeres jóvenes, debe revisarse la SLE o intervalo libre de cáncer de mama. Este periodo comprende desde la fecha del diagnóstico o del fin del tratamiento17 hasta la presentación de diferentes eventos clínico-patológicos que hacen las veces de puntos finales para determinar el tiempo transcurrido. Estos puntos sustitutos o subrogados se utilizan para reducir el número de pacientes que participan en un ensayo clínico, así como su duración y su costo. Los eventos que se han evaluado como puntos finales son: la recurrencia invasiva del tumor mamario (ipsilateral, contralateral, local o regional), la metástasis, y la formación de un tumor secundario en otro órgano o sistema18. El desenlace oncológico en este periodo es determinante para el pronóstico19.
Los factores asociados a la SLE son: la etapa clínica al momento del diagnóstico20, el tipo de tratamiento20,21, el subtipo inmunohistoquímico y la edad, aunque recientemente se han incluido también los marcadores genéticos tumorales22.
Entre las mujeres con cáncer de mama en las etapas I y II, tratadas quirúrgicamente, la SLE a diez años es de hasta 80%, con mejor pronóstico para el subtipo luminal. Entre las mujeres con cáncer en la etapa III, la SLE a diez años es de 55% para los tumores luminales, de 50% para triple negativo, y de 30% para tumores HER2 (+)20.
La edad es un factor pronóstico independiente. La SLE entre las mujeres menores de 40 años es significativamente menor que entre las mujeres de más de 40 años (73% vs 57%, a 60 meses de seguimiento p=0.032)17 y tienen un riesgo de muerte hasta 52% mayor que las mujeres de más de 40 años14. Los factores pronósticos –como el índice de masa corporal (IMC) bajo– se comportan de forma diferente a lo observado entre las mujeres mayores; en este grupo de edad se les confiere un peor pronóstico3,12,23.
TratamientoEl tratamiento del cáncer de mama es multidisciplinario (cirugía, radioterapia, quimioterapia, hormonoterapia, terapia biológica y terapia de apoyo) y debe seguir en las mujeres menores de 40 años el mismo protocolo que para las mujeres de más de 40 años3. La elección del tipo de tratamiento se basa, sobre todo, en dos aspectos: la etapa clínica al momento del diagnóstico y el subtipo inmunohistoquímico3,24–26. Los principales fármacos del tratamiento quimioterapéutico son las antraciclinas (doxorrubicina y epirrubicina), los taxanos (paclitaxel y docetaxel), los antimetabolitos (5-fluoracilo y metotrexato) y los alquilantes, como la ciclofosfamida. El principal fármaco de la hormonoterapia es el tamoxifeno. Se utilizan, además, inhibidores de la aromatasa (como el exemestano). En los tumores HER2 (+) se usan agentes biológicos como el trastuzumab19,25.
El tratamiento del cáncer de mama –con cirugía, quimioterapia, terapia endocrina o radioterapia– tiene el potencial de afectar la salud fisiológica (incluyendo la fertilidad, la menopausia prematura y la homeostasis ósea) y psicológica de las mujeres jóvenes11,27. De aquí que se deba tomar en cuenta el apoyo psicosocial personalizado, la consejería genética, y la derivación a un servicio especializado para la preservación de la reserva ovárica y la fertilidad, así como el abordaje de las alteraciones sexuales y la imagen corporal 27–29.
La respuesta al tratamiento del cáncer de mamaEntre las mujeres con cáncer de mama, la respuesta al tratamiento farmacológico varía en términos de eficacia y eficiencia. Hay, por consiguiente, subconjuntos de mujeres con perfiles de riesgo-beneficio diferentes con respecto a cada fármaco30. Estos perfiles pueden ir desde aquellos en que la dosis es insuficiente para alcanzar la respuesta terapéutica deseada, hasta los que presenten efectos secundarios tóxicos graves o incluso la muerte31. Esta variabilidad puede tener causas farmacocinéticas –como la absorción, la distribución, la metabolización y la excreción del fármaco– o bien farmacodinámicas –de la interacción fármaco-receptor–. La variabilidad puede deberse también a determinantes genéticos (farmacogenéticos), ambientales o patológicos. Otras causas son el cumplimiento o la adherencia al tratamiento y las ineficiencias del sistema de salud, tales como errores o retrasos en la prescripción31,32.
La farmacogenética es el estudio de las diferencias en las respuestas individuales a los medicamentos, atribuibles a la variación alélica de los genes que actúan sobre el metabolismo de los fármacos. En otras palabras, es “la Influencia de la herencia sobre la respuesta a los fármacos”, y tiene como objetivo principal identificar las variantes genéticas que puedan usarse para predecir el resultado de los agentes terapéuticos33. El citocromo P450 es uno de los principales blancos de los estudios farmacogenéticos.
El citocromo P450 (CYP450) y su efecto en el metabolismo de los fármacosLos principales genes involucrados en el metabolismo de –y la respuesta a– los fármacos pertenecen a la superfamilia del CYP450. Estos codifican las enzimas que controlan el metabolismo de más de 80% de los fármacos de prescripción. Se han descrito variaciones genéticas en este citocromo que modifican el metabolismo de los fármacos34. De acuerdo con la actividad metabolizadora de las enzimas del CYP450, se han identificado cuatro fenotipos metabolizadores:
- 1)
El metabolizador extensivo (ME), que es el más común en Occidente, sirve de referencia en la administración de los fármacos para obtener la respuesta terapéutica deseada; es decir, a partir de este fenotipo se calculan las dosis y los periodos de administración.
- 2)
El metabolizador intermedio (MI), que es la segunda variedad más frecuente; procesa los fármacos un poco más lentamente que el ME.
- 3)
El metabolizador pobre (PM) que corresponde solo a una parte reducida de la población; no es capaz de metabolizar los fármacos de manera adecuada; suele tener altas concentraciones plasmáticas del fármaco y, por lo común, produce toxicidad.
- 4)
El metabolizador ultrarrápido (MU), también poco frecuente, metaboliza los fármacos más rápidamente que los demás fenotipos. Los pacientes con este fenotipo suelen necesitar dosis altas del fármaco para tener el efecto terapéutico deseado, y suelen tener más bajas concentraciones plasmáticas del fármaco31,34,35.
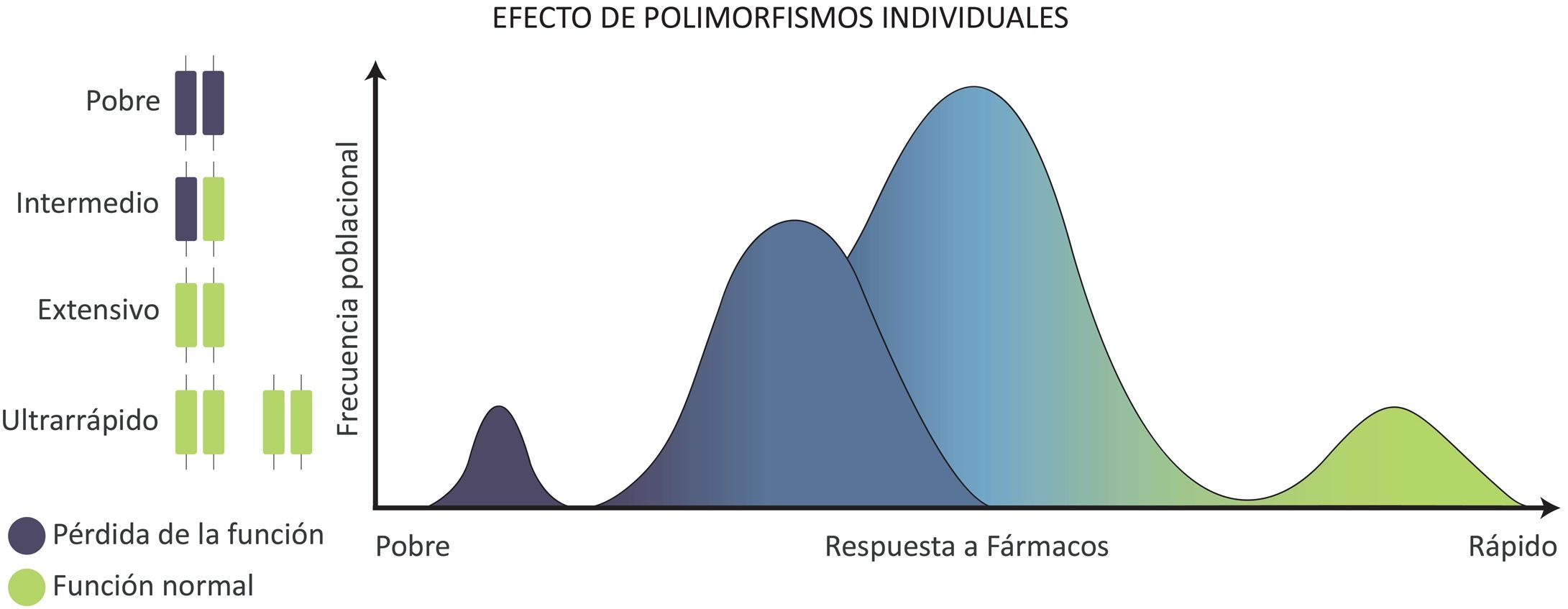
El modelo de frecuencia de estos fenotipos de metabolizadores según la distribución de los alelos de un gen se puede observar en la figura 2.
Distribución de los fenotipos y genotipos de metabolizadores según el efecto monogénico de CYP450. Se observa que la mayor parte de la población hispana posee un fenotipo metabolizador extensivo o intermedio, mientras que solo una pequeña parte de la población posee fenotipos de metabolizadores pobres o ultrarrápidos.
El CYP450 es el sistema enzimático metabolizador más importante en el ser humano, y comprende un amplio grupo de proteínas “hem” con actividad enzimática. Apareció evolutivamente como un mecanismo de defensa para metabolizar los xenobióticos, que son compuestos químicos extraños en la naturaleza, como la mayoría de los fármacos, alcoholes y carcinogénicos33. El CYP450 se encuentra en la gran mayoría de los seres vivos, y se han encontrado cerca de 7 700 isoformas34. Representa de entre 70 y 80% del metabolismo de fase I de todos los fármacos33. Su función principal es transformar los compuestos liposolubles en metabolitos hidrosolubles para su acción o excreción por el riñón36.
De acuerdo con su nomenclatura, las siglas CYP corresponden a la superfamilia de los genes del citocromo 450, las sigue un número (la familia), una letra (la subfamilia) y un número más (el gen específico). Por ejemplo, CYP3A436–38. Para cada gen específico, el alelo silvestre se identifica como *1, y las variables alélicas se numeran de manera secuencial según hayan sido identificadas (*2, *3, etc.)39. Este sistema enzimático se encuentra en el interior de las células, específicamente en el retículo endoplásmico liso, la mitocondria y la membrana celular37.
Las enzimas del CYP450 intervienen en el metabolismo de la mayoría de los fármacos antineoplásicos empleados para tratar el cáncer de mama: los agentes alquilantes, los taxanos y el tamoxifeno40. A continuación se presentan los principales fármacos utilizados en la quimioterapia, la terapia hormonal y la terapia biológica, así como su mecanismo de acción y su vía metabólica (tabla 1).
Principales vías metabólicas de los fármacos utilizados para el tratamiento del cáncer de mama
| Fármaco | Mecanismo de acción | Vía metabólica |
|---|---|---|
| Doxorrubicina (Adriamicina) | Rompe el ADN al inhibir la topoisomerasa II, se intercala en el ADN e inhibe la DNA polimerasa. | Metabolismo hepático Carbonil reductasa (CBR) |
| Docetaxel y Paclitaxel | Aumenta la formación y estabilización de los microtúbulos, deteniendo la mitosis. | Metabolismo hepático de fase I CYP3A4, CYP2B6, CYP2C8 |
| Ciclofosfamida | Sufre metabolismo hepático, produciendo metabolitos alquilantes que impiden la duplicación del ADN. | Metabolismo hepático de fase ICYP2B6, CYP2C19CYP3A4 |
| Metotrexato | Rompe el ADN al inhibir la topoisomerasa II, se intercala en el ADN e inhibe la DNA polimerasa. | Metabolismo hepático de fase ICarbonil reductasa (CBR) |
| Fluoracilo | Antimetabolito de la pirimidina, que cuando se convierte en nucleótido activo inhibe la timidilato sintetasa, bloqueando así la síntesis de ADN en fase S. | Metabolismo hepático de fase IDihidrofolato reductasa(DHFR)Dihidropirimidina deshidrogenasa (DPD) |
| Tamoxifeno | Bloqueador temporal selectivo de receptores de estrógenos en célula neoplásica. También efecta los factores de crecimiento celular, lo factores de crecimiento epidérmico y los factores TGFα y β | Metabolismo hepático de fase ICYP3A4, CYP3A5, CYP2D6, CYP2C19 |
| Exemestano | Inhibidor esteroideo irreversible de la aromatasa estructuralmente relacionado con el sustrato natural androstenediona. | Metabolismo hepático de fase ICYP3A4 |
| Trastuzumab | Anticuerpo IgG1 humanizado anti –Her2, Herceptin, dirigido contra el dominio extracelular del receptor del factor de crecimiento Her2 (p185Her2) | Información limitada |
Fuente: Skeel RT. Quimioterapia del cáncer (edición en español de: Handbook of Cancer Chemotherapy), octava edición, Marbán libros S.L. Madrid España 2013. 19
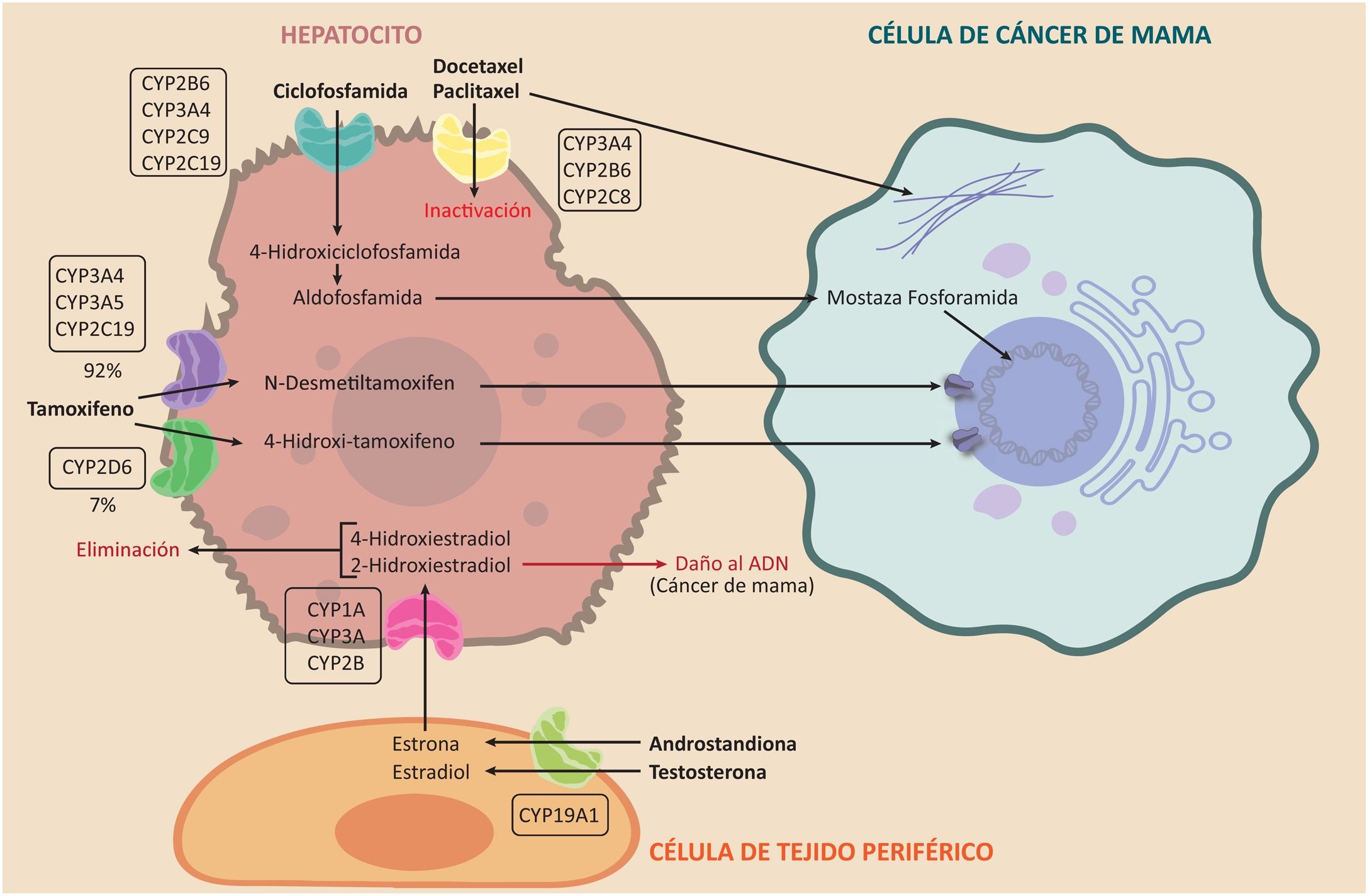
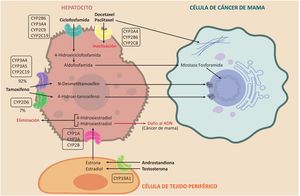
La ciclofosfamida es un profármaco que se utiliza en el tratamiento quimioterapéutico de muchos cánceres, tales como el cáncer de mama y los linfomas. Debe pasar por el metabolismo hepático para poder actuar como alquilante a través de las enzimas CYP2B6, CYP3A4, CYP3A5, CYP2C9 y CYP2C19, para convertirse en 4-hidroxiciclofosfamida. Esta se convierte, a su vez, en aldofosfamida, pasa al torrente sanguíneo y llega a la célula tumoral donde se transforma en mostaza fosforamida, que es el metabolito activo40. Sus principales efectos adversos son la urotoxicidad, la neurotoxicidad y la mielosupresión19. Este proceso se esquematiza en la figura 341.
Efecto del sistema del CYP450 sobre el metabolismo de los estrógenos y los fármacos ciclofosfamida, taxanos y tamoxifeno. Se aprecia la participación de las enzimas del CYP450 en activación de los pro-fármacos tamoxifeno y ciclofosfamida y en la inactivación de los taxanos, además la participación en la vía metabólica de los estrógenos para formación del 2-Hidroxiestradial con potencial carcinogénico.
El tamoxifeno es de gran importancia para prevenir y tratar el cáncer de mama hormonosensible, por ser un inhibidor de los receptores de estrógenos. Los metabolitos de este profármaco son los responsables de su actividad. Es metabolizado en 92% por las enzimas CYP3A4, CYP3A5 y CYP2C19 a N–desmetiltamoxifeno, mientras que 7% se metaboliza por CYP2D6 a 4-hidroxi- tamoxifeno, que es de 30 a 100 veces más potente como antiestrógeno que el N-desmetiltamoxifen. De esta forma, la enzima CYP2D6 también ha demostrado su importancia clínica en el metabolismo del tamoxifeno 42 (véase la fig. 3).
Actividad del citocromo P450 sobre los taxanosTanto el paclitaxel como el docetaxel se administran por vía intravenosa y son fármacos activos, a diferencia del tamoxifeno y la ciclofosfamida. Los taxanos sufren el metabolismo de fase I por las enzimas CYP3A4 y CYP2B6; sin embargo, una fracción pequeña del paclitaxel también es metabolizada por la CYP2C8. La función de estas enzimas es inactivar el fármaco; de esta forma la efectividad de los taxanos es inversamente proporcional a la velocidad con que son inactivados por las enzimas del CYP450. Su mecanismo de acción es estabilizar los microtúbulos evitando la mitosis, para luego causar la muerte celular43 (véase la fig. 3).
Actividad del citocromo P450 sobre los estrógenosLos estrógenos se sintetizan en los tejidos periféricos –incluyendo el hígado– por medio de una enzima conocida como aromatasa, a la que codifica el gen CYP19A144,45. Esta enzima transforma los andrógenos: androstendiona y testosterona en estrona y estradiol, respectivamente. En el hígado, estos se biotransforman, a su vez, mediante las enzimas CYP1A, CYP3A y CYP2B, en 4-hidroxiestradiol y 2-hidroxiestradiol, para su posterior eliminación. Sin embargo, este último es un metabolito capaz de dañar el ADN46,47. Esta ruta metabólica es independiente de la aromatasa y es un importante campo de estudio del riesgo, el pronóstico y el tratamiento del cáncer de mama48–50 (véase la fig. 3).
Los genes del citocromo P450El CYP3A4El CYP3A4 es un gen de 27.2 kilobases (kb) ubicado en el brazo largo del cromosoma 7 (7q21.1). Se expresa en el intestino, el hígado y la próstata, entre otros tejidos, y codifica para cuatro variantes de proteínas con actividad enzimática51. La enzima CYP3A4 es la más importante del sistema del CYP450 y está involucrada en el metabolismo de aproximadamente 30% de todos los fármacos52. Se han identificado 347 SNP en este gen, 25 de los cuales son de relevancia clínica51. Entre los más importantes por disminuir su actividad enzimática están *1B, *1F, *6, *8, *11, *12, *13, * 14, *16, *17 y *18, y *22, que aumenta la actividad enzimática52.
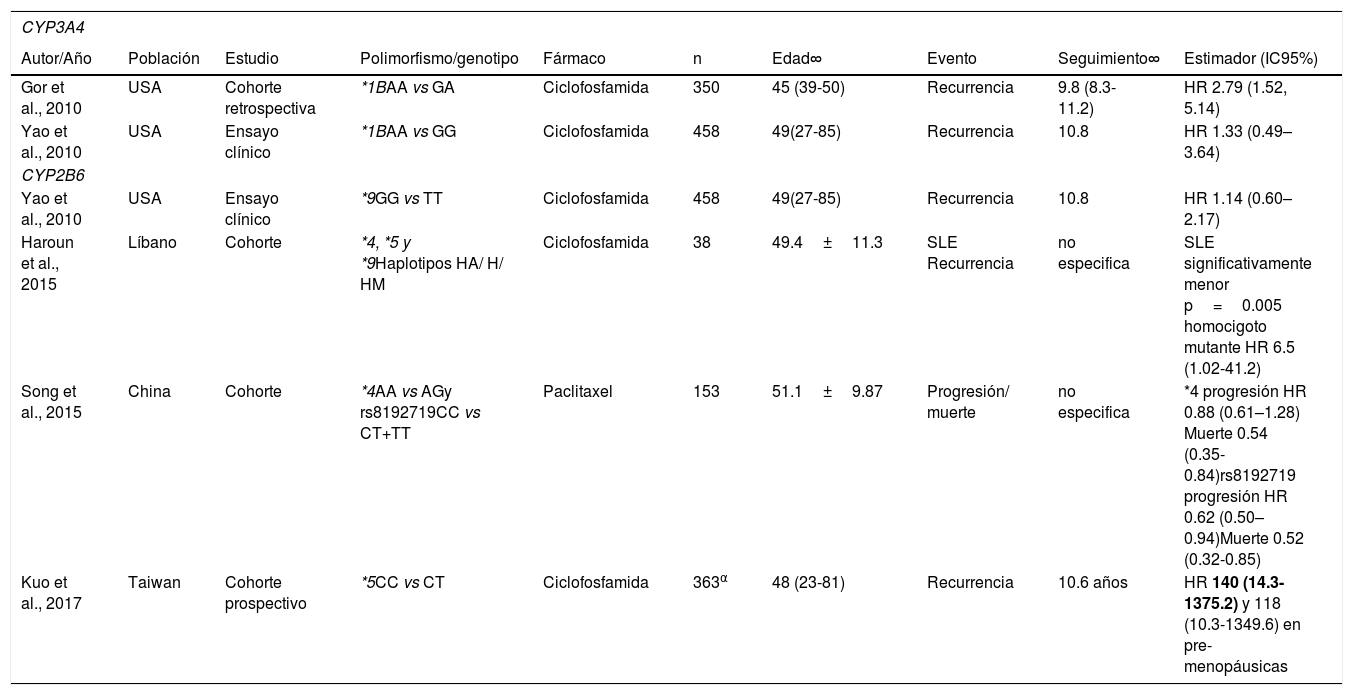
El único polimorfismo del CYP3A4 que se ha asociado significativamente a la SLE de las mujeres con cáncer de mama es el CYP3A4*1B, cuya frecuencia global se describe en la tabla 2. Gor et al. (2010) encontraron que las mujeres que tenían el genotipo heterocigoto en CYP3A4*1B, y recibieron quimioterapia adyuvante basada en la ciclofosfamida, tuvieron una SLE más breve que aquellas con el genotipo homocigoto ancestral, y un mayor riesgo instantáneo de recurrencia (HR 2.79, IC95% 1.52 -5.14)53. De forma similar, Yao et al. (2010) relatan en su ensayo clínico –sobre mujeres con cáncer de mama tratadas con ciclofosfamida– haber observado un incremento en el riesgo instantáneo de recurrencia (homocigoto ancestral vs homocigoto mutante HR 1.33 IC95% 0.49–3.64). Sin embargo, sus resultados no fueron estadísticamente significativos54. Ambos estudios se hicieron con población estadounidense (véase la tabla 3).
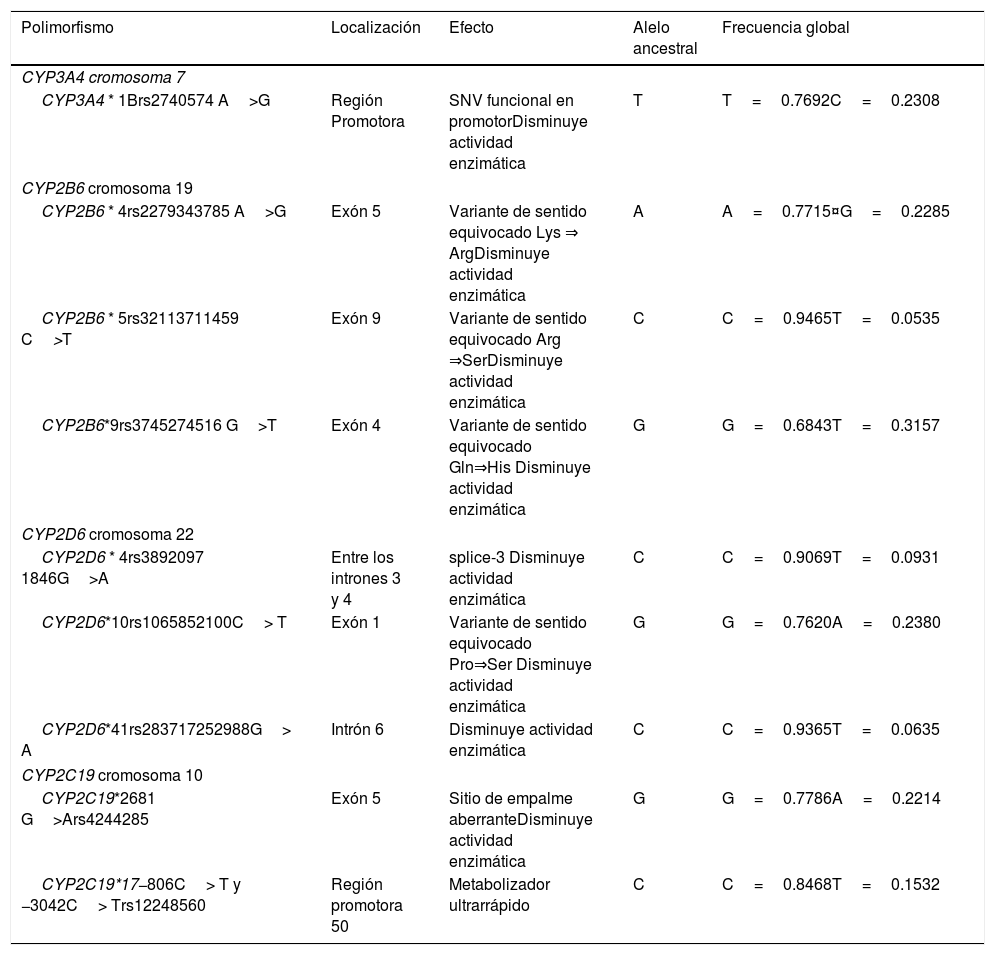
Polimorfismos funcionales del CYP450 asociados a la SLE en cáncer de mama
| Polimorfismo | Localización | Efecto | Alelo ancestral | Frecuencia global |
|---|---|---|---|---|
| CYP3A4 cromosoma 7 | ||||
| CYP3A4 * 1Brs2740574 A>G | Región Promotora | SNV funcional en promotorDisminuye actividad enzimática | T | T=0.7692C=0.2308 |
| CYP2B6 cromosoma 19 | ||||
| CYP2B6 * 4rs2279343785 A>G | Exón 5 | Variante de sentido equivocado Lys ⇒ ArgDisminuye actividad enzimática | A | A=0.7715¤G=0.2285 |
| CYP2B6 * 5rs32113711459 C>T | Exón 9 | Variante de sentido equivocado Arg ⇒SerDisminuye actividad enzimática | C | C=0.9465T=0.0535 |
| CYP2B6*9rs3745274516 G>T | Exón 4 | Variante de sentido equivocado Gln⇒His Disminuye actividad enzimática | G | G=0.6843T=0.3157 |
| CYP2D6 cromosoma 22 | ||||
| CYP2D6 * 4rs3892097 1846G>A | Entre los intrones 3 y 4 | splice-3 Disminuye actividad enzimática | C | C=0.9069T=0.0931 |
| CYP2D6*10rs1065852100C> T | Exón 1 | Variante de sentido equivocado Pro⇒Ser Disminuye actividad enzimática | G | G=0.7620A=0.2380 |
| CYP2D6*41rs283717252988G> A | Intrón 6 | Disminuye actividad enzimática | C | C=0.9365T=0.0635 |
| CYP2C19 cromosoma 10 | ||||
| CYP2C19*2681 G>Ars4244285 | Exón 5 | Sitio de empalme aberranteDisminuye actividad enzimática | G | G=0.7786A=0.2214 |
| CYP2C19*17−806C> T y −3042C> Trs12248560 | Región promotora 50 | Metabolizador ultrarrápido | C | C=0.8468T=0.1532 |
Fuente Base de datos de los 1000 genomas https://www.ncbi.nlm.nih.gov/snp¤Frecuencias de estudio ALFA https://www.ncbi.nlm.nih.gov/snp/docs/gsr/alfa/
Características y resultados de los estudios incluidos
| CYP3A4 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Autor/Año | Población | Estudio | Polimorfismo/genotipo | Fármaco | n | Edad∞ | Evento | Seguimiento∞ | Estimador (IC95%) |
| Gor et al., 2010 | USA | Cohorte retrospectiva | *1BAA vs GA | Ciclofosfamida | 350 | 45 (39-50) | Recurrencia | 9.8 (8.3-11.2) | HR 2.79 (1.52, 5.14) |
| Yao et al., 2010 | USA | Ensayo clínico | *1BAA vs GG | Ciclofosfamida | 458 | 49(27-85) | Recurrencia | 10.8 | HR 1.33 (0.49–3.64) |
| CYP2B6 | |||||||||
| Yao et al., 2010 | USA | Ensayo clínico | *9GG vs TT | Ciclofosfamida | 458 | 49(27-85) | Recurrencia | 10.8 | HR 1.14 (0.60–2.17) |
| Haroun et al., 2015 | Líbano | Cohorte | *4, *5 y *9Haplotipos HA/ H/ HM | Ciclofosfamida | 38 | 49.4±11.3 | SLE Recurrencia | no especifica | SLE significativamente menor p=0.005 homocigoto mutante HR 6.5 (1.02-41.2) |
| Song et al., 2015 | China | Cohorte | *4AA vs AGy rs8192719CC vs CT+TT | Paclitaxel | 153 | 51.1±9.87 | Progresión/ muerte | no especifica | *4 progresión HR 0.88 (0.61–1.28) Muerte 0.54 (0.35-0.84)rs8192719 progresión HR 0.62 (0.50–0.94)Muerte 0.52 (0.32-0.85) |
| Kuo et al., 2017 | Taiwan | Cohorte prospectivo | *5CC vs CT | Ciclofosfamida | 363α | 48 (23-81) | Recurrencia | 10.6 años | HR 140 (14.3-1375.2) y 118 (10.3-1349.6) en pre-menopáusicas |
∞ Se muestra media±desviación estándar o mediana (rango) SLE=Supervivencia libre de enfermedad HA=Homocigoto ancestral, H=Heterocigoto, HM=Heterocigoto mutante. α Este estudio contaba con 250 pacientes pre-menopáusicas
El CYP2B6 es un gen de 27.14kb, ubicado en el brazo lago del cromosoma 19 (19q13.2)55,56, que codifica para una proteína de acción enzimática del mismo nombre y un peso de 56.1 kDa57. Su enzima es la más importante de la subfamilia CYP2B e interviene en el metabolismo de 7 a 8% de los fármacos, que suman más de 20036. Se conoce a detalle su papel en el metabolismo de aproximadamente 40 medicamentos, de los cuales 15 actúan como sustratos –entre los que se encuentran la ciclofosfamida, la ifosfamida y el tamoxifeno–, 16 como inhibidores y 5 como inductores55,58,59.
La enzima CYP2B6 también interviene en el metabolismo de los estrógenos. Es probable que su enzima sea también la más importante en el metabolismo de las toxinas ambientales, como los pesticidas organofosforados, el alcohol y el tabaco, entre otras sustancias, lo que explica su presencia en piel, el tracto gastrointestinal, la mucosa nasal y pulmonar, y el riñón. Al igual que los demás componentes del sistema enzimático del CYP450, se expresa principalmente en el hígado55.
Actualmente se conocen 38 polimorfismos del CYP2B6, de los cuales 19 son funcionales60. La variante más común en las poblaciones estudiadas hasta la fecha es el CYP2B6*6 (que alberga dos cambios de aminoácidos, 516 G>T, rs3745274, junto con 785 A>G, rs2279343, ubicadas en el exón 4 y el exón 5 respectivamente), que está involucrado con una disminución de la actividad enzimática. Su frecuencia poblacional varía entre 15 y 65%, dependiendo de la etnia. El segundo polimorfismo más importante es el CYP2B6*18 (983T>C, rs28399499) –ubicado en el exón 7–, que se ha relacionado con una disminución de la actividad de la enzima.
Otros polimorfismos de importancia son el CYP2B6*4 (516 A>G aislada rs2279343, ubicado en el exón 5), CYP2B6*5 (459 C>T rs3211371, ubicado en exón 9), que disminuyen la actividad enzimática. Finalmente, el CYP2B6*22.-82T>C rs34223104, ubicado en la región promotora, aumenta la actividad enzimática55. Los polimorfismos de este gen que tienen importancia en el tratamiento del cáncer de mama se resumen en la tabla 2.
En un estudio realizado en China con mujeres con cáncer de mama metastásico, tratadas con docetaxel+tiotepa, Song et al. (2015) encontraron que el genotipo AG del CYP2B6*4 (rs2279343 c.785A> G) había tenido una media de supervivencia global 5 meses mayor que los homocigotos AA, (0.54 IC95% 0.35-0.84, ajustado por edad, receptores de estrógenos HER2Neu y quimioterapia) en mujeres con metástasis hepática, debido probablemente a la disminución de la actividad enzimática del CYP2B6, que provoca, a su vez, mayores concentraciones del fármaco activo. Sin embargo, no se encontró una asociación estadísticamente significativa con la supervivencia libre de progresión (HR 0.88 IC95% 0.61–1.28), mientras que el genotipo CC del rs8192719 del CYP2B6 se asoció a una mayor supervivencia tanto global como libre de progresión, en comparación con los genotipos CT+TT (HR 0.62 IC95% 0.5-0.94 y HR 0.52 IC95% 0.32-0.85 respectivamente)61.
Si bien los polimorfismos del CYP2B6 han mostrado tener un efecto protector en las quimioterapias con paclitaxel, están asociados a un mal pronóstico en las mujeres tratadas con ciclofosfamida. En su investigación de mujeres con cáncer de mama tratadas con quimioterapia combinada con ciclofosfamida, en Líbano, Haroun et al. (2015) estudiaron los polimorfismos CYP2B6*4, *5 y *9 en conjunto, y encontraron que las mujeres con el genotipo homocigoto mutante habían tenido un periodo libre de recurrencia más breve con respecto a las mujeres con el genotipo homocigoto ancestral, p=0.005, con un HR de 6.5 IC95% 1.02-41.262. En un estudio similar realizado en Taiwan con mujeres con cáncer de mama que tenían receptores hormonales positivos y fueron tratadas con ciclofosfamida, Kuo et al. (2017) encontraron que el CYP2B6*5 se asociaba al incremento del riesgo instantáneo de recurrencia (HR 140 IC95%14.3-1375.2 en toda la muestra, y HR 118 IC95% 10.3-1349.6 en las mujeres pre-menopáusicas)63. Sin embargo, en un ensayo clínico realizado en Estados Unidos con mujeres con cáncer de mama tratadas con ciclofisfamida, Yao et al. (2010) no encontraron una asociación estadísticamente significativa del CYP2B6*9 con la recurrencia de la enfermedad (genotipo GG vs TT HR 1.14 IC95% 0.60-2.17)54 (véase la tabla 3).
El CYP2D6El CYP2D6 es un gen de 4.38kb, ubicado en el brazo largo del cromosoma 22 (22q13.2), y tiene nueve exones. Se expresan cuatro transcritos de ARN de 1190-1684 pb –en el cerebro, el hígado, el bazo y el sistema reproductor– y codifica para cuatro isoformas protéicas51. Después de la enzima CYP3A4, la CYP2D6 es la segunda más importante del sistema CYP450, pues metaboliza de 20 a 25% de los medicamentos de prescripción, y más de 60% de los psicotrópicos actuales36,51,64. El gen es sumamente polimorfo en la población: se conocen 141 variantes alélicas, que dan como resultado los cuatro fenotipos de metabolizadores. Actualmente se sabe que interviene en el metabolismo de 982 medicamentos51. Entre los polimorfismos de relevancia clínica se conocen: CYP2D6*3,*4, *5, *9, *10, *17 y *41, que disminuyen su acción enzimática, y el CYP2D6*2xn (gen duplicado) que la incrementa64–66.
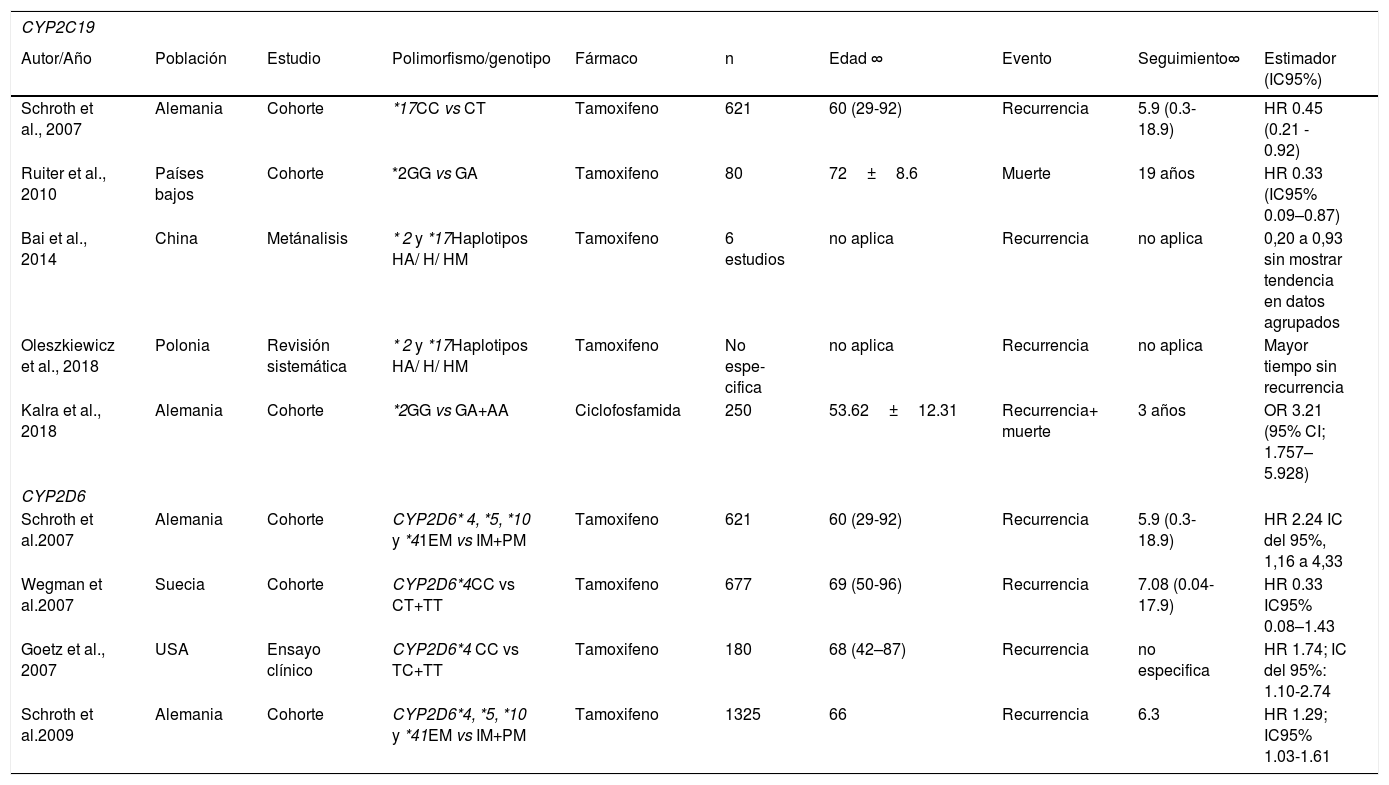
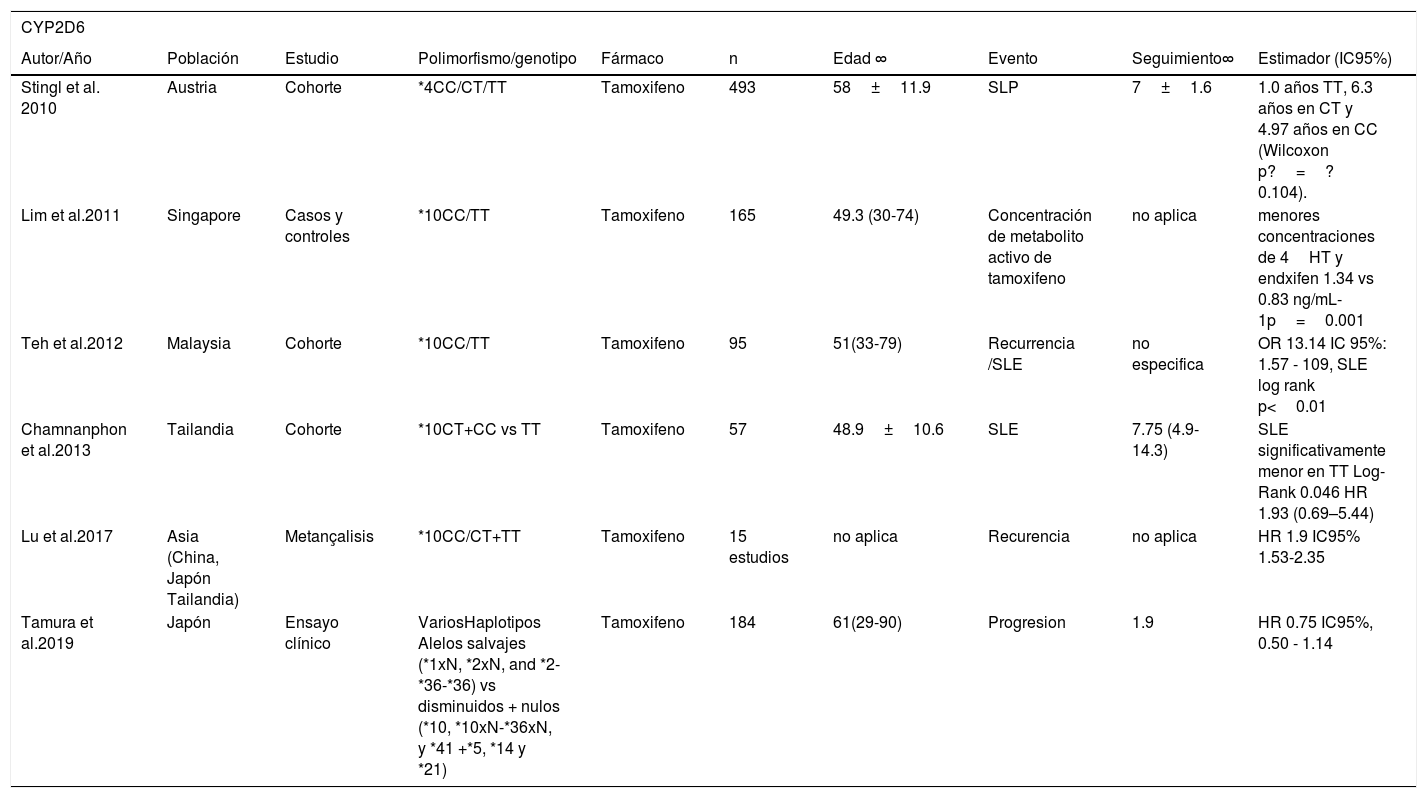
Diversos autores han estudiado el efecto de los polimorfismos del CYP2D6 en las mujeres con cáncer de mama por su efecto en el metabolismo del tamoxifeno. Sus frecuencias globales se describen en la tabla 2. Goetz et al. (2007) observaron que las mujeres estadounidenses con los genotipos MI y MP del CYP2D6*4 (CT+TT) tuvieron un tiempo de recurrencia más breve (HR=1.91 IC95% 1.05,3.45) y una peor SLE (HR 1.74 IC95% 1.10-2.74) que las mujeres ME (CC)67. De forma similar, Stingl et al. (2010) observaron que las mujeres austriacas portadoras del genotipo homocigoto mutante TT tuvieron una supervivencia libre de progresión más breve que aquellas con el genotipo homocigoto ancestral CC (1.00 vs 4.97 años p=0.104). Sin embargo esta diferencia no fue estadísticamente significativa68. En Suecia, Wegman et al. (2007) obtuvieron resultados en el sentido opuesto, pues observaron una disminución en el riesgo de recurrencia en mujeres portadoras de los genotipos CT y TT, en comparación con portadoras del CC del CYP2D6*4 (HR 0.33 IC95% 0.08–1.43). Este resultado tampoco fue estadísticamente significativo69.
En un estudio del CYP2D6*10 de mujeres tailandesas, Chamnanphon et al. (2013) observaron una menor SLE en las mujeres portadoras del genotipo homocigoto mutante (TT), en comparación con las portadoras del alelo silvestre (CT+CC) (log Rank p=0.046), y un mayor riesgo de recurrencia (HR 3.48 IC95% 0.86, 14.07). Pero este resultado no fue estadísticamente significativo70. En Malasia, Teh et al. (2012) observaron que el genotipo TT estaba asociado con un aumento del riesgo de recurrencia (OR 13.14 IC95% 1.57-109) y una SLE más breve (p<0.01) que el genotipo CC71. En Singapur, Lim et al. (2011) encontraron que las mujeres con el genotipo TT tenían menores concentraciones del fármaco activo 4 hidroxitamoxifen y endoxifen que el genotipo CC (0.83 vs 1.34 ng/mL p=0.001)72. Estos datos son consistentes con el metaanálisis de Lu et al. (2017) de estudios con poblaciones asiáticas (China, Japón y Tailandia), en los que observaron que los genotipos CT+TT se asociaron a un mayor riesgo instantáneo de recurrencia (HR 1.93 IC95% 1.53-2.35)73.
Otros polimorfismos del CYP2D6 se han estudiado de forma conjunta. Por ejemplo, las dos investigaciones de Schroth et al. (2007 y 2009), en Alemania, estudiaron los *4. *5, *10 y *41, en mujeres con cáncer de mama tratadas con tamoxifeno, clasificándolas en MI y MP, según presentaran uno o dos alelos mutantes de cualquiera de los polimorfismos. Las compararon con las portadoras de dos alelos ancestrales (ME), y observaron un incremento del riesgo instantáneo de recurrencia en las mujeres IM+PM (2.24 IC95% 1.16-4.33 y 1.29 IC95% 1.03-1.61)74,75. Sin embargo, en un estudio similar de los polimorfismos *5 *10, *14 *21 *36, *41 en la población Japonesa, Tamura et al. (2019) no encontraron una asociación significativa con la progresión en mujeres con cáncer de mama avanzado tratadas con tamoxifeno (HR 0.75 IC95% 0.50-1.14)76 (véase la tabla 3).
El CYP2C19El gen CYP2C19, ubicado en el brazo largo del cromosoma 10, es el más polimorfo e importante de la subfamilia del CYP2C. Interviene en el metabolismo de aproximadamente 7% de los fármacos36 y tiene 90.21kb. Cuenta con 9 exones mapeados en 10q24.1q23.3, y se expresa en células hepáticas. Este gen está relacionado con el metabolismo de más de 500 medicamentos.
Si bien se han detectado 541 SNP51, los polimorfismos más importantes son el *2 (rs4244285) y el *3(rs4986893), que disminuyen su actividad enzimática, y el *17 (rs12248560) que la incrementa34,77. Sus frecuencias globales se describen en la tabla 2. Se ha observado que los polimorfismos *2 y *17 están asociados a una disminución del riesgo de recurrencia y muerte en las mujeres con cáncer de mama tratadas con tamoxifeno. En un estudio de mujeres alemanas, Schroth et al. (2007) observaron que el genotipo homocigoto mutante TT del CYP2C19*17 se asociaba a una disminución del riesgo de recurrencia con respecto a las mujeres portadoras de homocigoto ancestral CC (HR, 0.45 IC95% 0.21-0.92) 74. De forma similar, en los Países Bajos, Ruiter et al. (2010) encontraron que el genotipo GA de CYP2C19*2 se asociaba a una supervivencia global más prolongada que la del homocigoto ancestral GG (HR 0.33 IC95% 0.09-0.87)78. En su metaanálisis con población asiática, en un estudio por haplotipos, Bai et al. (2014) evaluaron estos dos polimorfismos juntos, y observaron que los heterocigotos y los homocigotos mutantes estaban asociados a una disminución de la recurrencia con HR de 0.2 a 0.9, aunque no mostraron una tendencia en datos agregados79. De la misma forma, en una revisión sistemática, Oleszkiewicz et al. (2018) observaron que los heterocigotos y los homocigotos mutantes de estos dos polimorfismos estaban asociados a un mayor tiempo sin recurrencia80.
A diferencia del efecto protector del CYP2C19*2, observado en las mujeres tratadas con tamoxifeno, en las mujeres tratadas con ciclofosfamida se ha observado una asociación con un mal pronóstico. Kalra et al. (2018) observaron en mujeres alemanas tratadas con quimioterapia adyuvante con adriamicina-ciclofosfamida, que las portadoras del alelo A en CYP2C19*2 tenían una supervivencia más breve y un mayor riesgo de recurrencia y de metástasis que las mujeres del genotipo homocigoto ancestral GG (OR 3.21 IC95% 1.76–5.93)81 (véase la tabla 3).
Características y resultados de los estudios incluidos (Continuación)
| CYP2D6 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Autor/Año | Población | Estudio | Polimorfismo/genotipo | Fármaco | n | Edad ∞ | Evento | Seguimiento∞ | Estimador (IC95%) |
| Stingl et al. 2010 | Austria | Cohorte | *4CC/CT/TT | Tamoxifeno | 493 | 58±11.9 | SLP | 7±1.6 | 1.0 años TT, 6.3 años en CT y 4.97 años en CC (Wilcoxon p?=?0.104). |
| Lim et al.2011 | Singapore | Casos y controles | *10CC/TT | Tamoxifeno | 165 | 49.3 (30-74) | Concentración de metabolito activo de tamoxifeno | no aplica | menores concentraciones de 4HT y endxifen 1.34 vs 0.83 ng/mL-1p=0.001 |
| Teh et al.2012 | Malaysia | Cohorte | *10CC/TT | Tamoxifeno | 95 | 51(33-79) | Recurrencia /SLE | no especifica | OR 13.14 IC 95%: 1.57 - 109, SLE log rank p<0.01 |
| Chamnanphon et al.2013 | Tailandia | Cohorte | *10CT+CC vs TT | Tamoxifeno | 57 | 48.9±10.6 | SLE | 7.75 (4.9-14.3) | SLE significativamente menor en TT Log-Rank 0.046 HR 1.93 (0.69–5.44) |
| Lu et al.2017 | Asia (China, Japón Tailandia) | Metançalisis | *10CC/CT+TT | Tamoxifeno | 15 estudios | no aplica | Recurencia | no aplica | HR 1.9 IC95% 1.53-2.35 |
| Tamura et al.2019 | Japón | Ensayo clínico | VariosHaplotipos Alelos salvajes (*1xN, *2xN, and *2-*36-*36) vs disminuidos + nulos (*10, *10xN-*36xN, y *41 +*5, *14 y *21) | Tamoxifeno | 184 | 61(29-90) | Progresion | 1.9 | HR 0.75 IC95%, 0.50 - 1.14 |
∞ Se muestra media±desviación estándar o mediana (rango) SLE=Supervivencia libre de enfermedad SLP=Supervivencia libre de progresión
Debido a la amplia variabilidad biológica de los entornos clínicos en que se administra la terapia contra el cáncer, las directrices actuales del Consorcio para la implementación de la farmacogenética (CPIC, por sus siglas en inglés) se centran en el papel del genotipo CYP2D6 en el tratamiento adyuvante del cáncer de mama con receptores a estrógenos (RE) positivos, utilizando los criterios de valoración de recidiva, supervivencia sin recidiva, SLE, supervivencia sin recidiva a distancia, supervivencia específica del cáncer de mama y supervivencia global82. Para los criterios de valoración clínicos de recurrencia y SLE, la evidencia se calificó como moderada para los MP de CYP2D6, con un mayor riesgo de recurrencia y menor SLE82.
El uso del genotipo CYP2D6 tiene un beneficio potencial para guiar el uso del tamoxifeno: en las mujeres con genotipos asociados a un mayor riesgo de recurrencia y SLE (ME y MP) podría recurrirse al ajuste de las dosis y de los agentes administrados. Dado que se ha demostrado que los tratamientos farmacológicos alternativos (inhibidores de la aromatasa con supresión de la función ovárica o sin ella) son superiores al tamoxifeno83, y que los MP del CYP2D6 que cambiaron de tamoxifeno a anastrozol no presentaron un mayor riesgo de recurrencia84, se esperaría que el riesgo de utilizar los resultados de la genotipificación del CYP2D6 para guiar el tratamiento hormonal sea bajo.
La variabilidad farmacogenética en otros genes del citocromo P450 –como el CYP2C9 y el CYP3A4 o el CYP3A5– se asocia con un ligero efecto sobre las concentraciones plasmáticas de 4-hidroxitamoxifeno y endoxifeno, con consecuencias poco claras sobre la eficacia clínica del tamoxifeno. El genotipo CYP2C19 también se ha asociado con el resultado de la terapia donde los ME y los MP mostraron una mayor supervivencia (HR 0.70) y se describieron efectos directos sobre las concentraciones plasmáticas del endoxifeno78,85–87. Mientras que los polimorfismos del gen CYP2B6 se han asociado a efectos protectores en las quimioterapias con paclitaxel y a un aumento del riesgo de recurrencia en las mujeres tratadas con ciclofosfamida54,61–63. Sin embargo, aún no se cuenta con suficiente evidencia para recomendar la genotipificación de estos genes.
DiscusiónEn las mujeres jóvenes, el cáncer de mama es la primera causa de muerte por tumores malignos, y la segunda en mortalidad general. Existe evidencia de que la edad igual o menor a 40 años se asocia con una menor SLE y una menor supervivencia global. Estas mujeres presentan con mayor frecuencia características inmunohistoquímicas de mal pronóstico y son diagnosticadas en las etapas clínicas avanzadas, en comparación con las mujeres mayores de 40 años88.
La efectividad de los tratamientos quimioterapéutico y hormonal en las mujeres con cáncer de mama difiere interindividualmente. Parte de esta variación, se atribuye a los polimorfismos en los genes del CYP450. Existe evidencia suficiente de que algunos polimorfismos funcionales de los genes CYP3A4, CYP2C19, CYP2D6 y CYP2B6 se han asociado a modificaciones en la SLE, ya sea incrementándola o disminuyéndola (véase la tabla 3).
Características y resultados de los estudios incluidos (Continuación)
| CYP2C19 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Autor/Año | Población | Estudio | Polimorfismo/genotipo | Fármaco | n | Edad ∞ | Evento | Seguimiento∞ | Estimador (IC95%) |
| Schroth et al., 2007 | Alemania | Cohorte | *17CC vs CT | Tamoxifeno | 621 | 60 (29-92) | Recurrencia | 5.9 (0.3-18.9) | HR 0.45 (0.21 - 0.92) |
| Ruiter et al., 2010 | Países bajos | Cohorte | *2GG vs GA | Tamoxifeno | 80 | 72±8.6 | Muerte | 19 años | HR 0.33 (IC95% 0.09–0.87) |
| Bai et al., 2014 | China | Metánalisis | * 2 y *17Haplotipos HA/ H/ HM | Tamoxifeno | 6 estudios | no aplica | Recurrencia | no aplica | 0,20 a 0,93 sin mostrar tendencia en datos agrupados |
| Oleszkiewicz et al., 2018 | Polonia | Revisión sistemática | * 2 y *17Haplotipos HA/ H/ HM | Tamoxifeno | No espe-cifica | no aplica | Recurrencia | no aplica | Mayor tiempo sin recurrencia |
| Kalra et al., 2018 | Alemania | Cohorte | *2GG vs GA+AA | Ciclofosfamida | 250 | 53.62±12.31 | Recurrencia+ muerte | 3 años | OR 3.21 (95% CI; 1.757–5.928) |
| CYP2D6 | |||||||||
| Schroth et al.2007 | Alemania | Cohorte | CYP2D6* 4, *5, *10 y *41EM vs IM+PM | Tamoxifeno | 621 | 60 (29-92) | Recurrencia | 5.9 (0.3-18.9) | HR 2.24 IC del 95%, 1,16 a 4,33 |
| Wegman et al.2007 | Suecia | Cohorte | CYP2D6*4CC vs CT+TT | Tamoxifeno | 677 | 69 (50-96) | Recurrencia | 7.08 (0.04-17.9) | HR 0.33 IC95% 0.08–1.43 |
| Goetz et al., 2007 | USA | Ensayo clínico | CYP2D6*4 CC vs TC+TT | Tamoxifeno | 180 | 68 (42–87) | Recurrencia | no especifica | HR 1.74; IC del 95%: 1.10-2.74 |
| Schroth et al.2009 | Alemania | Cohorte | CYP2D6*4, *5, *10 y *41EM vs IM+PM | Tamoxifeno | 1325 | 66 | Recurrencia | 6.3 | HR 1.29; IC95% 1.03-1.61 |
∞ Se muestra media±desviación estándar o mediana (rango) SLE=Supervivencia libre de enfermedad HA=Homocigoto ancestral, H=Heterocigoto, HM=Heterocigoto mutante. EM=Metabolizadores extensivos IM=Metabolizadores intermedios PM=Metabolizadores pobres
Aún es poca la información sobre el efecto de los genes del CYP450 en el pronóstico de las mujeres jóvenes con cáncer de mama. Desde el punto de vista clínico, es importante conocer los modificadores de la respuesta al tratamiento para que cada mujer pueda recibir la mejor estrategia para combatir su enfermedad. Esto podría ser poco factible tanto por el costo económico89 como por la falta de infraestructura de los sistemas de salud de los países en vías de desarrollo. Si bien estos factores pueden representar una limitación, la evidencia actual revela que el estudio de los polimorfismos CYP450 puede tener implicaciones positivas en la SLE de las mujeres con cáncer de mama. Esperemos que en un futuro próximo haya evidencia suficiente que pueda utilizarse en la práctica médica.
Más de 50 pruebas genéticas disponibles presentan los resultados de las variantes genéticas funcionales más importantes en la respuesta a los fármacos. Las pruebas de paneles genéticos –especialmente las de la respuesta a los fármacos– incluyen, en su mayoría, los genes del CYP450. Casi todos estos paneles buscan variantes funcionales específicas, por lo que no obtienen la secuencia completa de los genes90. Para seleccionar una prueba, es importante considerar los reportes genéticos que informen sobre los alelos que se incluyeron. Es relevante también tener Información adicional sobre el número de copias, ya que esto permite identificar deleciones o duplicaciones en estos genes. Estas dos consideraciones evitarán que se atribuyan a un genotipo silvestre deleciones genéticas o variantes que no se probaron.
Si bien aún no existen guías de práctica para el uso generalizado de pruebas genéticas de la respuesta a los fármacos en el tratamiento del cáncer de mama, el CPIC ha hecho recomendaciones para la genotipificación del CYP2D6 en las mujeres tratadas con tamoxifeno82. Como en toda prueba diagnóstica, el genotipo CYP2D6 constituye solo una de las múltiples piezas de información que los médicos deben considerar al elegir el tratamiento para cada paciente.
ConclusiónHay poca evidencia clínica que permita estimar el efecto de los genes evaluados en esta revisión en las mujeres jóvenes con cáncer de mama. Dado que la susceptibilidad genética a esta neoplasia es mayor en las mujeres jóvenes, su estudio en este grupo de edad es prioritario para sentar las bases de los tratamientos quimioterapéuticos individualizados.
Por otro lado, si bien la quimioterapia y la hormonoterapia mejoran el pronóstico de las mujeres jóvenes, tienen un costo económico elevado71 y repercusiones negativas en su vida laboral, social y reproductiva. El tratamiento individualizado basado en el estudio de estos genes podría disminuir las consecuencias negativas del tratamiento al reducir los efectos adversos y alargar su supervivencia. Permitiría, además, optimizar los recursos en salud y aumentar la calidad de vida de las mujeres con este padecimiento.
Consideraciones éticasPara la realización de este trabajo se respetaron las fuentes de información haciendo cita y referencias de estas en el documento. Al ser un artículo de revisión, no se utilizó información de pacientes, ni se experimentó con animales ni personas.
FinanciaciónEl presente manuscrito fue realizado con apoyo de las becas del Consejo Nacional de Ciencia y Tecnología de México (CONACYT) S0008-2016-1/ 272823 y por el apoyo del Instituto Mexicano del Seguro Social (IMSS) con número de expediente 99182564.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.