P-30 - ANÁLISIS DE LA COMPOSICIÓN MICROBIANA FECAL EN PACIENTES RECIÉN DIAGNOSTICADOS DE ENFERMEDAD INFLAMATORIA INTESTINAL EN FUNCIÓN DE LA LOCALIZACIÓN
1Hospital Universitario de La Princesa, Instituto de Investigación Sanitaria Princesa (IIS-Princesa), Universidad Autónoma de Madrid (UAM) y Centro de Investigación Biomédica en Red de Enfermedades Hepáticas y Digestivas (CIBERehd), Madrid. 2Centro Nacional de Investigaciones Cardiovasculares Carlos III (CNIC), Madrid. 3Hospital Universitario Central de Asturias e ISPA, Oviedo. 4Hospital Universitario Marqués de Valdecilla e IDIVAL, Santander. 5Hospital General Universitario de Alicante, ISABIAL y CIBERehd, Alicante. 6Hospital Galdakao-Usansolo, Vizcaya. 7Hospital Clínico Universitario de Valladolid. 8Hospital Universitario de Gran Canaria Dr. Negrín, Las Palmas de Gran Canaria. 9Hospital Universitario Reina Sofía e IMIBIC, Córdoba. 10Hospital Universitario La Fe, Health Research Institute La Fe, Valencia. 11Complexo Hospitalario Universitario de Santiago de Compostela. 12Hospital Universitario de Fuenlabrada. 13Hospital General Universitario de Ciudad Real. 14Hospital San Jorge, Huesca. 15Hospital Universitari Son Espases, Palma de Mallorca. 16Hospital Universitari Mutua Terrassa. Centro de Investigación Biomédica en Red de Enfermedades Hepáticas y Digestivas (CIBERehd), Madrid.
Introducción: La disbiosis es clave en la enfermedad inflamatoria intestinal (EII), en la que los pacientes pueden presentar daño en diferentes partes del intestino.
Objetivos: Analizar la microbiota de pacientes con enfermedad de Crohn (EC) y colitis ulcerosa (CU) frente a controles sanos (CS), basado en la localización de la afectación.
Métodos: Se secuenció la microbiota fecal de 104 pacientes con EC y 145 pacientes con CU de reciente diagnóstico (< 1 mes) y antes del inicio de ningún tratamiento para la EII, y 49 CS mediante el método de shotgun, analizando los resultados con Cutadapt, Kraken2 y Bracken. Con los índices de Shannon (α- diversidad) y Bray-Curtis (β-diversidad) se evaluó la diversidad dentro del mismo grupo y entre grupos, respectivamente.
Resultados: Los pacientes con EC mostraron menos α-diversidad comparado con CS, siendo inferior en aquellos con localización ileocólica (L3). En los pacientes con CU, aquellos con afectación extensa (E3) presentaron una α-diversidad menor comparado con CS y con las otras localizaciones. La β-diversidad en los pacientes con EC reveló diferencias entre las comparativas de los subgrupos ileal (L1) vs. L3, y cólico (L2) vs. L3, sugiriendo una mayor diversidad en L3; no hubo diferencias en los pacientes con CU. Los pacientes con EC revelaron una disminución de Phocaeicola coprocola y varias especies de Adlercreutzia, y un aumento de Shigella flexneri, en comparación con los CS (tabla 1). En los pacientes con CU se observó un aumento en Toxoplasma gondii y Gemella morbillorum en los tres subgrupos de localización comparado con los CS. Sin embargo, varias especies de Adlercreutzia y Alistipes estaban disminuidas en E3 comparado con los CS (tabla 2).
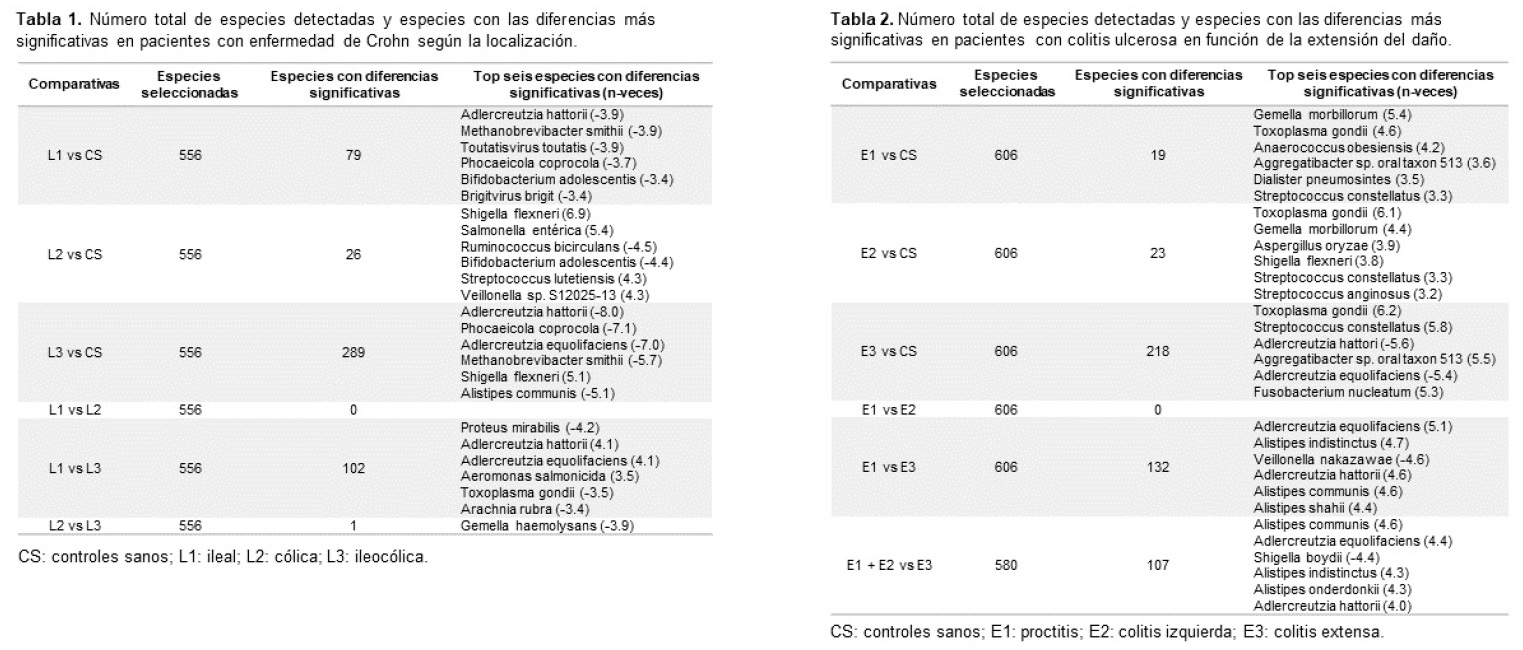
Conclusiones: En pacientes recién diagnosticados de EC y CU existen diferencias en la microbiota dependiendo de la localización de la enfermedad, detectándose una reducción de géneros beneficiosos como Adlercreutzia y Alistipes, y un aumento de especies patógenas.









